基于全基因组序列的弯曲菌特征分析
孙 磊,杨臻辉,恽 茜,黄金林
(1.扬州大学 信息工程学院,江苏 扬州 225127;2.扬州大学 人工智能学院,江苏 扬州 225127; 3.江苏省人兽共患病学重点实验室,江苏 扬州 225009; 4.江苏省动物重要疫病与人兽共患病防控协同创新中心,江苏 扬州 225009
弯曲菌(Campylobacterspp)是一类能够引起人类腹泻的人兽共患病病原菌[1]。因弯曲菌感染引起的肠炎病例数仅次于沙门氏菌和志贺氏菌[2]。在已发现的弯曲菌种类中,空肠弯曲菌和结肠弯曲菌是引起人类腹泻的主要致病菌,90%以上的病例由这两种病原引起[3],而其中空肠弯曲菌和结肠弯曲菌分别约占90%和10%[4]。空肠弯曲菌和结肠弯曲菌广泛存在于自然界,通过传统流行病学方法并不能准确确定传染源[5]。在WHO食品安全工作计划中,弯曲菌被划列为重点检测的食源性致病菌之一,许多国家也相继开展了对弯曲菌的监测。
目前弯曲菌检测主要针对空肠弯曲菌。传统检测方法包括前增菌、细菌分离和种的鉴定等步骤,其工作强度较大且耗时长,需要约5~6天才能完成检测[6]。同时,这些传统检测方法所依赖的关键生化反应—马尿酸盐水解试验可能导致假阴性和假阳性结果[7],从而影响后续分析。近年来一些基于常规或定量PCR的方法被用于弯曲菌的检测。荧光定量PCR方法较常规PCR具有更好的灵敏度,但仍存在不能有效区分细菌的死活状态以及不能获得活的培养细菌等问题[8-10]。同时,这些PCR方法所使用的试剂昂贵,且实验步骤多,容易产生样本间的交叉污染,从而导致假阳性和假阴性结果。
近年来全基因组测序技术(Whole-genome sequencing,WGS)开始被用于弯曲菌研究。测序数据在经处理和分析后可用来表征弯曲菌的不同种系,或用来快速识别群落的基因型特征,例如毒力和耐药性等[11]。同时,基于全基因组测序技术的弯曲菌研究也产生了大量数据(如存储于美国生物信息技术中心NCBI的弯曲菌全基因组序列等),而这些数据可用于进一步的信息挖掘。因此,本文将基于弯曲菌的全基因组序列,利用相关生物信息学方法,对弯曲菌的基因组序列、基因注释、耐药基因、多位点序列分型(Mutilocus sequence typing,MLST)和簇状规则间隔回文重复(Clustered regularly interspaced palindromic repeats,CRISPR)-Cas(CRISPR-associated)系统等进行分析,以挖掘出能够准确区分空肠弯曲菌和结肠弯曲菌的高分辨力特征,以帮助建立能够快速、准确地检测弯曲菌的生物信息学方法。
1 材料与方法
1.1 菌株的选择
本研究共包含120株空肠弯曲菌(空弯)和22株结肠弯曲菌(结弯),其菌株的全基因组序列下载自NCBI(https://www.ncbi.nlm.nih.gov/,按关键字“Campylobacter complete genome”搜索,结果中仅下载全基因组核心菌株)。以下实验过程均在这142株弯曲菌上展开。
1.2 全基因组序列分析
基因组序列长度和GC含量是生物最基本的遗传特征[12-13]。本文通过BioPython[14]获得空肠弯曲菌和结肠弯曲菌的基因组序列长度和GC含量信息。利用Python[15]的Matplotlib模块将以上信息进行可视化。
1.3 基因注释信息分析
为了对弯曲菌全基因组的基因信息进行识别和标记,采用Prokka软件[16]对空肠弯曲菌和结肠弯曲菌进行基因注释,后通过自编脚本从注释结果中提取密码子序列(Codon Sequence,CDS)、转运RNA(tRNA)、转运-信使RNA(tmRNA)、核糖体RNA(rRNA)以及外显子(Exon)的数量和密度信息。
1.4 耐药基因分析
耐药性弯曲菌菌株的比例在全世界范围内正在快速增加[17]。通过在线软件ResFinder 3.2[18](https://cge.cbs.dtu.dk/services/ResFinder/)查找空肠弯曲菌和结肠弯曲菌基因组中的耐药基因并进行比较。ResFinder采用默认参数,设置%ID的阈值为90%,最小长度比例为60%。
1.5 多位点序列分型分析
MLST方法通过测定多个管家基因(housekeeping genes)的核苷酸序列的变异情况对菌株类型进行表征。利用在线软件MLST 2.0(https://cge.cbs.dtu.dk/services/MLST/)[19]对全部弯曲菌菌株进行多位点序列分型,接着利用自编脚本提取每一株弯曲菌的等位基因谱,然后分析空肠弯曲菌和结肠弯曲菌之间等位基因谱的差异。
1.6 CRISPR-Cas系统分析
为了分析空肠弯曲菌和结肠弯曲菌在CRISPR-Cas系统方面的差异,利用在线软件CRISPRCasFinder(https://crisprcas.i2bc.paris-saclay.fr/CrisprCasFinder/)[20]分别对空肠弯曲菌和结肠弯曲菌中的CRISPR-Cas系统进行分析,并利用自编脚本从CRISPR-Cas系统中提取重复序列。根据Clustal X 2.1软件[21]对重复序列的比对结果,将菌株间重复的重复序列剔除,得到无冗余的重复序列。再利用Blast软件[22]将每一条无冗余的重复序列分别与空肠弯曲菌和结肠弯曲菌全基因组序列进行比对,以获得能够标识空肠弯曲菌/结肠弯曲菌的特异性序列。
2 结果与分析
2.1 全基因组序列分析
全基因组序列分析结果表明,结肠弯曲菌的基因组序列长度(均值:172.09 kbp)显著大于空肠弯曲菌(均值:166.29 kbp)(Wilcoxon秩和检验[23],p-value = 0.000 033), 如图1a箱线图[24]所示。图中,绿色三角形代表均值,红色横线代表中位数。另外,结肠弯曲菌的GC含量(均值:31.38%)也显著高于空肠弯曲菌(均值:30.43%)(Wilcoxon秩和检验,p-value = 0),见图1b。
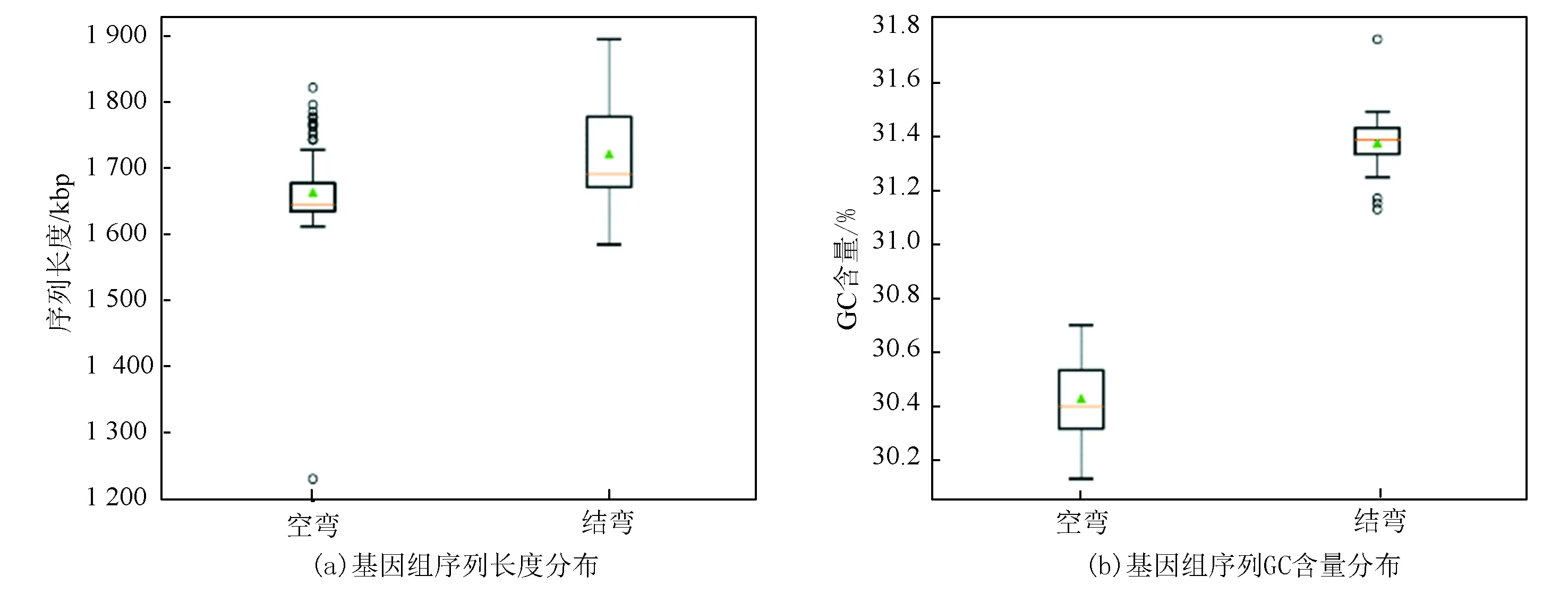
图1 空肠弯曲菌和结肠弯曲菌的基因组序列特征比较Fig.1 Feature comparison of genome sequences of Campylobacter jejuni and Campylobacter coli
2.2 基因组注释结果分析
利用Prokka软件对弯曲菌全基因组进行基因注释后,分别得到120份空肠弯曲菌和22份结肠弯曲菌的基因注释文件(GFF格式)。通过自编脚本从注释结果中统计出每个菌株的CDS、exon、rRNA、tRNA以及tmRNA的数量。其中,每一株空肠弯曲菌/结肠弯曲菌仅含有1个tmRNA。
统计分析结果表明,空肠弯曲菌和结肠弯曲菌在CDS数量方面没有显著差异(空肠均值:1 696.99,结肠均值1 728.18;Wilcoxon秩和检验,p-value =0.215 891),但在exon数量(空肠均值:51.92,结肠均值54.27;Wilcoxon秩和检验,p-value = 0.021 432)、rRNA数量(空肠均值:6.96,结肠均值8.27;Wilcoxon秩和检验,p-value = 0.014 076)以及tRNA数量(空肠均值:41.96,结肠均值43.09;Wilcoxon秩和检验,p-value = 0.033 78)方面,空肠弯曲菌和结肠弯曲菌之间存在显著差异(见图2)。

图2 空肠弯曲菌和结肠弯曲菌的基因注释特征(绝对数量)的比较Fig.2 Feature comparison of gene annotation (absolute count) of Campylobacter jejuni and Campylobacter coli
由于结肠弯曲菌和空肠弯曲菌在基因组序列长度方面存在显著差异,本文又对两种弯曲菌基因组上CDS、exon、rRNA及tRNA的数量密度(个数/序列长度)进行了比较(见图3)。统计分析结果表明,空肠弯曲菌的CDS密度(均值:1.021个/千碱基对)显著大于结弯肠弯曲菌(均值:1.003个/千碱基对)(Wilcoxon秩和检验,p-value = 0.000 37),而两种菌在exon、rRNA及tRNA的密度方面没有显著差异。

图3 空肠弯曲菌和结肠弯曲菌的基因注释特征(数量密度)的比较Fig.3 Feature comparison of gene annotation (count density) of Campylobacter jejuni and Campylobacter coli
2.3 耐药基因分析结果
经ResFinder分析发现,142株弯曲菌共含有blaOXA-193、blaOXA-450、blaOXA-451、blaOXA-452、blaOXA-453、blaOXA-489、blaOXA-61、blaOXA-460、blaOXA-465、blaOXA-447、blaOXA-184、blaOXA-461、aph(2'')-lf、aadE-Cc、cat、tet(O)等16种耐药基因。其中,含耐药基因的空肠弯曲菌和结肠弯曲菌的菌株数(比例)分别为106株(88.33%)和15株(68.18%),而发现最多的耐药基因为blaOXA(OXA β-lactamases, OXA β-内酰胺酶)型[25]。已有的研究表明,β-内酰胺类(Beta-lactam)抗生素对弯曲菌的功效有限,而对抗此类抗生素的耐药性可能是由弯曲菌产生的β-内酰胺酶所介导的[17]。表1展示了含耐药基因的弯曲菌数量及占比。
统计分析结果表明,本文所采用的空肠弯曲菌和结肠弯曲菌菌株在耐药基因分布上没有显著差异(卡方检验,p-value=0.055 839)。尽管如此,两种弯曲菌菌株耐药基因的区别仍值得讨论。例如,空肠弯曲菌对除了aadE-Cc之外的大部分耐药基因均有包含,但没有发现任何结肠弯曲菌含有blaOXA-465、blaOXA-447、blaOXA-184、blaOXA-461、cat、tet(O)等6种耐药基因。特别是在空肠弯曲菌中占比高达45%的耐药基因blaOXA-447在结肠弯曲菌中没有被发现,并且含有blaOXA-447的空肠弯曲菌不含其它任何blaOXA基因。
2.4 多位点序列分型结果
MLST方法一般测定6~10个管家基因内部400~600 bp片段的核苷酸序列(即MLST等位基因),并根据每个等位基因位点的序列被发现的顺序赋予一个等位基因编号,而每个菌株的等位基因及编号按照指定顺序排列后便构成其等位基因谱(Allelic profile)或序列型(Sequence type, ST)[26]。弯曲菌的MLST分析一般涵盖aspA、glnA、tkt、uncA、gltA、glyA、pgm等7个等位基因。对142株弯曲菌的等位基因谱进行去冗余处理后,共得到38种空肠弯曲菌和15种结肠弯曲菌的等位基因谱(见表2)。其等位基因编号的分布如图4所示。

表1 含有耐药基因的空肠弯曲菌和结肠弯曲菌的菌株数量比较Table 1 Count comparison of Campylobacter jejuni and Campylobacter coli strains containing resistance genes
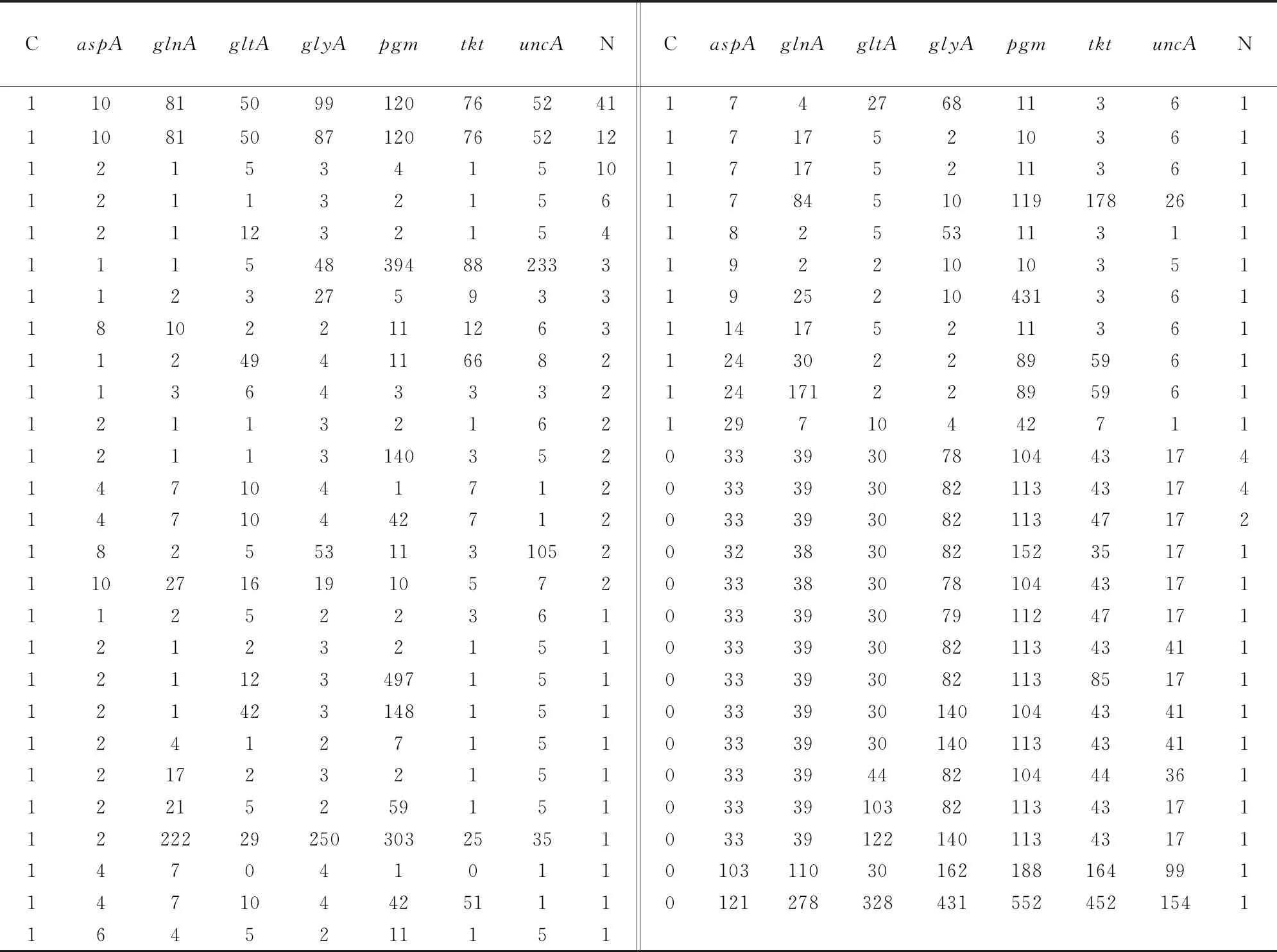
表2 空肠弯曲菌和结肠弯曲菌的等位基因谱列表Fig.2 List of allelic profiles of Campylobacter jejuni and Campylobacter coli

图4 空肠弯曲菌和结肠弯曲菌的等位基因编号分布图Fig.4 Distribution of allelic numbers of Campylobacter jejuni and Campylobacter coli
从表2和图4可见,空肠弯曲菌和结肠弯曲菌的样本菌株之间没有任何相同的等位基因谱及编号。这表明从MLST角度看,两种菌分属不同的家族。另外,全部空肠弯曲菌样本所覆盖的等位基因编号范围显著大于结肠弯曲菌(Wilcoxon秩和检验,p-value = 0.001 745)。
2.5 CRISPR-Cas系统的分析
一个完整的CRISPR-Cas系统包括CAS基因(Cas Genes)、先导序列(Leader sequence)、重复序列(Repeats/DR)以及间隔序列(Spacers)。通过CRISPRCasFinder分析发现大部分空肠弯曲菌(113/120,94.17%)含有至少一个CRISPR-Cas系统,而结肠弯曲菌中仅12个菌株(12/22,54.55%)含有CRISPR-Cas系统(见表3)。统计分析结果表明,空肠弯曲菌和结肠弯曲菌在CRISPR-Cas系统数量的分布方面具有显著差异(卡方检验,p-value=0.000 000 225 7)。
从弯曲菌样本的CRISPR-Cas系统中提取重复序列(DR序列)后,利用ClustalX 2.1对重复序列进行比对,再将冗余序列去除,最终得到66条无冗余的重复序列(见图5)。
为了找出能够标识空肠弯曲菌/结肠弯曲菌的特异性重复序列,利用Blast软件将66条重复序列分别与空肠弯曲菌和结肠弯曲菌基因组一一进行比对。结果发现重复序列NZ_CP017859_1(见图6)在105株空肠弯曲菌中存在(105/120,87.5%),而仅在1株结肠弯曲菌中存在(1/22,4.5%)。

表3 弯曲菌的CRISPR-Cas系统统计表Table 3 Statistics of CRISPR-Cas systems of Campylobacter

图5 66条无冗余重复序列(部分截图)Fig.5 Sixty-six non-redundant repeat sequences (partial snapshot)
为了进一步验证NZ_CP017859_1的分辨力,本文将重复序列NZ_CP017859_1与江苏省人兽共患病学重点实验室的自有菌株基因组(数据尚未公开)进行Blast比对,结果发现自有的全部22株空肠弯曲菌中有20株含有NZ_CP017859_1序列(20/22,90.9%),而全部27株结肠弯曲菌均不含此序列。

图6 重复序列NZ_CP017859_1Fig.6 A repeat sequence NZ_CP017859_1
3 结 论
利用生物信息学方法对弯曲菌全基因组进行了包括序列、基因注释、耐药基因、多位点序列分型以及CRISPR-Cas系统等分析。研究发现,结肠弯曲菌的基因组序列长度显著大于空肠弯曲菌,同时结肠弯曲菌的GC含量也显著高于空肠弯曲菌。基因注释结果表明,每株弯曲菌仅含一个tmRNA。结肠弯曲菌在exon、tRNA和rRNA三个特征的绝对数量上显著大于空肠弯曲菌,但两种菌的CDS含量接近。同时,空肠弯曲菌的CDS密度显著高于结肠弯曲菌,而exon、tRNA和rRNA在两种弯曲菌基因组上的密度相近。耐药基因分析结果表明,空肠弯曲菌和结肠弯曲菌在含耐药基因菌株的比例方面没有显著差异,但发现有相当一部分(45%)空肠弯曲菌含有blaOXA-447且不含其它blaOXA基因,可以推测这部分空肠弯曲菌菌株产生-内酰胺酶的机制会比较特殊,并且可能依赖于blaOXA-447。MLST分析发现空肠弯曲菌和结肠弯曲菌有着截然不同的等位基因谱,且前者的编号范围较宽。CRISPR-Cas系统分析表明,大部分空肠弯曲菌(94.16%)含有CRISPR-Cas系统,但对于结肠弯曲菌只有54.55%。另外,研究发现重复序列NZ_CP017859_1普遍存在于空肠弯曲菌中,而极少结肠弯曲菌含有此序列,故利用重复序列NZ_CP017859_1进行空肠弯曲菌鉴定具有较高的敏感性和特异性。综上,可认为序列长度、GC含量、exon、tRNA和rRNA的数量、CDS密度、等位基因谱和CRISPR重复序列NZ_CP017859_1可以作为区分空肠弯曲菌和结肠弯曲菌的特征,其中重复序列NZ_CP017859_1具有较高的高分辨力。这些特征可以结合支持向量机和深度学习等机器学习/人工智能方法,以提高空肠弯曲菌和结肠弯曲菌鉴别的准确性。同时,通过增加结肠弯曲菌的样本数量,构建平衡样本集,能够进一步优化预测模型。

