毛竹SWEET基因家族的全基因组鉴定与分析
李新然,张智俊*,喻珮瑶,陈家璐,马瑞芳,刘笑雨
(1.浙江农林大学 省部共建亚热带森林培育国家重点实验室,杭州 311300; 2.浙江农林大学 林业与生物技术学院,杭州 311300)
毛竹(Phyllostachysedulis)是我国非常重要的经济类植物,具有适应环境性强、产量高、生长周期短的特点,是世界上高生长最快的禾本科植物[1]。近年来,竹类植物的高生长机理一直是研究的热点问题。已有研究表明,竹笋生长发育过程中的许多生物过程如植物激素、细胞的生长与发育、代谢与能量、光合作用等都影响着竹类植物的高生长[1]。作为光合作用的主要产物,糖类为竹笋高生长过程中细胞的分裂和伸长提供大量能量。因此,对糖类转运相关基因的研究,将有助于深入探究竹类植物高生长的分子机理。
SWEET蛋白是近年发现的一类不依赖环境pH值,顺浓度梯度双向跨膜运输的低亲和糖转运蛋白[2]。SWEET家族成员分布非常广泛,高等真核生物中普遍存在。另外,在原生动物、后生动物、真菌、细菌,古生菌中也有分布[3-4]。由SWEET基因编码的膜蛋白均具有一定数量的保守跨膜结构域,被命名为MtN3/saliva[3]。该结构域最早在苜蓿(MedicagoSativa)根部结瘤素(nodulin)中被发现[5]。目前已有研究对主要的SWEET蛋白做系统发育分析,并分为三大分支。第1分支为植物类SWEET基因,大部分蛋白含有 2 个MtN3/saliva跨膜结构域水;第2分支为动物类SWEET基因,具有 2 个特定MtN3/saliva结构域;第3分支是从细菌到古生菌(球菌属)及线虫的某些SWEET蛋白, 该分支的SWEET蛋白由具有3个跨膜阿尔法螺旋的一个MtN3/saliva结构域组成[2]。自SWEET基因家族发现以来,相继在拟南芥(Arabidopsisthaliana)、葡萄(Vitisvinifera)、番茄(Solanumlycopersicum)、木薯(Manihotesculenta)[6-9]等植物中发现了数量不等的SWEET基因家族成员。SWEET基因家族成员参与许多生理过程。有研究表明,拟南芥中的RPG1 (ATSWEET8/AT5g40260)编码的膜蛋白对拟南芥小孢子外壁的形成是必须的[10];在水稻(Oryzasativa)中该基因家族成员也涉及到生殖发育[11];拟南芥位于质膜上的蛋白质SAG29(AtSWEETl5)蛋白,能在高盐环境下调节细胞活性[12];SWEET基因也参与衰老过程的调控,水稻中OsSWEET5的过表达会使植株在幼苗阶段表现出生长延迟和过早衰老, 而该基因敲除植株并没有引起表型的变化[13]。Chu 等 研 究 发 现 水稻OsSWEET11(Xa13)突变的植株能够抵抗白粉病[14],这表明SWEET基因在行使糖转运蛋白功能的同时也参与了寄主与病原菌的互作, 在促进植物免疫中也起到重要作用。但目前尚未有SWEET家族在竹类植物中的研究报道。
随着毛竹基因组测序的完成,使得在基因组水平上研究毛竹SWEET家族的信息成为可能。本研究拟鉴定出毛竹SWEET基因家族成员,通过蛋白质理化性质、系统进化关系、基因结构、启动子元件、蛋白互作网络、GO注释分析,并结合相关转录组数据挖掘,发现SWEET家族在毛竹不同组织以及不同激素水平下的表达情况,为进一步研究SWEET基因家族的结构与功能、揭示毛竹快速生长的分子机制打下基础。
1 材料与方法
1.1 毛竹SWEET家族成员鉴定及理化性质分析
在ftp://parrot.genomics.cn/gigadb/pub/10.5524/100001_101000/100498/站点下载毛竹的基因组序列、基因编码区序列(Coding sequence, CDS)、蛋白序列以及基因组注释(General feature format, GFF)在内的数据文件。在Pfam数据库(http://pfam.xfam.org/)中以SWEET隐马尔可夫模型(HMM)PF03083为模板[15]下载结构域数据,以此为种子模型利用HMMER3软件(http://hmmer.janelia.org/)检索本地毛竹蛋白数据库,筛选E值设置为小于等于1×10-20,对检索结果进行排序整理,去重处理,初步获得候选基因家族成员。进一步通过TBtools软件从毛竹全基因组数据库中,提取得到毛竹SWEET基因家族成员的基因、CDS、蛋白fasta序列以及基因基因结构和位置信息。利用在线工具ProtParam(https://web.expasy.org/protparam/)、ProtScale(https://web.expasy.org/protscale/)分析SWEET基因家族各成员理化性质。利用TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)在线工具对基因家族各成员跨膜区进行分析。
1.2 SWEET基因家族进化分析
拟南芥(Arabidopsisthaliana)和水稻(Oryzasativa)全基因组信息分别从拟南芥数据库TAIR10 (http://www.arabidopsis.org/index.jsp)以及水稻数据库(http://rice.plantbiology.msu.edu)中下载,并用HMMER3软件对相应本地数据库进行检索,经过筛选后得到对应的拟南芥和水稻的SWEET基因家族蛋白序列。对毛竹、拟南芥、水稻SWEET基因家族成员蛋白序列进行ClustalW多重比对,用MEGA 7.0 (http://www.megasoftware.net)软件邻位连接法构建种内和种间系统进化树,自检值取1 000次抽样。
1.3 SWEET基因家族染色体分布及种内、种间共线性分析
利用Tbtools软件(v0.66836)中的BLAST模块将毛竹基因组所有蛋白序列自身比对及毛竹与水稻、毛竹与拟南芥基因组蛋白序列两两比对,结合三个物种全基因组染色体位置信息,利用MC ScanX[16]获取SWEET家族染色体分布和种间共线性关系,并用Circos和Multipe synteny plot分别对种内和种间的共线性结果进行可视化。
1.4 SWEET基因家族基序和保守结构域预测
通过在线网站NCBI Conserved Domain (https://www.ncbi.nlm.nih.gov/cdd/)和MEME(http://meme.sdsc.edu/meme/meme-intro.html)对SWEET家族成员的保守结构域(domain)和基序(Motif)进行预测[17]。
1.5 SWEET基因蛋白质互作网络预测分析
在Search Tool for the Retrieval of Interacting Genes/ Proteins(STRING)数据库(https://string-db.org/)中上传SWEET基因家族蛋白序列,选择拟南芥蛋白库进行比对,根据已知的蛋白质互作关系,SWEET基因家族的蛋白互作信息进行评估和预测。
1.6 SWEET基因家族GO富集分析
对毛竹SWEET基因家族的30个基因构建基因集,利用在线软件(https://www.i-sanger.com/)对毛竹的差异表达基因集SWEET参与的生物功能进行GO(Gene ontology)注释富集预测分析。
1.7 SWEET基因家族成员基因结构及启动子特征分析
依据毛竹全基因组的GFF注释文件基因位置信息,分析毛竹SWEET家族的基因结构,同时绘制基因结构图;提取毛竹SWEET基因上游1 500 bp序列作为启动子序列信息,利用在线工具PlantCare(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)[18]预测毛竹SWEET基因的顺式作用元件,并将预测结果整理筛选出顺式作用元件,在TBtools上进行启动子位置可视化。
1.8 SWEET基因家族表达分析
选取Ensemble数据库中毛竹不同部位(叶、花、竹鞭、根)的4组转录组数据(登录号ERR105067,ERR105069,ERR105073,ERR105075),以及选取NCBI SRA数据库中毛竹幼苗根组织经过赤霉素(Gibberellin, GA)、萘乙酸(Naphthalene acetic acid, NAA)处理的共12组转录组数据(登录号SRR613113, SRR613114, SRR613115, SRR613116,SRR613117, SRR613118; SRR5710702, SRR5710701,SRR5710700, SRR5710699, SRR5710698, SRR5710697),分别计算毛竹SWEET基因的表达丰度TPM (Transcripts per million reads)值,以log2 TPM值利用TBtools Amazing Heatmap绘制基因表达热图。
2 实验结果与分析
2.1 SWEET基因家族的鉴定
通过植物SWEETPfam (PF03083)模型搜索出68个家族成员,通过E值≤1×10-20筛选初步得到34个成员,再结合保守结构域、基因结构、染色体定位等特征,进一步筛选去除重复转录本,最终得到30个SWEET基因家族成员(见表1)。按照这些基因在染色体图中出现的位置顺序,依次命名为PeSWEET1-PeSWEET30。SWEET基因家族的30个成员的氨基酸序列及理化性质分析如表1所示。家族成员中蛋白分子量最大的为63.85 kD,蛋白分子量最小的为25.11 kD。氨基酸序列长度最长为229 aa,最短的长度为582 aa。等电点位于4.96~9.39之间,其中23个家族蛋白为碱性蛋白(理论等电点>7),有6个为酸性蛋白(理论等电点<7)。所有家族成员蛋白的平均疏水指数都大于零,均为疏水蛋白。 跨膜结构域预测结果显示所以家族成员均具有跨膜结构域,且有21个家族成员蛋白含有7个跨膜结构域。
2.2 SWEET基因家族分类与进化树构建
选取30个水稻SWEET家族成员与15个拟南芥和20个水稻SWEET家族成员的氨基酸序列,进行NJ法系统聚类分析,以分析毛竹SWEET基因家族的进化分类,参考SWEET家族在其它物种之间的分类)[9],毛竹SWEET家族可分为四个亚家族(I-IV) (见图1)。其中I亚家族的毛竹SWEET家族成员数量最多,为17个。各个亚族中拟南芥SWEET基因的氨基酸序列自成一个支,水稻和毛竹的ZF-HD基因的氨基酸序列聚成一支,表明SWEET亚族中毛竹与水稻的亲缘关系较近,与拟南芥亲缘关系较远。
2.3 毛竹SWEET家族染色体定位及共线性分析
毛竹SWEET基因家族的染色体分布状况如图2a所示,30个家族成员分布在15条染色体上,不同的染色体基因分布密度不同,并且有4对基因发生了并联复制。从单条染色体看,scaffold_14上分布的家族成员最多,出现了4个SWEET基因。毛竹与拟南芥、水稻之间的共线性关系如图2b所示,仅有5个SWEET同源蛋白基因出现在拟南芥染色体中,有25个毛竹SWEET基因可以在7条水稻染色体上找到对应的旁系同源基因。由此可见,毛竹与水稻之间的亲缘关系较近。此外,有9个水稻中的SWEET家族成员在毛竹中有对应的两个同源拷贝,个别家族成员有3个拷贝,且仅有3个SWEET基因在毛竹中没有发现相对应的拷贝。由此推断,SWEET家族在演化过程中可能存在全基因组多倍化事件。

图1 毛竹(Phyllostachys edulis),拟南芥(Arabidopsis thaliana),水稻(Oryza sative ) SWEET家族系统进化树Fig.1 Phylogentic tree of SWEET gene family from Phyllostachys edulis(Pe), Arabidopsis thaliana(At), and Oryza sative(Os)
2.4 毛竹SWEET家族基序及保守结构域分析
依据NCBI Conserved Domain进一步分析毛竹SWEET基因家族的保守结构域如图3a,该家族所有成员均含有MtN3_slv结构域。PeSWEET4、5、6、13、14、18、19、22、24、26除了具有典型的MtN3_slv结构域外,还含有PQ-loopsuperfamily结构域,且多位于N端。PQ-loopsuperfamily与MtN3_slv在氨基酸序列上十分接近,都具有糖运输功能[19]。其余的基因家族成员均含有两个MtN3_slv结构域。由此可知,毛竹SWEET基因家族MtN3_slv结构域具有一定的保守性,但该结构域的分布位置和数量具有一定分化。
利用在线工具MEME分析30个毛竹SWEET基因家族成员(见图3b),发现该家族成员含有3~7个数量不等的基序。其中PeSWEET18只含有3个基序,只有PeSWEET18不含有Motif 4。Motif 1、Motif 2、Motif 3在30个SWEET基因家族成员中均有出现,说明这三个基序为高度保守结构。
2.5 毛竹SWEET家族蛋白质互作PPI网络分析
利用分子互作网络检索工具 STRING 数据库预测SWEET基因家族的蛋白互作特性(见图4)。整个蛋白互作网络共有9个节点,节点间存在18组蛋白互作关系。如图可知,每个蛋白节点与其他蛋白节点都有一定的互作关系,其中PeSWEET12、PeSWEET15位于整个SWEET基因家族蛋白互作网络的中心,分别与7个家族成员蛋白产生相互作用。

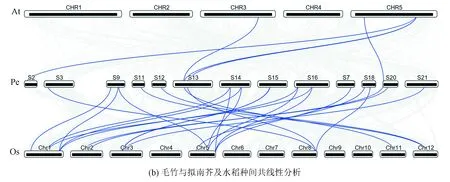
图2 毛竹SWEET家族染色体分布及种间共线性分析Fig.2 Chromosome distribution and inter-species collinearity analysis of SWEET family in Phyllostachys edulis

图3 毛竹SWEET基因家族结构域分析和保守元件分析Fig.3 Anaylsis of conserved domain and motif elements of SWEET gene family in Phyllostachys edulis

图4 毛竹SWEET家族蛋白互作网络预测图Fig.4 Protein-protein interaction network of SWEET gene family in Phyllostachys edulis
2.6 毛竹SWEET家族基因GO功能注释
利用GO对SWEET家族基因进行功能注释分析,GO结构显示该家族基因的功能主要富集于生物进程(Biological process, BP)、细胞组分(Cellular component, CC)和分子功能(Molecular function, MF) 3大类共20个小类上(见图5)。在BP分类中,所有家族成员均具有定位类别(Localization)和单有机体过程类别(Single-organism process)。在CC分类中,细胞膜(Membrane )以及膜组件类别(Membrane part)占比例最多,为所有成员共有,其次为细胞(Cell)以及细胞部分类别(Cell part)各占比百分之九十。在MF分类中SWEET家族基因的分子功能主要富集在转运活性(Transporter activity)功能。GO富集结果表明,30个毛竹SWEET基因对毛竹体内物质的转运起重要作用。
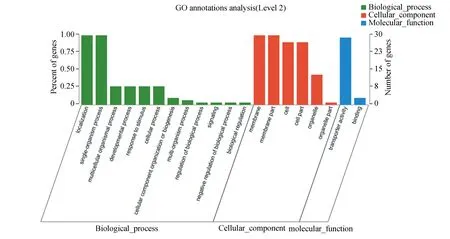
图5 毛竹SWEET基因家族的GO注释分析Fig.5 GO annotation analysis of SWEET familly in phyllostachys edulis
2.7 毛竹SWEET家族基因结构及启动子特征分析
提取30条毛竹SWEET基因上游1 500 bp的核苷酸序列,进行启动子顺势作用元件预测,结果表明除启动子核心元件TATA-box、CAAT-box以外,各家族成员上还存在多种作用元件(见图6a)。如光响应相关元件( Light cycle-related element) G-box,环境胁迫相关元件( Environmental stress-related element) ARE和MBS,以及响应脱落酸元件ABRE、响应乙烯元件ERE等激素应答相关元件( Hormonal responses-related element),同时还有生长发育相关元件( Development related element) CGTCA-motif和TGACG-motif。分析毛竹SWEET家族的基因结构(见图6b),各毛竹SWEET基因具内含子(Intron)数量3~7个不等。其中多数基因含5个内含子。
2.8 毛竹SWEET家族基因表达模式分析
SWEET基因家族对植物糖分的转运起着重要的作用,根据相关转录组数据,使用TBtools绘制表达热图(见图7),以探究毛竹SWEET基因的潜在功能。研究毛竹SWEET在幼苗不同激素(赤霉素、萘乙酸)水平下以及毛竹不同部位(叶、花、鞭、根)的表达情况。图7a所示,对毛竹幼苗赤霉素处理后PeSWEET20、PeSWEET24、PeSWEET26表达量明显下调,PeSWEET23表达量明显上调,其余家族基因表达量变化不明显。类似的,幼苗施加萘乙酸处理后PeSWEET07、PeSWEET12表达量明显上调,PeSWEET24、PeSWEET26表达量明显下调。说明了不同基因对不同种类激素的响应不同,推测这些基因可能受激素调控。并且PeSWEET02、PeSWEET03、PeSWEET13、PeSWEET16、PeSWEET21对两种激素处理均无反应。由图7b可知,PeSWEET03、PeSWEET07、PeSWEET16、PeSWEET21在毛竹的花中特异性表达,推测这些基因与毛竹的生殖活动有关。PeSWEET20在根中特异性表达,此外PeSWEET14、PeSWEET19、PeSWEET22、PeSWEET27、PeSWEET29在毛竹的四个部位均有一定量的表达,其中PeSWEET19在根中具有极高的表达量,表达丰度值高达232.8。
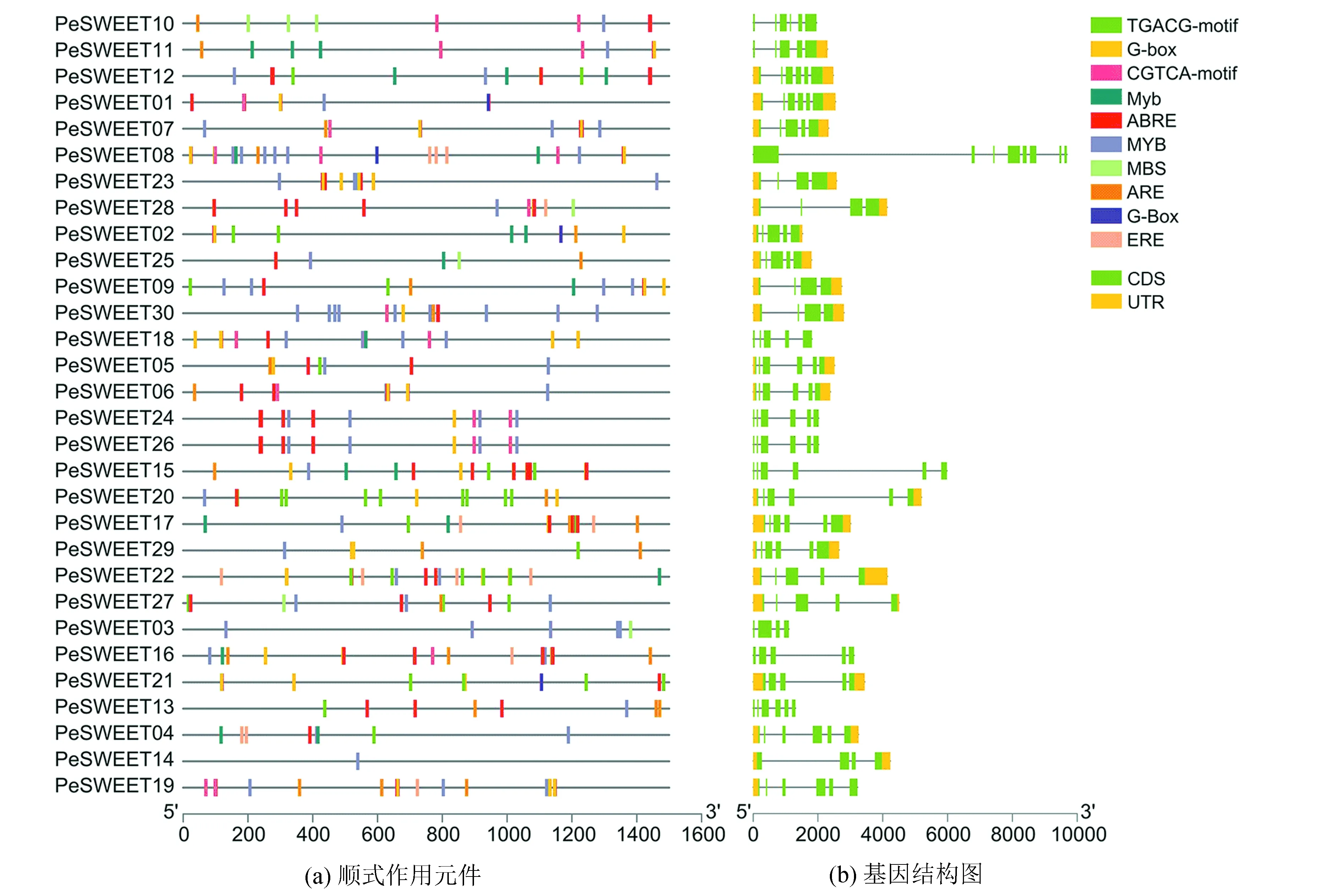
图6 毛竹SWEET基因家族启动子区顺式作用元件及基因结构图Fig.6 Structures analysis and cis-acting regulatory elements identified in the promoter region of SWEET gene family in Phyllostachys edulis

图7 毛竹SWEET基因家族表达分析Fig.7 Gene expression analysis of SWEET gene family in Phyllostachys edulis
3 讨 论
SWEET基因家族成员普遍存在于各种生物中,可涉及生物体生长发育、生殖、寄主与病原菌的互作、非生物应激反应等生理过程[2]。不同植物中的SWEET基因数量不同,本研究通过生物信息学的数据分析,从毛竹转录因子数据库中找到了30个候选的SWEET基因,而Han等(2015)统计的拟南芥、水稻以及番茄中的SWEET基因家族成员数量分别为17、21和29个[8],均少于毛竹。但毛竹中SWEET基因总数明显少于大豆中的52个[20],这可能与不同物种间基因组序列差异有关。本研究提取的SWEET基因家族与 Chen 等(2012)分类方法相一致[21],共分为4个亚族。结合GO功能注释分析,表明该家族所以成员均具有糖类转运的功能。种间共线性分析结果显示,毛竹的SWEET家族成员与同为单子叶植物的水稻之间存在着大量的共线性,有染色体水平的基因加倍现象,而拟南芥作为双子叶植物与毛竹的SWEET家族成员之间共线性不显著。表明了在系统进化的过程中,双子叶植物与单子叶植物的进化分支存在差异。每个家族成员都含有MtN3/saliva结构域,表明SWEET家族在进化中相对保守。
基因的表达大部分与上游启动子有关。经启动子分析显示,SWEET基因启动子存在许多生长以及逆境响应相关顺式作用元件,预示其可能参与毛竹生长发育以及抗逆过程;SWEET基因表达模式分析结果显示,SWEET基因可响应激素调控,这与SWEET基因启动子上存在有相应激素调控元件有关;另外,某些基因在花中特异性高效表达,表明这些基因在毛竹生殖过程中起一定作用。综上说明SWEET蛋白除了具有的糖转运功能外,该家族基因还可能参与毛竹的生长发育,花的生殖发育以及胁迫反应过程。
通过生物信息学分析,确定了毛竹基因组中SWEET基因家族成员及染色体分布情况,获得了SWEET家族结构、功能、进化等特征。结合转录组数据分析,揭示了SWEET基因家族的表达特征,证实毛竹SWEET家族可响应激素调控并参与毛竹的生长发育与生殖发育过程。这些研究提供了毛竹SWEET的重要信息,为今后SWEET基因家族在毛竹快速高生长过程中的分子功能和作用机制研究奠定了坚实基础。

