环状RNA翻译能力研究进展
郑帅龙,李利,张红平
综 述
环状RNA翻译能力研究进展
郑帅龙,李利,张红平
四川农业大学动物遗传育种研究所,成都 611130
随着高通量测序技术和翻译组学研究的快速发展,对环状RNA (circular RNA, circRNA)翻译能力的研究日益成为热点。已有研究表明,circRNA自身可以翻译为蛋白,其蛋白功能与人类疾病发生发展有着密切联系,而且其有望成为mRNA的理想替代品,未来可被广泛地应用在蛋白质工程。本文系统综述了circRNA来源、形成方式和主要特征、翻译蛋白的方式、翻译能力的鉴定和功能验证,归纳了近年来circRNA翻译在人类疾病中的研究进展及其在蛋白质工程的应用,并对后续研究关注的问题进行了展望,以期为相关领域的研究提供参考。
环状RNA;蛋白翻译;鉴定方法
环状RNA(circular RNA)是一类不具有5ʹ端m7G帽和3ʹ端poly(A)尾、以共价键首尾连接的环形RNA。20世纪70年代,研究者首次在马铃薯()块茎类病毒中发现了circRNA[1],随后在四膜虫()、古细菌()、人类()、小鼠()和大鼠()等物种中也相继发现了circRNA[2~6]。circRNA由于其在组织中表达量低,起初认为是RNA错误剪切产生的副产物,没有引起足够重视。但随着circRNA相关研究深入开展,人们发现circRNA在生物体内发挥着重要的作用,比如circRNA自身可以翻译为蛋白。1986年,Kos等[7]首次发现肝炎病毒(hepatitis virus)中的circRNA可以翻译为122个氨基酸的多肽。随后,人工构建的外源circRNA分别在大肠杆菌()、兔()网织红细胞裂解液和果蝇() S2细胞中成功地表达出蛋白[8~10]。近年来,国内外学者发现细胞内源性circRNA也可以在一些翻译起始因子的作用下招募核糖体翻译蛋白,并且蛋白产物功能与人类疾病的发生发展有着密切联系[11~14]。自此,circRNA翻译能力的研究迅速成为了焦点。本文结合国内外circRNA翻译能力的最新研究,对circRNA的翻译方式、翻译能力的鉴定和实验验证方法等进行了系统总结,旨在为深入揭示circRNA的作用机制提供参考。
1 circRNA概述
1.1 circRNA来源、环化方式和主要特征
circRNA由pre-mRNA经可变剪切(alternative splicing, AS)后产生,AS事件主要包括外显子跳跃(exon skipping, ES)、3ʹ端可变剪切(alternative 3ʹ splicing site, A3SS)、5ʹ端可变剪切(alternative 5ʹ splicing site, A5SS)和内含子保留(intron retention, IR)等。AS后生成的circRNA由于ICFs(intronic circRNA fragments)的不同,可分为由外显子形成的exon circRNA、由内含子形成的ciRNA (circular intronic RNA)和由外显子与内含子共同形成的EIciRNA (exon- intron circRNA)[15]。circRNA的环化方式主要有套索驱动环化、内含子配对驱动环化和RNA结合蛋白驱动环化3种(图1)[16~18]。circRNA呈共价环状结构,在生物体内非常稳定,不易被RNase R (ribonuclease R)降解[19],Jeck等[20]发现细胞内由外显子环化生成的circRNA的半衰期超过48 h。circRNA在不同的物种间具有保守性,Venø等[21]研究发现小鼠大脑中有15%~20%的circRNA的剪切位点与猪脑中circRNA的剪切位点高度保守。circRNA表达具有组织特异性和时空特异性,在人、小鼠和猪的不同组织中,皮质和小脑中circRNA的水平最高;在猪胚胎发育过程中,猪大脑中circRNA的表达水平呈动态变化,60天时表达量最高[21~24]。
1.2 circRNA作用机制
circRNA在生物体内表达量很低,其主要在转录及转录后水平等多个层面调控机体的生长发育、细胞增殖分化、细胞凋亡和疾病的发生等过程。其主要的作用机制有:(1) circRNA的形成会影响其同源线性转录本的生成,环化生成的circRNA越多,其同源线性转录本生成量就越少(图2A)[24]。(2)调控亲本基因的表达。EIciRNA与U1小核糖核蛋白形成复合体作用于其亲本基因启动子区,和RNA polymerase II发生相互作用,促进亲本基因表达(图2B)[25]。(3)调节其同源线性转录本的剪切。拟南芥SEP3基因转录生成的circSEP3,可与SEP3基因强烈结合,形成RNA:DNA杂合体,导致转录暂停,影响线性转录本的生成(图2C)[26]。(4)充当miRNA 分子海绵,调控下游靶基因的表达。如CDR1as可竞争miR-7靶向位点,作为miR-7的分子海绵参与基因表达调控;circFGFR4在牛成肌细胞中可以竞争性结合miR-107,促进成肌细胞分化(图2D)[27,28]。(5) circRNA可以结合相应的蛋白,通过调控蛋白对下游靶基因的作用来发挥作用(图 2E)[29]。(6) circRNA自身可以翻译为蛋白(图2F)[11~14]。(7) circRNA发挥假基因效应。可以反转录生成cDNA,cDNA可以进入细胞核内,破坏基因组DNA的完整性(图2G)[30]。(8) circRNA可以作为生物标志物,诊断早期癌症(图2H)[31]。
2 circRNA翻译方式
真核生物mRNA的翻译依赖于m7G帽结构的核糖体扫描机制(m7G cap-dependent scanning mechanism),其过程主要包括43S前起始复合物的组装、43S前起始复合物与mRNA 5ʹ末端m7G帽的结合、起始密码子的选择、60S核糖体亚基的加入和80S亚基的形成等[32]。而circRNA由于其共价环状结构[33],不能通过上述机制翻译蛋白,需要依赖一些翻译启动元件的参与启动翻译。目前已经发现的circRNA的翻译方式主要有核糖体介入位点(internal ribosome entry site,IRES)介导的翻译、滚环扩增翻译、由UTR (untranslated region)翻译激活元件介导的翻译和m6A修饰介导的翻译。
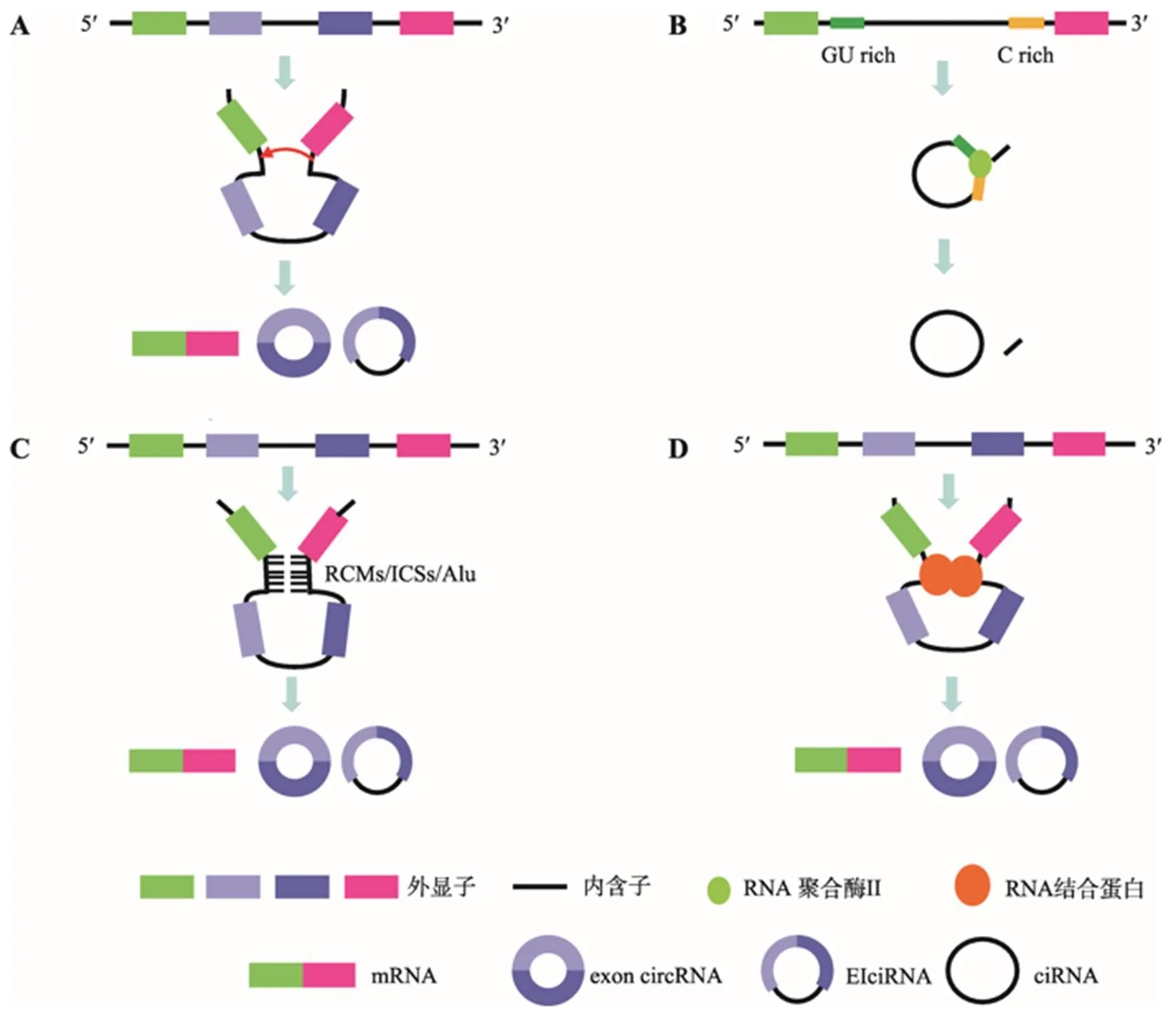
图1 circRNA的环化方式
A:套索驱动环化。当mRNA前体(pre-mRNA)进行GU/AG剪切时,可以跨外显子进行剪切,形成含有外显子和内含子的套索中间体,然后加工形成circRNA。B:内含子套索驱动环化。一些内含子的5ʹ端附近含有7 nt的GU富集序列,3ʹ端附近含有11 nt的C富集序列,它们可以在RNA polymerase II作用下形成一个套索内含子,进一步剪切形成ciRNA。C:内含子配对驱动环化。某些外显子两侧的内含子序列中含有反向互补序列,如RCMs (reverse complementary matches)、ICSs (intronic complementary sequences)及Alu元件,它们之间互补配对形成RNA双链,促进circRNA的形成。D:RNA结合蛋白驱动环化。一些RNA结合蛋白可以结合到外显子侧翼的内含子序列上,促进circRNA的形成。
2.1 核糖体介入位点介导的翻译
IRES是circRNA起始密码子ATG上游的一段长度150~250 bp的序列,能够折叠成类似tRNA的结构,可以被eIF4G2 (eukaryotic initiation factor 4G2)识别,驱动cirRNA翻译蛋白(图3A)。1995年,Chen等[8]将含有IRES的circRNA转入兔网织红细胞裂解液中,检测到了其翻译产物。Perriman等[9]将含有GFP (green fluorescent protein)标签序列和IRES的circRNA转染进大肠杆菌后,检测到了完整的GFP蛋白。2015年,Wang等[10]将人工构建的迷你基因(minigene)转染进果蝇S2细胞内,其转录本可以在细胞内自我环化形成带有GFP标签序列和IRES的circRNA,该circRNA成功地表达出了GFP蛋白。
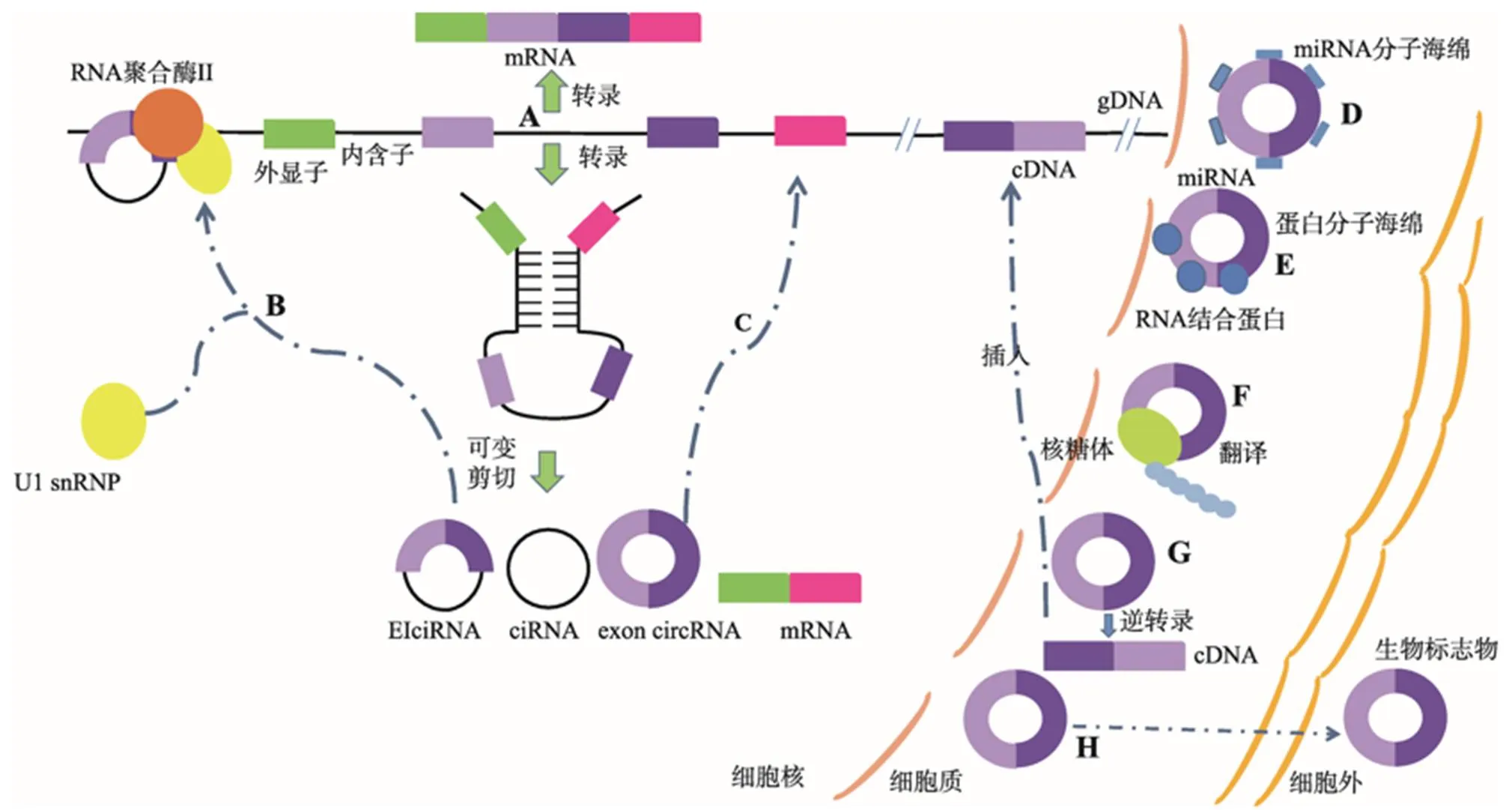
图2 circRNA的作用机制
A:circRNA的形成会影响其同源线性转录本的生成;B:可调控亲本基因的表达;C:调节其同源线性转录本的剪切;D:充当miRNA的分子海绵;E:充当RNA结合蛋白的分子海绵;F:可以翻译蛋白或多肽;G:衍生假基因;H:可以作为生物标记物。
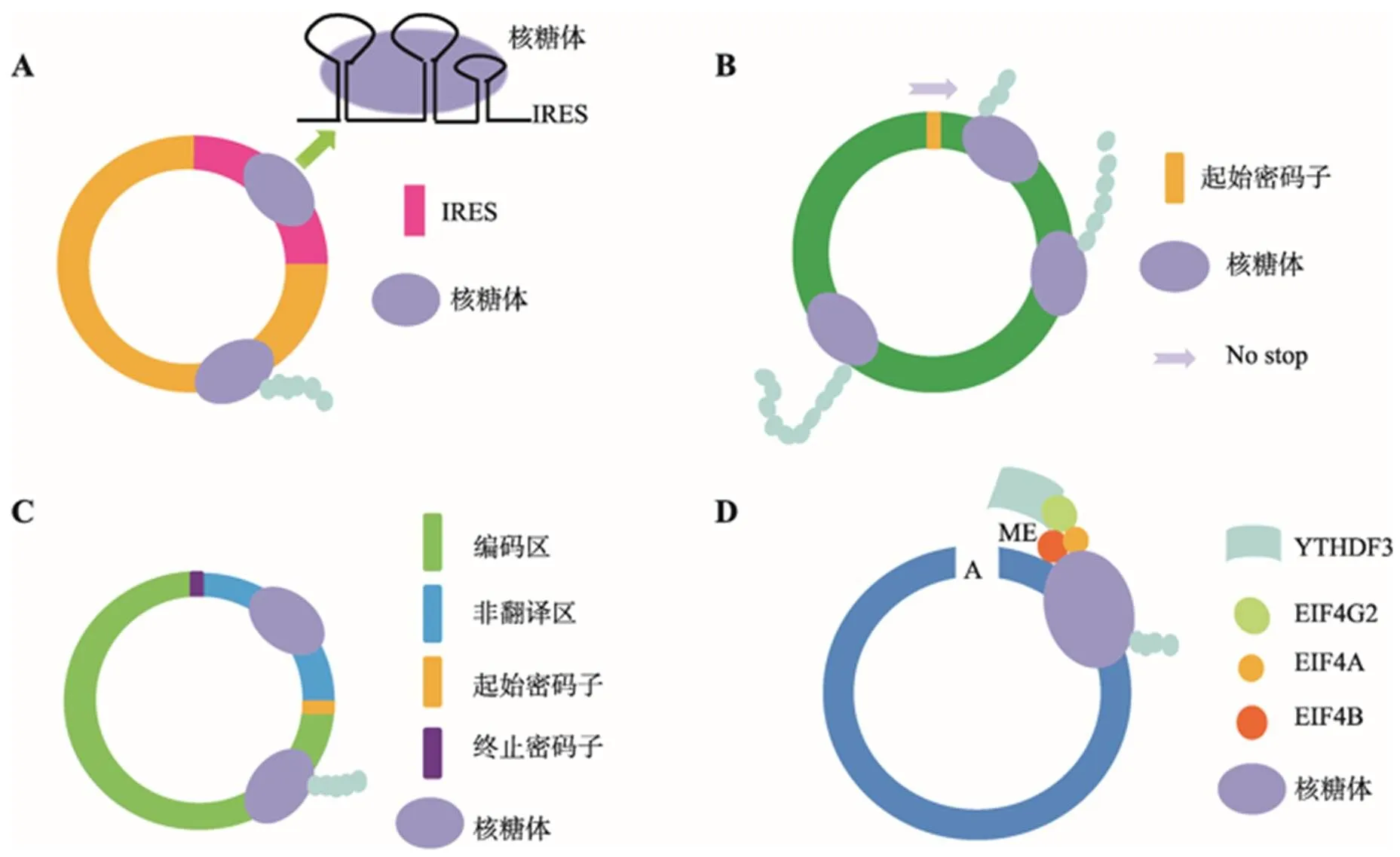
图3 circRNA的翻译方式
A:核糖体介入位点介导circRNA翻译;B:滚环扩增翻译;C:UTR翻译激活元件介导circRNA翻译;D:m6A修饰介导circRNA 翻译。
2.2 滚环扩增翻译
DNA滚环扩增(rolling circle amplification, RCA)是噬菌体感染病毒后进行自我复制的一种方式,可以对环状DNA分子进行无限单链扩增。部分circRNA由于缺失终止密码子,导致其具有无限开放阅读框,可以像RCA一样滚环翻译蛋白(图3B)。Perriman等[9]构建了一种含有无限阅读框的circRNA,发现其能模拟RCA在大肠杆菌内进行翻译,且产物分子量是其同源线性mRNA的100倍。Abe等[34]将含有无限阅读框的circRNA转入兔网织红细胞裂解液中,发现其也可以进行翻译。
2.3 由UTR翻译激活元件介导的翻译
部分circRNA由pre-mRNA反向剪切时,其剪切位点位于UTR区,导致生成的circRNA与其线性同源转录本具有部分相同的UTR序列,UTR可募集核糖体,驱动circRNA翻译蛋白(图3C)。4EBP (eukaryotic initiation factor 4E binding protein)可与翻译起始因子eIF4E (eukaryotic initiation factor 4E)结合,阻止了翻译起始复合物eIF4F(eukaryotic initiation factor 4F)的形成,抑制mRNA的翻译[35]。Pamudurti等[11]发现果蝇大脑中部分circRNA的UTR区结合有核糖体,并且检测到了生成的蛋白产物,为排除同源线性mRNA翻译干扰,过表达4EBP抑制线性mRNA翻译,发现circRNA的蛋白生成量未受影响,表明circRNA可由UTR驱动进行翻译。
2.4 m6A修饰介导的翻译
RNA甲基化修饰约占所有RNA修饰的60%以上,N6-甲基腺嘌呤(N6-methyladenosine, m6A)是RNA甲基化的一种,其通常发生在RRm6ACH (R = G或A; H = A, C或U)基序的A碱基上。m6A修饰同时受到甲基转移酶(METTL3、METTL4和WTAP等)、去甲基化酶(FTO和ALKBH5等)和一些m6A修饰结合蛋白阅读器(YTHDF1/2/3和ELAVL1等)的共同调控。Yang等[13]发现一些circRNA的UTR存在保守的m6A基序,其经甲基转移酶修饰后可以被YTHDF3蛋白识别,随后YTHDF3蛋白与翻译起始因子eIF4G2结合,eIF4G2招募翻译起始因子eIF4A (eukaryotic initiation factor 4A)和eIF4B (eukaryotic initiation factor 4B)组成翻译起始复合物eIF4 (eukaryotic initiation complex 4),启动circRNA翻译(图3D)。2019年,Zhao等[36]在被HPV16 (human papillomavirus 16)病毒感染的组织中发现了表达异常的circE7,其经m6A修饰后可以翻译E7蛋白,E7蛋白可以促进人乳头瘤细胞的增殖。
3 circRNA翻译能力的鉴定与功能验证
3.1 circRNA翻译能力鉴定的方法和工具
近年来,随着高通量测序和生物信息学的不断发展,在不同物种的组织和细胞中鉴定出了大量的circRNAs。但是,以往绝大多数circRNA鉴别方法仅实现了对BSJ (back-spliced junction)的预测,无法对circRNA转录本全长进行预测和重建,导致绝大部分鉴定出的circRNA仍然缺乏全长序列信息,给具有潜在翻译能力的circRNA的筛选造成了障碍。基于以上问题,Gao等[15]开发了CIRI-AS工具,实现了对个体内AS事件的高效检测,可以对具有相同BSJ的circRNAs的ICF进行预测,为进一步实现对circRNA全长的重建提供了可能。随后,其团队发布了CIRI-full方法,根据circRNA的识别特征—反向重叠区(reverse overlap)实现了对circRNA全长序列的重建和相对丰度的预测[37]。最近,其团队开发了CIRIquant工具,实现了在转录组数据中对circRNA的准确识别和定量,对环状RNA差异表达分析方法进行了改进,并提供了高效的分析流程[38]。这3个分析工具从根本上解决了在转录组数据中对circRNA精确识别、全长预测和重建的问题,实现了circRNA保守性和表达差异的高效分析,为后续具有潜在翻译能力的circRNA的准确筛选提供了可能。
circRNA必须具备开放阅读框以及一些翻译调控元件才可以启动翻译,获得具有全长序列信息的circRNA后,需要利用生物信息学工具(表1)对其翻译能力进行多方面预测:(1) ORF (open reading frame)预测。ORFfinder可以对circRNA序列进行搜索,列出所有潜在的ORFs,并可以推导出其氨基酸序列。通常筛选含有跨越剪切位点的ORF的circRNA,因为具有跨剪切位点ORF的circRNA与其同源线性转录本具有编码序列差异,便于后期的验证[39]。(2) circRNA翻译能力的评估。CPC2和CPAT可根据序列特征从大量候选转录本中快速识别编码和非编码转录本,对circRNA翻译潜力进行评估[40,41];它们预测的分值越接近1,则表明circRNA翻译蛋白的可能性越大,通常综合两者预测结果确定候选circRNA。(3) circRNA翻译启动方式预测。circRNA大部分由IRES或经m6A修饰后启动翻译,因此需要预测circRNA序列是否含有IRES或m6A基序。IRESite对文献报道和实验证实的IRES进行了汇总,当输入circRNA序列后,IRESite可以对circRNA上所有潜在的IRES进行评分并给出其在序列上的相应位置。IRESfinder基于k-mer特征识别真核细胞中RNA序列上的IRES,相比于IRESite有着较高的精确性[42,43],SRAMP软件可基于序列特征对哺乳动物中circRNA的m6A基序进行预测[44],m6Apred软件可对酿酒酵母()中circRNA的m6A基序进行预测[45]。(4)蛋白产物功能预测。pfam 32.0可用于蛋白功能预测,将circRNA的ORF理论产物序列输入pfam 32.0数据库后,数据库可根据序列同源性检索其潜在蛋白结构域,预测蛋白功能[46]。
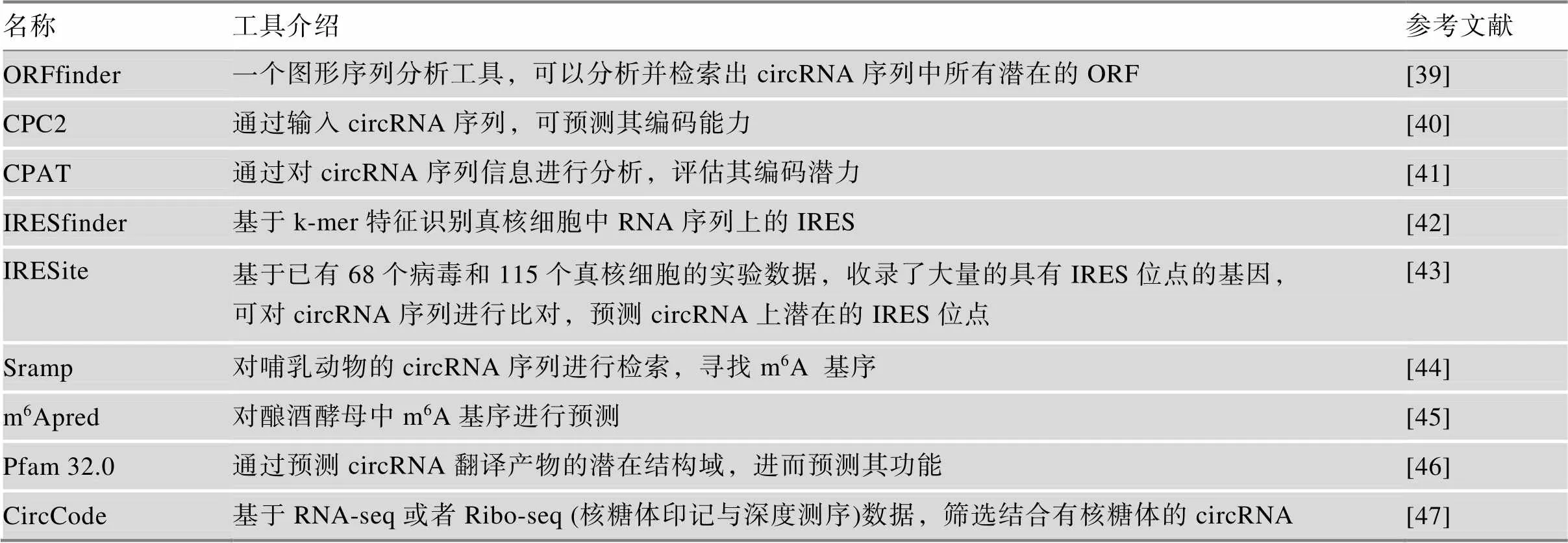
表1 研究circRNA翻译能力常用的预测工具
借助计算机科学、深度测序和生物信息学分析,研究人员研发了多个人性化、可视化的circRNA数据库(表2)。它们将多个物种的circRNA的序列、可变剪切的方式、亚细胞定位、甲基化修饰和编码能力等信息进行了汇总。2016年,Chen等[48]开发了circRNAdb数据库,其是首个汇总人类可编码蛋白的circRNA信息的数据库,可以根据基因名称查询相应circRNA的ORF、蛋白特征、外显子数目、IRES和m6A基序等信息。但是,几大数据库也存在一些不足,需要进一步完善,比如各数据库对circRNA没有进行统一命名,不便于跨数据库查询circRNA信息,各数据库具有物种局限性,对非模式生物的circRNA信息收录很少。
3.2 circRNA翻译能力的功能验证
利用生物信息学预测工具对circRNA序列进行预测分析后,需要从多方面对circRNA翻译能力进行功能验证:(1)核糖体印记与深度测序技术筛选结合有核糖体的circRNA。被核糖体包裹的RNA片段不易被Rnase降解,对该部分片段进行测序后,利用CircCode将片段序列信息与circRNA序列信息进行比对,筛选出结合有核糖体的circRNA[59,60]。 (2)体外circRNA的ORF翻译能力验证。构建含有circRNA的ORF和Flag标签的载体,将载体转入体外转录翻译系统,利用Flag抗体检测有无Flag融合蛋白生成,验证ORF是否可以在体外编码。 (3)细胞内circRNA的翻译能力验证。在circRNA的ORF终止密码子前插入Flag标签,构建含有Flag标签的circRNA过表达载体,转染细胞后,利用Flag抗体检测有无融合蛋白的生成或利用抗Flag标签蛋白的免疫荧光探针进行检测。(4)根据ORF理论蛋白产物序列设计特异性抗体,通过免疫组化或Western Blot进一步检测circRNA是否在体内进行翻译,后续通过免疫沉淀并结合质谱分析验证产物序列是否与理论蛋白序列一致[12,61]。(5)验证circRNA与翻译起始因子的相互作用。利用RNA结合蛋白免疫沉淀 (RNA binding protein immunoprecipitation, RIP)和蛋白质体外结合实验(RNA pull-down)双向验证circRNA与IRES介导翻译相关的eIF4G2和ITAF(IRES trans-acting factors)的相互作用或与m6A介导翻译相关的YTHDF1/2/3的相互作用[62,63]。(6)验证特定刺激或蛋白对circRNA翻译的影响,例如对细胞进行热应激或饥饿处理,以及对eIF4E、4E-BP、FOXO (forkhead box O)、FTO (fat mass and obesity associated)和METTL3/14等蛋白进行过表达或敲低处理,检测circRNA的蛋白产物是否受到影响[11~14]。circRNA翻译功能验证中常用的实验方法如表3。

表2 研究circRNA翻译能力常用的数据库
4 circRNA翻译在人类疾病领域研究进展
近年来,关于circRNA翻译能力研究的报道较多,大部分报道集中在癌症肿瘤领域,人们发现circRNA翻译的蛋白可以影响肿瘤的生长和转移。circFBXW7在恶性胶质瘤中高度表达,进一步研究发现circFBXW7可以被IRES介导翻译FBXW7- 185aa蛋白,FBXW7-185aa协同母基因编码的FBXW7蛋白调控原癌基因C-Myc蛋白的稳定性,抑制恶性胶质瘤的发生[14]。长非编码RNA lincPINT的第2外显子通过自身环化形成了circPINT,其序列上的IRES可驱动其翻译PINT-87aa蛋白,PINT- 87aa蛋白结合聚合酶相关因子复合物PAF1,抑制多种致癌基因的转录,进而抑制恶性胶质瘤的发生[64]。来自致癌基因的circβ-catenin,可以由IRES介导翻译β-catenin-370aa蛋白,β-catenin-370aa蛋白竞争性结合GSK3β蛋白,避免了β-catenin蛋白与GSK3β蛋白结合引起的磷酸化后泛素裂解反应的发生,导致β-catenin蛋白持续激活Wnt/β-catenin通路,促进肝癌细胞的生长增殖和转移[65];circGprc5a可以翻译circGprc5a-peptide,促进膀胱癌细胞的生长和转移[66]。此外,还有一些circRNA的蛋白产物也参与某些肿瘤的生长和转移过程(表4)。
除癌症肿瘤外,研究者对杜氏型肌营养不良症(duchenne muscular dystrophy, DMD)和扩张型心肌病(dilated cardiomyopathy, DCM)等疾病研究时也有很多发现。circZNF609在DMD组织中表达量升高,其是由ZNF609基因的第二外显子独自环化形成,参与调节肌细胞增殖,circ-ZNF609可由IRES驱动翻译蛋白,但其蛋白产物的详细功能机制仍然未知[12]。Van Heesch等[67]在患有DCM的人心肌组织中发现多个circRNA表达量异常,通过核糖体印记与深度测序技术并结合质谱检测发现其中6个circRNA可以进行翻译,包括CDR1as、circANKH、circASXL1、circCSNK1G3、circKIAA1429和circSIPA1L1,为后续科学研究提供了非常有价值的信息。
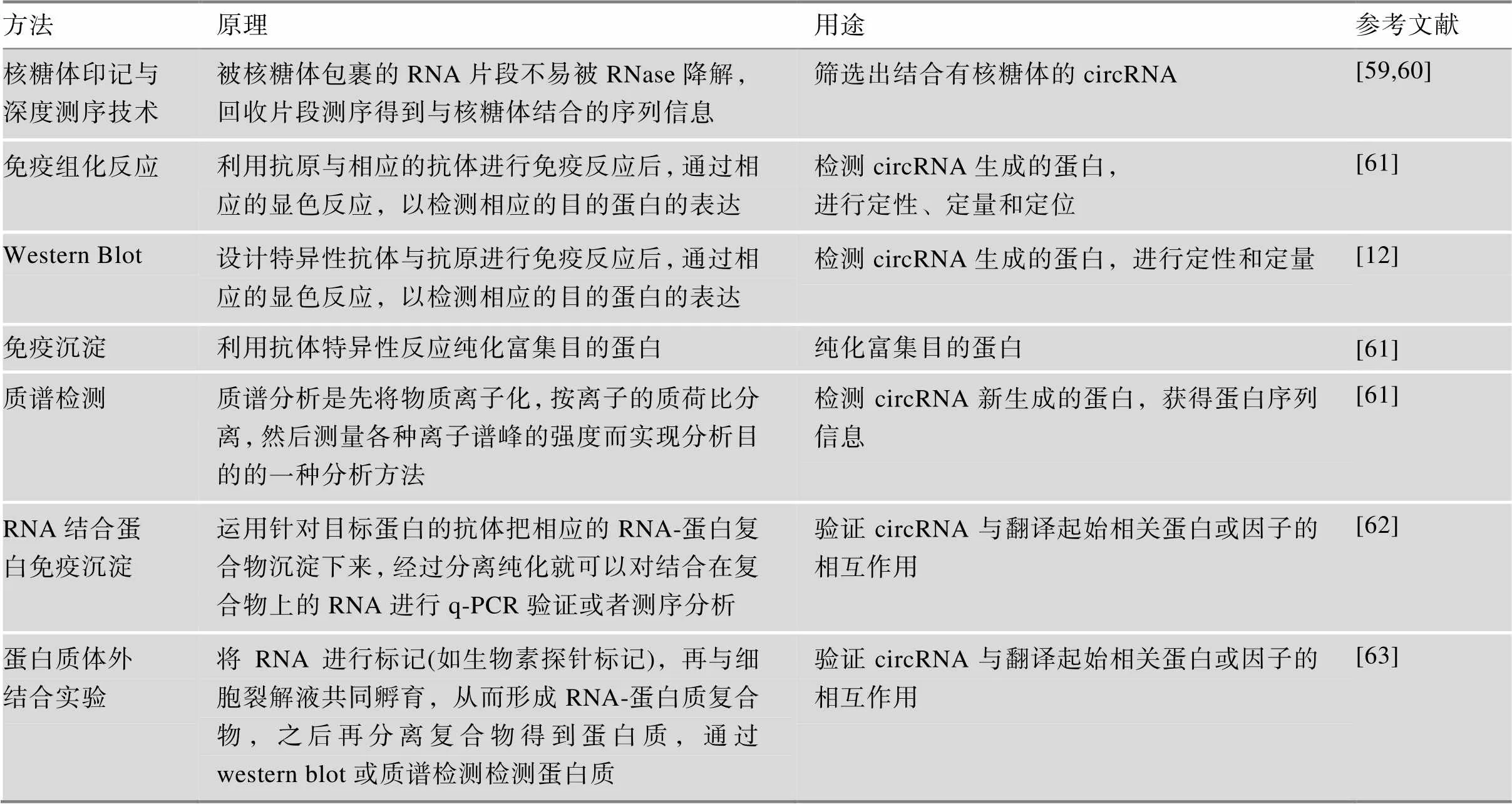
表3 研究circRNA翻译常用的实验方法
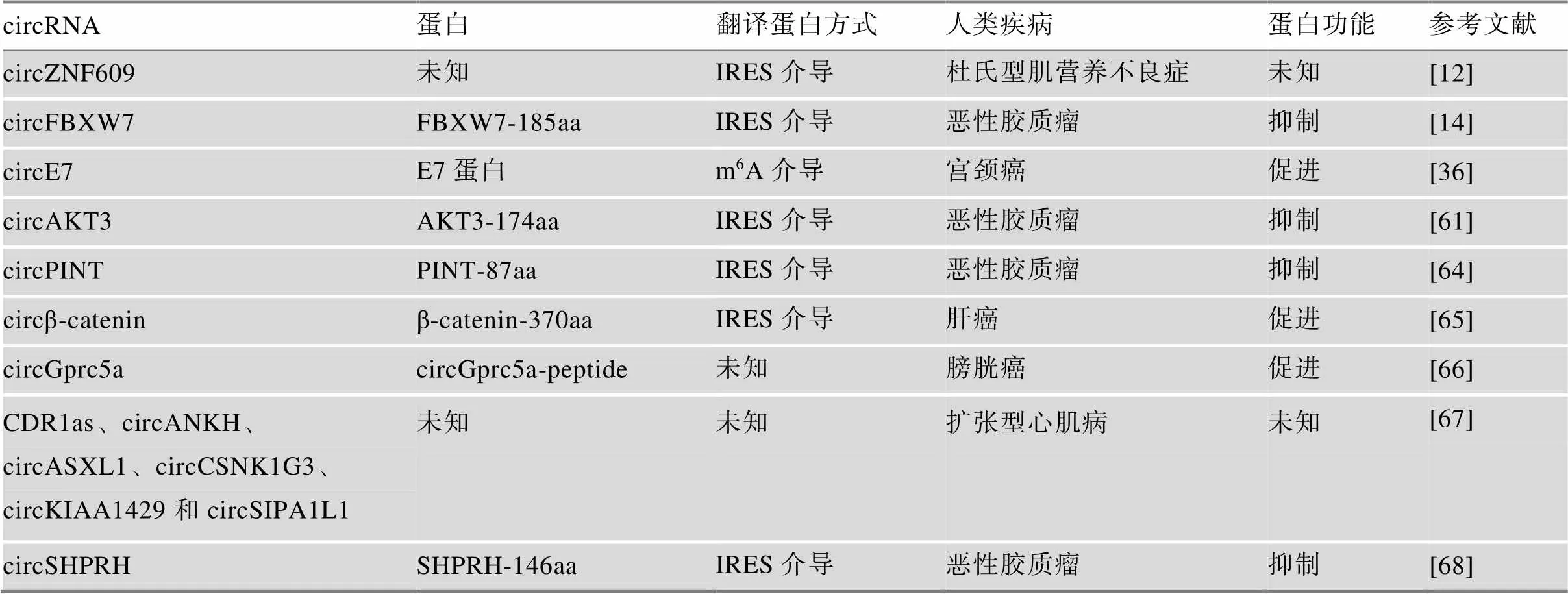
表4 circRNA翻译在人类疾病中的研究进展
5 circRNA翻译在蛋白质工程中的应用
已有大量研究发现真核细胞内源性circRNA具有许多生物学功能,部分circRNA来源于外显子,通过可变剪切机制产生,具有编码蛋白的功能[11~14]。相比于线性mRNA,内源性circRNA缺乏核酸外切酶介导降解的游离末端,可以抵抗RNA酶降解。Wesselhoeft等[69]通过设计同源互补序列辅助剪切,有效地环化长达5 kb的pre-mRNA,延长了mRNA的蛋白表达的持续时间,经过高效液相色谱法(high performance liquid chromatography, HPLC)纯化后翻译出了数量多、活性高和稳定性强的蛋白质。该研究首次在真核细胞中利用由外源mRNA环化产生的circRNA翻译蛋白,意味着未来circRNA有望成为线性mRNA的理想替代品,对于蛋白质工程的发展具有重要意义。
自然界中存在很多RNA病毒,机体为有效抵御这些病毒的感染,进化出了复杂的免疫系统,其中识别外源RNA分子的受体是激活相关免疫机制的触发器。已知生物体内RNA识别受体主要有RIG-I (retinoic acid–inducible protein I)、TLR-3 (toll-like receptor 3)、TLR-7 (toll-like receptor 7)和TLR-8 (toll-like receptor 8)等分子,它们可以识别不同的RNA及其降解产物。以往利用外源mRNA转染细胞表达蛋白时,需要对RNA进行假尿苷修饰(ψ)、N1-甲基-假尿苷(m1ψ)和N5-甲氧基-尿苷修饰(5moU)等修饰,降低这些RNA感受器分子介导的免疫效应。Wesselhoeft等[70]将mRNA环化成circRNA后,注射入小鼠后不会被TLR3/7/8识别,避免诱发TLR/ RIG-I介导的免疫反应,可以在小鼠组织中高效地翻译蛋白,不需要再对mRNA进行复杂修饰,为circRNA应用于蛋白质工程提供了新的依据。
6 结语与展望
受益于高通量测序和翻译组学的不断发展与运用,circRNA翻译研究近年来取得了一定的进展。目前已完成了对翻译启动方式的初步解析并开展了蛋白产物功能研究,形成了生物信息学、翻译组学和蛋白质组学相结合的研究体系。医学上,人们发现circRNA的蛋白产物与人类疾病有着密切的联系,比如许多恶性肿瘤单靠传统的化疗没有很好的治疗效果,而circRNA翻译的蛋白对部分肿瘤有着显著的抑制作用,未来可以根据此特点开发新的癌症治疗途径。蛋白质工程中,研究人员已利用由外源线性mRNA环化生成的circRNA在生物体内高效地生产活性蛋白,但现有的纯化方式不能获得高纯度的circRNA,未来若广泛投入实际生产,需要进一步改进纯化方式,提高其纯度。
当然,目前circRNA翻译能力的研究仍然不够成熟,还有很多问题急待解决。比如:(1) circRNA是否存在其他的翻译方式,仍然是科学家们未来持续研究的问题;(2) circRNA蛋白产物的详细功能机制仍待进一步探究,需要结合蛋白质组学,探究其蛋白产物的详细功能机制;(3)非模式生物circRNA缺乏相应注释信息,未来需加快对非模式生物circRNA的信息注释并开发相应的数据库进行系统收录;(4)生物信息学工具仍需进一步完善,目前预测工具的算法存在一定的局限性,预测的准确性仍待提高;(5)相关实验技术和设备仍待改进,比如核糖体图谱与深度测序不够成熟,质谱检测不能检测10 kDa以下的小肽,造成某些circRNA翻译产物无法被检测,给circRNA翻译能力研究带来了障碍。
总之,circRNA翻译能力研究开启了circRNA研究领域的新篇章,未来随着研究的不断深入,将会推动生命科学、医学和蛋白质工程多领域发展。
[1] Sanger HL, Klotz G, Riesner D, Gross HJ, Kleinschmidt AK. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures., 1976, 73(11): 3852– 3856.
[2] Grabowski PJ, Zaug AJ, Cech TR. The intervening sequence of the ribosomal RNA precursor is converted to a circular RNA in isolated nuclei of tetrahymena., 1981, 23(2): 467–476.
[3] Kjems J, Garrett RA. Novel splicing mechanism for the ribosomal RNA intron in the archaebacterium desulfurococcus mobilis., 1988, 54(5): 693–703.
[4] Zaphiropoulos PG. Exon skipping and circular RNA formation in transcripts of the human cytochrome P-450 2C18 gene in epidermis and of the rat androgen binding protein gene in testis., 1997, 17(6): 2985– 2993.
[5] Capel B, Swain A, Nicolis S, Hacker A, Walter M, Koopman P, Goodfellow P, Lovell-Badge R. Circular transcripts of the testis-determining gene Sry in adult mouse testis., 1993, 73(5): 1019–1030.
[6] Zaphiropoulos PG. Circular RNAs from transcripts of the rat cytochrome P450 2C24 gene: correlation with exon skipping., 1996, 93(13): 6536– 6541.
[7] Kos A, Dijkema R, Arnberg AC, van der Meide PH, Schellekens H. The hepatitis delta (δ) virus possesses a circular RNA., 1986, 323(6088): 558–560.
[8] Chen CY, Sarnow P. Initiation of protein synthesis by the eukaryotic translational apparatus on circular RNAs., 1995, 268(5209): 415–417.
[9] Perriman R, Ares M. Circular mRNA can direct translation of extremely long repeating-sequence proteins., 1998, 4(9): 1047.
[10] Wang Y, Wang Z. Efficient backsplicing produces translatable circular mRNAs., 2015, 21(2): 172–179.
[11] Pamudurti NR, Bartok O, Jens M, Ashwalfluss R, Stottmeister C, Ruhe L, Hanan M, Wyler E, Perezhernandez D, Ramberger E, Shenzis S, Samson M, Dittmar G, Landthaler M, Chekulaeva M, Rajewsky N, Kadener S. Translation of circRNAs., 2017, 66(1): 9–21.e7.
[12] Legnini I, Di Timoteo G, Rossi F, Morlando M, Briganti F, Sthandier O, Fatica A, Santini T, Andronache A, Wade M, Laneve P, Rajewsky N, Bozzoni I. Circ-ZNF609 is a circular RNA that can be translated and functions in myogenesis., 2017, 66(1): 22–37.
[13] Yang Y, Fan XJ, Mao MW, Song XW, Wu P, Zhang Y, Jin YF, Yang Y, Chen LL, Wang Y, Wong CC, Xiao XS, Wang ZF. Extensive translation of circular RNAs driven by N6-methyladenosine., 2017, 27(5): 626–641.
[14] Yang YB, Gao XY, Zhang ML, Yan S, Sun CJ, Xiao FZ, Huang NN, Yang XS, Zhao K, Zhou HK. Novel role of FBXW7 circular RNA in repressing glioma tumorigenesis., 2018, 110(3): 304–315.
[15] Gao Y, Wang JF, Zheng Y, Zhang JY, Chen S, Zhao FQ. Comprehensive identification of internal structure and alternative splicing events in circular RNAs., 2016, 7: 12060.
[16] Chen LL, Yang L. Regulation of circRNA biogenesis., 2015, 12(4): 381–388.
[17] Zhang Y, Zhang XO, Chen T, Xiang JF, Yin QF, Xing YH, Zhu S, Yang L, Chen LL. Circular intronic long noncoding RNAs., 2013, 51(6): 792–806.
[18] Conn SJ, Pillman KA, Toubia J, Conn VM, Salmanidis M, Phillips CA, Roslan S, Schreiber AW, Gregory PA, Goodall GJ. The RNA binding protein quaking regulates formation of circRNAs., 2015, 160(6): 1125–1134.
[19] Suzuki H, Tsukahara T. A view of pre-mRNA splicing from RNase R resistant RNAs., 2014, 15(6): 9331–9342.
[20] Jeck WR, Sorrentino JA, Wang K, Slevin MK, Burd CE, Liu JZ, Marzluff WF, Sharpless NE. Circular RNAs are abundant, conserved, and associated with ALU repeats., 2013, 19(2): 141–157.
[21] Venø MT, Hansen TB, Venø ST, Clausen BH, Grebing M, Finsen B, Holm IE, Kjems J. Spatio-temporal regulation of circular RNA expression during porcine embryonic brain development., 2015, 16(1): 245.
[22] Rybak-Wolf A, Stottmeister C, Glažar P, Jens M, Pino N, Giusti S, Hanan M, Behm M, Bartok O, Ashwal-Fluss R, Herzog M, Schreyer L, Papavasileiou P, Ivanov A, Öhman M, Refojo D, Kadener S, Rajewsky N. Circular RNAs in the mammalian brain are highly abundant, conserved, and dynamically expressed., 2015, 58(5): 870–885.
[23] Szabo L, Morey R, Palpant NJ, Wang PL, Afari N, Jiang C, Parast MM, Murry CE, Laurent LC, Salzman J. Statistically based splicing detection reveals neural enrichment and tissue-specific induction of circular RNA during human fetal development., 2015, 16(1): 126.
[24] You XT, Vlatkovic I, Babic A, Will T, Epstein I, Tushev G, Akbalik G, Wang MT, Glock C, Quedenau C, Wang X, Hou JY, Liu HY, Sun W, Sambandan S, Chen T, Schuman EM, Chen W. Neural circular RNAs are derived from synaptic genes and regulated by development and plasticity., 2015, 18(4): 603–610.
[25] Li ZY, Huang C, Bao C, Chen L, Lin M, Wang XL, Zhong GL, Yu B, Hu WC, Dai LM, Zhu PF, Chang ZX, Wu QF, Zhao Y, Jia Y, Xu P, Liu HJ, Shan G. Exon-intron circular RNAs regulate transcription in the nucleus., 2015, 22(3): 256–264.
[26] Conn VM, Hugouvieux V, Nayak A, Conos SA, Capovilla G, Cildir G, Jourdain A, Tergaonkar V, Schmid M, Zubieta C, Conn SJ. A circRNA from SEPALLATA3 regulates splicing of its cognate mRNA through R-loop formation., 2017, 3(5): 17053.
[27] Xu HY, Guo S, Li W, Yu P. The circular RNA Cdr1as, via miR-7 and its targets, regulates insulin transcription and secretion in islet cells., 2015, 5(1): 12453.
[28] Li H, Wei XF, Yang JM, Dong D, Hao D, Huang YZ, Lan XY, Plath M, Lei CZ, Ma Y, Lin FP, Bai YY, Chen H. CircFGFR4 promotes differentiation of myoblasts via binding miR-107 to relieve its inhibition of Wnt3a., 2018, 11: 272–283.
[29] Feng Y, Hu AP, Li D, Wang JQ, Guo YH, Liu Y, Li HJ, Chen YJ, Wang XJ, Huang K, Zheng LD, Tong QS. Circ-HuR suppresses HuR expression and gastric cancer progression by inhibiting CNBP transactivation., 2019, 18(1): 158.
[30] Dong R, Zhang XO, Zhang Y, Ma XK, Chen LL, Yang L. CircRNA-derived pseudogenes., 2016, 26(6): 747–750.
[31] Chen GW, Shi YT, Liu MM, Sun JY. circHIPK3 regulates cell proliferation and migration by sponging miR-124 and regulating AQP3 expression in hepatocellular carcinoma., 2018, 9(2): 175.
[32] Haimov O, Sinvani H, Dikstein R. Cap-dependent, scanning-free translation initiation mechanisms., 2015, 1849(11): 1313–1318.
[33] Liu XQ, Gao YB, Zhao LZ, Cai YC, Wang HY, Miao M, Gu LF, Zhang HX. Biogenesis, research methods, and functions of circular RNAs., 2019, 41(6): 469–485.刘旭庆, 高宇帮, 赵良真, 蔡宇晨, 王汇源, 苗苗, 顾连峰, 张航晓. 环状RNA的产生、研究方法及功能. 遗传, 2019, 41(6): 469–485.
[34] Abe N, Matsumoto K, Nishihara M, Nakano Y, Shibata A, Maruyama H, Shuto S, Matsuda A, Yoshida M, Ito Y, Abe H. Rolling circle translation of circular RNA in living human cells., 2015, 5: 16435.
[35] Ruggero D, Sonenberg N. The Akt of translational control., 2005, 24(50): 7426–7434.
[36] Zhao JW, Lee EE, Kim J, Yang R, Chamseddin B, Ni CY, Gusho E, Xie Y, Chiang CM, Buszczak M, Zhan XW, Laimins L, Wang RC. Transforming activity of an oncoprotein-encoding circular RNA from human papillomavirus., 2019, 10(1): 2300.
[37] Zheng Y, Ji PF, Chen S, Hou LL, Zhao FQ. Reconstruction of full-length circular RNAs enables isoform-level quantification., 2019(1), 11: 2.
[38] Zhang JY, Chen S, Yang JW, Zhao FQ. Accurate quantification of circular RNAs identififies extensive circular isoform switching events., 2020, 11: 90.
[39] Stothard P. The sequence manipulation suite: JavaScript programs for analyzing and formatting protein and DNA sequences., 2000, 28(6): 1102–1104.
[40] Kong L, Zhang Y, Ye ZQ, Liu XQ, Zhao SQ, Wei LP, Gao G. CPC: assess the protein-coding potential of transcripts using sequence features and support vector machine., 2007, 35: 345–349.
[41] Wang LG, Park HJ, Dasari S, Wang SQ, Kocher JP, Li W. CPAT: coding-potential assessment tool using an alignment- free logistic regression model., 2015, 41(6): e74.
[42] Zhao J, Wu J, Xu T, Yang Q, He J, Song X. IRESfinder: Identifying RNA internal ribosome entry site in eukaryotic cell using framed k-mer features.,2018,45(7): 403–406.
[43] Mokrejs M, Vopálenský V, Kolenaty O, Masek T, Feketová Z, Sekyrová P, Skaloudová B, Kríz V, Pospísek M. IRESite: the database of experimentally verified IRES structures., 2006, 34: 125–130.
[44] Zhou Y, Zeng P, Li YH, Zhang Z, Cui Q. SRAMP: prediction of mammalian N6-methyladenosine (m6A) sites based on sequence-derived features., 2016, 44(10): e91.
[45] Wei LY, Chen HR, Su R. M6APred-EL: A sequence-based predictor for identifying N6-methyladenosine sites using ensemble learning., 2018, 12: 635–644.
[46] Finn RD, Bateman A, Clements J, Coggill P, Eberhardt RY, Eddy SR, Heger A, Hetherington K, Holm L, Mistry J, Sonnhammer ELL, Tate J, Punta M. Pfam: the protein families database., 2013, 42(1): 222– 230.
[47] Sun P, Li GL. CircCode: a powerful tool for identifying circRNA coding ability., 2019, 10: 981.
[48] Chen XP, Han P, Zhou T, Guo XJ, Song XF, Li Y. circRNADb: a comprehensive database for human circular RNAs with protein-coding annotations., 2016, 6: 34985.
[49] Liu M, Wang Q, Shen J, Yang BB, Ding XM. Circbank: a comprehensive database for circRNA with standard nomenclature., 2019, 7: 899–905.
[50] Glažar P, Papavasileiou P, Rajewsky N. CircBase: a database for circular RNAs., 2014, 20(11): 1666– 1670.
[51] Xia SY, Feng J, Chen K, Ma YB, Gong J, Cai FF, Jin YX, Gao Y, Xia LJ, Chang H, Wei L, Han L, He CJ. CSCD: a database for cancer-specific circular RNAs., 2017, 46(D1): 925–929.
[52] Xia SY, Feng J, Lei LJ, Hu J, Xia LJ, Wang J, Xiang Y, Liu LJ, Zhong S, Han L, He CJ. Comprehensive characterization of tissue-specific circular RNAs in the human and mouse genomes., 2016, 18(6): 984–992.
[53] Dong R, Ma XK, Li GW, Yang L. CIRCpedia v2: an updated database for comprehensive circular RNA annotation and expression comparison., 2018, 16(4): 226–233.
[54] Panda AC, Dudekula DB, Abdelmohsen K, Gorospe M. Analysis of circular RNAs using the web tool CircInteractome., 2018, 1724: 43–56.
[55] Liu YC, Li JR, Sun CH, Andrews E, Chao RF, Lin MF, Weng SL, Hsu SD, Huang CC, Cheng C, Liu CC, Huang HD. CircNet: a database of circular RNAs derived from transcriptome sequencing data., 2016, 44(1): 209–215.
[56] Chu Q, Zhang X, Zhu X, Liu C, Mao L, Ye C, Zhu QH, Fan L. PlantcircBase: a database for plant circular RNAs., 2017, 10(8): 1126–1128.
[57] Ghosal S, Das S, Sen R, Basak P, Chakrabarti J. Circ2Traits: a comprehensive database for circular RNA potentially associated with disease and traits., 2013, 4: 283.
[58] Ji PF, Wu WY, Chen S, Zheng Y, Zhou L, Zhang JY, Cheng H, Yan J, Zhang SG, Yang PH, Zhao FQ. Expanded expression landscape and prioritization of circular RNAs in mammals., 2019, 26(12): 3444–3460.e5.
[59] Ingolia NT, Lareau LF, Weissman JS. Ribosome profiling of mouse embryonic stem cells reveals the complexity and dynamics of mammalian proteomes., 2011, 147(4): 789–802.
[60] Ingolia NT, Ghaemmaghami S, Newman JRS, Weissman JS. Genome-wide analysisof translation with nucleotide resolution using ribosome profiling., 2009, 324(5924): 218–223.
[61] Xia X, Li XX, Li FY, Wu XJ, Zhang ML, Zhou HK, Huang NN, Yang XS, Xiao FZ, Liu DW, Yang LX, Zhang N. A novel tumor suppressor protein encoded by circular AKT3 RNA inhibits glioblastoma tumorigenicity by competing with active phosphoinositide-dependent Kinase-1., 2019, 18(1): 131.
[62] Tenenbaum SA, Lager PJ, Carson CC, Keene JD. Ribonomics: identifying mRNA subsets in mRNP complexes using antibodies to RNA-binding proteins and genomic arrays., 2002, 26(2): 191–198.
[63] Marín-Béjar O, Huarte M. RNA pulldown protocol fordetection and identification of RNA-associated proteins., 2015, 1206: 87–95.
[64] Zhang ML, Zhao K, Xu XP, Yang YB, Yan S, Wei P, Liu H, Xu JB, Xiao FZ, Zhou HK, Yang XS, Huang NN, Liu JL, He KJ, Xie KP, Zhang G, Huang SY, Zhang N. A peptide encoded by circular form of LINC-PINT suppresses oncogenic transcriptional elongation in glioblastoma., 2018, 9(1): 4475.
[65] Liang WC, Wong CW, Liang PP, Shi M, Cao Y, Rao ST, Tsui SKW, Waye MMY, Zhang Q, Fu WM, Zhang JF. Translation of the circular RNA circβ-catenin promotes liver cancer cell growth through activation of the Wnt pathway., 2019, 20(1): 84.
[66] Gu CH, Zhou NC, Wang ZY, Li GR, Kou YP, Yu SL, Feng YJ, Chen L, Yang JJ, Tian FY. CircGprc5a promoted bladder oncogenesis and metastasis through Gprc5a- targeting peptide., 2018, 13: 633–641.
[67] Van Heesch S, Witte F, Schneider-Lunitz V, Schulz JF, Adami E, Faber AB, Kirchner M, Maatz H, Blachut S, Sandmann CL, Kanda M, Worth CL, Schafer S, Calviello L, Merriott R, Patone G, Hummel O, Wyler E, Obermayer B, Mücke MB, Lindberg EL, Trnka F, Memczak S, Schilling M, Felkin LE, Barton PJR, Quaife NM, Vanezis K, Diecke S, Mukai M, Mah N, Oh S-J, Kurtz A, Schramm C, Schwinge D, Sebode M, Harakalova M, Asselbergs FW, Vink A, de Weger RA, Viswanathan S, Widjaja AA, Gärtner-Rommel A, Milting H, dos Remedios C, Knosalla C, Mertins P, Landthaler M, Vingron M, Linke WA, Seidman JG, Seidman CE, Rajewsky N, Ohler U, Cook SA, Hubner N. The translational landscape of the human heart., 2019, 178(1): 242–260.
[68] Zhang ML, Huang NN, Yang XS, Luo JY, Yan S, Xiao FZ, Chen WP, Gao XY, Zhao K, Zhou HK, Li ZQ, Ming L, Xie B, Zhang N. A novel protein encoded by the circular form of the SHPRH gene suppresses glioma tumorigenesis., 2018, 37(13): 1805–1814.
[69] Wesselhoeft RA, Kowalski PS, Anderson DG. Engineering circular RNA for potent and stable translation in eukaryotic cells., 2018, 9(1): 2629.
[70] Wesselhoeft RA, Kowalski PS, Parker-Hale FC, Huang YX, Bisaria N, Anderson DG. RNA circularization diminishes immunogenicity and can extend translation duration., 2019, 74(3): 508–520.
Progress on translation ability of circular RNA
Shuailong Zheng, Li Li, Hongping Zhang
With the rapid development of high-throughput sequencing technology and translatome studies, the translational ability of circular RNA (circRNA) has gradually attracted much attention. Previous studies have shown that circRNA itself can be translated into proteins, whose function is closely related to the occurrence and development of human diseases. And it is expected to become an ideal substitute for mRNA, which can be widely used in protein engineering in the future. In this review, we systematically summarize the sources, biogenesis and features of circRNA, the driving models of circRNA translation,identification and functional verification of circRNA translation. We also sum up the latest research progress of circRNA translation in human diseases and its application in protein engineering, and make prospective anticipations for future research directions, which may offer more theoretical references for related researches in this field.
circular RNA; protein translation; identification methods
2020-01-07;
2020-03-24
国家自然科学基金项目(编号:31772578,31672402)和四川省科技支撑计划项目(编号:2016NYZ0045)资助[Supported by the National Natural Science Foundation of China (Nos. 31772578,31672402), and the Science and Technology Program of Sichuan Province (No. 2016NYZ0045)]
郑帅龙,在读硕士研究生,专业方向:动物遗传育种。E-mail: 382581626@qq.com
李利,博士,教授,博士生导师,研究方向:动物遗传育种。E-mail: lily@sicau.edu.cn张红平,博士,教授,博士生导师,研究方向:动物遗传育种。E-mail: zhp@sicau.edu.cn
10.16288/j.yczz.19-354
2020/4/2 13:18:10
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20200402.1034.003.html
(责任编委: 赵方庆)

