代谢组学技术发展及其在农业动植物研究中的应用
田菁,王宇哲,3,闫世雄,孙帅,贾俊静,胡晓湘
综 述
代谢组学技术发展及其在农业动植物研究中的应用
田菁1,2,王宇哲1,2,3,闫世雄4,孙帅4,贾俊静4,胡晓湘1,2
1. 中国农业大学农业生物技术国家重点实验室,北京 100193 2. 中国农业大学生物学院,北京 100193 3. 中国农业大学动物科学技术学院,北京 100193 4. 云南农业大学动物科学技术学院,昆明 650201
代谢组学是依赖灵敏、稳定的分析流程和数据库,利用色谱–质谱联用、核磁共振技术对生物体内以及生物样品所有的小分子代谢物进行鉴定和定量分析的学科,在医学、食品科学、畜牧学、植物学等领域得到广泛应用。代谢组学方法可将代谢物种类和含量的变化与生物表型变化建立更直接的联系,因此代谢组学逐渐成为继基因组学、转录组学、蛋白组学后对复杂性状系统解析的新的研究手段。本文介绍了代谢组学常用分析策略、检测平台和常用数据库。在此基础上,综述了代谢组学在农业动物重要经济性状代谢分子鉴定、疾病诊断、肉品质及动物制品安全检测等领域取得的进展,并总结了利用代谢组学、转录组学和基因组学等多组学研究在动植物重要性状的发育、形成和解析等领域取得的最新成果。代谢组学与其他多组学方法整合分析,可以更全面地阐述各类复杂性状的遗传机制,有助于完善“突变–基因–表达–代谢–表型”的完整生物学过程,为复杂性状的机理解析提供新方法,为新型农业育种提供新思路。
代谢组学; 广泛靶向代谢组学;代谢组检测平台;农业动物;多组学联合分析
代谢组是指生物体或细胞内所有小分子代谢物的集合,其分子量通常小于1000 Da,参与生物体的新陈代谢,维持生物体正常生长发育[1]。代谢组学来源于“代谢组”一词,1997年Oliver等[2]正式提出了代谢组学(metabolomics)概念,主要是研究细胞、生物流体、组织或生物体中发生的代谢过程,是一个静态的概念,对限定条件下的特定生物样品中所有代谢物定性、定量分析,又称为代谢物组学;随后Nicholson等[3]在1999年从另一角度定义代谢组学(metabonomics),即生物体对病理生理应激或遗传修饰引起的代谢反应的定量测定,是生物系统进行整体及其动态变化规律的研究,通常应用于人类营养和对药物或疾病的反应研究中,他们应用代谢组学在疾病诊断和药物筛选等研究[4,5]中取得进展。代谢组学是研究细胞、组织和器官中所有小分子代谢物集合的一门学科,通过对小分子物质进行定性定量分析指示生物体的生化状态,同时也可以寻找代谢物与机体生理病理、遗传环境的变化等之间的相互关联[6]。
随着组学时代的到来,代谢组学已经成为除基因组学、转录组学、蛋白质组学外的重要组学分支,共同组成系统生物学[7]。与其他组学相比,代谢组学具有以下特点:(1)小分子代谢物是基因组下游产物[8],可以直接、准确地反映生物体的生理病理状态;(2)代谢组是与表型组最接近的组学,可以利用代谢物的变化表征生物体不同的表型特征,得到更精确的表型数据;(3)代谢物在各个生物体中是类似的,所以代谢组学研究中采用的技术更容易在各个领域中通用。本文将从代谢组学的常用分析策略、检测平台的发展、代谢组学在农业动物不同研究领域中的应用及代谢组与其他多组学联合应用等4个方面进行综述,以期更好地理解代谢组学的发展现状和应用前景。
1 代谢组学常用分析策略
对代谢物进行检测的目的不同,选择的策略也存在较大差异,目前常用的代谢组学研究策略包括非靶向代谢组学(non-targeted metabolomics)、靶向代谢组学(targeted metabolomics)和拟靶向/广泛靶向代谢组学(pseudotargeted/widely targeted metabolomics)等。
1.1 非靶向代谢组学
非靶向代谢组学又被称为发现代谢组学,对生物样本的内源性物质进行全面系统的测定分析。主要特点为对样本中代谢物分析的无偏向性,可以对样本小分子物质整体轮廓进行表征和筛选,通过生物信息方法进行差异分析或通路分析。目前非靶向代谢组学被广泛应用于生物标志物发现[9]、疾病诊断[10]、食品风味和安全[11,12]等多个领域。
常见的代谢组学分析样本包括血浆、血清、尿液、组织和细胞等[7],往往需要使用多个生物学重复来减小样本个体差异带来的误差。样品前处理即代谢物的提取对代谢物的检测分析尤为重要,其中对小分子代谢物的累计富集和去除干扰测定的杂质物质是关键步骤[13]。目前主要的样本前处理方法包括液-液萃取(liquid-liquid extraction, LLE)、固相萃取(solid-phase extraction, SPE)、超临界流体萃取(supercritical-fluid extraction, SFE)、加速溶剂萃取(pressurized-liquid extraction, PLE)、微波辅助萃取(microwave-assisted extraction, MAE)和蛋白质沉淀等[14]。
代谢组数据一般采用多元数据分析策略,通常包括主成分分析(principal-component analysis, PCA)、偏最小二乘判别分析(partial least squares discrimination, PLS-DA)和正交偏最小二乘判别分析(orthogonal partial least squares discrimination, OPLS-DA)等[15],结合检验和变量权重重要性排序(variable importance projection, VIP)值筛选差异代谢物或潜在生物标志物[16],同时利用筛选得到的代谢物可以进行代谢通路富集、聚类等分析开展后续研究。其主要分析流程如图1所示。
由于复杂样本中代谢物质种类多、每种代谢物含量差异大且性质复杂,很难有一种提取方法可以将其中所有的代谢物提取出来[17],同时非靶向代谢组学通常使用低分辨检测平台进行检测。因此,受到以上条件的限制,某些代谢物信息容易丢失(尤其低丰度代谢物),代谢物的鉴定和定量的准确性有待验证,需要开发适用性更强的流程来满足目前对于代谢组学研究中的要求。
1.2 靶向代谢组学
靶向代谢组学分析是对关注的几个、几十个或者几类已知的目标化合物进行测定的技术。通常是利用非靶向代谢组学发现差异代谢物,再使用靶向代谢组学进一步系统验证[18]。其主要特点是对关注代谢物进行准确鉴定和绝对定量,在分析上更具有针对性,与非靶向代谢组学优势互补,被主要应用于药物开发[7]、代谢疾病诊断及机制研究[19,20]等多个研究领域。
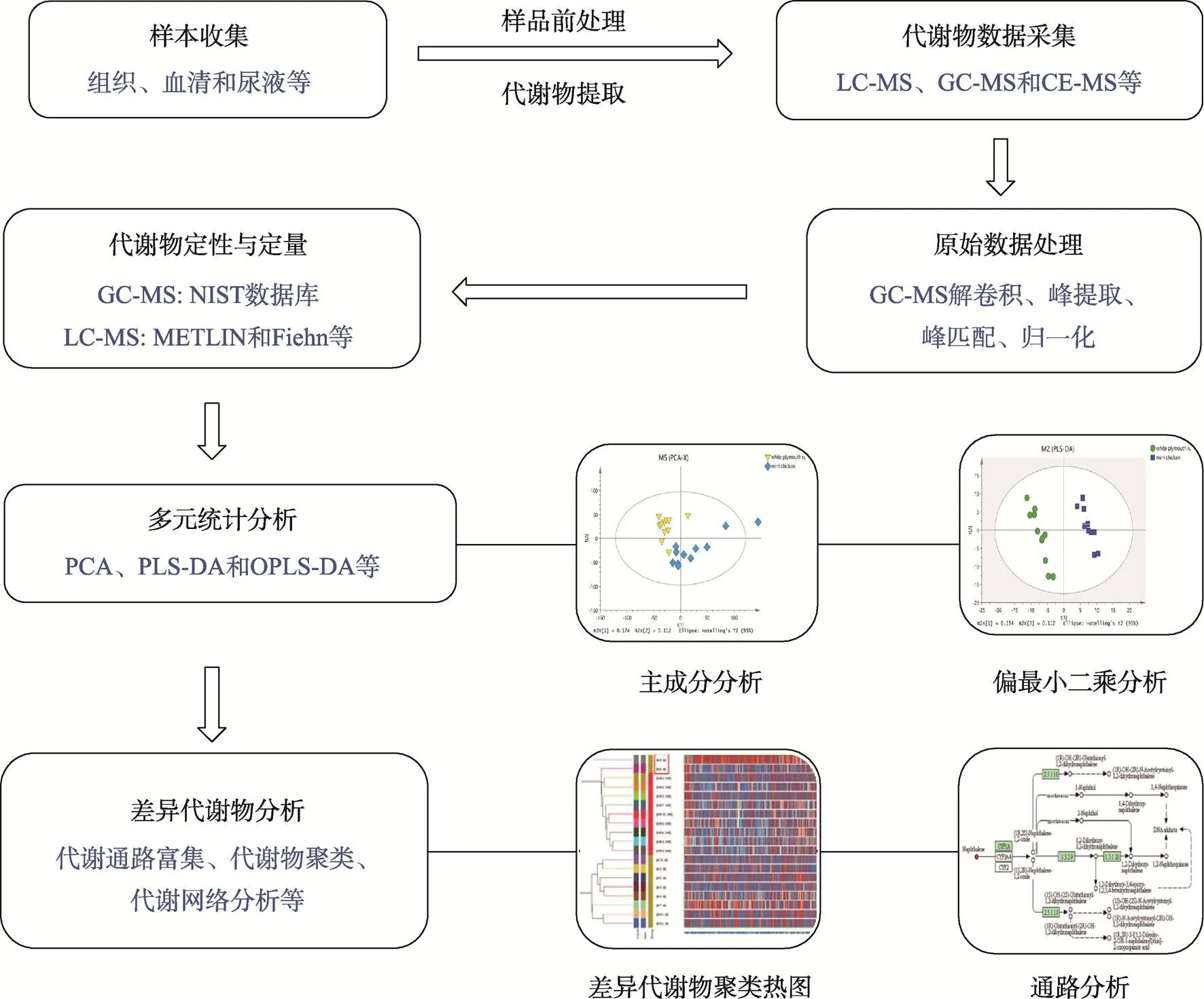
图1 非靶向代谢组学主要分析流程
靶向代谢组学分析样本与非靶向代谢组学类似,但在前处理时可以根据要检测代谢物的特性选择合适的代谢物提取方法,使得检测更具可靠性和准确性。由于靶向代谢组学中只对关注的已知代谢物进行分析及生物学功能阐述,因此数据处理流程相对于非靶向代谢组学来说更加简单方便。在检测到关注的代谢物后,进行通路富集及该代谢物在生物体中合成机制等研究。
1.3 拟靶向/广泛靶向代谢组学
拟靶向/广泛靶向代谢组学[21]分析是一种结合非靶向代谢组学和靶向代谢组学优点的新方法,可以同时定性和精确定量数百种已知代谢物并对数千种已知及未知代谢物进行定量。通常使用超高相液相质谱–高分辨质谱联(ultra performance liquid chromatography high resolution mass spectrometer, UPLC- HRMS)和超相液相质谱–三重四级杆质谱联用(UPLC-MS/MS)平台实现代谢物信息采集。其主要由两部分搭建而成:第一部分是通过传统质谱或高分辨质谱进行代谢物采集并建立二级谱图(MS/MS)数据库,目前主要包括全扫描和ddMS2模式结合方法[22]、SWATH-MS方法[23]和基于多离子监控模式–增强碎片离子(multiple ion monitoring enhanced product ions, MIM-EPI)方法[24,25]等;第二部分是将待测样本进行多反应监测(multiple reaction monitoring, MRM)模式[26]扫描后与第一部分中建立的数据库进行比对,对待测样本中代谢物进行定性及准确定量,实现拟靶向/广泛靶向代谢组的目的。该方法兼具了非靶向代谢组学和靶向代谢组学各自的优点,已经被广泛用于代谢组学研究中。其主要分析流程如图2所示。
2 代谢组学检测平台发展及常用数据库
由于代谢物具有种类繁多、性质差异大、浓度范围分布广等特点,代谢组学发展出不同的检测平台[27],例如核磁共振(nuclear magnetic resonance, NMR)平台、液相色谱–质谱联用(liquid chromatography-mass spectrometry, LC-MS)平台、气相色谱–质谱联用(gas chromatography-mass spetrometry, GC- MS)平台、毛细管电泳–质谱联用(capillary electrophoresis-mass spectrometry, CE-MS)平台等。NMR是最早应用于代谢组学研究的平台,主要应用于代谢物结构的解析,但其检测灵敏度低,目前已经不能满足现在代谢组学研究。质谱具有高灵敏性的特点,已经成为代谢组学研究的主要分析技术,LC-MS[28]、GC-MS[29]及CE-MS[30]已经普遍应用于代谢组学研究。近年来随着高分辨质谱仪的推出及应用,对代谢物检测荷质比(m/z)可以精确到小数点后第4位,同时进行二级谱MS/MS信息的采集,利用同位素峰比例信息,进一步提升了代谢物定性的准确性,为代谢组学发展提供更好的技术平台。目前市场上主流的高分辨质谱仪主要分为四级杆飞行时间质谱(time of flight mass spectrometer, QTOF-MS)和静电场轨道阱质谱(electrostatic obitrap mass spectrometer, Obitrap-MS),其性能特点见表1。
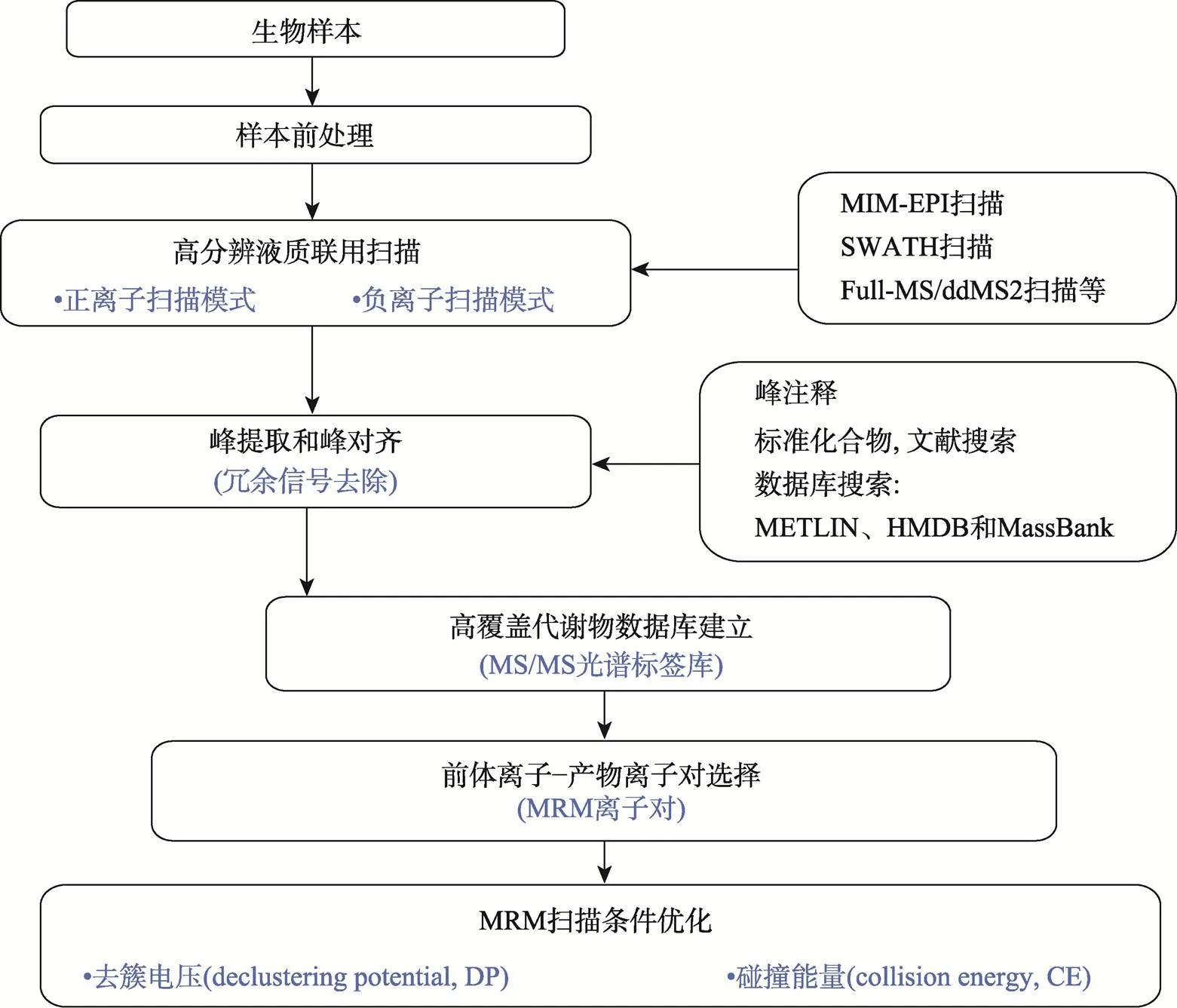
图2 广泛靶向代谢组一般分析流程
代谢物定性的准确性不仅取决于质谱仪的检测分辨能力,对应的代谢物数据库也至关重要。随着代谢组学研究的发展,代谢物数据库的构建逐渐完善。GC-MS检测到的代谢物通常参考NIST和FiehnLib数据库进行代谢物定性;LC-MS通常使用METLIN、HMBD数据库及部分自建库;脂质物质的种类繁多、结构特殊,通常使用脂质组专用的数据库LipidSearch、LMSD等进行比对确认。通过多元统计分析得到关注的代谢物或差异代谢物后往往还需要进行通路富集分析,揭示影响复杂生物过程的多种因素,通常使用KEGG和Reactome等数据库进行分析。随着代谢组学逐步发展,将多种数据库资源整合已经成为趋势,进一步提高数据资源利用率,其中跨库原始数据检索平台-MetabolomeXchange (http://www.metabolomexchange.org/site)于2015年由多家机构共同建立,为数据库资源的整合和扩展提供快捷途径。代谢组学中常用数据库见表2。
3 代谢组学在农业动物研究领域中的应用
农业动物具有重要的经济价值,为人类提供肉、蛋、奶等农副产品,是人类日常营养物质获取的重要来源。随着代谢组学技术的发展与应用,目前已经在农业动物差异性状表征与重要经济性状代谢分子鉴定、农业动物疾病诊断、畜禽肉质风味及动物制品安全检测等多个领域中得到广泛应用。
3.1 代谢组在农业动物差异性状表征、重要经济性状代谢分子鉴定中的应用
通过对不同品种(系)、性别畜禽的血清、尿液和组织等生物样本进行代谢物检测,可以筛选出一些小分子作为区分该品种(系)、性别的生物标志物,为不同品种(系)、性别畜禽差异性状表征提供理论依据。Bovo等[41]利用靶向代谢组学方法检测来自意大利的两个商品猪品种—大白猪(Large White)和杜洛克猪(Duroc)血浆和血清中180种代谢物,其中鞘磷脂、生物胺(犬尿氨酸和乙酰鸟氨酸)等代谢物在两个猪种中有很大差异,可以作为区别两品种猪的生物标记物;He等[42]检测了肥胖型猪—宁乡猪(Ningxiang strain)和瘦肉型杂交猪(Duroc×Landrace×Large Yorkshire strain)的血清代谢物,在肥胖型猪中胰岛素、胰高血糖素、脂质、肌醇和丙氨酸等代谢物含量均高于瘦肉型猪,但其血清中尿素和葡糖糖的含量较低;Ji等[43]利用LC-MS检测了来航鸡(Leghorn)、Fayoumi瘦肉型鸡和商业肉鸡的脂肪组织中92种代谢物,与商业肉鸡相比,其他两个品种鸡脂肪组织中有47种代谢物发生变化,其中肉碱、乙酰肉碱、鸟苷、胞嘧啶、腺苷、磷酸戊糖等多种代谢物含量有2倍以上的改变;Beauclercq等[44]对肌糖原含量不同(pHu–和pHu+)的两个品系鸡的血清和肌肉进行代谢物检测,在pHu–中鉴定到碳水化合物丰度高,pHu+的代谢物与肌糖原分解和氧化应激相关,最终鉴定到15个代谢标志物用于鸡肉质鉴定;Bovo等[45]对阉割公猪和母猪的血浆进行靶向代谢组学检测,共检测到132个代谢物,其中85种代谢物可以体现出两组猪之间的差异,在阉割公猪中酰基肉碱、生物胺、氨基酸分解等与脂质沉积相关的代谢物丰度高。
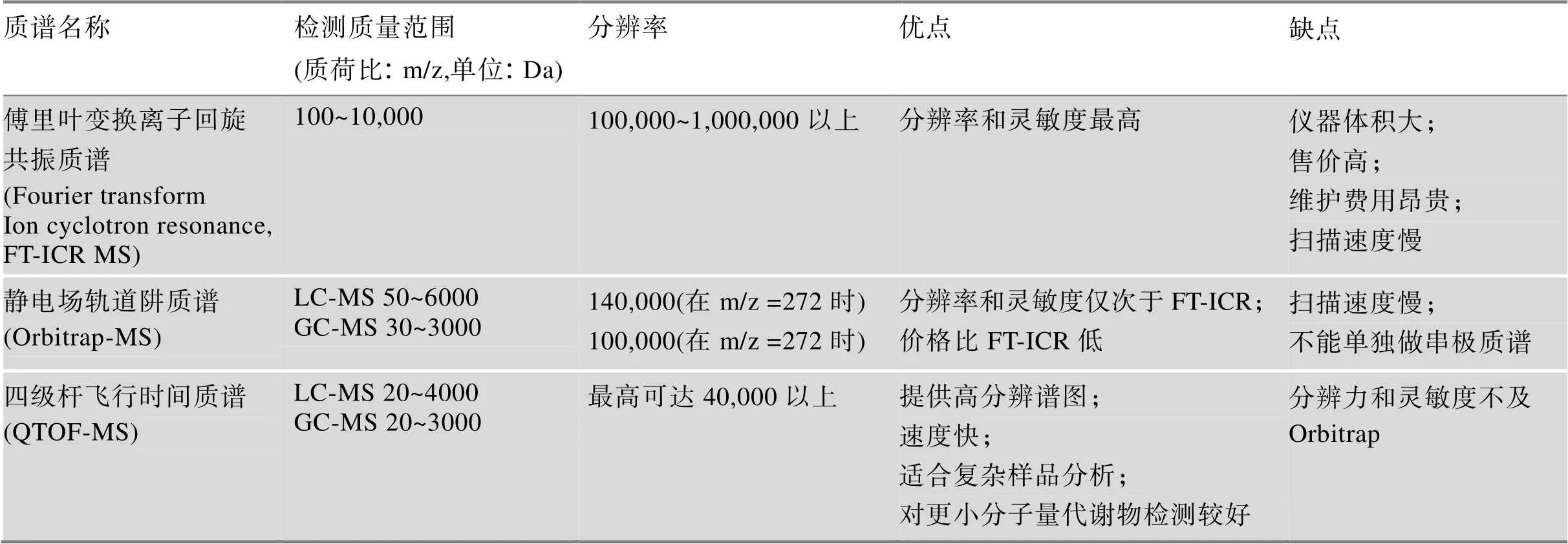
表1 不同高分辨质谱仪性能特点
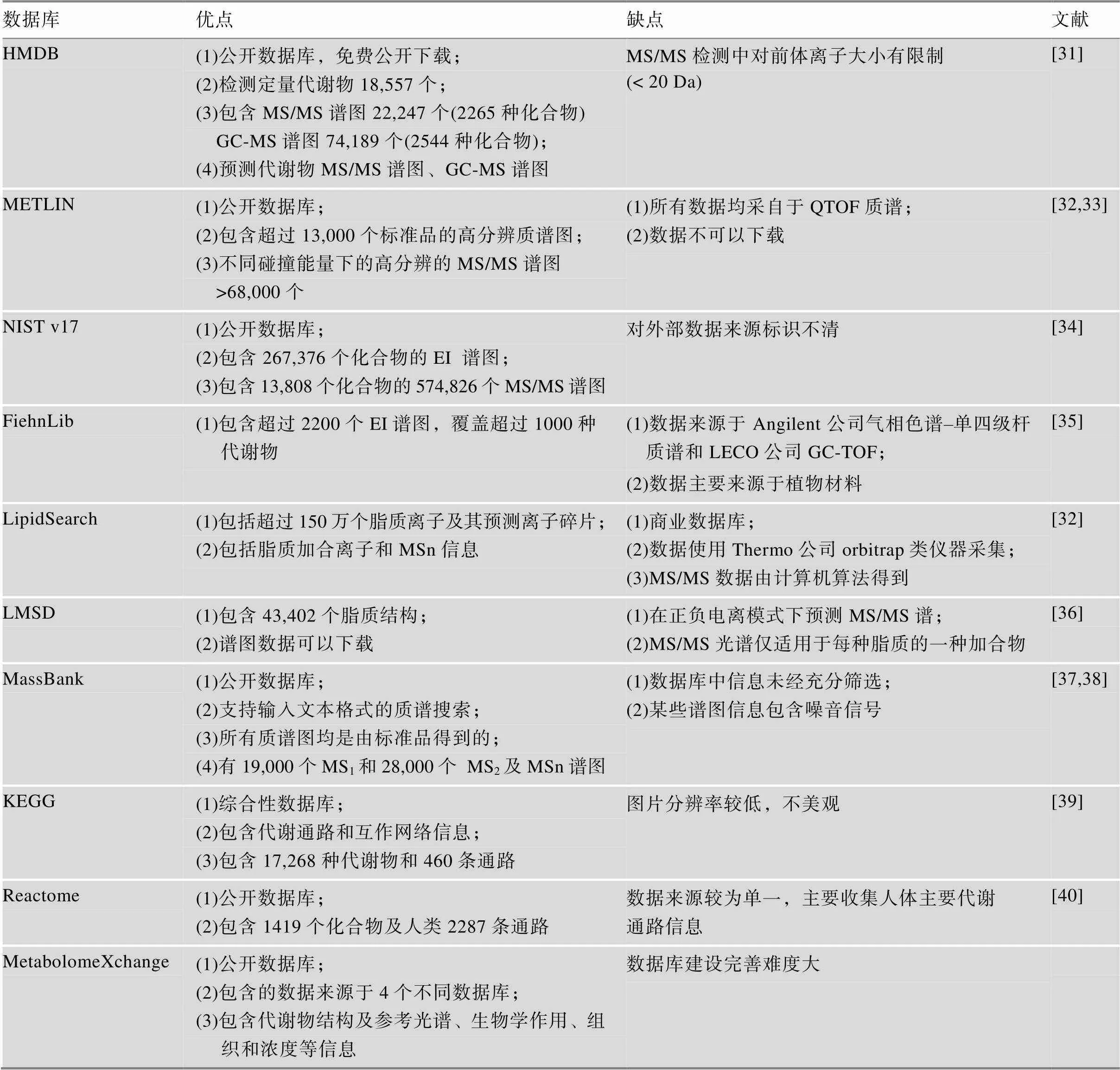
表2 代谢组学中常见数据库特点
代谢小分子物质可以反映生物体的生理状态和表型,利用代谢物表型可以更加准确地表征复杂表型。Rohart等[46]利用NMR对大白猪、长白猪(Landrace)和皮特兰猪(Pietrain)的血浆进行代谢物检测,结果显示肌酸酐、缬氨酸、柠檬酸、β-丙氨酸、乳酸、丙氨酸和异亮氨酸等代谢物与猪的瘦肉率性状相关,可以用于猪瘦肉率预测;Picone等[47]对大白猪、长白猪、杜洛克母猪的初乳代谢物进行检测,乳糖可以区分3种母猪品种,醋酸盐、牛磺酸、二甲胺和顺式-酸酯与仔猪体重增加和存活率有关;Karisa等[48]利用NMR测定了16头纯种安格斯牛(Angus)和10头杂交肉牛血浆代谢组,共检测到45种代谢物,其中肌酸、肉毒碱和马尿酸与肉牛剩余采食量(residual feed intake, FRI)性状相关,解释了32%的FRI表型变异,可以作为肉牛选育的分子标志物;Sun等[49]对饲喂苜蓿和玉米秸秆的泌乳牛的瘤胃液、牛奶、血清和尿液进行代谢物检测,在每种生物流体中分别发现55、8、28和31种差异代谢物,其差异代谢物主要参与甘氨酸、丝氨酸、苏氨酸、酪氨酸等氨基酸代谢通路,可以作为提高奶牛产奶量和乳蛋白质质量的生物标记物;Beauclercq等[50]使用NMR检测鸡血清、回肠和盲肠代谢物,利用氮校正表观代谢能(apparent metabolisable energy corrected to zero nitrogen retention, AMEn)来评估鸡对饲料的消化效率(digestible energy, DE),发现与AMEn关联最强的代谢物在血清中是脯氨酸,回肠中是富马酸盐,盲肠中是葡糖糖,为鸡DE的预测提供新的见解。对农业动物重经济性状进行标志代谢物筛选,将标志代谢物应用于标记辅助选择可提高选择准确性。
3.2 代谢组在农业动物疾病诊断中的应用
在畜禽养殖业发展过程中,疾病的发生往往对畜禽生长、经济效益产生很大的影响,但是很多疾病的防治及治疗难度很大,而机体在发生疾病前后往往会引起体内代谢物水平的改变,应用代谢组学可以进行早期诊断的特点,通过检测血液、尿液等生物样本鉴定与疾病相关的生物标志物。Gong等[51]对感染猪瘟后3天和7天龄的断奶仔猪血清进行代谢物检测,代谢物主要富集在色氨酸分解代谢、苯丙氨酸代谢、脂肪代谢、核苷酸代谢等关键代谢通路上,同时观察到与肠道微生物相关的代谢物,对仔猪猪瘟疾病认识提供新的见解;Welle等[52]将猪结肠与猪流行性乙型痢疾杆菌共培养以模拟猪感染痢疾杆菌的肠道环境,并对暴露8 h的结肠外植体进行代谢物检测,结果显示感染流行性乙型痢疾的外植体上皮细胞明显坏死,l-瓜氨酸和IL-1α水平升高,为猪痢疾病理生理机制解析提供科学参考;Hailemariam等[53]对12头奶牛过渡期的4个时间点的120种血浆代谢物进行测定,肉毒碱、丙酰肉碱、溶血磷脂酰胆碱酰基(C14: 0)3种代谢物发生显著改变,利用这3种血浆代谢物就可以对在围产期可能患病的奶牛进行预测;Zhang等[54]对生产前8周和4周奶牛、产后患有酮症的奶牛以及生产后4周和8周的奶牛靶向检测检测128种血清代谢物,在整个检测周期中氨基酸、甘油磷脂、鞘脂、酰基肉碱和生物胺等代谢物水平发生显著变化,鉴定到可能作为酮症风险预测性生物标志物,为奶牛酮症预测提供科学依据;Shen等[55]利用LC-MS对患有腹水综合征的鸡和健康鸡的肝脏代谢物进行检测,结合血液生化和病理学结果表明,牛磺酸脱氧胆酸、胆酸葡萄糖醛酸、甘胆酸、LysoPC(15: 0)和牛磺胆酸被鉴定为腹水综合征的潜在生物标志物,且代谢物与脂质代谢紊乱有关,有助于了解鸡腹水综合征疾病的机制;Lu等[56]对处于热应激状态的鸡和健康鸡的257种血清代谢物进行了检测,其中78种代谢物存在显著差异且热应激肉鸡处于负能量平衡状态,不能有效的调动脂肪,影响肉鸡生长。以上研究证明代谢组可以对农业动物生产疾病引起的机体代谢物变化进行综合分析,筛选到的标志代谢物可用于疾病诊断和预测等。
3.3 代谢组在畜禽肉质风味及动物制品安全检测中的应用
通过动物育种后产生的优良品种主要为人类提供丰富的食物,而影响食物风味的挥发性物质主要是分子量小于1000 Da的小分子物质。以往宏观的表型测定方法无法表征这类特殊表型,随着代谢组检测平台以及代谢物数据库的日益完善,可以对该类物质进行准确测定并深入研究,为动物产品风味改良提供代谢物表型。Romarathnam等[12]研究表明醛类(烯醛、二烯醛)是鸡脂肪受热时的特征香味物;Wasseriwan等[57]研究认为杂环化合物风味阈值低,是烤肉中最重要的风味呈味物,主要包括呋喃、噻吩、噻唑、吡啶、吡咯和吡嗪类化合物;Liu等[58]利用GC-O-MS对4种北京烤鸭的鸭皮和鸭胸肉检测到42种挥发性香味物质,主要分为醛、酮、醇、酸、酚、含硫化合物和含氮化合物7类,通过香气重组和遗漏实验及感官评价最终确定了9种对烤鸭风味有显著贡献的香味化合物:2-糠基硫醇、二甲基三硫醚、己醛、庚醛、辛醛、壬醛、甲硫氨酸、1-辛烯-3-醇和(E, E)-2,4-癸二烯醛。
食品品质与人类健康密切相关,食品安全越来越受到人们关注,代谢组便可以对食品中含量甚微或难分离的化学污染物以及肉品掺假进行快速、准确的检测。Liu等[59]利用HPLC-MS/MS建立了快速检测猪肉、鸡蛋、牛奶等多种农产品中丙硫菌唑–硫脲的方法;Yin等[60]利用HPLC-MS/MS开发建立了快速简便检测猪肉中210种药物的残留方法,对食品安全检测和兽药使用具有重要意义;Trivedi等[61]对市场购买的猪肉碎、牛肉碎馅进行不同百分比混合样制备并进行代谢组测定,鉴定到15种差异代谢物用于鉴定区分猪肉和牛肉不同掺假比例的混合样品。
4 代谢组与其他组学联合分析研究应用
代谢物是一切生命活动的物质基础,代谢组被认为是与表型组最为接近的组学,可以利用代谢物的变化对复杂的性状进行量化表征,形成代谢物分子表型。目前主要的农业经济性状大多为复杂的数量性状,而传统的全基因组关联分析(genome wide association study, GWAS)对这类主效基因的鉴定往往停留在QTL的水平,其中一个关键因素就是生长表型为典型复合性状,其基础调控途径和代谢通路相对应的一级表型在几年前难以评估和测定,导致基因型–表型无法直接对应。使用代谢物含量作为分子表型与基因组进行关联分析可以提高检测效力和检测精度,有望对影响复杂性状主效基因的致因突变的发掘提供新的解决手段。此外,利用转录组技术对不同时空状态下的基因表达进行检测,可以得到大量差异基因和众多调控网络,同时差异积累代谢物信息可以辅助时序表达的基因进行共表达分析,对基因功能解析、代谢通路和分子生化机制的完善提供研究基础。截至目前,利用代谢组作为基因组、转录组等其他组学与表型之间的桥梁,形成“突变–基因–调控–表达–代谢–表型”的关系网络,已经在番茄()[62]、黄瓜(L.)[63]、水稻(L.)[64]等农艺植物性状研究中被广泛应用。
在农艺作物番茄中,Tieman等[62]利用GC-MS对398份番茄检测影响其风味的代谢物35个并与全基因组200多万个SNP进行mGWAS分析,找到影响番茄果糖、葡萄糖、类胡萝卜衍生挥发物的多个位点,同时提出利用具有甜味感知的挥发性物质进行番茄风味改良的建议;Zhu等[65]利用番茄的代谢组(442份材料、980种代谢物)、基因组(610份材料,2000多万SNP)、转录组(399份材料)数据进行多组学联合分析,利用鉴定到的3526个mGWAS信号和9万多个eQTL位点构建“代谢物-SNP-基因”网络,对番茄果实重量、甾体糖生物碱生物合成驯化、番茄颜色差异等进行综合解析,揭示了番茄以外观/味觉为导向的育种对番茄代谢组成的影响。总而言之,研究者找到了可以表征番茄风味的代谢物,同时也鉴定了影响上述代谢物的SNP位点,为番茄风味丢失历史以及风味改良提供了科学依据,而番茄风味的多组学分析也为农业动物的遗传育种提供了很好的借鉴。
在农业动物上利用代谢组学方法进行复杂性状遗传机制的研究正在兴起,目前主要集中在猪、牛等大型家养动物重要经济性状研究中[66,67],通过使用代谢物小分子作为表型后与基因组、转录组等多组学进行关联分析,可以更加精确地鉴定影响肉质、乳质或生长性能相关的遗传标记或者遗传规律,为农业动物分子育种提供理论支持。Son等[68]利用靶向方法测定了480只商业杜洛克猪的皮下脂肪中不同长链脂肪酸,与全基因组3.9万个SNP进行关联分析后,在14号染色体117.6~124.6 Mb检测到一个显著关联区间,其中与不同脂肪酸关联SNP位于、和等多个基因上,可用于脂肪酸去饱和水平的遗传选择;Welzenbach等[69]对97头杜洛克和皮特兰杂交猪背最长肌的126个代谢物和35种蛋白质进行检测同时利用60 K Illumina芯片进行基因分型,富集分析发现共有10条通路,其中鞘脂代谢和糖酵解/糖异生通路对肉滴水损失有影响,GWAS分析发现在18号染色体中与滴水损失、蛋白质“磷酸甘油酸突变酶2”和代谢产物甘氨酸相关的SNP,同时鉴定到与滴水损失相关的候选基因区间;Melzer等[70]利用GC-MS对1305头荷斯坦奶牛(Holstein)泌乳开始21天和120天的乳汁进行代谢组测定,共检测到187种已知代谢物和3种未知代谢物,然后利用全基因组4万多个SNP进行关联分析,分别鉴定到与牛奶脂肪含量、pH值和蛋白质分别相关的11种、10种和16种代谢物,同时找到与上述性状相关的具有遗传效应的SNP位点,为牛奶品质改良提供理论基础;Widmann等[71]对237头F2代牛的血浆进行221种代谢物的靶向检测,同时测定了牛出生后6~9月的剩余采食量(RFI)、饲料转化率(feed conversion ratio, FCR)、日常能量摄入(daily energy intake, dEI)等评估饲料效率的指标,与全基因组4万多个SNP进行GWAS分析,发现位点是影响RFI最显著的位点且与精氨酸含量显著相关,通过构建“表型–代谢型–基因型”网络寻找到对RFI有显著调控作用的候选基因(和),为农业动物饲料效率研究提供新的研究思路;Shi等[72]为研究影响鸡腹水综合征(broiler ascites syndrome, AS)的代谢标志物和差异表达基因,选择抗AS鸡和AS鸡利用LC-MS测定其血清代谢组及肝脏转录组,在实验组和对照组中鉴定到15个生物标志物,结合转录组数据表明甘油磷脂的代谢在抗AS肉鸡发育中起重要作用。更多相关研究见表3。
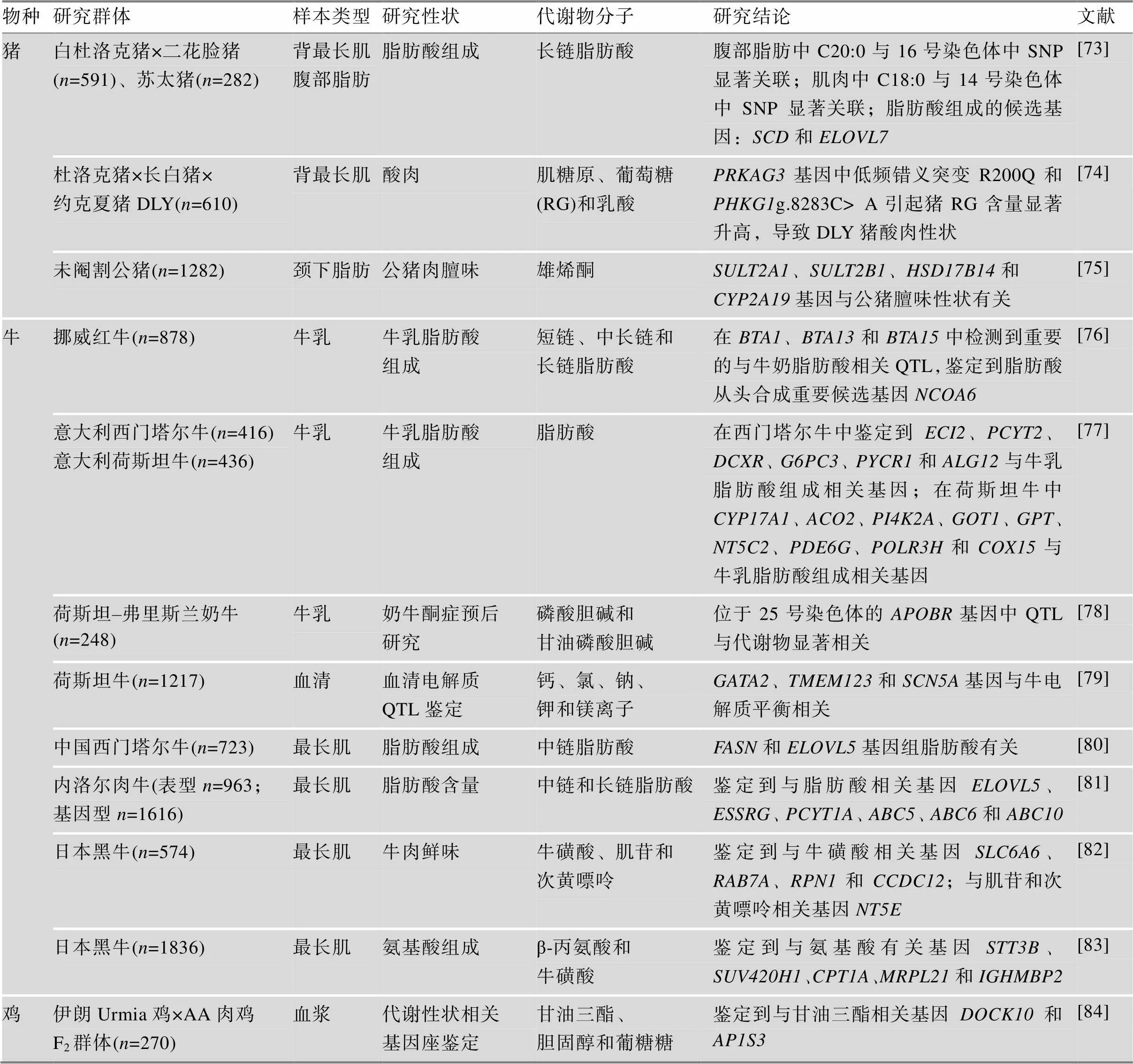
表3 代谢组与多组学联合分析在农业动物研究结果汇总
总之,通过使用代谢物小分子作为表型与基因组、转录组等多组学进行联合关联分析,可以更加精确地鉴定影响畜禽重要经济性状,例如肉质、乳质或生长性能相关的功能基因或遗传标记,进一步阐明复杂数量性状的遗传机制,为新型农业动物分子育种提供全面的理论支持。
5 结语与展望
在代谢组学近20年的发展过程中,已经被广泛应用于环境污染物识别、疾病诊断、医药研制开发、食品营养科学等领域[85]。代谢组在动植物育种研究中也发挥了巨大功效,随着近年来代谢物检测仪器的发展、代谢物数据库的完善以及不同代谢组分析策略的开发应用,代谢组学必将会在农业动植物重要复杂经济性状解析中占有不可或缺的地位。
目前,代谢组研究仍有许多问题亟待解决。就研究对象而言,人们已经将目光从较为简单地研究个体转向大样本复杂性状研究,由于代谢组会受到遗传、不同生理状态、环境刺激的影响,选择能准确表征当下状态的样本进行研究尤为重要;就代谢物检测而言,虽然近年来代谢组学技术平台得到长足进步,但是其在代谢物检测灵敏度及覆盖度上仍存在局限性,因此需要发展多平台联合检测技术实现全代谢谱的获取;就数据处理而言,不同于其他组学,代谢组数据为高维数据,需要使用多元统计分析进行信息挖掘,但是其数据处理能力还是有限,随着人工智能的兴起,有助于降低整体数据的维度从而对代谢组数据信息深入解读提供新思路。
组学时代的到来,代谢组学已经发展成了除基因组、转录组和蛋白组以外不可或缺的部分,多组学整合分析逐渐成为研究热点,有助于建立的“突变–基因–表达–代谢–表型”完整生物学网络,成为人类复杂疾病解析、动植物重要经济性状研究以及生命活动过程中复杂调控网络研究等多领域研究中新的工具和方法。
[1] Qiu DY, Huang LQ. Metabolomics--an important part of functional genomics., 2004, 2(2): 165–177.邱德有, 黄璐琦. 代谢组学研究–功能基因组学研究的重要组成部分. 分子植物育种, 2004, 2(2): 165–177.
[2] Oliver SG. Yeast as a navigational aid in genome analysis. 1996 Kathleen Barton-Wright Memorial Lecture., 1997, 143(5): 1483–1487.
[3] Nicholson JK, Lindon JC, Holmes E. 'Metabonomics': understanding the metabolic responses of living systems to pathophysiological stimulimultivariate statistical analysis of biological NMR spectroscopic data., 1999, 29(11): 1181–1189.
[4] Nicholson JK, Connelly J, Lindon JC, Holmes E. Metabonomics: a platform for studying drug toxicity and gene function., 2002, 1(2): 153–161.
[5] Lindon JC, Holmes E, Bollard ME, Stanley EG, Nicholson JK. Metabonomics technologies and their applications in physiological monitoring, drug safety assessment and disease diagnosis., 2004, 9(1): 1–31.
[6] Nicholson JK, Wilson ID. Opinion: understanding 'global' systems biology: metabonomics and the continuum of metabolism., 2003, 2(8): 668–676.
[7] Fan SC, Gao Y, Zhang HZ, Huang M, Bi HC. Untargeted and targeted metabolomics and their applications in discovering drug targets., 2017, 41(4): 263–269.范仕成, 高悦, 张慧贞, 黄民, 毕惠嫦. 非靶向和靶向代谢组学在药物靶点发现中的应用. 药学进展, 2017, 41(4): 263–269.
[8] Harrigan GG, Goodacre R. Metabolic Profiling: Its Role in Biomarker Discovery and Gene Function Analysis,2003.
[9] Bhute VJ, Bao X, Dunn KK, Knutson KR, McCurry EC, Jin G, Lee WH, Lewis S, Ikeda A, Palecek SP. Metabolomics identifies metabolic markers of maturation in human pluripotent stem cell-derived cardiomyocytes., 2017, 7(7): 2078–2091.
[10] Naviaux RK, Naviaux JC, Li K, Bright AT, Alaynick WA, Wang L, Baxter A, Nathan N, Anderson W, Gordon E. Metabolic features of chronic fatigue syndrome., 2016, 113(37): E5472–E5480.
[11] Lai BW, Liu B, Liang YK. Research progress on food fraud using non-targeted metabolomics based on high-resolution mass spectrometry., 2019, 35(2): 192–197.赖博文, 刘玢, 梁永康. 基于高分辨质谱的非靶向代谢组学在食品造假鉴定中的研究进展. 生物技术通报, 2019, 35(2): 192–197.
[12] Ramarathnam N, Rubin LJ, Diosady LL. Studies on meat flavor. 4. Fractionation, characterization, and quantitation of volatiles from uncured and cured beef and chicken., 1993, 41(6): 939–945.
[13] Zhang TL, Zhang AH, Qiu S, Yang SQ, Wang XJ, Current trends and innovations in bioanalytical techniques of metabolomics., 2016, 46(4): 342–351.
[14] Dettmer K, Aronov PA, Hammock BD. Mass spectrometry-based metabolomics., 2007, 26(1): 51–78.
[15] Li H, Jiang Y, He FC. Recent development of metabonomics and its applications in clinical research., 2008, 30(4): 389–399.李灏, 姜颖, 贺福初. 代谢组学技术及其在临床研究中的应用.遗传, 2008, 30(4): 389–399.
[16] Ke CF, Zhang T, Wu XY, Li K. A statistical method for metabolomics data analysis.,2014, 31(2): 357–359.柯朝甫, 张涛, 武晓岩, 李康. 代谢组学数据分析的统计学方法.中国卫生统计, 2014, 31(2): 357–359.
[17] Feng L, Cao FR, Liu XM, Pan RL, Liao YH, Chang Q. Sample collection and preparation of untargeted metabolomics., 2014, 12(12): 1217–1221.冯利, 曹芳瑞, 刘新民, 潘瑞乐, 廖永红, 常琪. 非靶向代谢组学生物样品采集和制备方法探讨. 中南药学, 2014, 12(12): 1217–1221.
[18] Mathé EA, Patterson AD, Haznadar M, Manna SK, Krausz KW, Bowman ED, Shields PG, Idle JR, Smith PB, Anami K, Kazandjian DG, Hatzakis E, Gonzalez FJ, Harris CC. Noninvasive urinary metabolomic profiling identifies diagnostic and prognostic markers in lung cancer., 2014, 74(12): 3259–3270.
[19] Gong LX, Chi HL, Wang J, Zhang HJ, Liu YL. Application of targeted metabolomics technology in nutritional diseases., 2017, 38(18): 323–327.龚凌霄, 迟海林, 王静, 张慧娟, 刘英丽. 靶向代谢组学技术在营养性疾病研究中的应用. 食品工业科技, 2017, 38(18): 323–327.
[20] Liu WL, Wang CJ, Yao L, Ai D, Zhu Y. Effects of n-3 PUFA on early nonalcoholic fatty liver disease: a mechanism study based on targeted metabolomics., 2015, (10): 1877–1877.刘雯丽, 王春炅, 姚柳, 艾玎, 朱毅. n-3 PUFA对早期非酒精性脂肪性肝病的作用—基于靶向代谢组学的机制研究. 中国病理生理杂志, 2015, (10): 1877–1877.
[21] Sawada Y, Akiyama K, Sakata A, Kuwahara A, Otsuki H, Sakurai T, Saito K, Hirai MY. Widely targeted metabolomics based on large-scale MS/MS data for elucidating metabolite accumulation patterns in plants., 2009, 50(1): 37–47.
[22] Xuan Q, Hu C, Yu D, Wang L, Zhou Y, Zhao X, Li Q, Hou X, Xu G. Development of a high coverage pseudotargeted lipidomics method based on ultra-high performance liquid chromatography-mass spectrometry., 2018, 90(12): 7608–7616.
[23] Zha H, Cai Y, Yin Y, Wang Z, Li K, Zhu ZJ. SWATHtoMRM: Development of high-coverage targeted metabolomics method using SWATH technology for biomarker discovery., 2018, 90(6): 4062–4070.
[24] Yao M, Ma L, Humphreys WG, Zhu MS. Rapid screening and characterization of drug metabolites using amultiple ion monitoring-dependent MS/MS acquisition method on a hybrid triple quadrupole-linear ion trap mass spectrometer., 2008, 43(10): 1364–1375.
[25] Chen W, Gong L, Guo ZL, Wang WS, Zhang HG, Liu XQ, Yu SB, Xiong LZ, Luo J. A novel integrated method for large-scale detection, identification, and quantification of widely targeted metabolites: application in the study of rice metabolomics., 2013, 6(6): 1769–1780.
[26] Matsuda F, Okazaki Y, Oikawa A, Kusano M, Nakabayashi R, Kikuchi J, Yonemaru J, Ebana K, Yano M, Saito K. Dissection of genotype-phenotype associations in rice grains using metabolome quantitative trait loci analysis., 2012, 70(4): 624–636.
[27] Zhang FX, Wang GD. Current metabolomics platforms: technical composition and applications., 2019, 41(9): 883–892.张凤霞, 王国栋. 现代代谢组学平台建设及相关技术应用.遗传, 2019, 41(9): 883–892.
[28] Evans CR, Karnovsky A, Kovach MA, Standiford TJ, Burant CF, Stringer KA. Untargeted LC-MS metabolomics of bronchoalveolar lavage fluid differentiates acute respiratory distress syndrome from health., 2014, 13(2): 640–649.
[29] Ciborowski M, Lipska A, Godzien J, Ferrarini A, Korsak J, Radziwon P, Tomasiak M, Barbas C. Combination of LC-MS- and GC-MS-based metabolomics to study the effect of ozonated autohemotherapy on human blood., 2012, 11(12): 6231–6241.
[30] Ramautar R, Somsen GW, De Jong GJ. CE-MS in metabolomics., 2009, 30(1): 276–291.
[31] Wishart DS, Feunang YD, Marcu A, Guo AC, Liang K, Vázquez-Fresno R, Sajed T, Johnson D, Li C, Karu N, Sayeeda Z, Lo E, Assempour N, Berjanskii M, Singhal S, Arndt D, Liang Y, Badran H, Grant J, Serra-Cayuela A, Liu Y, Mandal R, Neveu V, Pon A, Knox C, Wilson M, Manach C, Scalbert A. HMDB 4.0: the human metabolome database for 2018., 2018, 46(D1): D608–D617.
[32] Vinaixa M, Schymanski EL, Neumann S, Navarro M, Salekf RM, Yanes O. Mass spectral databases for LC/MS- and GC/MS-based metabolomics: State of the field and future prospects., 2015, 78: 23–35.
[33] Smith CA, O'Maille G, Want EJ, Qin C, Trauger SA, Brandon TR, Custodio DE, Abagyan R, Siuzdak G. METLIN: a metabolite mass spectral database., 2005, 27(6): 747–751.
[34] NIoSa T. NIST/EPA/NIH Mass Spectral Library with Search Program v17. 2017.
[35] Kind T, Wohlgemuth G, Lee DY, Lu Y, Palazoglu M, Shahbaz S, Fiehn O. FiehnLib: mass spectral and retention index libraries for metabolomics based on quadrupole and time-of-flight gas chromatography/mass spectrometry., 2009, 81(24): 10038–10048.
[36] Sud M, Fahy E, Cotter D, Brown A, Dennis EA, Glass CK, Merrill AHJ, Murphy RC, Raetz CR, Russell DW, Subramaniam S. LMSD: LIPID MAPS structure database., 2007, 35: D527–D532.
[37] Horai H, Arita M, Kanaya S, Nihei Y, Ikeda T, Suwa K, Ojima Y, Tanaka K, Tanaka S, Aoshima K, Oda Y, Kakazu Y, Kusano M, Tohge T, Matsuda F, Sawada Y, Hirai MY, Nakanishi H, Ikeda K, Akimoto N, Maoka T, Takahashi H, Ara T, Sakurai N, Suzuki H, Shibata D, Neumann S, Iida T, Tanaka K, Funatsu K, Matsuura F, Soga T, Taguchi R, Saito K, Nishioka T. MassBank: a public repository for sharing mass spectral data for life sciences., 2010, 45(7): 703–714.
[38] Song J, Wu YB, Zhou YH, Liu BJ, Wang N, Hao ZF, Wu YQ. Comparation and utilization of crop-omics databases., 2018, 40(7): 534–545.宋洁, 吴永波, 周跃恒, 柳波娟, 王楠, 郝转芳, 吴元奇. 作物组学数据库的比较和应用. 遗传, 2018, 40(7): 534–545.
[39] Laboratories K. KEGG: Kyoto Encyclopedia of Genes and Genomes, 2019.
[40] Jassal B, Matthews L, Viteri G, Gong C, Lorente P, Fabregat A, Sidiropoulos K, Cook J, Gillespie M, Haw R, Loney F, May B, Milacic M, Rothfels K, Sevilla C, Shamovsky V, Shorser S, Varusai T, Weiser J, Wu G, Stein L, Hermjakob H, D'Eustachio P. The reactome pathway knowledgebase., 2020, 48(D1): D498– D503.
[41] Bovo S, Mazzoni G, Galimberti G, Calo DG, Fanelli F, Mezzullo M, Schiavo G, Manisi A, Trevisi P, Bosi P, Dall'Olio S, Pagotto U, Fontanesi L. Metabolomics evidences plasma and serum biomarkers differentiating two heavy pig breeds., 2016, 10(10): 1741–1748.
[42] He QH, Ren PP, Kong XF, Wu YN, Wu GY, Li P, Hao FH, Tang HR, Blachier F, Yin YL. Comparison of serum metabolite compositions between obese and lean growing pigs using an NMR-based metabonomic approach., 2012, 23(2): 133–139.
[43] Ji B, Middleton JL, Ernest B, Saxton AM, Lamont SJ, Campagna SR, Voy BH. Molecular and metabolic profiles suggest that increased lipid catabolism in adipose tissue contributes to leanness in domestic chickens., 2014, 46(9): 315–327.
[44] Beauclercq S, Nadal-Desbarats L, Hennequet-Antier C, Collin A, Tesseraud S, Bourin M, Le Bihan-Duval E, Berri C. Serum and muscle metabolomics for the prediction of ultimate pH, a key factor for chicken-meat quality., 2016, 15(4): 1168–1178.
[45] Bovo S, Mazzoni G, Calò DG, Galimberti G, Fanelli F, Mezzullo M, Schiavo G, Scotti E, Manisi A, Samoré AB, Bertolini F, Trevisi P, Bosi P, Dall'Olio S, Pagotto U, Fontanesi L. Deconstructing the pig sex metabolome: Targeted metabolomics in heavy pigs revealed sexual dimorphisms in plasma biomarkers and metabolic pathways., 2015, 93(12): 5681–5693.
[46] Rohart F, Paris A, Laurent B, Canlet C, Molina J, Mercat MJ, Tribout T, Muller N, Iannuccelli N, Villa-Vialaneix N, Liaubet L, Milan D, San Cristobal M. Phenotypic prediction based on metabolomic data for growing pigs from three main European breeds., 2012, 90(13): 4729–4740.
[47] Picone G, Zappaterra M, Luise D, Trimigno A, Capozzi F, Motta V, Davoli R, Nanni Costa L, Bosi P, Trevisi P. Metabolomics characterization of colostrum in three sow breeds and its influences on piglets' survival and litter growth rates., 2018, 9(1): 23.
[48] Karisa BK, Thomson J, Wang Z, Li C, Montanholi YR, Miller SP, Moore SS, Plastow GS. Plasma metabolites associated with residual feed intake and other productivity performance traits in beef cattle., 2014, 165(1): 200–211.
[49] Sun HZ, Wang DM, Wang B, Wang JK, Liu HY, Guan LL, Liu JX. Metabolomics of four biofluids from dairy cows: potential biomarkers for milk production and quality., 2015, 14(2): 1287–1298.
[50] Beauclercq S, Nadal-Desbarats L, Hennequet-Antier C, Gabriel I, Tesseraud S, Calenge F, Le Bihan-Duval E, Mignon-Grasteau S. Relationships between digestive efficiency and metabolomic profiles of serum and intestinal contents in chickens., 2018, 8(1): 6678.
[51] Gong WJ, Jia JJ, Zhang BK, Mi SJ, Zhang L, Xie XM, Guo HC, Shi JS, Tu CC. Serum metabolomic profiling of piglets infected with virulent classical swine fever virus., 2017, 8: 731.
[52] Welle T, Hoekstra AT, Daemen IAJJM, Berkers CR, Costa MO. Metabolic response of porcine colon explants to in vitro infection by Brachyspira hyodysenteriae: a leap into disease pathophysiology., 2017, 13(7): 83.
[53] Hailemariam D, Mandal R, Saleem F, Dunn SM, Wishart DS, Ametaj BN. Identification of predictive biomarkers of disease state in transition dairy cows., 2014, 97(5): 2680–2693.
[54] Zhang GS, Dervishi E, Dunn SM, Mandal R, Liu P, Han B, Wishart DS, Ametaj BN. Metabotyping reveals distinct metabolic alterations in ketotic cows and identifies early predictive serum biomarkers for the risk of disease., 2017, 13(4): 43.
[55] Shen Y, Shi S, Tong H, Guo Y, Zou J. Metabolomics analysis reveals that bile acids and phospholipids contribute to variable responses to low-temperature- induced ascites syndrome., 2014, 10(6): 1557–1567.
[56] Lu Z, He X, Ma B, Zhang L, Li J, Jiang Y, Zhou G, Gao F. Serum metabolomics study of nutrient metabolic variations in chronic heat-stressed broilers., 2018, 119(7): 771–781.
[57] Wasseriwan AE. Symposium on meat flavor. Chemical basis for meat flavor: A review., 2010, 44(1): 6–11.
[58] Liu H, Wang Z, Zhang D, Shen Q, Pan T, Hui T, Ma J. Characterization of key aroma compounds in beijing roasted duck by gas chromatography-olfactometry-mass spectrometry, odor-activity values, and aroma-recombination experiments., 2019, 67(20): 5847–5856.
[59] Liu H, Yao G, Liu X, Liu C, Zhan J, Liu D, Wang P, Zhou Z. Approach for pesticide residue analysis for metabolite prothioconazole-desthio in animal origin food., 2017, 65(11): 2481–2487.
[60] Yin Z, Chai T, Mu P, Xu N, Song Y, Wang X, Jia Q, Qiu J. Multi-residue determination of 210 drugs in pork by ultra-high-performance liquid chromatography-tandem mass spectrometry., 2016, 1463: 49–59.
[61] Trivedi DK, Hollywood KA, Rattray NJ, Ward H, Trivedi DK, Greenwood J, Ellis DI, Goodacre R. Meat, the metabolites: an integrated metabolite profiling and lipidomics approach for the detection of the adulteration of beef with pork., 2016, 141(7): 2155–2164.
[62] Tieman D, Zhu G, Resende MF, Jr., Lin T, Nguyen C, Bies D, Rambla JL, Beltran KS, Taylor M, Zhang B, Ikeda H, Liu Z, Fisher J, Zemach I, Monforte A, Zamir D, Granell A, Kirst M, Huang S, Klee H. A chemical genetic roadmap to improved tomato flavor., 2017, 355(6323): 391–394.
[63] Shang Y, Ma Y, Zhou Y, Zhang H, Duan L, Chen H, Zeng J, Zhou Q, Wang S, Gu W, Liu M, Ren J, Gu X, Zhang S, Wang Y, Yasukawa K, Bouwmeester HJ, Qi X, Zhang Z, Lucas WJ, Huang S. Plant science. Biosynthesis, regulation, and domestication of bitterness in cucumber., 2014, 346(6213): 1084–1088.
[64] Chen W, Gao Y, Xie W, Gong L, Lu K, Wang W, Li Y, Liu X, Zhang H, Dong H, Zhang W, Zhang L, Yu S, Wang G, Lian X, Luo J. Genome-wide association analyses provide genetic and biochemical insights into natural variation in rice metabolism., 2014, 46(7): 714–721.
[65] Zhu G, Wang S, Huang Z, Zhang S, Liao Q, Zhang C, Lin T, Qin M, Peng M, Yang C, Cao X, Han X, Wang X, van der Knaap E, Zhang Z, Cui X, Klee H, Fernie AR, Luo J, Huang S. Rewiring of the fruit metabolome in tomato breeding., 2018, 172(1–2): 249–261.
[66] Zhou M, Jing JH, Mao RH, Guo J, Wang ZP. Applications of metabonomics in animal genetics and breeding.2019, 41(2): 111–124.周萌, 景军红, 毛瑞涵, 郭静, 王志鹏. 代谢组学在家养动物遗传育种中的应用. 遗传, 2019, 41(2): 111–124.
[67] Liu Y, Jin L, Li MZ. Research and application of metabolomics in agricultural animals., 2018, 54(6): 1–5.刘瑞, 金龙, 李明洲. 代谢组学在农业动物中的研究与应用. 中国畜牧杂志, 2018, 54(6): 1–5.
[68] Son VM, Enger EG, Grove H, Ros-Freixedes R, Kent MP, Lien S, Grindflek E. Genome-wide association study confirm major QTL for backfat fatty acid composition on SSC14 in Duroc pigs., 2017, 18(1): 369.
[69] Welzenbach J, Neuhoff C, Heidt H, Cinar MU, Looft C, Schellander K, Tholen E, Große-Brinkhaus C. Integrative analysis of metabolomic, proteomic and genomic data to reveal functional pathways and candidate genes for drip loss in pigs., 2016, 17(9): 1426.
[70] Melzer N, Wittenburg D, Repsilber D. Integrating milk metabolite profile information for the prediction of traditional milk traits based on SNP information for Holstein cows., 2013, 8(8): e70256.
[71] Widmann P, Reverter A, Weikard R, Suhre K, Hammon HM, Albrecht E, Kuehn C. Systems biology analysis merging phenotype, metabolomic and genomic data identifies Non-SMC Condensin I Complex, Subunit G (NCAPG) and cellular maintenance processes as major contributors to genetic variability in bovine feed efficiency., 2015, 10(4): e0124574.
[72] Shi SR, Shen YR, Zhang S, Zhao ZH, Hou ZC, Zhou HJ, Zou JM, Guo YM. Combinatory evaluation of transcriptome and metabolome profiles of low temperature-induced resistant ascites syndrome in broiler chickens., 2017, 7(1): 2389.
[73] Yang B, Zhang WC, Zhang ZY, Fan Y, Xie XH, Ai HS, Ma JM, Xiao SJ, Huang LS, Ren J. Genome-wide association analyses for fatty acid composition in porcine muscle and abdominal fat tissues., 2013, 8(6): e65554.
[74] Liu XX, Zhou LS, Xie XH, Wu ZZ, Xiong XW, Zhang ZY, Yang J, Xiao SJ, Zhou MQ, Ma JW, Huang LS. Muscle glycogen level and occurrence of acid meat in commercial hybrid pigs are regulated by two low-frequency causal variants with large effects and multiple common variants with small effects., 2019, 51(1): 46.
[75] Duijvesteijn N, Knol EF, Bijma P. Boar taint in entire male pigs: a genomewide association study for direct and indirect genetic effects on androstenone., 2014, 92(10): 4319–4328.
[76] Olsen HG, Knutsen TM, Kohler A, Svendsen M, Gidskehaug L, Grove H, Nome T, Sodeland M, Sundsaasen KK, Kent MP, Martens H, Lien S. Genome-wide association mapping for milk fat composition and fine mapping of a QTL for de novo synthesis of milk fatty acids on bovine chromosome 13., 2017, 49(1): 20.
[77] Palombo V, Milanesi M, Sgorlon S, Capomaccio S, Mele M, Nicolazzi E, Ajmone-Marsan P, Pilla F, Stefanon B, D'Andrea M. Genome-wide association study of milk fatty acid composition in Italian Simmental and Italian Holstein cows using single nucleotide polymorphism arrays., 2018, 101(12): 11004–11019.
[78] Tetens J, Heuer C, Heyer I, Klein MS, Gronwald W, Junge W, Oefner PJ, Thaller G, Krattenmacher N. Polymorphisms within the APOBR gene are highly associated with milk levels of prognostic ketosis biomarkers in dairy cows., 2015, 47(4): 129–137.
[79] Gan QF, Li YR, Lund M, Su GS, Liang XW. Genome-wide association study identifies loci linked to serum electrolyte traits in Chinese Holstein cattle., 2019, 50(6): 744–748.
[80] Zhu B, Niu H, Zhang WQ, Wang ZZ, Liang YH, Guan L, Guo P, Chen Y, Zhang LP, Guo Y, Ni HM, Gao X, Gao HJ, Xu LY, Li JY. Genome wide association study and genomic prediction for fatty acid composition in Chinese Simmental beef cattle using high density SNP array., 2017, 18(1): 464.
[81] Lemos MVA, Chiaia HLJ, Berton MP, Feitosa FLB, Aboujaoud C, Camargo GMF, Pereira ASC, Albuquerque LG, Ferrinho AM, Mueller LF, Mazalli MR, Furlan JJM, Carvalheiro R, Gordo DM, Tonussi R, Espigolan R, de Oliveira Silva RM, de Oliveira HN, Duckett S, Aguilar I, Baldi F. Genome-wide association between single nucleotide polymorphisms with beef fatty acid profile in Nellore cattle using the single step procedure., 2016, 17: 213.
[82] Uemoto Y, Ohtake T, Sasago N, Takeda M, Abe T, Sakuma H, Kojima T, Sasaki S. Effect of two non-synonymous ecto-5'-nucleotidase variants on the genetic architecture of inosine 5'-monophosphate (IMP) and its degradation products in Japanese Black beef., 2017, 18(1): 874.
[83] Sasago N, Takeda M, Ohtake T, Abe T, Sakuma H, Kojima T, Sasaki S, Uemoto Y. Genome-wide association studies identified variants for taurine concentration in Japanese Black beef., 2018, 89(8): 1051–1059.
[84] Javanrouh-Aliabad A, Vaez Torshizi R, Masoudi AA, Ehsani A. Identification of candidate genes for blood metabolites in Iranian chickens using a genome-wide association study., 2018, 59(4): 381–388.
[85] Goldansaz SA, Guo AC, Sajed T, Steele MA, Plastow GS, Wishart DS. Livestock metabolomics and the livestock metabolome: A systematic review., 2017, 12(5): e0177675.
Metabolomics technology and its applications in agricultural animal and plant research
Jing Tian1,2, Yuzhe Wang1,2,3, Shixiong Yan4, Shuai Sun4, Junjing Jia4, Xiaoxiang Hu1,2
Metabolomics is a discipline that uses Chromatography-Mass Spectrometry and Nuclear Magnetic Resonance techniques to identify and quantify all small molecule metabolites in living organisms and biological samples, which relies on sensitive, stable analytical procedures and improving databases. Metabolomics has been widely used in medicine, food science, crop and farm animal research, and other fields. Metabolomics can establish a more direct relationship between changes in the type and content of metabolites and phenotypes. Metabolomics has gradually become a new research method for the analysis of genetic mechanisms of complex traits following genomics, transcriptomics, and proteomics with the advances in omics technology. In this review, we firstly introduce common analytical strategies, metabolomics platforms, and metabolomics databases. Then, we review the application of metabolomics in metabolic molecular identification of important economic traits in agricultural animals, disease diagnosis, meat quality and safety detection of animal products. We also introduce the latest achievements in the development, formation and analysis of important traits of animals and plants by using metabolomics, transcriptome, and genomics. Overall, the integrated analysis of metabolomics and other omics can comprehensively explain the genetic mechanism of all kinds of complex traits and help to improve the complete biological process of “mutation-gene-expression-metabolism-phenotype”. Metabolomics provides a new method for the mechanism analysis of complex characters and a novel idea for new agricultural breeding.
metabolomics; widely targeted metabolomics; metabolome detection platform; agricultural animal; multi-omics association analysis
2019-11-04;
2020-03-13
国家自然科学基金项目(编号:31902143, 31961133003, U1702232)资助[Supported by the National Natural Science Foundation of China (Nos. 31902143, 31961133003, U1702232)]
田菁,在读博士研究生,专业方向:动物遗传育种。E-mail: 18604841591@163.com
王宇哲,博士,博士后,研究方向:动物数量遗传与功能基因组。E-mail: yuzhe891@163.com胡晓湘,博士,教授,研究方向:动物遗传育种。E-mail: huxx@cau.edu.cn
10.16288/j.yczz.19-287
2020/4/2 13:34:28
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20200402.1033.002.html
(责任编委: 李明洲)

