基于CRISPR/Cas9系统在全基因组范围内筛选功能基因及调控元件研究进展
刘思远,易国强,唐中林,3,陈斌
综 述
基于CRISPR/Cas9系统在全基因组范围内筛选功能基因及调控元件研究进展
刘思远1,2,易国强2,唐中林2,3,陈斌1
1. 湖南农业大学动物科学技术学院,长沙 410128 2. 中国农业科学院农业基因组研究所,岭南现代农业科学与技术广东省实验室深圳分中心,农业部农业基因数据分析重点实验室,深圳 518120 3. 中国农业科学院北京畜牧兽医研究所,北京 100193
CRISPR/Cas9系统是一种近年来被广泛应用于基因组编辑的强大工具。通过将CRISPR/Cas9系统中的Cas9蛋白突变后,使其失去剪切活性而成为dCas9 (nuclease-dead Cas9),再结合基因功能丧失(loss-of-function, LOF)、基因功能激活(gain-of-function, GOF)以及非编码功能基因鉴定技术即可实现全基因组高通量的功能基因及调控元件靶向鉴定和筛选。目前,该技术已被广泛应用于疾病免疫机理、药物靶点筛选和动物遗传育种等研究,为生命医学和基础科学带来了全新高效的技术方法和研究思路。本文综述了基于CRISPR/Cas9技术在全基因组中高通量筛选功能基因及调控元件的方法及研究进展,重点阐述了CRISPR/Cas9系统在动物细胞中筛选功能性基因的方法,以期为基因编辑及相关研究领域提供参考。
CRISPR/Cas9;全基因组筛选;功能基因;调控元件
成簇的规律间隔的短回文重复序列及其相关蛋白(clustered regularly interspaced short palindromic repeats/CRISPR-associated 9, CRISPR/Cas9)具有低成本、高效率和操作便捷等优点,已被广泛应用于遗传改良、分子育种和分子生物学等基因组编辑领域[1~3]。CRISPR/Cas9系统是一种存在于多数古生菌中的免疫系统[4~6],该系统在微生物中抵御外源DNA的感染,微生物利用Cas9酶切割外源基因序列使入侵序列被破坏并失活,随后将捕获的片段储存于本身基因组中组成新的间隔区(spacer),并保持免疫记忆[7]。基于Cas9蛋白结合启动子前导区转录生成的CRISPR RNAs (crRNA)等元件形成的复合体可识别基因序列上原间隔物相邻基序(protospacer adjacent motif, PAM)位点,并进行靶向剪切的工作模式,研究者只需合成一条长度在22 nt左右与目的序列互补的向导RNA(single guide RNA, sgRNA),引导Cas9复合体结合到基因组上不同的PAM位点就可达到对基因组进行靶向切割的目的[8]。CRISPR/ Cas9系统切割DNA后会导致双碱基键断裂(double- strand break, DSB),从而产生移码突变和碱基缺失等现象,随后引发DNA修复机制,即通过非同源末端连接(non-homologous end joining, NHEJ)途径直接修复缺口,或利用同源直接修复(homology directed repair, HDR)在断裂处引入同源粘性末端的DNA片段[9,10]。
由于CRISPR/Cas9系统在编码区与非编码区均可精准打靶目的基因片段,因此可实现全基因组敲除、大片段基因敲入和调控基因表达等功能[11,12]。目前,在全基因组范围内筛选表型是基因功能研究的热点方向,将CRISPR/Cas9系统与全基因组鉴定和后续的功能分析技术相结合,可以在分子、细胞和个体层面对生物进行全基因组范围的功能筛选,从而获得新的功能基因或特异性遗传位点,加快后续研究进展[13]。
本文综述了利用CRISPR/Cas9系统在全基因组中高通量筛选功能基因和调控元件的方法及其研究进展,并阐述了该方法在筛选功能性长链非编码RNA、癌症基因、生长发育性状相关的功能基因及调控元件等相关领域中的应用情况。同时剖析了这种方法存在的问题和未来发展方向,以期为后续筛选和验证动物功能基因及调控元件提供研究方法和参考。
1 基于CRISPR/Cas基因编辑技术的全基因组筛选工具
CRISPR/Cas9系统利用sgRNA的引导性和Cas9蛋白的定点识别及切割功能,可以直接对靶标基因进行靶向切割,从而干扰基因表达。在此基础上,科研人员开发出了CRISPR-Cas9全基因组敲除文库、CRISPR干扰系统(CRISPR interference, CRISPRi)与CRISPR基因转录激活系统(CRISPR activation, CRISPRa)。其中,CRISPR-Cas9全基因组敲除文库是通过全基因组的打靶sgRNA,利用CRISPR/Cas9系统在细胞群内高通量的靶向敲除目的基因,从而获得单基因敲除的单克隆细胞库。后两种系统都需要利用突变后失去核酸内切酶活性的Cas9 (nuclease- dead mutants of Cas9, dCas9)酶,并使其与sgRNA共同靶向基因组特定位点的功能[14]。其中,CRISPR 干扰系统是利用sgRNA引导dCas9在打靶基因的转录起始位点(transcription start sites, TSS)融合转录抑制因子KRAB (Krüppel-associated box),从而可以高效的抑制靶标目的基因的表达,导致基因功能丧失(loss-of-function, LOF),该系统也被称作CRISPRi系统[15]。相反,CRISPRa系统则可以通过dCas9在TSS位点招募转录激活因子如VP64、p65、Rta以及相关蛋白形成“dCas-X”的复合体,进而显著促进内源靶标基因的表达,促进内源基因的高水平转录,使基因呈现基因功能激活(gain-of-function, GOF)[16]。因此,通过sgRNA引导dCas9靶向基因的两种调控手段,可实现对全基因组范围内基因的精准调控[17,18]。目前,CRISPR/Cas9技术及CRISPR-dCas9系统已经应用于高通量筛选编码基因、启动子及增强子和长链非编码RNA等序列的功能。2013年,Gilbert等[14]通过测试CRISPR-dCas9系统中dCas9蛋白与cCas9、dCas9-KRAB、dCas9-CS和dCas9- WRPW等4种不同的融合蛋白工作效率,并在GFP随机整合的人胚肾细胞(HEK293)中产生了不同程度的荧光蛋白表达沉默,有效证明了dCas9-KRAB融合蛋白所导致的基因沉默效率最高。同时,通过RNA-seq实验表明CRISPRi在真核细胞中介导的转录抑制具有高度特异性。Ganguly等[19]利用CRISPRi系统在嗜热杆菌()中准确地抑制了中央代谢乳酸脱氢酶和磷酸转乙酰酶基因的表达。Joung等[11]在2017年通过CRISPR/Cas9系统在人的细胞系中分别开发出全基因组敲除文库和转录激活元件筛选系统,Konermann等[20]通过优化改造后的CRISPRa系统促进了基因在细胞内的转录激活,并同时激活了多个基因的高表达以及高效上调了靶标lncRNAs的转录本,最终大规模筛选了抵抗BRAF抑制剂的激活基因,在细胞层面证明了CRISPRa系统作为上调基因表达的转录激活工具的诸多优势。
2 全基因组功能基因筛选原理和技术流程
设计一个全基因组功能筛查的首要条件是如何针对全基因组设计特异性高的sgRNA库,并将其包装入可稳定转染的病毒载体中[21],其次需要选择合适的受体样本进行功能分析和验证[22]。Liu等[23]在2015年报道了一种名为“CRISPR-ERA”的sgRNA在线设计网站,可在全基因组中预测高效而特异的sgRNA,用于CRISPR系统介导的基因编辑、抑制和激活。2017年,Zhao等[24]建立了一种名为“CRISPR- offinder”的基因组sgRNA自定义设计软件,可针对不同实验目的设计sgRNA并评估其打靶效率。
在细胞层面的全基因组功能筛查流程可分为以下几个步骤:(1)确定表型与基因筛选范围;(2)构建全基因组敲除或激活基因的sgRNA文库;(3)包装慢病毒文库,通过低感染复数(multiplicity of infection, MOI)的全基因组慢病毒文库并感染目的细胞,构建稳定表达sgRNA的细胞文库并获得稳定表达株[25];(4)筛选细胞表型:对转染后的细胞施加抗生素或药物等压力并保留能存活的细胞(阳性筛选)、挑选死亡细胞(阴性筛选)或细胞增殖能力和筛选标记基因等;(5)分别提取筛选后细胞的基因组并建库;(6)利用高通量测序手段获得细胞文库中的sgRNA序列信息,并筛选目的性状的关联基因等步骤[11,26]。CRISPR/ Cas9全基因组功能筛选系统具体工作流程如图1所示。
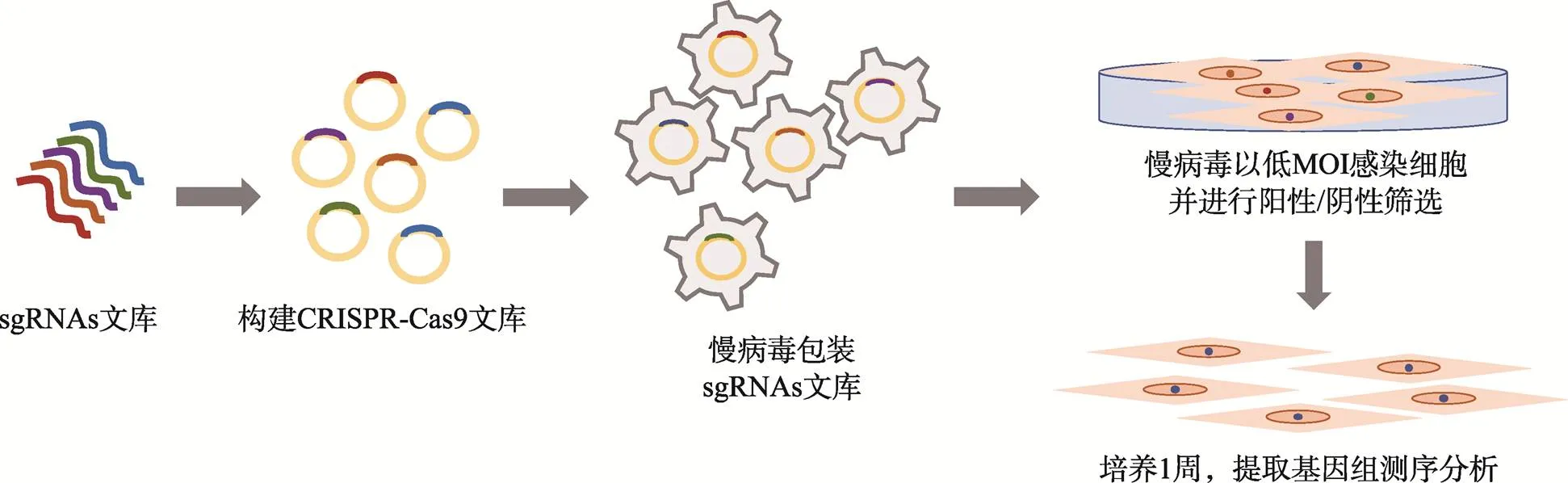
图1 CRISPR/Cas9全基因组功能筛选系统工作流程
3 CRISPR/Cas9鉴定不同功能基因及调控元件
3.1 挖掘功能性长链非编码RNA
随着全基因组测序成本降低,结合第二代和第三代基因测序手段,利用高通量数据筛选全基因组中功能基因的实验方法变得愈来愈流行。全基因组功能基因筛查是最为全面的基因检测及验证手段之一。RNA干扰(RNA interference, RNAi)是一种已标准化的基因沉默工具,靶标基因组中成熟的RNA,可高效且低成本的抑制靶标基因表达,但RNAi无法将干扰覆盖到编码基因本身,只能影响基因在转录后的表达但不能抑制基因表达,且存在较高的错配率与其他mRNA的抑制效应,因此干扰效果不全面[27,28]。而CRISPRi可在转录起始区域发挥阻止转录的作用,此外CRISPRi还可以靶标细胞核内的转录本,RNAi试剂则很难做到这一点。因此,利用CRISPRi进行基因组高通量功能元件筛选比以往的RNAi和cDNA文库等基因表达干扰手段更有优势。
长链非编码RNA (long non-coding RNAs, lncRNAs)是一种长度为200 nt以上的转录本,虽然不能编码为蛋白质,但对动物的生长发育、细胞功能、疾病和植物驯化机制等方面有着重要的影响和调控作用[29~31]。2017年,Liu等[15]对7种细胞系中的16,401个lncRNAs基因座进行了高通量CRISPR打靶干扰,筛选出细胞生长过程中必须存在的499个lncRNA基因座位点,并且这些具有生长调控功能的lncRNA存在细胞类型特异性。2019年,Cai等[32]通过CRISPRi对人表皮细胞中2263个lncRNA进行了表达筛选,并新鉴定了9个具有调节角质细胞增殖功能的候选lncRNA,其中PRANCR具有重要的调控皮肤表皮稳态的作用。Esposito等[33]也汇总了近年来CRISPR相关筛选技术在发现新癌基因lncRNA具有很大的应用潜力。Liu等[34]在2018年从10,996个lncRNA中鉴定出230个对于慢性粒细胞白血病K562细胞的细胞生长至关重要的lncRNA,并验证了该方法的稳健性和特异性。CRISPRi为鉴定全基因组功能性lncRNA提供了一种高效手段。
3.2 筛选生长发育性状的功能基因
转录组学和比较基因组学一直是挖掘影响机体发育过程关键基因和转录调控机制的常用方法。2007年,Tang等[35]利用长标签基因表达系列分析的方法绘制了中外不同猪种在多个胚胎发育期骨骼肌发育的转录组图谱,证实了通城猪和长白猪相比有更慢的肌肉生长速度和更为复杂的分子形成机制。Li等[36]还利用基因表达芯片技术比较了中国梅山猪背最长肌和比目鱼肌的转录组差异,发现差异表达基因在TGF-beta、Wnt和MAPK等信号通路富集。多组学分析技术只能预测肌肉生长发育阶段中潜在的相关基因和蛋白靶标位点,无法直接获得与预测基因相对应表型数据,因此,为了能精确的筛选出与表型直接相关的基因并同时进行验证,基于CRISPR/Cas9的全基因组功能筛选是更为有效的方法。为寻找到影响肌细胞生成的新调控元件,Bi等[37]在2017年利用结合CRISPR技术和全基因组在小鼠成肌细胞融合和成肌纤维形成过程中所需基因的功能丧失筛选技术,鉴定出了一种名为Myomixer的肌肉特异性肽,该蛋白对于胚胎发生过程中融合和骨骼肌形成起着至关重要的作用。是人类和小鼠破骨细胞形成所必需的基因,与牙齿、头骨、长骨塑性和软骨等组织的形成密切相关。MacLeod等[38]利用dCas9融合KRAB的CRISPRi系统抑制了小鼠的表达,并成功建立了淋巴结发育失败和骨质增生等表型。与野生型病理表型相比较,CRISPRi转基因小鼠具有更明显的病理表型。这些结果表明,CRISPRi所介导的基因LOF方法可以有效的抑制动物基因组上的靶基因表达,并有望搭建动物生长发育过程中的相关表型,对以后研究不同细胞类型的特异性功能丧失有极大的帮助。
3.3 筛选与疾病相关的功能基因
基于CRISPR/Cas9系统的全基因组功能筛查方法已广泛应用于医学、免疫学和药理学等领域[39],研究者不仅建立了基因定点突变细胞模型[40],还成功筛选出人急性髓细胞性白血病等疾病的潜在治疗靶点。令人惊喜的是,为了进一步建立小鼠等多种哺乳动物的疾病模型,科研人员正迅速将全基因组功能基因筛选技术应用于实验动物中。Chen等[41]针对肿瘤生长和转移过程中进行了全基因组CRISPR/ Cas9介导的全基因组基因的LOF规模筛选,在非转移性小鼠癌细胞系中构建了含有67,405种sgRNA的细胞文库,并在小鼠体内富集了文库靶向的基因,鉴定出624条sgRNAs高度靶向的基因在特定功能丧失或突变后会驱动肿瘤的生长和转移。Shi等[42]在2015年利用全基因组CRISPR/Cas9系统联合sgRNA鉴别了小鼠骨髓源永生化巨噬细胞(iBMDMs)中参与炎症激活因子caspase-1和细菌脂多糖受体caspase-11介导的细胞凋亡的宿主因子中发现了消皮素D (gasdermin D, GSDMD)底物蛋白,并验证了对GSDMD的切割介导细胞焦亡。Napier等[43]在鼠巨噬细胞细胞系中创建了一种CRISPR-Cas9全基因组文库的方法曾应用于筛选介导细胞死亡过程中的新介体[44],并筛选出Cpb1-C3- C3aR途径在促炎症传导、依赖细胞死亡过程和败血症中的新作用。
3.4 在其他研究领域的应用
全基因组基因编辑打靶技术目前广泛应用于癌症基因、药物靶点挖掘、微生物反应器制备、病毒感染机制以及CRISPRi/a系统的优化和改良等研究方面。通过CRISPR/Cas9系统及全基因组功能筛选工具建立的疾病模型,为深入研究癌症致病机理和关键功能基因提供了新思路[40,45]。2016年,Tzelepis等[46]通过优化CRISPR-Cas9系统,在人急性髓细胞性白血病细胞(acute myeloid leukemia, AML)中进行全基因组遗传脆弱性的隐性筛选,鉴定出一种新的潜在的治疗靶点,还确定了、和等其他几种已知的治疗靶标。同年,Zotova等[47]利用CRISPR-Cas9基因敲除(GeCKO)文库载体整合入CEM T细胞和Raji B细胞中并通过免疫荧光分离阳性单抗,并且新鉴定出肿瘤转移抑制因子CD82的新单抗BF4。为了结合实验目的和研究所需,科研人员针对Cas蛋白功能进行了不同的功能修饰,CRISPR/Cas9编辑系统慢慢趋于定制化[48]。Polstein等将dCas9蛋白与融合隐花色素2 (cryptochromes, CRY2)和碱性螺旋环螺旋蛋白1 (cryptochrome- interacting basic-helix-loop-helix 1, C1B1)后,便可通过sgRNA靶向转录激活区域并高效抑制内源基因的表达[49]。
在微生物基因编辑领域,2017年,Zhang等[50]通过改造后的CRISPR-Cas Cpf1蛋白(DNase-dead Cpf1 mutant, ddCpf1)系统在大肠杆菌中实现了一次性多重基因的调控,并通过RNA-Seq技术验证了该系统介导的基因表达抑制具有高特异性,有望在细胞和临床研究中取得下一步进展。经过不断的优化和改善,Li等[51]在2018年基于FnCRISPR-Cpf1系统建立了一种新型高效的链霉菌基因组编辑工具,补充了链霉菌菌株的多基因编辑领域里的技术空白,并有望利用与其他放线菌中药物活性天然产物的开发。在2019年,Depardieu等[52]建立了可应用于多种细菌中的CRISPRi筛选系统,这些应用均为日后建立大规模原核表达和筛选系统奠定了基础。
综上所述,基于CRISPR/Cas9和CRISPRi/a技术可以系统地建立全基因组目标区域的sgRNA文库和细胞文库,在动物体内和体外研究个体发育及疾病发生和进化过程等方面的功能基因筛选都是一种有效的方法。
4 结语与展望
近几年来,CRISPR/Cas9技术在各类基础科研中的便利性和重要性与日俱增[53~55]。同时,CRISPR/ Cas9全基因组功能筛选鉴定技术在农业精准育种、遗传改良、生命医学、分子治疗和多基因编辑等方面飞速发展,并取得了一系列成绩。例如,利用CRISPR/Cas9系统结合体细胞核移植(somatic cell nuclear transfer, SCNT)等技术已成功制备了斑马鱼()、小鼠()、猪()、黑腹果蝇()和食蟹猴()等基因编辑和转基因动物模型[56~60]。由于CRISPR/Cas9全基因组功能筛选在sgRNA文库和细胞文库搭建的前期需要花费大量的时间成本,目前该方法较多的应用于人癌症基因的摸索和疾病模型的搭建等研究领域或在动物细胞中进行功能验证。但在今后的研究中,CRISPR/Cas9全基因组功能筛选技术势必会普及到动植物的全基因组育种工作中,该技术也为人们理解基因遗传机制、三维基因组调控、生长发育调控和病毒等疾病的治病机理建立了新思路[61~63]。
单细胞测序是以单个细胞为单位进行的全基因组或转录组扩增的高通量测序手段,该技术可以揭示单个细胞的基因结构、基因表达状态以及细胞间的异质性,是目前研究肿瘤、细胞发育生物学、微生物学等生物学领域的热点[64~66]。如果在前期结合CRISPR基因编辑技术,对功能筛选后的单个基因编辑细胞进行高通量组分分析,可以获得相关性状更清晰的差异信息[67, 68]。因此,全基因组功能筛选技术在农业生物和相关学科的研究中还有很大的利用空间。
[1] Baliou S, Adamaki M, Kyriakopoulos AM, Spandidos DA, Panayiotidis M, Christodoulou I, Zoumpourlis V. CRISPR therapeutic tools for complex genetic disorders and cancer (Review)., 2018, 53(2): 443–468.
[2] Kruminis-Kaszkiel E, Juranek J, Maksymowicz W, Wojtkiewicz J. CRISPR/Cas9 technology as an emerging tool for targeting Amyotrophic Lateral Sclerosis (ALS)., 2018, 19(3): 906.
[3] Crispo M, Mulet AP, Tesson L, Barrera N, Cuadro F, Dos Santos-Neto PC, Nguyen TH, Creneguy A, Brusselle L, Anegon I, Menchaca A. Efficient generation of myostatin knock-out sheep using CRISPR/Cas9 technology and microinjection into zygotes., 2015, 10(8): e0136690.
[4] Jansen R, Embden JD, Gaastra W, Schouls LM. Identification of genes that are associated with DNA repeats in prokaryotes., 2002, 43(6): 1565–1575.
[5] Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product., 1987, 169(12): 5429–5433.
[6] Brouns SJJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJH, Snijders APL, Dickman MJ, Makarova KS, Koonin EV, Van der Oost J. Small CRISPR RNAs guide antiviral defense in prokaryotes., 2008, 321(5891): 960– 964.
[7] Pourcel C, Salvignol G, Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies., 2005, 151(Pt 3): 653– 663.
[8] Anders C, Niewoehner O, Duerst A, Jinek M. Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease., 2014, 513(7519): 569–573.
[9] Wyman C, Kanaar R. DNA double-strand break repair: all's well that ends well., 2006, 40: 363– 383.
[10] Mao Z, Bozzella M, Seluanov A, Gorbunova V. DNA repair by nonhomologous end joining and homologous recombination during cell cycle in human cells., 2008, 7(18): 2902–2906.
[11] Joung J, Konermann S, Gootenberg JS, Abudayyeh OO, Platt RJ, Brigham MD, Sanjana NE, Zhang F. Genome- scale CRISPR-Cas9 knockout and transcriptional activation screening., 2017, 12(4): 828–863.
[12] Chowdhury TA, Koceja C, Eisa-Beygi S, Kleinstiver BP, Kumar SN, Lin CW, Li K, Prabhudesai S, Joung K, Ramchandran R. Temporal and spatial post-transcriptional regulation of zebrafish tie1 mrna by long noncoding RNA during brain vascular assembly., 2018, 38(7): 1562–1575.
[13] Li HH, Huang CH. Functional genetic screening using CRISPR-Cas9 system., 2018, 34(4): 461– 472.李欢欢,黄承浩. 基于CRISPR-Cas9的功能基因筛选研究进展. 生物工程学报, 2018, 34(4): 461–472.
[14] Gilbert LA, Larson MH, Morsut L, Liu Z, Brar GA, Torres SE, Stern-Ginossar N, Brandman O, Whitehead EH, Doudna JA, Lim WA, Weissman JS, Qi LS. CRISPR- mediated modular RNA-guided regulation of transcription in eukaryotes., 2013, 154(2): 442–451.
[15] Liu SJ, Horlbeck MA, Cho SW, Birk HS, Malatesta M, He D, Attenello FJ, Villalta JE, Cho MY, Chen Y, Mandegar MA, Olvera MP, Gilbert LA, Conklin BR, Chang HY, Weissman JS, Lim DA. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells., 2017, 355(6320). pii:aah7111.
[16] Perez-Pinera P, Kocak DD, Vockley CM, Adler AF, Kabadi AM, Polstein LR, Thakore PI, Glass KA, Ousterout DG, Leong KW, Guilak F, Crawford GE, Reddy TE, Gersbach CA. RNA-guided gene activation by CRISPR-Cas9-based transcription factors., 2013, 10(10): 973– 976.
[17] Kampmann M. CRISPRi and CRISPRa screens in mammalian cells for precision biology and medicine., 2018, 13(2): 406–416.
[18] Chavez A, Scheiman J, Vora S, Pruitt BW, Tuttle M, P R Iyer E, Lin S, Kiani S, Guzman CD, Wiegand DJ, Ter-Ovanesyan D, Braff JL, Davidsohn N, Housden BE, Perrimon N, Weiss R, Aach J, Collins JJ, Church GM. Highly efficient Cas9-mediated transcriptional programming., 2015, 12(4): 326–328.
[19] Ganguly J, Martin-Pascual M, van Kranenburg R. CRISPR interference (CRISPRi) as transcriptional repression tool for Hungateiclostridium thermocellum DSM 1313., 2019, 13(2): 339–349.
[20] Konermann S, Brigham MD, Trevino AE, Joung J, Abudayyeh OO, Barcena C, Hsu PD, Habib N, Gootenberg JS, Nishimasu H, Nureki O, Zhang F. Genome- scale transcriptional activation by an engineered CRISPR- Cas9 complex., 2015, 517(7536): 583–588.
[21] Doench JG, Fusi N, Sullender M, Hegde M, Vaimberg EW, Donovan KF, Smith I, Tothova Z, Wilen C, Orchard R, Virgin HW, Listgarten J, Root DE. Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9., 2016, 34(2): 184–191.
[22] Sanjana NE, Shalem O, Zhang F. Improved vectors and genome-wide libraries for CRISPR screening., 2014, 11(8): 783–784.
[23] Liu HL, Wei Z, Dominguez A, Li YD, Wang XW, Qi LS. CRISPR-ERA: a comprehensive design tool for CRISPR- mediated gene editing, repression and activation., 2015, 31(22): 3676–3678.
[24] Zhao CZ, Zheng XG, Qu WB, Li GL, Li XY, Miao YL, Han XS, Liu XD, Li ZH, Ma YL, Shao QZ, Li HW, Sun F, Xie SS, Zhao SH. CRISPR-offinder: a CRISPR guide RNA design and off-target searching tool for user-defined protospacer adjacent motif., 2017, 13(12): 1470–1478.
[25] Chen C, Hao S, Bai Y, Zhang JP, Zhang JB, Cheng T. Establishment and optimization of genome-wide CRISPR/ Cas9-sgRNA screening system in THP1cell line for functional oncogenes and tumor suppressor genes., 2016, 46(7): 839–850.陈晨, 郝莎, 白杨, 张健萍, 张孝兵, 程涛. CRISPR/ Cas9-sgRNA全基因组文库筛选人单核细胞白血病功能性促癌/抑癌基因体系的建立与优化. 中国科学:生命科学, 2016, 46(7): 839–850.
[26] Morgens DW, Deans RM, Li A, Bassik MC. Systematic comparison of CRISPR/Cas9 and RNAi screens for essential genes., 2016, 34(6): 634–636.
[27] Schuster A, Erasimus H, Fritah S, Nazarov PV, van Dyck E, Niclou SP, Golebiewska A. RNAi/CRISPR screens: from a pool to a valid hit., 2019, 37(1): 38–55.
[28] Klann TS, Black JB, Chellappan M, Safi A, Song L, Hilton IB, Crawford GE, Reddy TE, Gersbach CA. CRISPR-Cas9 epigenome editing enables high-throughput screening for functional regulatory elements in the human genome., 2017, 35(6): 561–568.
[29] Wang Z, Yang Y, Li S, Li K, Tang Z. Analysis and comparison of long non-coding RNAs expressed in the ovaries of Meishan and Yorkshire pigs., 2019, 50(6): 660–669.
[30] Yu X, Wang Z, Sun H, Yang Y, Li K, Tang Z. Long non-coding MEG3 is a marker for skeletal muscle development and meat production traits in pigs., 2018, 49(6): 571–578.
[31] Zheng XM, Chen J, Pang HB, Liu S, Gao Q, Wang JR, Qiao WH, Wang H, Liu J, Olsen KM, Yang QW. Genome-wide analyses reveal the role of noncoding variation in complex traits during rice domestication., 2019, 5(12): eaax3619.
[32] Cai P, Otten AB, Cheng B, Ishii MA, Zhang W, Huang BB, Qu K, Sun BK. A genome-wide long noncoding RNA CRISPRi screen identifies PRANCR as a novel regulator of epidermal homeostasi., 2020, 30(1): 22– 34.
[33] Esposito R, Bosch N, Lanzós A, Polidori T, Pulido- Quetglas C, Johnson R. Hacking the cancer genome: Profiling therapeutically actionable long Non-coding RNAs using CRISPR-Cas9 screening., 2019, 35(4): 545–557.
[34] Liu Y, Cao ZZ, Wang YN, Guo Y, Xu P, Yuan PF, Liu ZH, He Y, Wei WS. Genome-wide screening for functional long noncoding RNAs in human cells by Cas9 targeting of splice sites., 2018, 36(12): 1203–1210.
[35] Tang ZL, Li Y, Wan P, Li XP, Zhao SH, Liu B, Fan B, Zhu MJ, Yu M, Li K. LongSAGE analysis of skeletal muscle at three prenatal stages in Tongcheng and Landrace pigs., 2007, 8(6): R115.
[36] Li Y, Xu ZY, Li HY, Xiong YZ, Zuo B. Differential transcriptional analysis between red and white skeletal muscle of Chinese Meishan pigs., 2010, 6(4): 350–360.
[37] Bi PP, Ramirez-Martinez A, Li H, Cannavino J, Mcanally JR, Shelton JM, Sánchez-Ortiz E, Bassel-Duby R, Olson EN. Control of muscle formation by the fusogenic micropeptide myomixer., 2017, 356(6335): 323–327.
[38] MacLeod RS, Cawley KM, Gubrij I, Nookaew I, Onal M, O'Brien CA. Effective CRISPR interference of an endogenous gene via a single transgene in mice., 2019, 9(1): 17312.
[39] Ahmad HI, Ahmad MJ, Asif AR, Adnan M, Iqbal MK, Mehmood K, Muhammad SA, Bhuiyan AA, Elokil A, Du XY, Zhao CZ, Liu XD, Xie SS. A Review of CRISPR- Based genome editing: Survival, evolution and challenges., 2018, 28: 47–68.
[40] Zhang K, Liu W, Liu XF, Chen YS, Liu XH, He ZY. Generation of cell strains containing point mutations in HPRT1 by CRISPR/Cas9., 2019, 41(10): 939–949.张楷, 刘蔚, 刘小凤, 陈瑶生, 刘小红, 何祖勇. 利用CRISPR/Cas9系统构建人HPRT1基因定点突变细胞株. 遗传, 2019, 41(10): 939–949.
[41] Chen S, Sanjana NE, Zheng K, Shalem O, Lee K, Shi X, Scott DA, Song J, Pan JQ, Weissleder R, Lee H, Zhang F, Sharp PA. Genome-wide CRISPR screen in a mouse model of tumor growth and metastasis., 2015, 160(6): 1246–1260.
[42] Shi JJ, Zhao Y, Wang K, Shi XY, Wang Y, Huang HW, Zhuang YH, Cai T, Wang FC, Shao F. Cleavage of GSDMD by inflammatory caspases determines pyroptotic cell death., 2015, 526(7575): 660–665.
[43] Napier BA, Monack DM. Creating a RAW264.7 CRISPR- Cas9 genome wide library., 2017, 7(10): 1–10.
[44] Napier BA, Brubaker SW, Sweeney TE, Monette P, Rothmeier GH, Gertsvolf NA, Puschnik A, Carette JE, Khatri P, Monack DM. Complement pathway amplifies caspase-11-dependent cell death and endotoxin-induced sepsis severity., 2016, 213(11): 2365–2382.
[45] Liu H, Li DM, Zhu LY, Lai LJ, Yan WY, Lu YS, Wei Y, Huang YQ, Fang M, Su YG, Yang F, Shu W. Research on the knockout of LMNA gene by CRISPR/Cas9 system in human cell lines., 2019, 41(1): 66–75.刘恒, 李东明, 朱兰玉, 赖乐锦, 闫婉云, 陆玉双, 韦伊, 黄月琪, 方媚, 苏元港, 杨芳, 舒伟. 利用CRISPR/Cas9敲除人源细胞系中LMNA基因的研究. 遗传, 2019, 41(1): 66–75.
[46] Tzelepis K, Koike-Yusa H, De Braekeleer E, Li Y, Metzakopian E, Dovey OM, Mupo A, Grinkevich V, Li M, Mazan M, Gozdecka, M, Ohnishi S, Cooper J, Patel M, McKerrell T, Chen B, Domingues AF, Gallipoli P, Teichmann S, Ponstingl H, McDermott U, Saez-Rodriguez J, Huntly BJP, Iorio F, Pina C, Vassiliou GS, Yusa K. A CRISPR dropout screen identifies genetic vulnerabilities and therapeutic targets in acute myeloid leukemia., 2016, 17(4): 1193–1205.
[47] Zotova A, Zotov I, Filatov A, Mazurov D. Determining antigen specificity of a monoclonal antibody using genome-scale CRISPR-Cas9 knockout library., 2016, 439: 8–14.
[48] Covarrubias S, Robinson EK, Shapleigh B, Vollmers A, Katzan S, Hanley N, Fong N, McManus MT, Carpenter S. CRISPR/Cas-based screening of long non-coding RNAs (lncRNAs) in macrophages with an NF-κB reporter., 2017, 292(51): 20911–20920.
[49] Polstein LR, Gersbach CA. A light-inducible CRISPR- Cas9 system for control of endogenous gene activation., 2015, 11(3): 198–200.
[50] Zhang XC, Wang JM, Cheng QX, Zheng X, Zhao GP, Wang J. Multiplex gene regulation by CRISPR-ddCpf1., 2017, 3: 17018.
[51] Li L, Wei K, Zheng G, Liu X. CRISPR-Cpf1-Assisted multiplex genome editing and transcriptional repression in streptomyces., 2018, 84(18): e00827–18.
[52] Depardieu F, Bikard D. Gene silencing with CRISPRi in bacteria and optimization of dCas9 expression levels., 2019, 172: 61–75.
[53] Li W, Teng F, Li TD, Zhou Q. Simultaneous generation and germline transmission of multiple gene mutations in rat using CRISPR-Cas systems., 2013, 31(8): 684–686.
[54] Huang JJ, Cao CW, Zheng GM, Zhao JG. Genome editing technologies drive the development of pig genetic improvement., 2017, 39(11): 1078–1089.黄娇娇, 曹春伟, 郑国民, 赵建国. 基因组编辑技术在猪遗传改良中的应用. 遗传, 2017, 39(11): 1078–1089.
[55] Li S, Yang YY, Qiu Y, Chen YH, Xu LL, Ding QR. Applications of genome editing tools in precision medicine research., 2017, 39(3): 177–188.李爽, 杨圆圆, 邱艳, 陈彦好, 徐璐薇, 丁秋蓉. 基因组编辑技术在精准医学中的应用. 遗传, 2017, 39(3): 177– 188.
[56] Wu YX, Liang D, Wang YH, Bai MZ, Tang W, Bao SM, Yan ZQ, Li DS, Li JS. Correction of a genetic disease in mouse via use of CRISPR-Cas9., 2013, 13(6): 659–662.
[57] Zuo EW, Cai YJ, Li K, Wei Y, Wang BA, Sun YD, Liu Z, Liu JW, Hu XD, Wei W, Huo XN, Shi LY, Tang C, Liang D, Wang Y, Nie YH, Zhang CC, Yao X, Wang X, Zhou CY, Ying WQ, Wang QF, Chen RC, Shen Q, Xu GL, Li JS, Sun Q, Xiong ZQ, Yang H. One-step generation of complete gene knockout mice and monkeys by CRISPR/Cas9- mediated gene editing with multiple sgRNAs., 2017, 27(7): 933–945.
[58] Yan S, Tu ZC, LIU ZM, Fan NN, Yang HM, Yang S, Yang WL, Zhao Y, Ouyang Z, Lai CD, Yang HQ, Li L, Liu QS, Shi H, Xu GQ, Zhao H, Wei HJ, Pei Z, Li SH, Lai LX, Li XJ. A huntingtin knockin pig model recapitulates features of selective neurodegeneration in huntington's disease., 2018, 173(4): 989–1002.e13.
[59] Kimura Y, Hisano Y, Kawahara A, Higashijima SI. Efficient generation of knock-in transgenic zebrafish carrying reporter/driver genes by CRISPR/Cas9-mediated genome engineering., 2014, 4: 6545.
[60] Tong XL, Fang CY, Gai TT, Shi J, Lu C, Dai FY. Applications of the CRISPR/Cas9 system in insects., 2018, 40(4): 266–278.童晓玲, 方春燕, 盖停停, 石津, 鲁成, 代方银. CRISPR/ Cas9系统在昆虫中的应用. 遗传, 2018, 40(4): 266–278.
[61] Liu PF, Wu Q. Probing 3D genome by CRISPR/Cas9., 2020, 42(1): 18–31.刘沛峰, 吴强. CRISPR/Cas9基因编辑在三维基因组研究中的应用. 遗传, 2020, 42(1): 18–31.
[62] Wang J, Huang J, Xu R. Seamless genome editing in Drosophila by combining CRISPR/Cas9 and piggyBac technologies., 2019, 41(5): 422–429.王珏, 黄娟, 许蕊. 利用CRISPR/Cas9和piggyBac实现果蝇基因组无缝编辑. 遗传, 2019, 41(5): 422–429.
[63] Tang LC, Gu F. Next-generation CRISPR-Cas for genome editing: focusing on the Cas protein and PAM., 2020, 42(3): 236–249.唐连超, 谷峰. CRISPR-Cas基因编辑系统升级:聚焦Cas蛋白和PAM. 遗传, 2020, 42(3): 236–249.
[64] Wen L, Tang FC. Single-cell sequencing in stem cell biology., 2016, 17: 71.
[65] Li L, Dong J, Yan LY, Yong J, Liu XX, Hu YQ, Fan XY, Wu XL, Guo HS, Wang XY, Zhu XH, Li R, Yan J, Wei Y, Zhao YY, Wang W, Ren YX, Yuan P, Yan ZQ, Hu BQ, Guo F, Wen L, Tang FC, Qiao J. Single-Cell RNA-Seq analysis maps development of human germline cells and gonadal niche interactions., 2017, 20(6): 858–873.
[66] Bian SH, Hou Y, Zhou X, Li XL, Yong J, Wang YC, Wang WD, Yan J, Hu BQ, Guo HS, Wang JL, Gao S, Mao yn, Dong J, Zhu P, Xiu DR, Yan LY, Wen L, Qiao J, Tang FC, Fu W. Single-cell multiomics sequencing and analyses of human colorectal cancer., 2018, 362(6418): 1060– 1063.
[67] Kanesaka Y, Okada M, Ito S, Oyama T. Monitoring single- cell bioluminescence of Arabidopsis leaves to quantitatively evaluate the efficiency of a transiently introduced CRISPR/Cas9 system targeting the circadian clock gene ELF3., 2019, 36(3): 187–193.
[68] Diaz-Hernandez ME, Khan NM, Trochez CM, Yoon T, Maye P, Presciutti SM, Gibson G, Drissi H. Derivation of notochordal cells from human embryonic stem cells reveals unique regulatory networks by single cell-transcriptomics., 2019, 235(6): 5241–5255.
Progress on genome-wide CRISPR/Cas9 screening for functional genes and regulatory elements
Siyuan Liu1,2, Guoqiang Yi2, Zhonglin Tang2,3, Bin Chen1
The CRISPR/Cas9 system is a powerful tool which has been extensively used for genome editing in the past few years. Nuclease-dead Cas9 (CRISPR/dCas9), a Cas9 protein mutant without splicing ability, along with loss-of- function (LOF), gain-of-function (GOF), or non-coding genes scanning approaches can reveal genome-scale functional determinants. CRISPR/Cas9 has been widely adopted to decipher disease mechanisms and pinpoint drug targets in the life science field, and also provide novel insights into animal genetics and breeding. In this review, we summarize the research progress in high-throughput CRISPR/Cas9 screening for revealing the functional genes and regulatory elements in the whole genome. We also highlight the applications of CRISPR/Cas9 system in the animal cells, providing a reference for gene editing and other related research in related fields.
CRISPR/Cas9; genome-wide screening; functional gene; regulatory elements
2020-01-02;
2020-04-08
湖南省生猪产业技术体系岗位专家项目(编号:2019-2021),深圳市技术攻关项目(编号:JSGG20180507182028625),转基因生物新品种培育重大专项(编号:2016ZX08006002-005)和广东省重点领域研发计划(现代种业)项目(编号:2018B020203002)资助[Supported by Position Expert of Hunan Province Pig Industry Technology System (No. 2019-2021), Shenzhen Key Technology Projects (No.JSGG20180507182028625), National Science and Technology Major Project of China (No. 2016ZX08006002-005), and the Key R&D Programmes of Guangdong Province (No. 2018B020203002)]
刘思远,在读博士研究生,专业方向:动物遗传育种与繁殖。E-mail: 515970802@qq.com易国强,研究员,研究方向:功能基因组和表观遗传学。E-mail: yiguoqiang@caas.cn刘思远和易国强为并列第一作者。
唐中林,研究员,博士生导师,研究方向:动物基因组与育种。E-mail: tangzhonglin@caas.cn陈斌,教授,博士生导师,研究方向:猪的遗传育种。E-mail: chenbin7586@126.com
10.16288/j.yczz.19-390
2020/4/24 13:09:02
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20200424.1053.002.html
(责任编委: 李明洲)

