2型糖尿病易感基因SNP位点对生活方式干预降低血糖应答效果的修饰效应
王玉琢,张一鸣,董晓莲,王学才,朱建福,王娜,江峰,陈跃,姜庆五,付朝伟
研究报告
2型糖尿病易感基因SNP位点对生活方式干预降低血糖应答效果的修饰效应
王玉琢1,张一鸣2,董晓莲2,王学才2,朱建福2,王娜1,江峰1,陈跃3,姜庆五1,付朝伟1
1. 复旦大学公共卫生学院流行病学教研室,公共卫生安全教育部重点实验室,国家卫生健康委员会卫生技术评估重点实验室(复旦大学),上海 200032 2. 浙江省德清县疾病预防控制中心,德清县 313200 3. 加拿大渥太华大学医学院,渥太华 K1N6N5
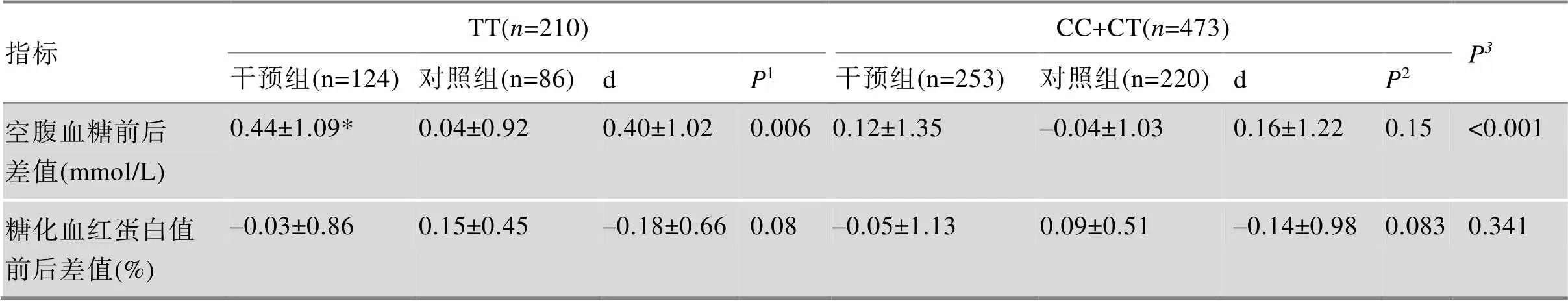
近年来的研究发现,一些2型糖尿病(type 2 diabetes mellitus, T2DM)易感基因位点不仅与T2DM的发病风险有关,还会影响生活方式干预效果。为进一步探究T2DM易感基因单核苷酸多态性(single nucleotide polymorphism, SNP)位点对生活方式干预降低高危人群血糖应答效果的修饰作用,本课题组在德清农村社区开展了生活方式干预试验(2017年6月~12月,对干预组的研究对象进行强化生活方式干预,对照组仅接受常规健康知识宣教),并对研究对象进行SNP基因分型。研究发现,对于rs9502570,干预组中的CC+CT基因型人群的空腹血糖降低值显著高于TT基因型(=0.031);干预组中CC+CT基因型人群的糖化血红蛋白值降低值为0.03%,TT基因型人群的糖化血红蛋白值升高了0.27% (=0.012);CC+CT和TT基因型干预组和对照组的空腹血糖和糖化血红蛋白前后差值差之间也有统计学差异(均为<0.001)。对于rs10811661,干预组中TT基因型人群的空腹血糖降低值显著高于CC+CT基因型(=0.021);TT和CC+CT基因型干预组和对照组的空腹血糖前后差值差之间亦有统计学差异(<0.001)。上述研究结果表明,rs9502570、rs10811661两个位点会在一定程度上修饰高危人群对T2DM生活方式干预降低血糖的应答效果,为今后进一步研制糖尿病高危人群个体化干预措施提供了客观依据。
2型糖尿病;生活方式干预;高危人群;单核苷酸多态性
遗传因素在2型糖尿病(type 2 diabetes mellitus, T2DM)的发生发展中起到非常重要的作用,约占糖尿病致病因素的40%[1]。随着全基因组关联研究(genome-wide association study, GWAS)的发展,研究者们发现了不少与T2DM相关的易感基因,截至2018年9月,已发现279个T2DM易感基因位点[2]。研究发现,一些T2DM易感基因位点不仅与T2DM的发病风险有关,还会影响个体对T2DM生活方式干预的易感性,进而影响生活方式干预的效果[3~5]。例如在Haupt等[6]的研究中,基因中的Q等位基因与生活方式干预后胰岛素敏感性的增加显著相关。目前关于T2DM易感基因对T2DM生活方式干预效果影响的研究并不多,在中国农村社区高危人群中开展的更少。因此,本研究通过分析多个T2DM易感基因单核苷酸多态性(single nucleotide polymorphism, SNP)对生活方式干预降低糖化血红蛋白、空腹血糖应答效果的影响,探讨不同基因型群体对生活方式干预的易感性,以期为T2DM高危人群个体化预防提供科学依据。
1 对象与方法
1.1 研究对象
采用随机整群抽样,在浙江省湖州市德清县农村社区中随机抽取了两个社区,其中M镇作为干预社区,Y镇作为对照社区,根据纳入与排除标准在两个社区中招募研究对象。研究对象纳入标准(需同时符合以下5项标准):(1)所选社区户籍居民;(2)年龄在18周岁及以上;(3)近期无迁出计划;(4)愿意配合调查及体检,并签署知情同意书;(5) T2DM高危人群:体质指数(body mass index, BMI)≥25 kg/m2和/或空腹血糖受损。研究对象排除标准(符合以下任一标准):(1)非所在社区户籍居民;(2)年龄不满18周岁;(3)不能提供知情同意书或配合体检;(4)近期计划迁出当地;(5)患有T2DM和严重心脑血管疾病。干预组纳入高危人群合格对象446人,随访了398人,最终有377人成功进行了SNP基因分型。对照组中纳入高危人群377人,随访了336人,最终有306人成功进行了SNP基因分型。本课题经由复旦大学公共卫生学院伦理学委员会批准(批准号:IRB# 07-06-0099和#2014-03-0480)。项目开始前由课题组成员向所有招募的参与者介绍本次研究的内容和研究目的,研究对象自愿参加并都签署了知情同意书。
1.2 生活方式干预
2017年6 ~12月,对干预组的研究对象进行生活方式干预。干预内容包括:开展4次健康教育主题讲座,发放健康教育材料。社区医生每月入户随访一次,了解研究对象的饮食、运动情况和体重、腰围等的变化情况并记录在随访调查表上,给出有针对性的生活方式指导意见,并与研究对象共同制定随访目标,干预时间为6个月。对照组仅接受德清县社区卫生服务中心给予的常规健康知识宣教。
1.3 T2DM易感基因位点的选择
在https://www.ebi.ac.uk/gwas/网站上搜索diabetes mellitus,得到279个T2DM易感基因。筛选策略如下:(1)截至2018年9月30日,GWAS报道的<5×10–8的T2DM阳性位点;(2)使用SNP info Web Server、SNP Predication Function等进行功能预测得到的功能性位点;(3)优先选择在亚洲人中验证的位点。最终,选取了与T2DM发病相关的41个基因中的43个位点,具体见表1。
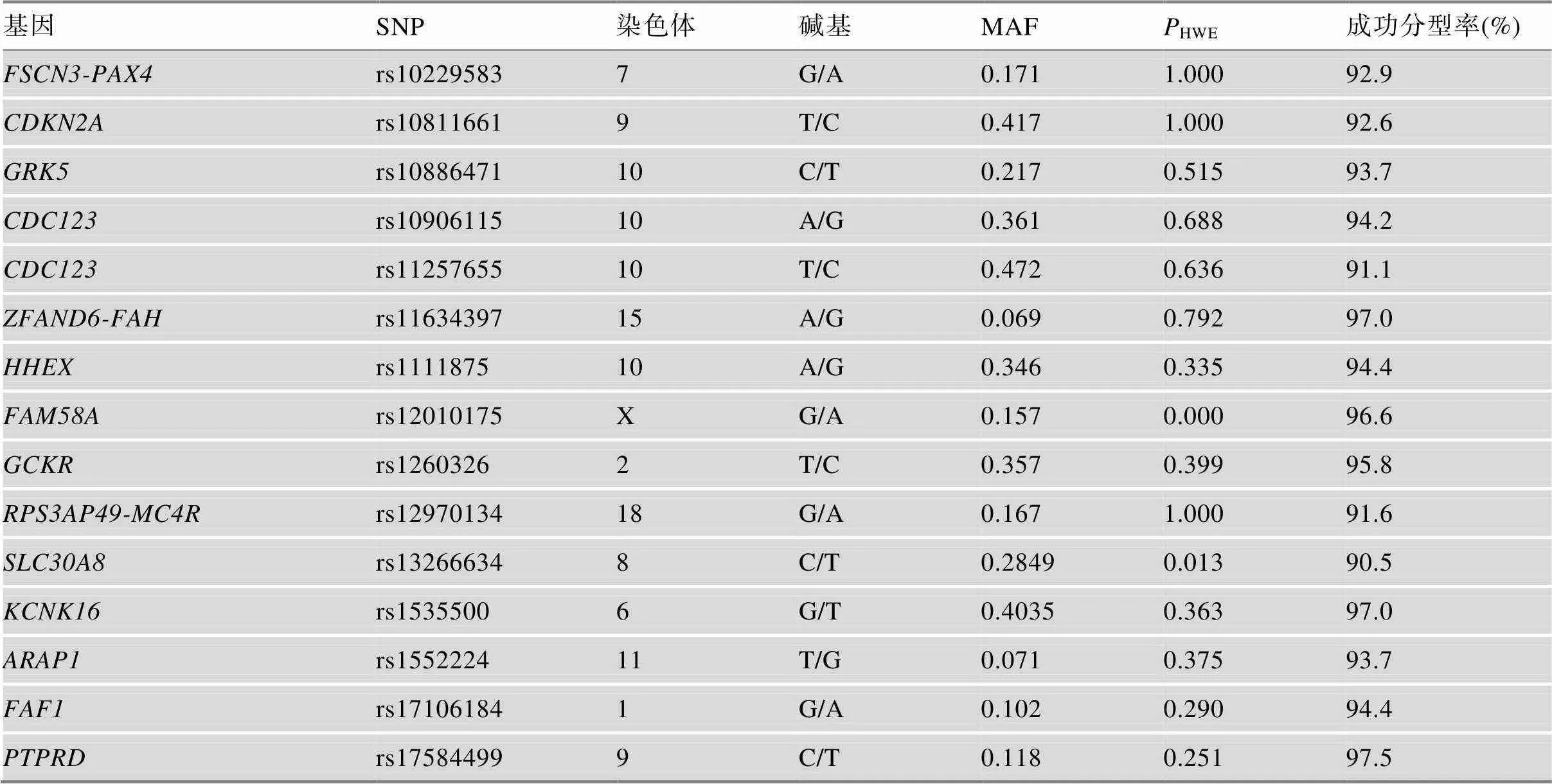
表1 43个基因位点的基本信息
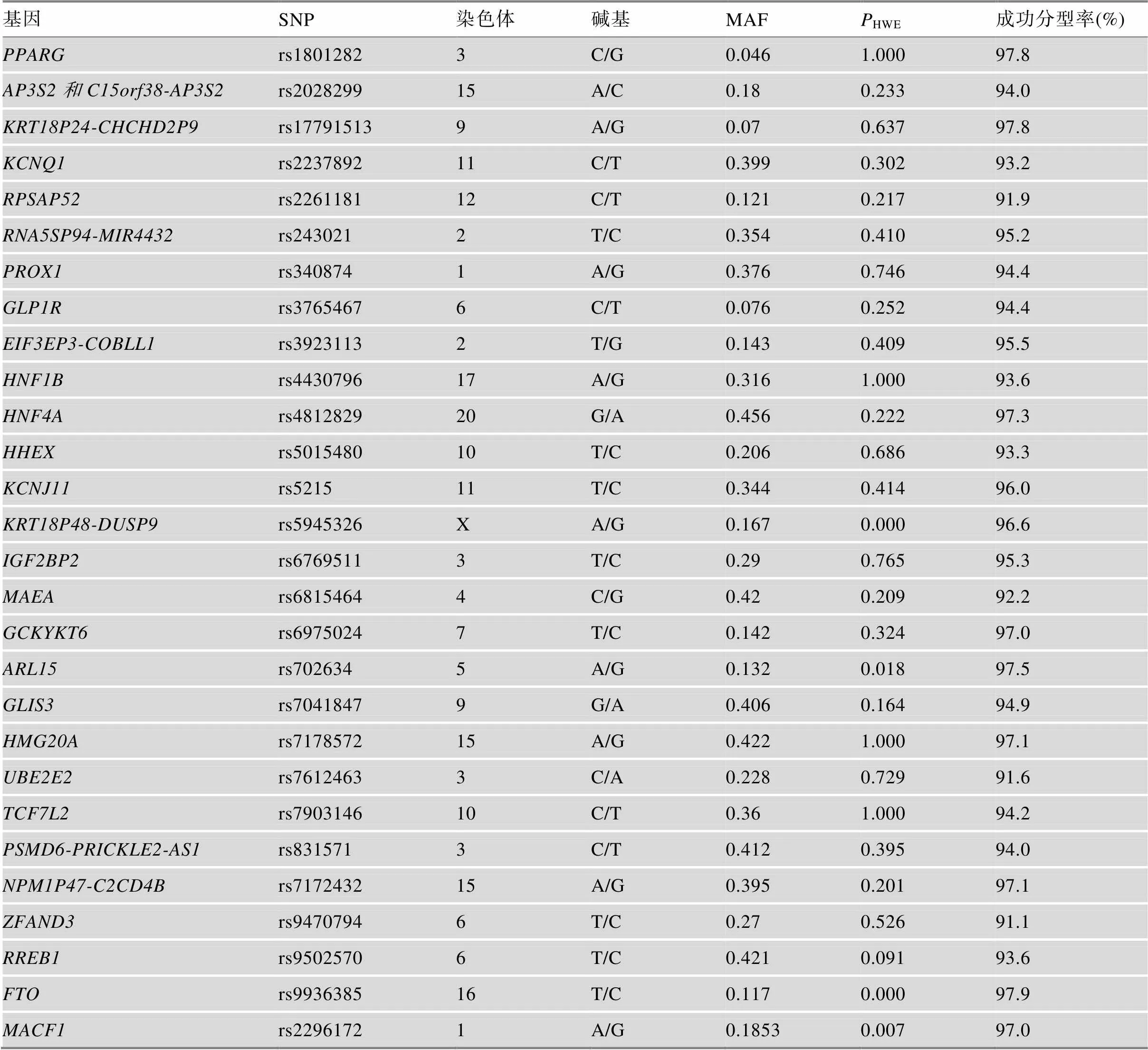
续表1
MAF为最小等位基因频率;HWE为哈德–温伯格平衡的显著性检验结果。
1.4 信息收集方法与内容
使用自制生活方式调查问卷,于干预前(2017年5月)由乡镇卫生院的医务人员(统一培训后)进行一对一的问卷调查。在干预前和干预后分别进行免费的体格检查包括身高、体重、腰围和血压,并抽取空腹8 h以上的静脉血,采用标准葡萄糖酶氧化法检测空腹血糖,采用自动生化检测仪检测糖化血红蛋白,由杭州迪安公司完成检测。
在德清队列血库中挑选本次研究高危人群的血凝块标本,用离心柱法提取DNA,使用SNP时间飞行质谱生物芯片系统(MassARRAY Sequenom平台)进行SNP基因分型[7]。
1.5 变量定义和疾病诊断标准
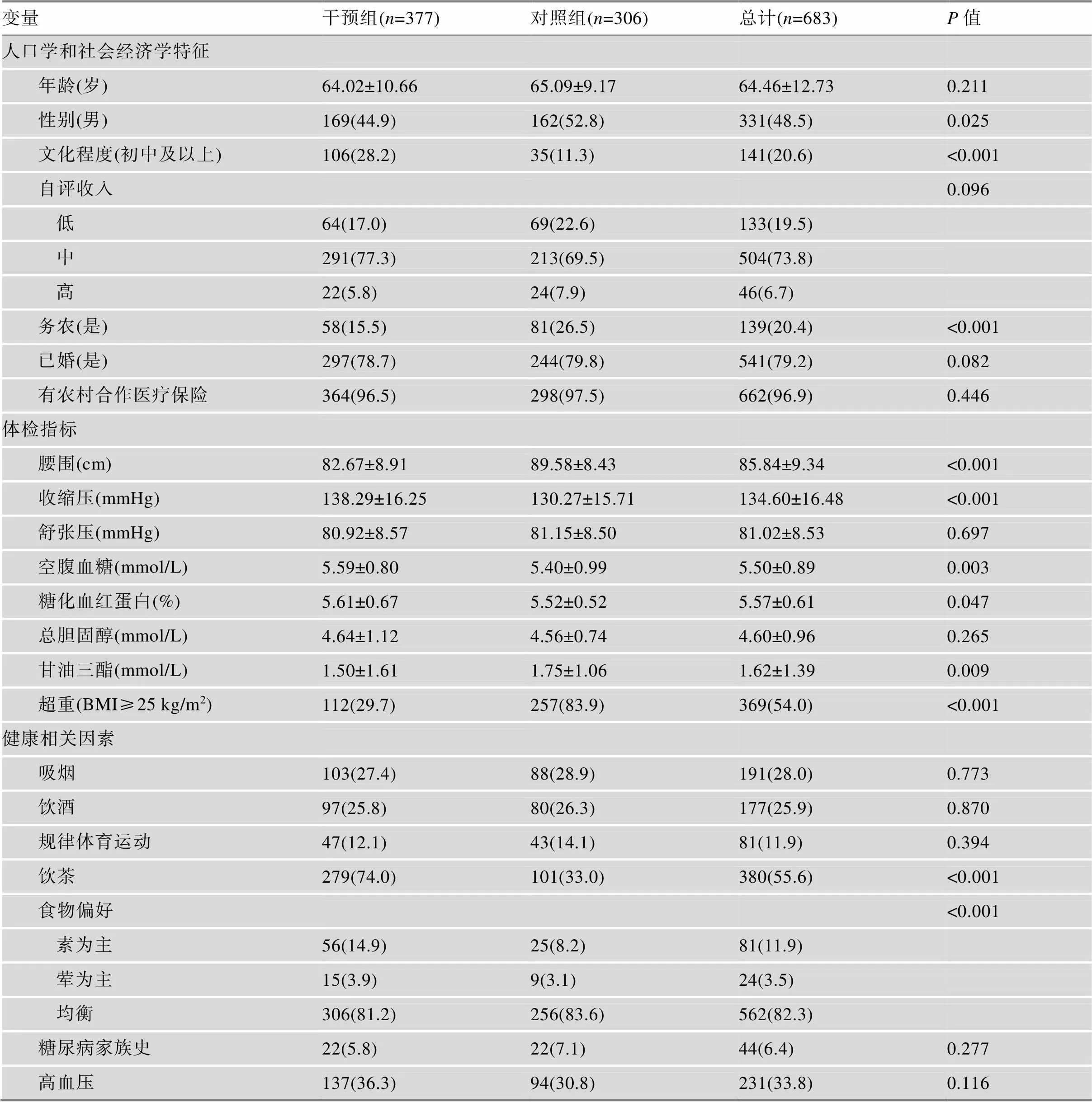
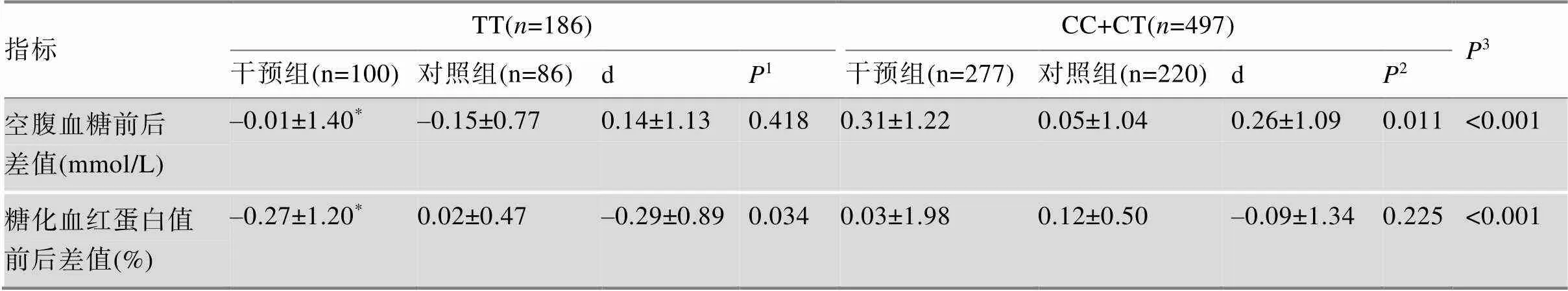
T2DM:指空腹血糖≥7.0 mmol/L和/或正在接受糖尿病治疗[8]。高血压:收缩压≥140 mmHg和/或舒张压≥90 mmHg,或者正在服用抗高血压药物或正在接受抗高血压治疗[9]。吸烟:每天吸烟1支及以上且持续时间超过3个月定义为吸烟[10]。饮酒:每周至少饮酒1次且持续时间超过3个月定义为饮 酒[11]。规律体育锻炼:每周至少运动1次,每次至少30 min以上中等强度活动且持续时间超过3个月[10]。饮茶:每天至少饮茶1次且持续时间超过3个月[10]。体质指数:BMI=体重/身高2(kg/m2),低体重:BMI<18.5 kg/m2,正常:18.5 kg/m2 采用Epidata3.1建立数据库,由复旦大学公共卫生学院研究生进行数据双录入。使用SPSS19.0、Plink、R studio软件进行统计分析。用均数±标准差描述正态分布(近似正态分布)型数据,比较组内差异使用配对检验,比较组间差异使用方差分析或检验,用频数和百分比对分类变量进行描述,用卡方检验来比较组间分布差异。对于非正态分布的计量资料使用Mann-Whitney U检验。使用协方差分析校正混杂因素对关联分析的影响。以<0.05为差异有统计学意义,并以检验的次数用Bonferroni法校正多重检验的值阈值为0.05/43。 干预组中男性169人,女性208人,平均年龄为(64.02±10.66)岁;对照组中男性162人,女性144人,平均年龄为(65.09±9.17)岁。在基线调查时,与对照组相比,干预组初中及以上文化程度者所占比例更高,务农的比例更低,腰围平均值更低,收缩压平均值更高,空腹血糖平均值更高,糖化血红蛋白值更高,甘油三酯的平均值较低,超重的比例更低,饮茶的人更多(表2)。 在调整了性别、年龄、文化程度、高血压、超重、T2DM家族史、吸烟、饮酒、饮茶、规律体育锻炼和饮食偏好等协变量后,43个SNP除了rs9502570和rs10811661外,其余SNP未发现对生活方式干预效果有统计学影响。如表3所示,对于rs9502570,干预后干预组的CC+CT基因型人群的空腹血糖平均值比TT基因型人群降低更多(0.31 mmol/L–0.01 mmol/L,=0.031),经Bonferroni校正后,差异无统计学意义(>0.05)。同时,干预组和对照组的空腹血糖前后差值差CC+CT基因型降低也高于TT基因型(0.26 mmol/L0.14 mmol/L,<0.001),经Bonferroni校正后,差异仍有统计学意义(<0.05) (表3)。干预后干预组的CC+CT基因型人群的糖化血红蛋白平均值降低了0.03%,而TT基因型的糖化血红蛋白平均值升高了0.27% (=0.012),经Bonferroni校正后,差异无统计学意义(>0.05)。CC+CT和TT基因型的干预组和对照组的糖化血红蛋白前后差值差之间也有统计学差异(–0.29 mmol/L–0.09 mmol/L,<0.001),经Bonferroni校正后,差异仍有统计学意义(<0.05)。未调整相关协变量的分析结果见附表1。 对于rs10811661,干预后干预组的TT基因型人群的空腹血糖平均值比CC+CT基因型人群降低更多(0.44 mmol/L0.12 mmol/L,=0.021)。经Bonferroni校正后,差异无统计学意义(>0.05) (表4)。同时,TT和CC+CT基因型的干预组和对照组的空腹血糖前后差值差之间有统计学差异(0.40 mmol/L0.16 mmol/L,<0.001)。经Bonferroni校正后,差异仍有统计学意义(<0.05)。但是,未发现干预组内和对照组内不同基因型的糖化血红蛋白值前后差值之间有统计学差异(>0.05)。未调整相关协变量的分析结果见附表2。 本研究发现基因附近的rs9502570与干预效果有关,具体表现在对空腹血糖变化值和糖化血红蛋白变化值的影响上。干预组中rs9502570的CC+CT基因型人群的空腹血糖平均值比TT基因型下降得更多,其对生活方式干预的净效应也更强,提示rs9502570的CC+CT基因型人群对生活方式干预降低空腹血糖的应答效果更敏感。同时,干预组中CC+CT基因型对象糖化血红蛋白值降低而TT基因型对象糖化血红蛋白值升高,并且CC+CT基因型对象的干预组和对照组糖化血红蛋白前后差值差之间亦有统计学差异,提示rs9502570的CC+CT基因型人群对生活方式干预降低糖化血红蛋白值的应答效果更敏感。rs9502570位于RREB1基因附近,RREB1基因与空腹血糖升高有强烈的相关性[13]。尽管目前rs9502570与糖尿病发病之间的生物学机制尚不清楚,但在一项关于GWAS研究的meta分析中指出该基因位点与T2DM的发病有关[14]。研究显示,基因中的风险等位基因可能与T2DM中的胰腺β细胞功能障碍有关[15],进而导致T2DM的发生。本研究首次发现基因附近的rs9502570位点会修饰个体对T2DM生活方式干预降低空腹血糖和糖化血红蛋白值的应答效果。 表2 干预组和对照组基线人群特征 本研究也发现基因附近的rs10811661与生活方式的血糖干预效果有关,具体表现在对空腹血糖值变化的影响上,即干预组中TT基因型人群的空腹血糖平均值比CC+CT基因型降低得更多,其生活方式干预的净效应更大,提示rs10811661位点的TT基因型人群对生活方式干预降低空腹血糖的应答效果更敏感。基因位于9p21号染色体区域,该区域是心血管疾病最强的遗传易感性位点[16],同样也与T2DM[17,18]、阿兹海默病[19]、青光眼[20]和牙周病[21]的发病密切相关。在法国Duesing等[22]、日本Takeuchi等[23]和中国Wu等[24]的研究中均发现位于基因附近的rs10811661位点与T2DM有很强的相关性。研究表明与胰岛β细胞功能受损有关,同时在葡萄糖毒性转导和胰岛β细胞再生能力中发挥一定作用,进而导致T2DM的发生[25]。本研究也是首次发现基因附近的rs10811661位点会修饰个体对T2DM生活方式干预降低空腹血糖的应答效果的研究。 表3 rs9502570的不同基因型在干预组和对照组空腹血糖、糖化血红蛋白值前后差值对比 d为干预组与对照组的差值,1表示TT基因型人群中干预组和对照组空腹血糖前后差值的协方差分析的显著性检验结果,2表示CC+CT基因型人群中干预组和对照组空腹血糖前后差值的协方差分析的显著性检验结果,*表示干预组内或对照组内TT和CC+CT基因型空腹血糖前后差值的协方差分析有显著性意义,3表示TT和CC+CT基因型干预组和对照组的空腹血糖前后差值差的协方差分析的显著性检验结果。以上协方差分析调整的变量有性别、年龄、文化程度、高血压、超重、T2DM家族史、吸烟、饮酒、饮茶、规律体育锻炼和饮食偏好。 表4 rs10811661的不同基因型在干预组和对照组的空腹血糖、糖化血红蛋白值前后差值对比 d为干预组与对照组的差值,1表示TT基因型人群中干预组和对照组空腹血糖前后差值的协方差分析的显著性检验结果,2表示CC+CT基因型人群中干预组和对照组协方差分析的显著性检验结果,*表示干预组内或对照组内TT和CC+CT基因型空腹血糖前后差值的协方差分析有显著性意义,3表示TT和CC+CT基因型干预组和对照组的空腹血糖前后差值差的协方差分析的显著性检验结果。以上协方差分析调整的变量有性别、年龄、文化程度、高血压、超重、T2DM家族史、吸烟、饮酒、饮茶、规律体育锻炼和饮食偏好。 本研究发现rs9502570、rs10811661两个位点会在一定程度上修饰个体对T2DM生活方式干预降低空腹血糖或糖化血红蛋白值的应答效果,这有助于解释不同基因型个体干预效果不同的原因,也可以在未来的糖尿病预防项目中预测不同基因型个体的干预效果。此外,易感基因位点与T2DM生活方式干预效果的关联研究可以为制定个体化的生活方式干预模式提供理论依据。本研究的局限性之一为研究对象仅在浙江省德清县农村社区内招募,可能会影响结果的外推。加之本研究的干预时间较短,其中长期效应有待今后继续研究。 综上所述,rs9502570的CC+CT基因型和TT基因型对生活方式干预降低空腹血糖和糖化血红蛋白的应答效果均不同,同时,rs10811661的TT基因型和CC+CT基因型仅对生活方式干预降低空腹血糖的应答效果不同,提示它们均可能修饰了我国农村糖尿病高危人群的生活方式干预效果,为今后研制进一步的糖尿病高危人群个体化干预措施提供了客观依据。 附表1和附表2详见文章电子版www.chinagene.cn。 [1] Kaprio J, Tuomilehto J, Koskenvuo M, Romanov K, Reunanen A, Eriksson J, Stengård J, Kesäniemi YA. Concordance for type 1 (insulin-dependent) and type 2 (non-insulin-dependent) diabetes mellitus in a population- based cohort of twins in Finland., 1992, 35(11): 1060–1067. [2] https://www.ebi.ac.uk/gwas/[EB/OL]. [2018.09]. [3] Kang JY, Sung SH, Lee YJ, Choi TI, Choi SJ. Impact of ENPP1 K121Q on change of insulin resistance after web-based intervention in korean men with diabetes and impaired fasting glucose., 2014, 29(10): 1353–1359. [4] Müssig K, Heni M, Thamer C, Kantartzis K, Machicao F, Stefan N, Fritsche A, Häring HU, Staiger H. The ENPP1 K121Q polymorphism determines individual susceptibility to the insulin-sensitising effect of lifestyle intervention., 2010, 53(3): 504–509. [5] Wang M, Pang C, Qu YL, Huang QY. Association and meta-analysis of ENPP1 K121Q with type 2 diabetes in Han Chinese., 2010, 32(8): 808–816.王敏, 彭婵, 屈亚莉, 黄青阳. 中国汉族人群ENPP1基因K121Q多态与2型糖尿病的关联及Meta分析. 遗传, 2010, 32(8): 808–816. [6] Haupt A, Thamer C, Heni M, Ketterer C, Machann J, Schick F, Machicao F, Stefan N, Claussen CD, Häring HU, Fritsche A, Staiger H. Gene variants of TCF7L2 influence weight loss and body composition during lifestyle intervention in a population at risk for type 2 diabetes., 2010, 59(3): 747–750. [7] Kim S, Misra A. SNP genotyping: technologies and biomedical applications., 2007, 9(1): 289–320. [8] Chinese Diabetes Society. Guidelines for the prevention and treatment of type 2 diabetes in China (2013 edition)., 2014, 30(10): 893–942.中华医学会糖尿病学分会. 中国2型糖尿病防治指南(2013年版). 中华内分泌代谢杂志, 2014, 30(10): 893– 942. [9] Writing Group of 2018 Chinese Guidelines for the Management of Hypertension, Liu LS. 2018 Chinese guidelines for the management of hypertension., 2019, 19(1): 1–44.《中国高血压防治指南》修订委员会, 刘力生. 中国高血压防治指南2018年修订版. 心脑血管病防治, 2019, 19(1): 1–44. [10] Fu CW. The Deqing cohort study on type 2 diabetes of adults in rural communities of China[Dissertation]. Fudan University, 2010.付朝伟. 农村社区成人2型糖尿病(德清队列)研究[学位论文]. 复旦大学, 2010. [11] Liu Y, Gen YM, Zhu YC. Chinese dietary guideline for residents evolution and enlightenments., 2017, 36(4): 21–25.刘越, 耿延敏, 朱亚成. 中国《居民膳食指南》演变及启示. 四川体育科学, 2017, 36(4): 21–25. [12] Obesity Working Group of China. Guidelines for prevention and control of overweight and obesity in Chinese adults., 2004, 26(1): 1–4.中国肥胖问题工作组. 中国成人超重与肥胖症预防与控制指南(节录). 营养学报, 2004, 26(1): 1–4. [13] Mahajan A, Taliun D, Thurner M, Robertson NR, Torres JM, Rayner NW, Payne AJ, Steinthorsdottir V, Scott RA, Grarup N, Cook JP, Schmidt EM, Wuttke M, Sarnowski C, Mägi R, Nano J, Gieger C, Trompet S, Lecoeur C, Preuss MH, Prins BP, Guo X, Bielak LF, Below JE, Bowden DW, Chambers JC, Kim YJ, Ng MCY, Petty LE, Sim X, Zhang W, Bennett AJ, Bork-Jensen J, Brummett CM, Canouil M, Ec Kardt KU, Fischer K, Kardia SLR, Kronenberg F, Läll K, Liu CT, Locke AE, Luan J, Ntalla I, Nylander V, Schönherr S, Schurmann C, Yengo L, Bottinger EP, Brandslund I, Christensen C, Dedoussis G, Florez JC, Ford I, Franco OH, Frayling TM, Giedraitis V, Hackinger S, Hattersley AT, Herder C, Ikram MA, Ingelsson M, Jørgensen ME, Jørgensen T, Kriebel J, Kuusisto J, Ligthart S, Lindgren CM, Linneberg A, Lyssenko V, Mamakou V, Meitinger T, Mohlke KL, Morris AD, Nadkarni G, Pankow JS, Peters A, Sattar N, Stančáková A, Strauch K, Taylor KD, Thorand B, Thorleifsson G, Thorsteinsdottir U, Tuomilehto J, Witte DR, Dupuis J, Peyser PA, Zeggini E, Loos RJF, Froguel P, Ingelsson E, Lind L, Groop L, Laakso M, Collins FS, Jukema JW, Palmer CNA, Grallert H, Metspalu A, Dehghan A, Köttgen A, Abecasis GR, Meigs JB, Rotter JI, Marchini J, Pedersen O, Hansen T, Langenberg C, Wareham NJ, Stefansson K, Gloyn AL, Morris AP, Boehnke M, McCarthy MI. Fine-mapping type 2 diabetes loci to single-variant resolution using high- density imputation and islet-specific epigenome maps., 2018, 50(11): 1505–1513. [14] Diabetes Genetics Replication And Meta-analysis (DIAGRAM) Consortium, Asian Genetic Epidemiology Network Type 2 Diabetes (AGEN-T2D) Consortium; South Asian Type 2 Diabetes (SAT2D) Consortium, Mexican American Type 2 Diabetes (MAT2D) Consortium, Type 2 Diabetes Genetic Exploration by Nex-generation sequencing in muylti-Ethnic Samples (T2D-GENES) Consortium, Mahajan A, Go MJ, Zhang W, Below JE, Gaulton KJ, Ferreira T, Horikoshi M, Johnson AD, Ng MC, Prokopenko I, Saleheen D, Wang X, Zeggini E, Abecasis GR, Adair LS, Almgren P, Atalay M, Aung T, Baldassarre D, Balkau B, Bao Y, Barnett AH, Barroso I, Basit A, Been LF, Beilby J, Bell GI, Benediktsson R, Bergman RN, Boehm BO, Boerwinkle E, Bonnycastle LL, Burtt N, Cai Q, Campbell H, Carey J, Cauchi S, Caulfield M, Chan JC, Chang LC, Chang TJ, Chang YC, Charpentier G, Chen CH, Chen H, Chen YT, Chia KS, Chidambaram M, Chines PS, Cho NH, Cho YM, Chuang LM, Collins FS, Cornelis MC, Couper DJ, Crenshaw AT, van Dam RM, Danesh J, Das D, de Faire U, Dedoussis G, Deloukas P, Dimas AS, Dina C, Doney AS, Donnelly PJ, Dorkhan M, van Duijn C, Dupuis J, Edkins S, Elliott P, Emilsson V, Erbel R, Eriksson JG, Escobedo J, Esko T, Eury E, Florez JC, Fontanillas P, Forouhi NG, Forsen T, Fox C, Fraser RM, Frayling TM, Froguel P, Frossard P, Gao Y, Gertow K, Gieger C, Gigante B, Grallert H, Grant GB, Grrop LC, Groves CJ, Grundberg E, Guiducci C, Hamsten A, Han BG, Hara K, Hassanali N, Hattersley AT, Hayward C, Hedman AK, Herder C, Hofman A, Holmen OL, Hovingh K, Hreidarsson AB, Hu C, Hu FB, Hui J, Humphries SE, Hunt SE, Hunter DJ, Hveem K, Hydrie ZI, Ikegami H, Illig T, Ingelsson E, Islam M, Isomaa B, Jackson AU, Jafar T, James A, Jia W, Jöckel KH, Jonsson A, Jowett JB, Kadowaki T, Kang HM, Kanoni S, Kao WH, Kathiresan S, Kato N, Katulanda P, Keinanen-Kiukaanniemi KM, Kelly AM, Khan H, Khaw KT, Khor CC, Kim HL, Kim S, Kim YJ, Kinnunen L, Klopp N, Kong A, Korpi-Hyövälti E, Kowlessur S, Kraft P, Kravic J, Kristensen MM, Krithika S, Kumar A, Kumate J, Kuusisto J, Kwak SH, Laakso M, Lagou V, Lakka TA, Langenberg C, Langford C, Lawrence R, Leander K, Lee JM, Lee NR, Li M, Li X, Li Y, Liang J, Liju S, Lim WY, Lind L, Lindgren CM, Lindholm E, Liu CT, Liu JJ, Lobbens S, Long J, Loos RJ, Lu W, Luan J, Lyssenko V, Ma RC, Maeda S, Mägi R, Männisto S, Matthews DR, Meigs JB, Melander O, Metspalu A, Meyer J, Mirza G, Mihailov E, Moebus S, Mohan V, Mohlke KL, Morris AD, Mühleisen TW, Müller-Nurasyid M, Musk B, Nakamura J, Nakashima E, Navarro P, Ng PK, Nica AC, Nilsson PM, Njølstad I, Nöthen MM, Ohnaka K, Ong TH, Owen KR, Palmer CN, Pankow JS, Park KS, Parkin M, Pechlivanis S, Pedersen NL, Peltonen L, Perry JR, Peters A, Pinidiyapathirage JM, Platou CG, Potter S, Price JF, Qi L, Radha V, Rallidis L, Rasheed A, Rathman W, Rauramaa R, Raychaudhuri S, Rayner NW, Rees SD, Rehnberg E, Ripatti S, Robertson N, Roden M, Rossin EJ, Rudan I, Rybin D, Saaristo TE, Salomaa V, Saltevo J, Samuel M, Sanghera DK, Saramies J, Scott J, Scott LJ, Scott RA, Segrè AV, Sehmi J, Sennblad B, Shah N, Shah S, Shera AS, Shu XO, Shuldiner AR, Sigurđsson G, Sijbrands E, Silveira A, Sim X, Sivapalaratnam S, Small KS, So WY, Stančáková A, Stefansson K, Steinbach G, Steinthorsdottir V, Stirrups K, Strawbridge RJ, Stringham HM, Sun Q, Suo C, Syvänen AC, Takayanagi R, Takeuchi F, Tay WT, Teslovich TM, Thorand B, Thorleifsson G, Thorsteinsdottir U, Tikkanen E, Trakalo J, Tremoli E, Trip MD, Tsai FJ, Tuomi T, Tuomilehto J, Uitterlinden AG, Valladares-Salgado A, Vedantam S, Veglia F, Voight BF, Wang C, Wareham NJ, Wennauer R, Wickremasinghe AR, Wilsgaard T, Wilson JF, Wiltshire S, Winckler W, Wong TY, Wood AR, Wu JY, Wu Y, Yamamoto K, Yamauchi T, Yang M, Yengo L, Yokota M, Young R, Zabaneh D, Zhang F, Zhang R, Zheng W, Zimmet PZ, Altshuler D, Bowden DW, Cho YS, Cox NJ, Cruz M, Hanis CL, Kooner J, Lee JY, Seielstad M, Teo YY, Boehnke M, Parra EJ, Chambers JC, Tai ES, McCarthy MI, Morris AP. Genome-wide trans-ancestry meta-analysis provides insight into the genetic architecture of type 2 diabetes susceptibility., 2014, 46(3): 234–244. [15] Kasuga K, Hata K, Tajima A, Ochiai D, Saisho Y, Matsumoto T, Arata N, Miyakoshi K, Tanaka M. Association of common polymorphisms with gestational diabetes mellitus in Japanese women: A case-control study., 2017, 64(4): 463–475. [16] Wellcome Trust Case Control Consortium. Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls., 2007, 447(7145): 661– 678. [17] Broadbent HM1, Peden JF, Lorkowski S, Goel A, Ongen H, Green F, Clarke R, Collins R, Franzosi MG, Tognoni G, Seedorf U, Rust S, Eriksson P, Hamsten A, Farrall M, Watkins H. Susceptibility to coronary artery disease and diabetes is encoded by distinct, tightly linked SNPs in the ANRIL locus on chromosome 9p., 2008, 17(6): 806–814. [18] Cugino D, Gianfagna F, Santimone I, de Gaetano G, Donati MB, Iacoviello L, Di Castelnuovo A. Type 2 diabetes and polymorphisms on chromosome 9p21: a meta-analysis., 2012, 22(8): 619–625. [19] Emanuele E, Lista S, Ghidoni R, Binetti G, Cereda C, Benussi L, Maletta R, Bruni AC, Politi P. Chromosome 9p21.3 genotype is associated with vascular dementia and Alzheimer's disease., 2011, 32(7): 1231– 1235. [20] Burdon KP, Macgregor S, Hewitt AW, Sharma S, Chidlow G, Mills RA, Danoy P, Casson R, Viswanathan AC, Liu JZ, Landers J, Henders AK, Wood J, Souzeau E, Crawford A, Leo P, Wang JJ, Rochtchina E, Nyholt DR, Martin NG, Montgomery GW, Mitchell P, Brown MA, Mackey DA, Craig JE. Genome-wide association study identifies susceptibility loci for open angle glaucoma at TMCO1 and CDKN2B-AS1., 2011, 43(6): 574–578. [21] Schaefer AS, Richter GM, Groessner-Schreiber B, Noack B, Nothnagel M, El Mokhtari NE, Loos BG, Jepsen S, Schreiber S. Identification of a shared genetic susceptibility locus for coronary heart disease and periodontitis., 2009, 5(2): e1000378. [22] Duesing K, Fatemifar G, Charpentier G, Marre M, Tichet J, Hercberg S, Balkau B, Froguel P, Gibson F, Strong association of common variants in the CDKN2A/ CDKN2B region with type 2 diabetes in French Europids., 2008, 51(5): 821–826. [23] Takeuchi F, Serizawa M, Yamamoto K, Fujisawa T, Nakashima E, Ohnaka K, Ikegami H, Sugiyama T, Katsuya T, Miyagishi M, Nakashima N, Nawata H, Nakamura J, Kono S, Takayanagi R, Kato N. Confirmation of multiple risk Loci and genetic impacts by a genome- wide association study of type 2 diabetes in the Japanese population., 2009, 58(7): 1690–1699. [24] Wu Y, Li HX, Loos RJF, Yu ZJ, Ye XW, Chen LH, Pan A, Hu FB, Lin X. Common variants in CDKAL1, CDKN2A/B, IGF2BP2, SLC30A8, and HHEX/IDE genes are associated with type 2 diabetes and impaired fasting glucose in a Chinese Han population., 2008, 57(10): 2834– 2842. [25] Steinthorsdottir V, Thorleifsson G, Reynisdottir I, Benediktsson R, Jonsdottir T, Walters GB, Styrkarsdottir U, Gretarsdottir S, Emilsson V, Ghosh S, Baker A,Snorradottir S, Bjarnason H, Ng MC, Hansen T, Bagger Y, Wilensky RL, Reilly MP, Adeyemo A, Chen Y, Zhou J, Gudnason V, Chen G, Huang H, Lashley K,Doumatey A, So WY, Ma RC, Andersen G, Borch-Johnsen K, Jorgensen T, van Vliet-Ostaptchouk JV, Hofker MH, Wijmenga C, Christiansen C, Rader DJ, Rotimi C, Gurney M, Chan JC, Pedersen O, Sigurdsson G, Gulcher JR, Thorsteinsdottir U, Kong A, Stefansson K. A variant in CDKAL1 influences insulin response and risk of type 2 diabetes., 2007, 39(6): 770–775. Modification effects of T2DM-susceptible SNPs on the reduction of blood glucose in response to lifestyle interventions Yuzhuo Wang1, Yiming Zhang2, Xiaolian Dong2, Xuecai Wang2, Jianfu Zhu2, Na Wang1, Feng Jiang1, Yue Chen3, Qingwu Jiang1, Chaowei Fu1 In recent years, it has been demonstrated that some susceptible gene loci of type 2 diabetes mellitus (T2DM) are not only associated with the susceptibility risk of T2DM, but also the modifying effects of lifestyle interventions. To further explore the modifying effects of the single nucleotide polymorphism (SNP) on the onset of T2DM and the reduction of blood glucose in response to lifestyle interventions among the high-risk population, we performed a lifestyle intervention study in two Deqing rural communities during the period from June to December in 2017. The intensive lifestyle interventions were conducted among the study subjects of the intervention group while those in the control group only received conventional and general health education. All participants were genotyped by the MassARRY system.This study showed that for SNP rs9502570, fasting blood glucose showed a significantly greater reduction for individuals with CC + CT genotype than those with TT genotype (=0.031). In the intervention group, the glycated hemoglobin A1C (HbA1C) decreased by 0.03% for those with CC+CT genotype, while HbA1C increased by 0.27% for those with TT genotype (=0.012). The difference in the reduction of fasting blood glucose and HbA1c between the intervention and control groups was also statistically significant between individuals with TT and those with CC+CT genotype. For SNP rs10811661, the reduction of fasting blood glucose was significantly higher in people with TT genotype than those with CC + CT genotype (0.44 mmol/L vs 0.12 mmol/L,=0.021). The difference in reduction of fasting blood glucose between the intervention group and control group was also statistically significant between TT and CC+CT genotype (<0.001). In summary, the SNP genotypes of both rs9502570 and rs10811661 could modify the effects of lifestyle interventions on reducing fasting blood glucose and HbA1C among the high risk rural population for T2DM. The present study has provided supporting evidence for future development of individualized intervention measures for high-risk population of T2DM. type 2 diabetes mellitus; lifestyle intervention; high-risk population; single nucleotide polymorphism 2019-12-05; 2020-02-26 国家自然科学基金项目(编号:81473038),上海市公共卫生重点学科建设计划项目(编号:15GWZX0801)和上海市公共卫生高端海外研修计划项目(编号:GWTD2015S04)资助[Supported by the National Natural Science Foundation of China (No.81473038), Shanghai Leading Academic Discipline Project of Public Health (No.15GWZK0801), and Shanghai 3-Year Public Health Action Plan (No.GWTD2015S04)] 王玉琢,在读硕士研究生,专业方向:遗传流行病学、慢性病流行病学。E-mail: wyzsally0323@163.com 付朝伟,博士,教授,研究方向:遗传流行病学、慢性病流行病学。E-mail: fcw@fudan.edu.cn 10.16288/j.yczz.19-304 2020/3/2 16:04:24 URI: http://kns.cnki.net/kcms/detail/11.1913.R.20200302.1104.002.html (责任编委: 吴志英)1.6 数据分析
2 结果与分析
2.1 研究对象基线特征
2.2 易感基因SNP对干预后血糖变化情况的影响
3 讨论
附录:

