基于生物信息学的脑胶质瘤预后风险长链非编码RNA筛选
俞盛健,麻怀露,陈王洋,丁晓飞,陈光,梁勇
(1.温州医科大学 第一临床医学院,浙江 温州 325035;2.台州学院 医学院,浙江 台州 318000;3.河北北方学院 研究生院,河北 张家口 075000)
胶质瘤是头部最常见的原发性恶性肿瘤[1]。目前,主要的脑胶质瘤治疗方式有手术、化疗、放疗、靶向治疗等,但是胶质瘤患者的预后仍较差,其中胶质母细胞瘤平均生存时间不超过18个月[2]。因此,探讨脑胶质瘤发生发展的潜在分子机制来发现有效的诊断和治疗靶点非常重要。长链非编码RNA(long non-coding RNA,lncRNA)是一类长度超过200个碱基的非编码RNA。虽然它不参与蛋白质的编码,但是近来许多研究发现lncRNA参与了体内多种生命活动,如胚胎干细胞的多样性、细胞周期的调节,以及癌症的发生发展等[3]。癌组织中lncRNA的差异表达可以通过调控DNA的突变,如改变增强子和启动子的活性或者染色质状态广泛影响转录来实现[4]。同样,lncRNA的差异表达也可以用来预测因组织DNA突变而导致的癌症发生。如今越来越多的研究证实lncRNA可以作为癌症的诊断标志物,甚至作为一种治疗选择[5]。例如在早期手术切除的乳腺癌中,lncRNA HOTAIR的过表达对于肿瘤转移和整体生存率具有很高的预测作用[6]。lncRNA PCA3作为前列腺癌患者尿中的一类生物标志物,与血清前列腺特异性抗原相比具有更高的特异性和预测价值,现已成为在前列腺癌诊断中一个非常有效可靠的指标[7]。
癌症基因图谱(the Cancer Genome Atlas,TCGA)是开放的公共数据平台,其包括30种癌症11 000例患者信息,对于这些巨大数据的挖掘分析,有助于研究者全面研究各种癌症发生的分子机制,确定新的治疗靶点和与肿瘤相关的分子标志物,从而提高对多种人类恶性疾病的诊断能力和治疗效果[8]。基因型组织表达(GTEx)数据库研究来自449名生前健康的人类捐献者的7 000多份尸检样本,GTEx对几乎所有转录基因的基因表达模式进行了观察,从而能够确定基因组中影响基因表达的特定区域。在本研究中,我们通过对联合GTEx和TCGA数据库中的脑胶质瘤样本进行基因转录水平的差异表达分析,构建了可用于预测患者预后的风险模型,并提取了可能与胶质瘤恶性进展有关的lncRNA靶点。
1 材料和方法
1.1 数据收集 从GDC Data Portal(https://portal.gdc.cancer.gov/)中下载人类脑胶质瘤的RNA-Seq数据和临床数据,时间截至2019-01-14。RNA-Seq数据包含lncRNA和mRNA,包括700个脑胶质瘤样本。按照TCGA官方指导说明中的方法,使用Data Transfer Toll进行数据下载。从UCSC Xena(https://xena.ucsc.edu/)中下载GTEx对应正常脑部组织表达数据,得到1 152个正常样本。
1.2 差异基因的筛选 利用edgeR包对脑胶质瘤以及相对应的正常样本进行分析,筛选出差异表达的lncRNA和mRNA。筛选标准为P<0.05,Fold Change>2。
1.3 生存分析以及多因素Cox模型的建立 根据lncRNA表达量的中位值将RNA表达量分为高表达组和低表达组,结合临床信息,通过survival包对lncRNA做Kaplan-Meier生存分析并画出生存曲线图。使用survival包对lncRNA做单因素Cox分析来挑选生存相关基因(survival related genes,SRGs),并且根据P值排序筛选出P值最小的前20个lncRNA,对挑选出的20个lncRNA进行多因素Cox模型的建立,其中剔除有共表达关系的lncRNA。根据风险值的中位数将患者分为高风险组和低风险组并画出风险生存曲线。最后通过ROC曲线评估多因素Cox模型的鉴别能力。
1.4 共表达基因预测 通过使用双侧Pearson相关系数检验lncRNA与蛋白编码基因的表达关系(|皮尔逊相关系数|>0.4,P<0.001)。符合条件的蛋白编码基因被认为是该lncRNA的共表达基因。
1.5 富集分析 根据前述所得的差异表达基因,通过DAVID在线软件(https://david.ncif-crf.gov/)和京都基因与基因组百科全书KEGG通路数据库(https://www.kegg.jp/)进行功能注释和通路富集分析。
1.6 蛋白相互作用网络构建 根据预测共表达基因中重复出现基因进行蛋白相互作用网络的构建。利用在线工具STRING11(https://string-db.org/)构建蛋白质的相互作用网络,统计互作网络各基因邻接节点数目。利用Cytoscape对获得的蛋白互作网络进行可视化,并通过MCODE插件对蛋白相互作用网络中各节点进行关联度分析(K值>5)。
1.7 患者标本和临床评估 患者标本和临床评估来源于具有脑胶质瘤组织学诊断,且在手术前未曾接受放疗和化疗的患者。从台州学院附属台州市立医院和台州市中心医院获取5对脑胶质瘤癌组织及癌旁组织。所有的样本均在手术切除后立即冷冻在液氮中。研究通过台州学院医学院伦理委员会批准,并获得患者的知情同意。
1.8 荧光实时定量PCR TRIzol试剂提取总RNA,Takara试剂盒完成反转录和qPCR检测,HOXA-AS2引物序列为5'-CCCGTAGGAAGAACCGATGA-3'(正)和5'-TTTAGGCCTTCGCAGACAGC-3'(反),GAPDH引物序列为5'-ACCACAGTCCATGCCATC-3'(正)和5'-TCCACCCTG TTGCTGTA-3'(反)。PCR扩增循环:95 ℃ 10 min;40循环的95 ℃ 10 s,60 ℃ 60 s。通过ΔΔCT法半定量HOXA-AS2相对水平。
1.9 统计学处理方法 应用R3.3.5软件进行相关图形制作,差异显著性的筛选阈值为log2foldchange(log2FC)不小于2,采用Kaplan-Meier法绘制生存曲线并应用log-rank方法比较生存率,多变量因素利用Cox回归进行生存分析,双侧Pearson相关系数检验lncRNA与蛋白编码基因的表达关系。采用双侧检验,P<0.05为差异有统计学意义。
2 结果
2.1 胶质瘤中差异表达的lncRNA和mRNA 通过比较脑胶质瘤样本和正常脑组织样本,获得差异表达的lncRNA 424个(211个lncRNA上调,213个lncRNA下调;见图1A),mRNA 3 827个(1 618个mRNA上调,2 209个mRNA下调;见图1B),差异有统计学意义(P<0.05)。
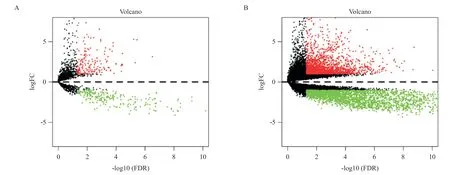
图1 脑胶质瘤和正常脑组织中差异表达RNA的火山图
2.2 生存分析以及多因素Cox模型的建立 运用单因素Cox分析探究差异表达的lncRNA与胶质瘤患者生存期的关系,挑选出统计学最显著的20个lncRNA(见表1)。利用这20个lncRNA,我们进行了多因素Cox分析。考虑到临床应用的经济性,我们剔除了有共表达关系的lncRNA,只保留其中的一个,最终得到包含9个lncRNA(见表2)的生存预后模型,并且进行生存曲线分析(见图2A)。为了测试模型的准确性,我们计算了模型的AUC值并进行ROC曲线分析(见图2B),AUC值达0.907,说明该模型能够相对准确地预测患者的预后。

表1 经单因素Cox模型挑选的lncRNA
2.3 胶质瘤中显著差异表达的lncRNA富集分析 对挑选出的9个lncRNA中的CRNDE、LINCOO994、LBX2-AS1、HOXA-AS2进行蛋白编码基因共表达关系的预测,以相关系数大于0.6为临界点,并对所得基因进行筛选,挑选出重复出现的基因进行富集分析。GO功能和KEGG通路富集分析结果显示(见图3),对其富集注释结果提示(见表3-4),这些基因功能主要富集在通道蛋白活性调控,分子黏附等方面;介导的信号转导通路主要集中于免疫缺陷病毒感染,神经信号转导相关通路。
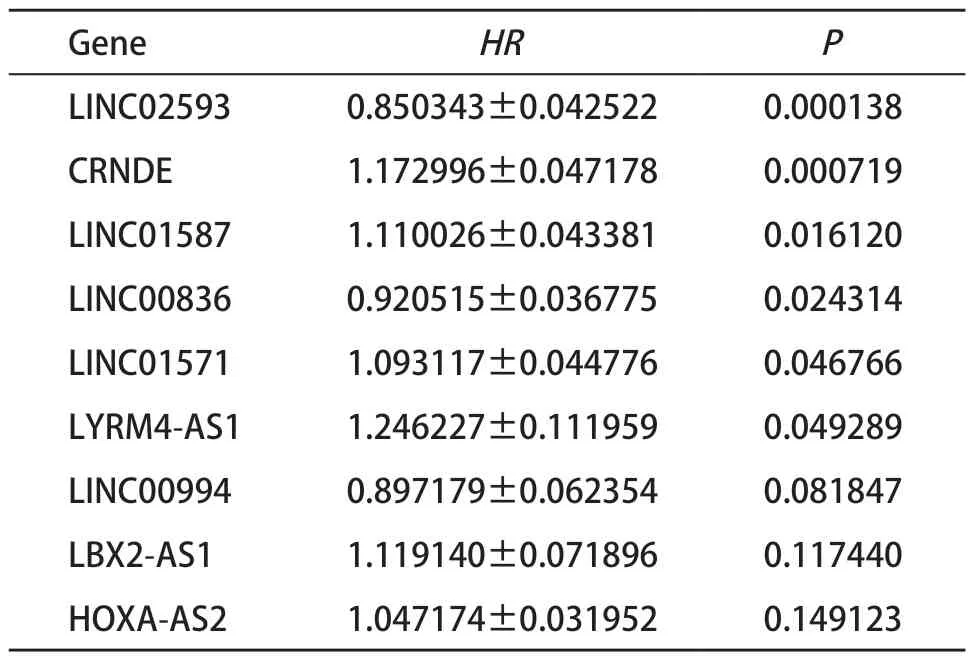
表2 经多因素Cox模型挑选的lncRNA
2.4 蛋白相互作用网络的构建和关键基因的筛选 在预测的共表达基因中,构建重复基因的蛋白相互作用网络,并与差异表达基因比对,发现33个上调基因,35个下调基因(见图4A)。对蛋白相互作用网络中各节点的邻接数进行统计(见图4B),其中KIF2C与CDC20的邻接节点最多,分别是16个和13个。使用MCODE插件筛选出网络中的关联度最紧密的2个基因簇(见图4C),其中一个基因簇包括KIF1A、KIF3A、KIF3C、SPTBN2、CAPZA1、KDELR2、KIF2C、KIF21B、TMED9、KDELR3、KDELR1。对其蛋白相互作用网络关键基因簇功能进行注释,该基因簇功能主要富集在逆行小泡介导转运,微管转运,ATP结合和细胞胞质中。另一个基因簇包括GABBR1、ADCY5、GNG12、HTR1A、ANXA1、SSTR2、GNG5,功能主要富集在细胞胞膜上。
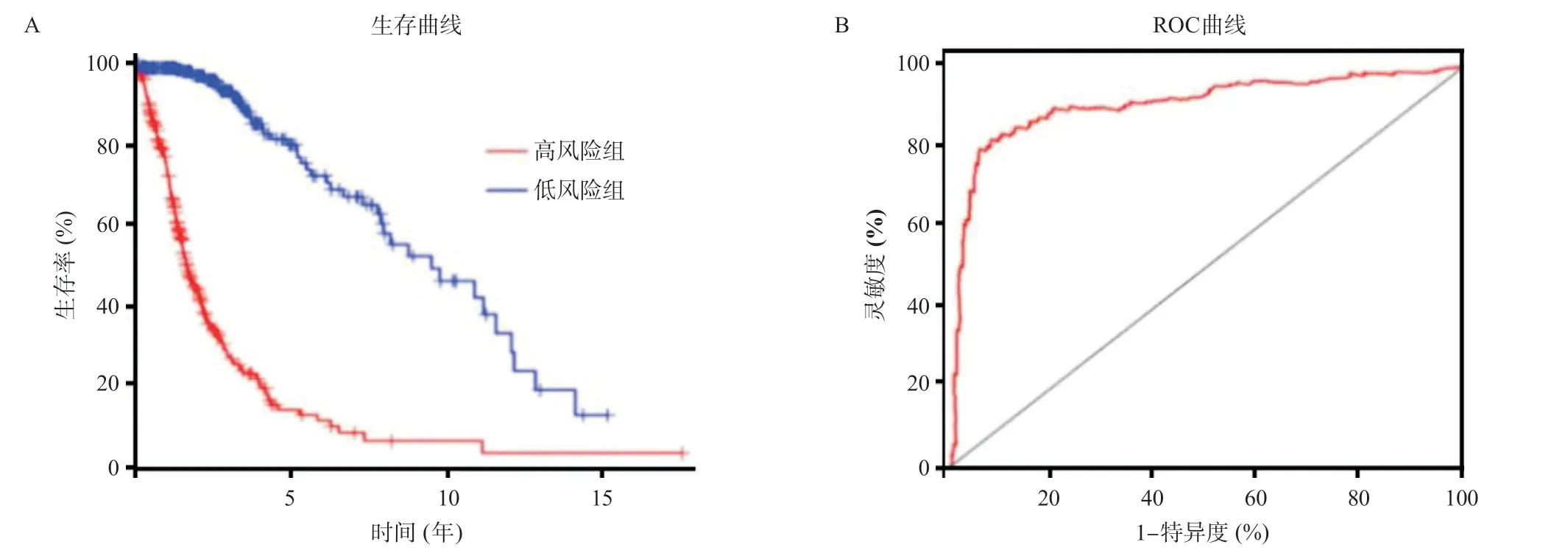
图2 Cox分析筛选出的差异基因以及构建的生存模型
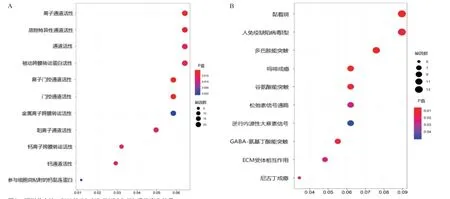
图3 预测共表达mRNA的GO(A)及KEGG(B)通路富集结果
2.5 HOXA-AS2在脑胶质瘤组织及正常脑组织中的表达 为进一步验证生物信息学分析结果,我们收集5例临床脑胶质瘤样本,抽提RNA,进行Real time PCR定量检测。结果如图5所示,HOXA-AS2在脑胶质瘤组织中表达水平明显高于正常脑组织(见图5)。
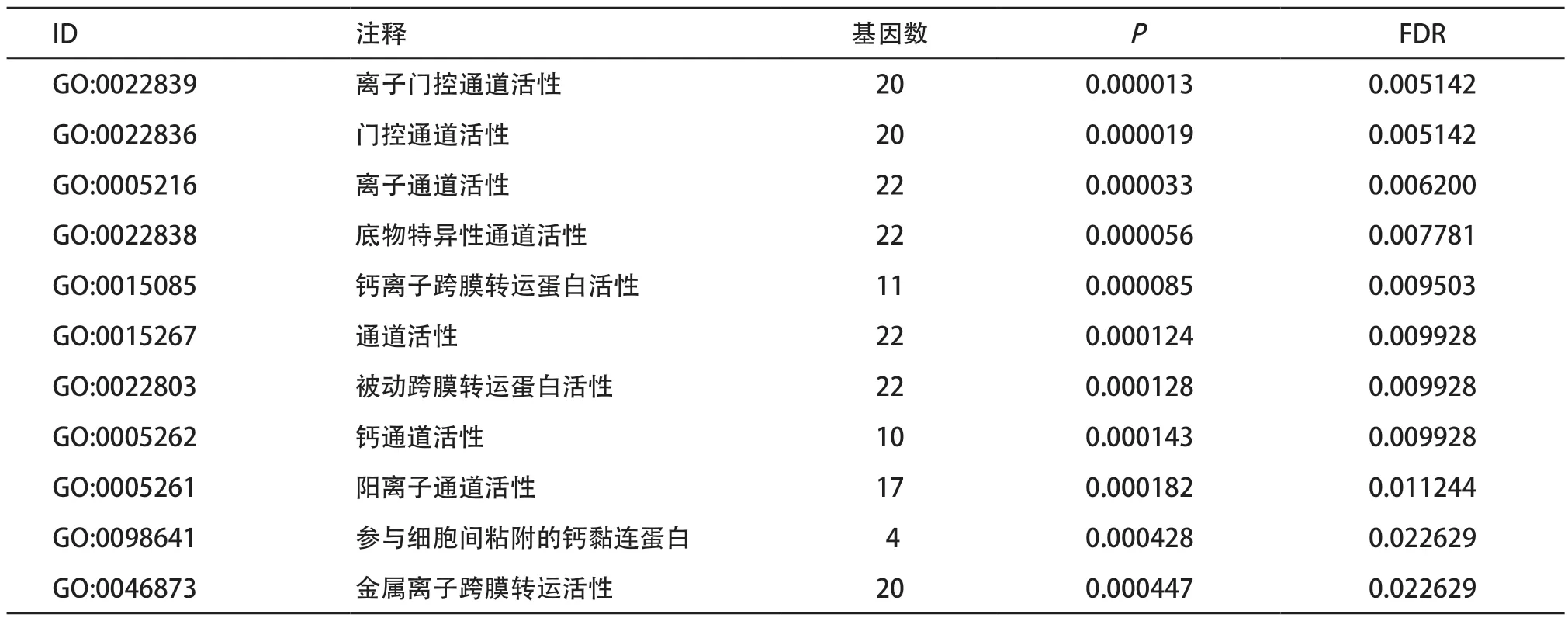
表3 GO富集注释结果
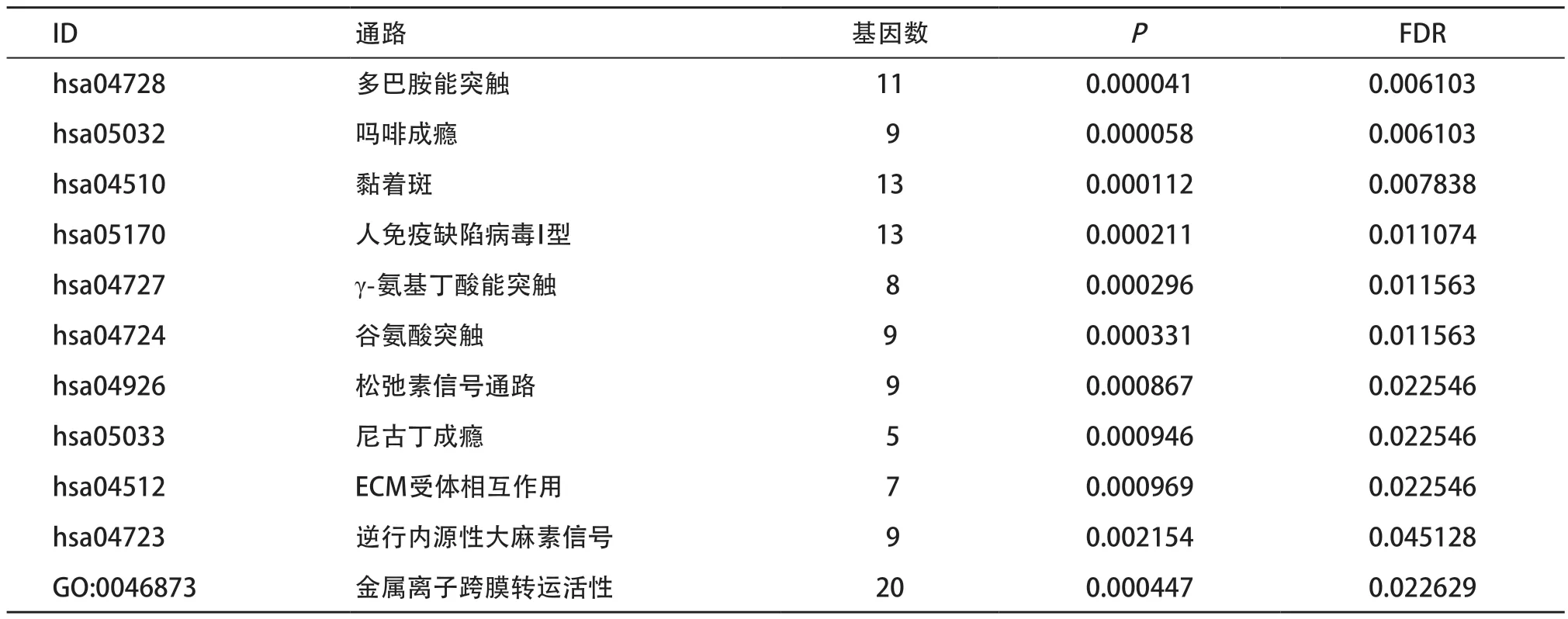
表4 KEGG通路富集注释结果
3 讨论
胶质瘤是最常见的脑肿瘤之一,在早期脑肿瘤中占50%~60%[9-10]。在WHO分类中,神经胶质瘤被归类为I-IV级。I级和II级星型细胞瘤和少突神经胶质瘤是低级别胶质瘤,III级和IV级星型细胞瘤和少突神经胶质瘤是高级别胶质瘤[11]。目前有证据表明lncRNA作为一类新的非编码基因调节因子,可能在胶质瘤的广泛的生物学过程中发挥重要作用,例如,通过充当癌基因或肿瘤抑制基因,它有助于胶质瘤的发生、进展和其他恶性表型的发展[12]。在本研究中,我们使用TCGA胶质瘤转录水平数据和临床数据,分析筛选出了424个差异表达的lncRNA,再对这些差异表达lncRNA进行单因素Cox分析,挑选出20个代表性的lncRNA,其中有研究发现DGCR9在胃癌组织中高表达,并且有利于胃癌细胞系AGS,MGC803的增殖、侵袭和糖代谢[13]。
HOTAIRM1是HOXA基因簇的反义链的转录产物,最近有研究显示其与急性髓性白血病、结直肠癌、多形性胶质母细胞瘤有关[14-16]。然而,HOTAIRM1在胃癌中又通过靶向miR-17-5p抑制胃癌细胞系的增殖和迁移并诱导其凋亡[17]。我们还通过多因素Cox分析构建了与脑胶质瘤患者生存相关的生存模型。通过绘制出ROC曲线和计算AUC值来确定该模型的准确性。最终AUC值达0.907,说明该模型能够相对准确地预测患者的预后,并且,我们对筛选出的CRNDE、LINCOO994、LBX2-AS1、HOXA-AS2这4个lncRNA进行了mRNA共表达的预测,利用这些mRNA的GO功能和KEGG通路富集结果来预测lncRNA可能参与的功能和通路。我们发现,这些基因主要在控制通道蛋白活性,分子黏附等功能富集。其中,有10个基因富集在控制钙通道活性的功能里。钙黏蛋白是钙依赖性细胞-细胞黏附分子,在神经系统发育成熟过程中起至关重要的作用,根据其结构和功能,可分为12个家族,包括经典钙黏蛋白、桥粒钙黏蛋白、T-钙黏蛋白、7D-钙黏蛋白、原钙黏蛋白、CDH15和23、脂肪、Dachsous、Flamingo、Celsr、钙调素和Ret[18]。在癌症中,E-钙黏蛋白作为侵袭抑制因子,常常被下调,而作为侵袭起始因子的N-钙黏蛋白则被上调[19]。原钙黏蛋白PCDH10甲基化抑制了肿瘤细胞的生长、迁移、侵袭和克隆形成[20]。基因还在免疫缺陷病毒感染,神经信号转导相关通路上富集。研究表明,HIV-1胞膜糖蛋白120可诱导胶质瘤细胞分泌基质金属蛋白酶[21]。另外,已经有报道称CRNDE在非小细胞肺癌、肝癌、宫颈癌和脑胶质瘤中起到促癌作用[22-25]。对中国胶质瘤基因组图谱数据库的RNA测序数据进行分析,发现LBX2-AS1与胶质瘤患者的预后和恶性进展有显著关系[26]。HOXA-AS2不仅在急性髓系白血病细胞对阿霉素的耐药性中起重要作用,也参与了肝癌的恶性进展过程[27-28]。
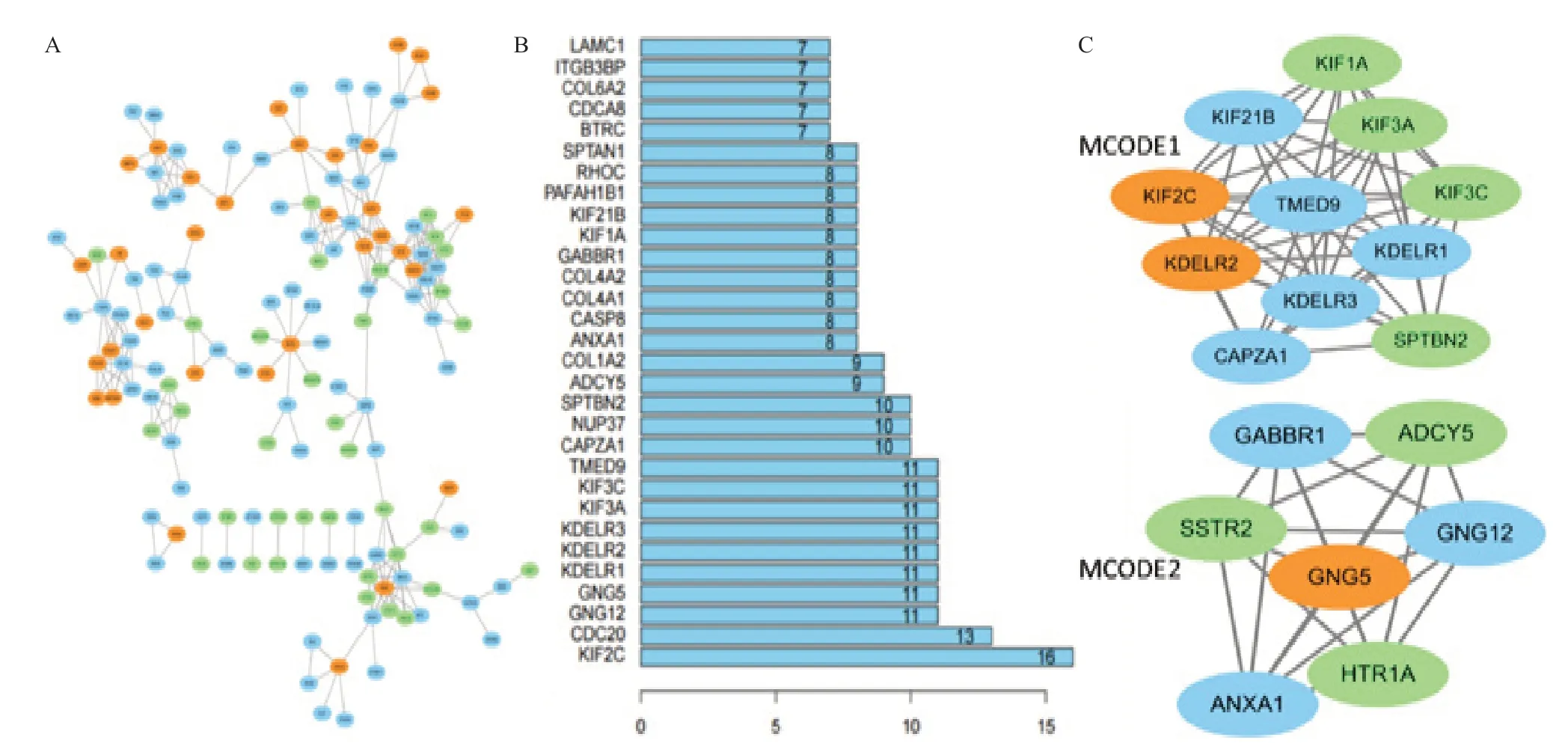
图4 预测共表达基因中重复基因的蛋白相互作用网络及关键基因簇分析
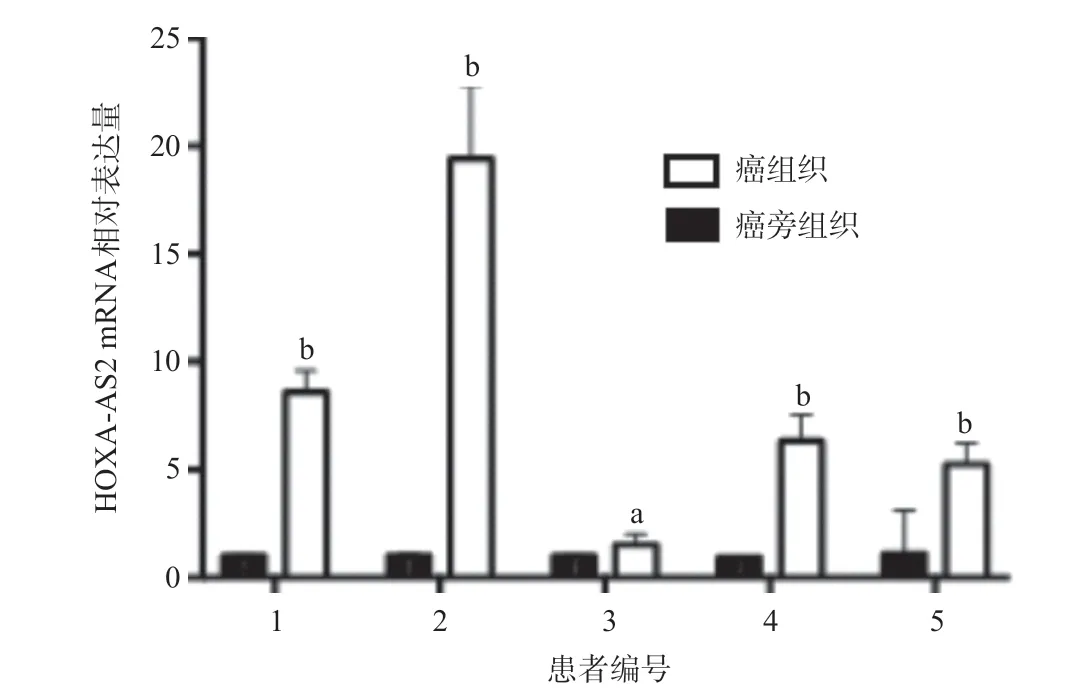
图5 HOXA-AS2在脑胶质瘤中的表达情况
综上所述,本研究利用生物信息学方法分析TCGA数据,筛选潜在的胶质瘤中表达差异显著的基因,通过Cox分析构建生存模型,并筛选出了可能为胶质瘤分子致病机制和分子靶向治疗提供参考的几个lncRNA和mRNA,包括CRNDE、LINCOO994、LBX2-AS1和HOXA-AS2。并在有限例数临床患者胶质瘤样本HOXA-AS2表达水平和病理进程相关性的分析中,进一步证实HOXA-AS2可能为诊断预警分子,但其潜在与胶质瘤分子机制的关系仍需临床及基础实验验证。

