CRISPR-Cas基因编辑系统升级:聚焦Cas蛋白和PAM
唐连超,谷峰
综 述
CRISPR-Cas基因编辑系统升级:聚焦Cas蛋白和PAM
唐连超,谷峰
温州医科大学附属眼视光医院,眼视光学和视觉科学国家重点实验室,温州 325027
以CRISPR-Cas (clustered regularly interspaced short palindromic repeats and CRISPR associated proteins)系统为代表的基因编辑技术的出现极大地促进了人类改造自然界物种的能力。在医疗、工业、农业等多个研究领域,基因编辑技术正在被广泛应用。Cas蛋白是CRISPR-Cas系统的功能蛋白,不同类型的Cas蛋白在其自身活性、识别位点、切割末端、RNA需求等方面具有不同的特性。PAM (protospacer adjacent motif)是靶位点附近的若干个碱基,对Cas蛋白识别靶序列至关重要,也是CRISPR-Cas系统发挥功效的关键特性之一。目前已有多种不同的PAM鉴定方法被报道。本文对Cas蛋白的寻找、Cas蛋白突变体筛选及PAM的确定方法(含PAM谱拓展)进行了综述,以期为新型基因编辑工具的发展和优化提供借鉴。
基因编辑;CRISPR-Cas系统;Cas蛋白;PAM;定向进化
成簇的规律间隔的短回文重复序列及CRISPR相关基因编码的系统(clustered regularly intersected short palindromic repeats and CRISPR associated proteins, CRISPR- Cas),即CRISPR-Cas系统,是继锌指核酸酶(zinc- finger nucleases, ZFNs)和转录激活因子样效应物核酸酶(transcription activator-like effector nucleases, TALENs)后出现的一种新型基因编辑技术,其操作简单,可精确编辑核酸序列[1,2]。CRISPR-Cas系统中Cas蛋白成员除了有双链DNA切割功能以外,还发现有其他特性,如:最新发现的C2c2 (class 2 candidate 2)/Cas13a能够对ssRNA (single-stranded RNA)进行切割,可于病毒检测[3~5];改造过的dCas (dead Cas)在融合其他功能蛋白后被用于基因表达调节或单碱基编辑[6~9];基于CRISPR-Cas系统的Prime Editing工具能以全新的方式进行基因编辑[10]。以CRISPR-Cas为主体的基因编辑技术正在生命科学领域引发一场技术革命[11~15]。
CRISPR-Cas系统遍布于多种细菌和古菌,通常由一组特征性CRISPR阵列和CRISPR相关(CRISPR associated, Cas)基因组成[16]。CRISPR阵列包含重复序列和间隔序列,重复序列(direct repeats)分散在可变的间隔序列(spacers)之间。间隔序列对应于外源遗传元件原型间隔区(protospacers)内的序列。Cas基因转录翻译为Cas蛋白,而CRISPR阵列经转录加工成更短的CRISPR RNA (crRNA),最后引导Cas蛋白降解靶核酸(图1)。
CRISPR-Cas系统产生切割必需3个条件:Cas蛋白、RNA和PAM[17,18]。Cas蛋白是发挥识别、切割的功能蛋白。而PAM被称为原型间区序列邻近基序,是CRISPR-Cas系统区分自我和入侵者的标记[19,20]。因此,Cas蛋白的寻找和PAM确定是CRISPR相关研究中的重要内容之一。本文对目前报道的Cas蛋白发现、定向进化和PAM的确定方法进行总结与展望。
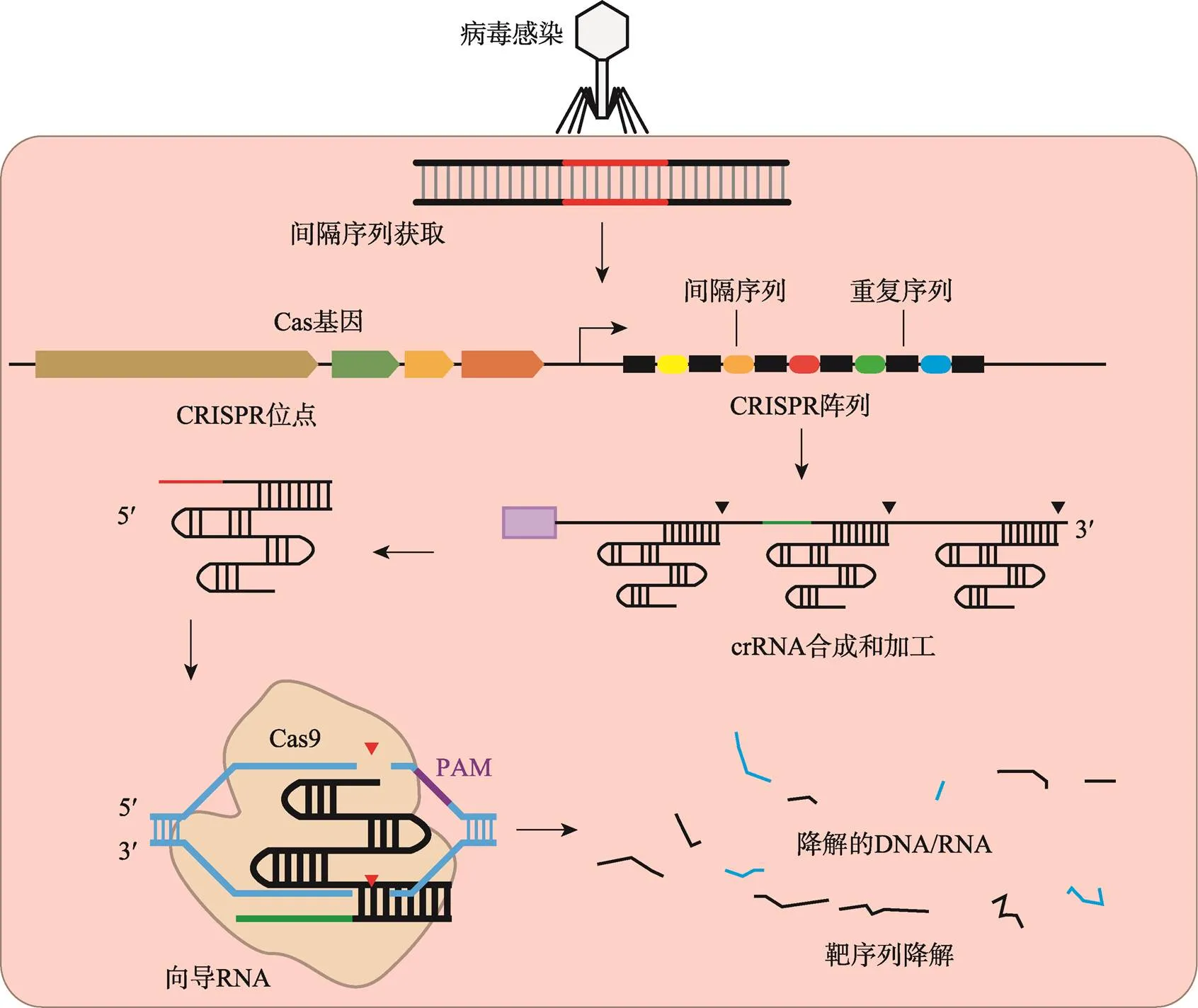
图1 微生物CRISPR系统在适应性免疫中的分子机制
微生物将从外源遗传物质中获得间隔序列(spacer),然后将其插入到自身的重复序列(direct repeat)中。当外源DNA再次入侵时,Cas蛋白结合crRNA后会靶向到该间隔序列所在区域。识别PAM序列后,Cas蛋白会将此外源DNA降解[13]。
1 新型Cas蛋白的发现与定向进化
Cas蛋白编码基因,最开始是通过生物信息学方法在CRISPR基因座附近搜索时被发现。Jansen等[16]定义了Cas1~Cas4共4种Cas蛋白。Jinek等[21]证明了双RNA引导的SpCas9 (Cas9)可用于dsDNA (double-stranded DNA)的切割。Mali等[22~24]迅速推动了SpCas9用于哺乳动物及人类基因编辑。随后,比SpCas9更小的Cas蛋白—SaCas9 (Cas9)被发现,它容易包装于AAV (Adeno-associated virus)并进行体内递送,在体内进行基因编辑时更具优势[25]。SpCas9是目前研究最充分并广泛用于基因编辑的Cas蛋白,但是它对5ʹ-NGG-3ʹPAM的要求也限制了其对靶向位点的选择。近期的研究发现了一类能够识别5ʹ-TTN-3ʹPAM的Cas蛋白,Cpf1 (CRISPR from Prevotella and Francisella 1,也被称为Cas12a),其在一定程度上扩大了CRISPR-Cas工具在DNA序列上的编辑范围[26]。此外CRISPR-Cas系统的脱靶也是其应用中不可忽略的限制因素[27]。针对上述缺陷,研究者相继发现新的相关蛋白:Cas蛋白突变体、Cas蛋白同系物和其他新的Cas蛋白(表1)。这些新发现或改造过的Cas蛋白不仅能够识别新的PAM,有一些还提高了基因编辑的效率或保真性。然而,要实现对靶序列的精准编辑,仍需要研发以“升级版”CRISPR-Cas为代表的新型基因编辑系统。
1.1 利用生物信息学发现Cas蛋白
CRISPR在古菌及细菌中广泛存在,因此在古菌或细菌中鉴定CRISPR和Cas蛋白是一种寻找Cas蛋白的方式。Schunder等[28]在土拉弗朗西斯菌诺维达菌株() U112基因组内鉴定了两种不同的CRISPR-Cas系统。他们获得多个菌株的基因组序列并分析了CRISPR-Cas系统的同源系统。随后,Zetsche等[29]探索了Cpf1组成的CRISPR系统的功能。
对已知的Cas蛋白进行比对是寻找Cas蛋白一种直接的方式。Shmakov等[30]设计了一个计算流程来展示微生物基因组序列多样性,以确定以前未检测到的2类CRISPR-Cas基因座(图2)。因为大型单亚基效应蛋白(如Cas9和Cpf1)的存在是II型和V型系统的判定特征,所以研究者使用作为确定候选基因座的关键词,寻找包含编码超过500个氨基酸大蛋白并且未分类的2类系统候选CRISPR-Cas基因座。最后,研究者发现了53个基因座具有2类CRISPR-Cas系统的特征,并根据推定的效应蛋白性质将其分成了3个不同的组(C2c1、C2c3和C2c2)[30]。
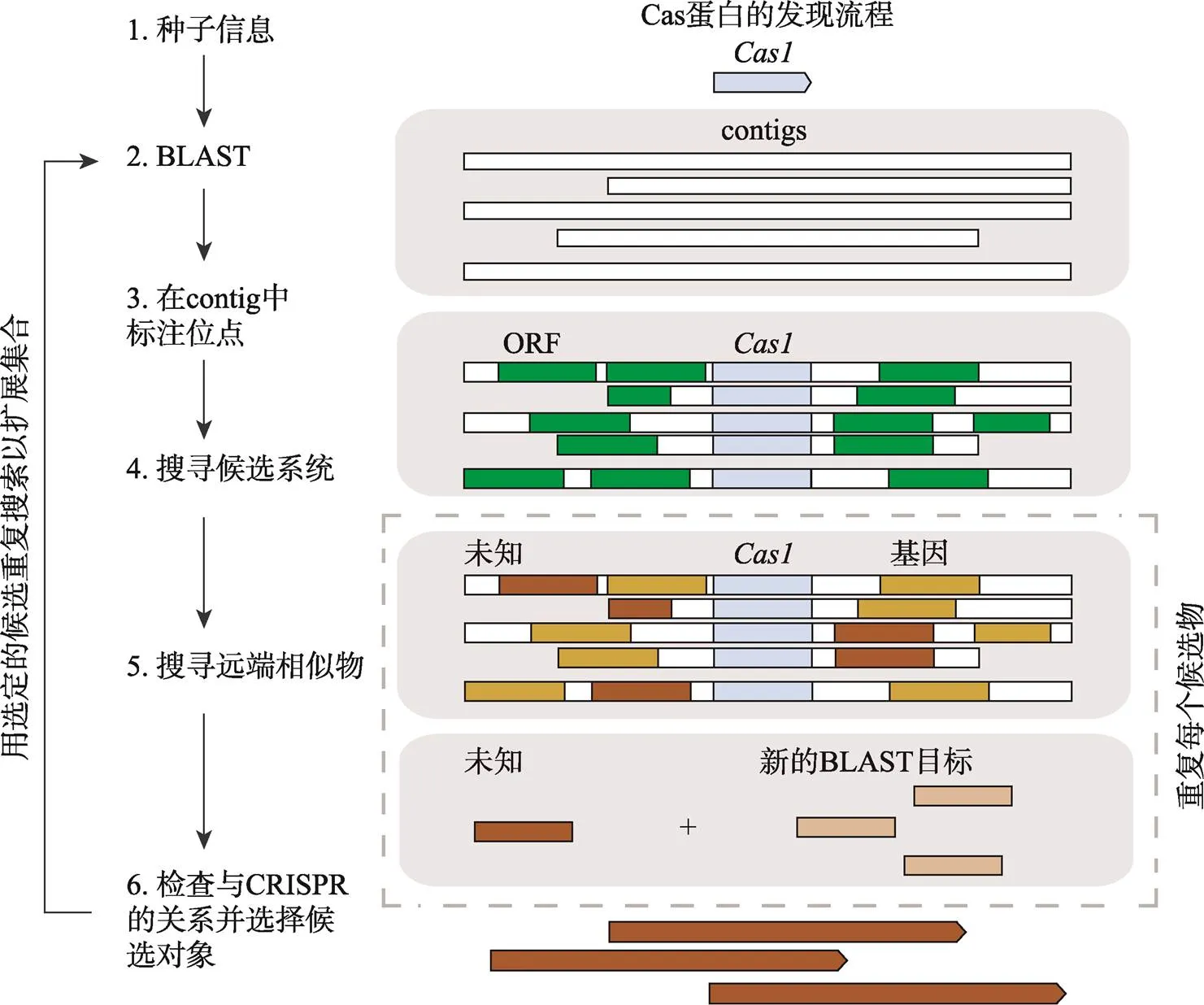
图2 用于识别推定新的2类CRISPR-Cas基因座的计算流程
通过设定条件进行比对,搜索尚未发现的Cas蛋白基因[30]。
Burstein等[31]在宏基因组数据库中比对了CRISPR-Cas系统的功能组件,在宏基因组中发现了2个未知的系统(CRISPR-CasX和CRISPR-CasY)。他们基于3个不同样品的宏基因组分析,首先使用HMM模型(Hidden Markov Model)在各种样品的组装重叠群中对已知的Cas蛋白进行比对;接下来,使用CRISPRFinder软件识别CRISPR阵列,然后使用了大于800个残基的未鉴定蛋白的条件进行筛选;最后将这些大蛋白作为潜在的2类Cas效应子进一步分析,进而发现了CRISPR-CasX和CRISPR-CasY系统。最近的研究结果表明CRISPR-CasX系统在人类细胞中具有基因编辑活性[32]。
1.2 利用质粒干扰耗尽筛选特定Cas蛋白
Gao等[33]使用盒式诱变构建了AsCpf1突变体并导入到大肠杆菌中,将携带有不同PAM的氨苄青霉素抗性质粒导入该细菌中。当在氨苄青霉素选择性培养基上生长时,细菌因抗性基因发生切割而死亡(图3)。通过将原始库的序列与存活细菌中携带AsCpf1质粒的DNA序列进行比较,确定了由于蛋白突变导致的PAM识别突变体。研究者利用此方法制作的AsCpf1 RR和AsCpf1 RVR突变体能分别特异性识别5ʹ-TYCV-3ʹ和5ʹ-TATV-3ʹ PAM,扩大了Cpf1在人类DNA上的靶向范围。
1.3 利用细菌选择性定向进化筛选识别不同PAM的Cas蛋白突变体
为寻找新的PAM特异性Cas9突变体,Kleinstiver等[34,35]使用了一种细菌选择性定向进化系统(bacterial selection-based directed evolution)。这个系统中含有SpCas9和毒性质粒,只有当SpCas9切割毒性基因后细菌才能存活,从而筛选出能够切割突变PAM的Cas9突变体(图4)。研究者对野生型和突变体R1335Q SpCas9的PAM相互作用域进行诱变,并使用NGA PAM目标位点对其进行筛选。来自两个库存活克隆的序列显示:最常见的氨基酸替换为D1135V/Y/N/E、R1335Q和T1337R。通过在人类细胞中切割测试这些突变的所有组合后,确定了不同于识别5ʹ-NGG-3ʹPAM的野生型Cas蛋白的两种突变体VQR和EQR,同时利用此方法还确定了VRER突变体。SpCas9的工程突变体VQR、EQR和VRER分别识别5ʹ-NGA-3ʹ、5ʹ-NGAG-3ʹ和5ʹ- NGCG-3ʹPAM。
1.4 利用噬菌体辅助连续进化筛选识别宽松PAM的Cas蛋白
Esvelt等[36,37]为了获得理想的SpCas9突变体,利用噬菌体辅助连续进化(phageassisted continuous evolution, PACE)进行筛选。具体的方法是将dCas9蛋白编码基因序列插入噬菌体基因组,并将dCas9的靶向活性与噬菌体的感染能力相偶联。因为具有结合活性的dCas9才能激活的表达,使噬菌体能够感染宿主大肠杆菌。该系统中的突变质粒(mutagenesis plasmids, MP)使大肠杆菌中的复制产生突变,导致储存于噬菌体基因组中的Cas蛋白基因在复制过程中产生大批量、持续性的突变,并在噬菌体感染过程中被筛选(图5)。研究者利用此方法获得了突变体xCas9 3.7,其具有更灵活的PAM适应性,能识别更广的PAM,包括5ʹ-NGG-3ʹ、5ʹ-NG-3ʹ、5ʹ-GAA-3ʹ和5ʹ-GAT-3ʹ[37]。
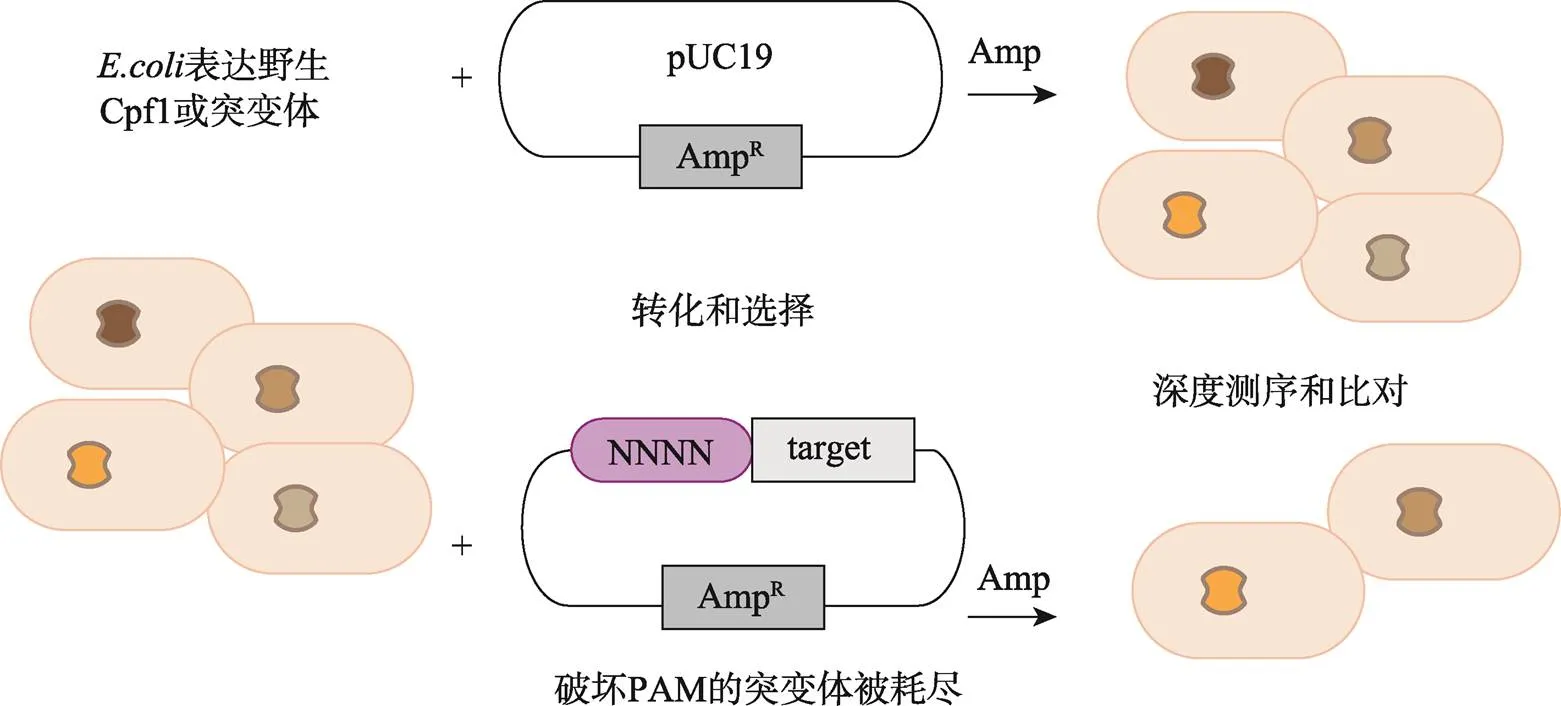
图3 用于鉴定改变PAM特异性突变体的质粒干扰耗尽筛选
利用抗性的丢失来区分对PAM识别[33]。
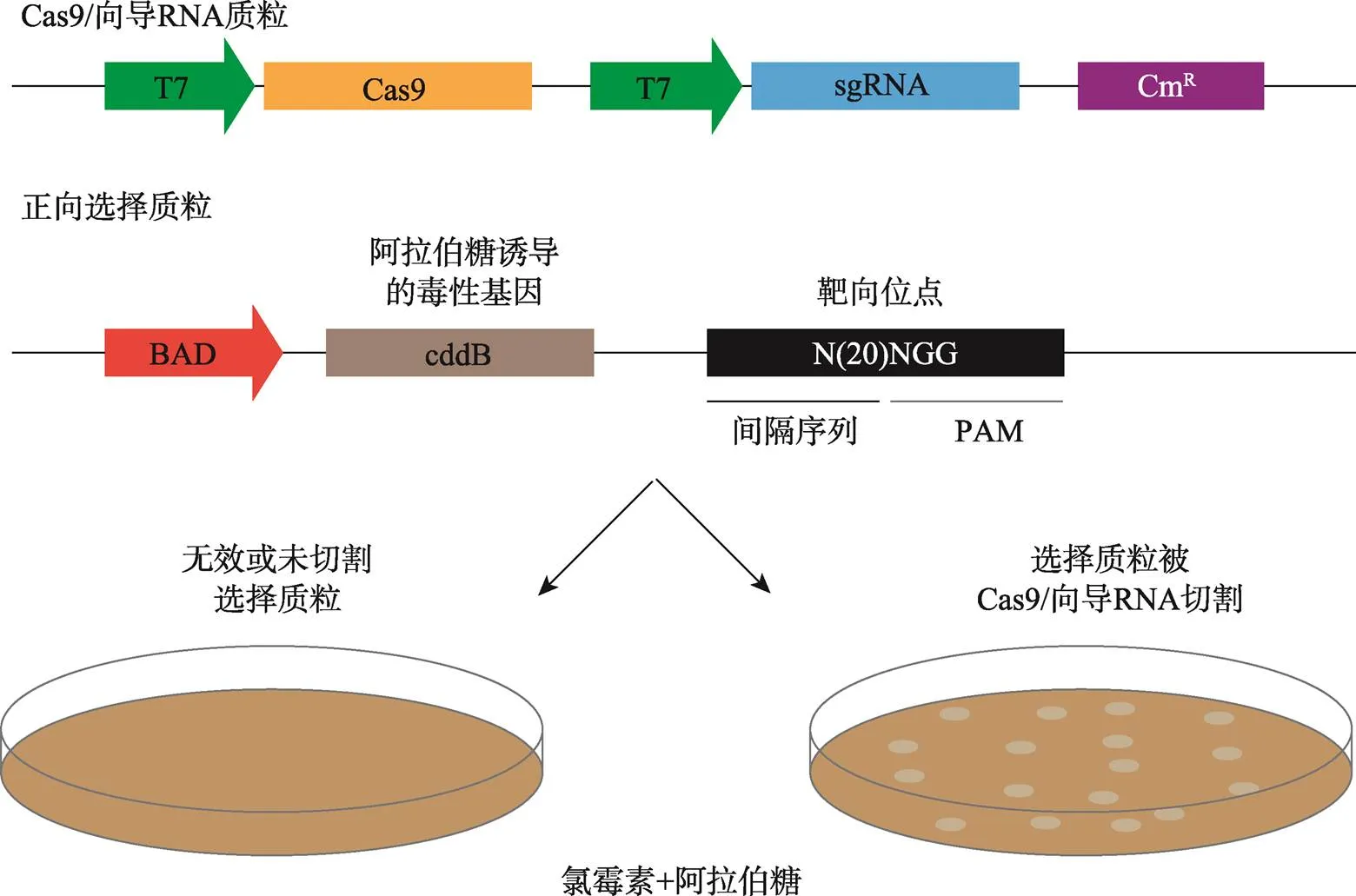
图4 用细菌选择性定向进化进行正向选择
细菌中的可被识别PAM的毒性质粒被切割后,细菌得以存活[34]。
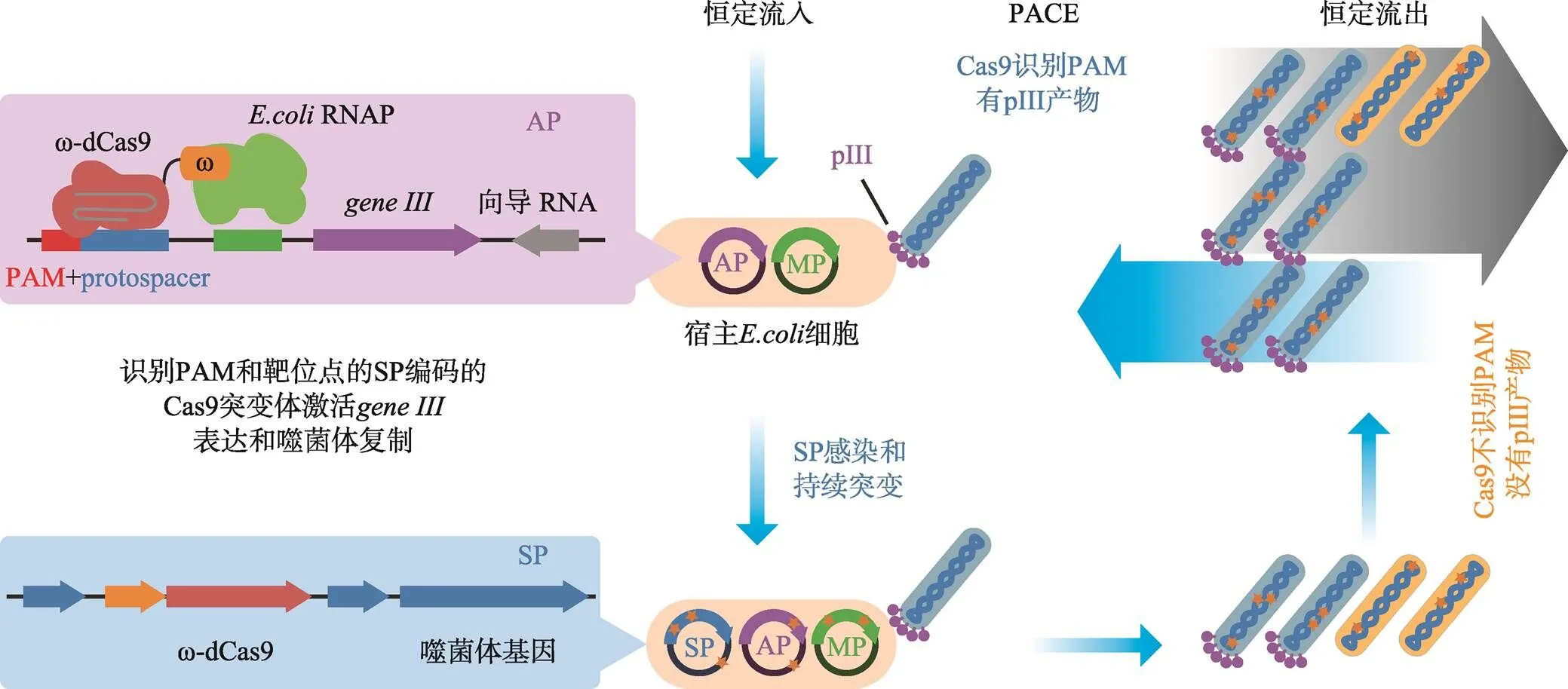
图5 具有扩大的PAM兼容性的Cas9突变体的PACE
选择性噬菌体(selection phage, SP)基因组中包含了Cas9突变体基因。辅助质粒(accessory plasmid, AP)含有靶序列,PAM和。Cas突变后若有结合能力,可以使得下游表达,噬菌体得以繁殖。这个过程中突变的有结合能力的Cas蛋白会被富集[37]。
1.5 利用晶体结构分析设计突变产生新的Cas蛋白
Nishimasu等[38]发现了SpCas9的突变体SpCas9-NG,其可以识别5ʹ-NG-3ʹ PAM,并且其活性能够达到SpCas9的70%~90%。通过对该突变体蛋白晶体结构分析,他们认为可以通过消除SpCas9 Arg1556与5ʹ-NGG-3ʹ PAM中第3个G之间的相互作用实现5ʹ-NG-3ʹ PAM的特异识别。然而,进行R1335A氨基酸突变的蛋白对5ʹ-TGG-3ʹ PAM几乎没有活性。所以,研究者通过引入突变R1335A/L1111R/ G1218R/A1322R/T1337R,恢复了其部分活性。根据先前报道的VQR和VRER突变体中所述的突变D1135V在蛋白与PAM双链体相互作用中的功能后,研究者在突变体中加入了R1335V的突变。此外,分子模拟也表明E1219F突变与第2个G的核糖部分形成疏水相互作用,R1335V突变稳定了E1219F突变体。将全部有利突变集合后的R1335V/L1111R/ D1135V/G1218R/E1219F/A1322R/T1337R突变体(VRVRFRR)恢复了其活性并完成预期假设的目标。这个识别5ʹ-NG-3ʹ PAM的SpCas9 VRVRFRR突变体被命名为SpCas9-NG,具有相对广泛的PAM识别特性[38]。
1.6 Cas蛋白发现方法的比较
为发现不同的Cas蛋白和相关突变体以实现DNA的精确编辑,科研人员采用了多种不同的方法和策略,以上仅仅列出了颇具代表性的几种研究方法:生物信息学挖掘、质粒干扰耗尽筛选法、细菌选择性定向进化、噬菌体辅助连续进化和以晶体结构为基础的定点突变法,这些方法的比较见表1。
相比于其他方法,生物信息学挖掘需要已知的序列信息,其操作简便快速,而且一次能够发现大量结构相似或者功能类似的蛋白。质粒干扰耗尽筛选法和细菌选择性定向进化是将以PAM识别为前提的切割与细菌的存亡相偶联,但是这两种方法需要在筛选前制作大量的Cas蛋白突变库,工作量大。本研究团队尝试进行毒性基因参与的细菌选择性定向进化,通过细菌能够完成筛选,但是转移到哺乳动物却无法重复结果,原因未明。以晶体结构为基础的定点突变法是根据Cas蛋白晶体结构设计氨基酸突变来制备突变体。除了获得晶体结构外,如何选择氨基酸进行突变也极其重要。利用PACE法筛选突变体可以实现同时进行突变和筛选。研究者利用该方法筛选出具有广泛PAM识别的xCas9 3.7,但是其筛选过程只利用了Cas蛋白的结合能力,没有利用其切割活性,所以筛选出的Cas蛋白更适合作为一种dCas9。近期的研究表明,xCas9对PAM的识别并没有原作者报道的那么广泛,其对TGA,TGT和TGC靶标的切割活性仅仅少量位点高于野生型,远不及SpCas9-NG[38]。本研究团队最近的研究结果表明,通过将T-RNA加入到sgRNA中能够明显提高高保真SpCas9 (HF-Cas9和eCas9) 的活性,但是不能显著提高xCas9的活性[39]。
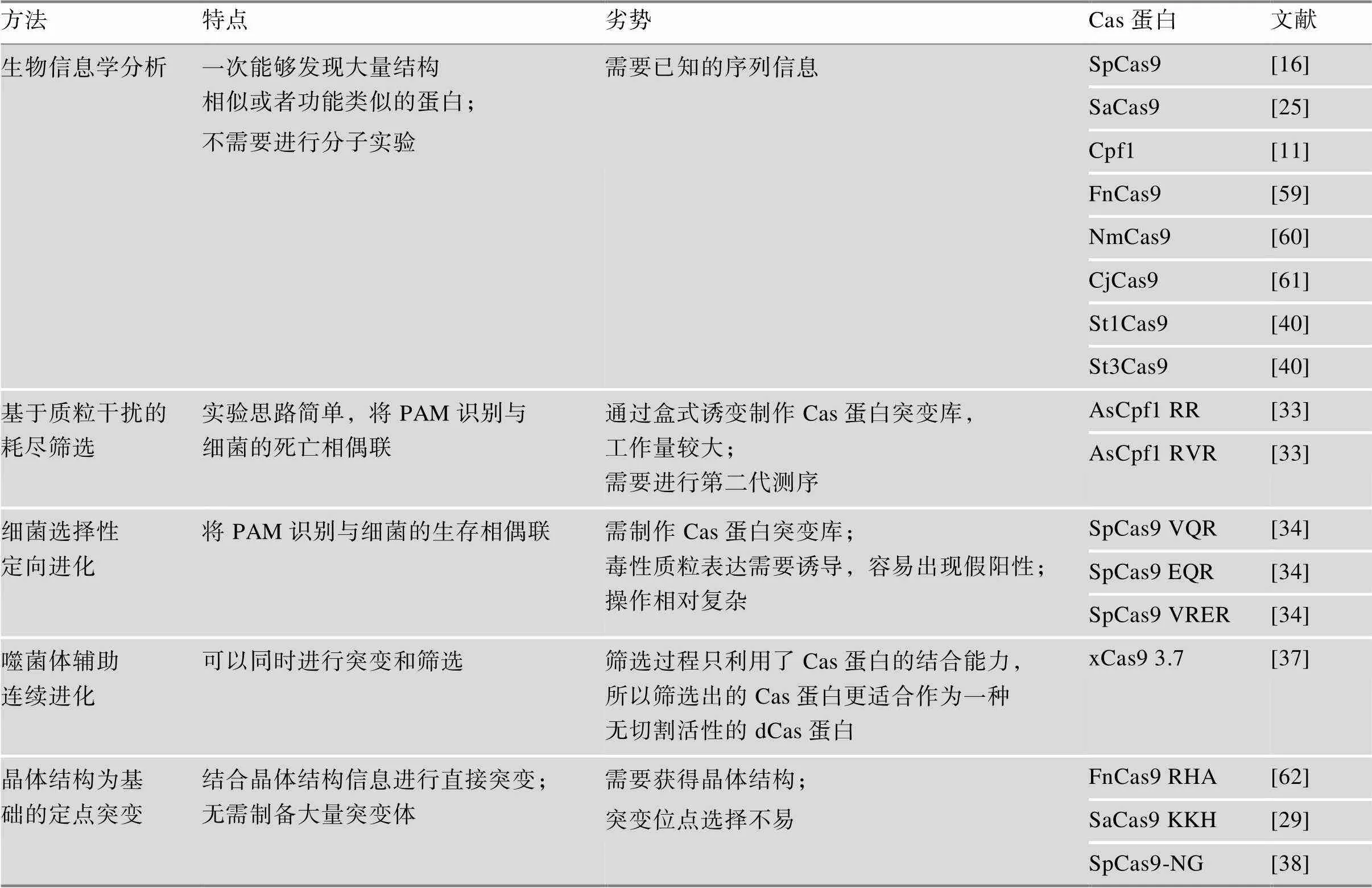
表1 不同Cas蛋白寻找方法与比较
2 PAM的确定
2007年,Horvath等和Deveau等[40,41]分别观察到,嗜热链球菌抵抗烈性噬菌体感染需要原型间隔区旁边的保守序列。2009年,Mojica等[42]通过生物信息学分析发现了多个CRISPR-Cas系统具有的相似序列,并将PAM确定为这类系统的普遍特征。研究者们对CRISPR-Cas系统PAM的研究表明,该系统使用这些侧翼序列来判断CRISPR阵列来自本身还是外来入侵者[20,21,43]。
相关的结构和生化分析揭示了PAM如何参与目标识别[44~46]。Cas蛋白以RNA为指导形成RNA-蛋白质-DNA相互作用并随后解链下游DNA序列。Cas蛋白“审查”靶序列的一条链和crRNA间隔区部分碱基的匹配程度。两条链之间的匹配度决定了是否启动切割。PAM在目标识别中起重要作用,其特征在不同类型的CRISPR-Cas系统之间存在差异。这些区别包括PAM的位置和具体序列组成。使用原型间隔区的非靶链作为参考,目标链通常用于指定PAM[44,47~49]。由于PAM位置的差异,有些PAM也被称为原型间隔区侧翼位点(protospacer flanking site, PFS)。
鉴定PAM是基因编辑工具研发过程中必不可少的环节。从PAM被发现开始[41],目前已经发现有多种方法可以用来解析PAM。
2.1 利用生物信息学方法确定PAM
生物信息学方法是根据已有的序列在数据库中寻找相似序列,首先在Cas蛋白来源的原核生物中找到CRISPR-Cas系统靶向的间隔序列,然后在数据库中比对找到spacer侧翼序列。CRISPRTarget即是为此开发的一款程序软件[50]。利用该程序可以将spacer序列以多种格式文件进行输入,将输入的序列在数据库中进行BLASTn搜索之后,可以获得候选的原型间隔区目标。在BLASTn搜索之后,该程序可以显示靶序列并对侧翼序列进行评分,然后通过精确匹配的种子(seed)区域筛选结果。此PAM确定方法的信息来自于基因组中本身存在的spacer,结果可靠。但是利用生物信息学方法确定PAM仍存在一定的缺陷:首先,该方法需要已知的spacer才能进行比对;其次,无法确保spacer来源的正确性以及有可能出现PAM偏好;另外,搜索的PAM可能只代表一个子集,无法代表所有PAM的集合。
2.2 将质粒库的切割与细菌的性状关联以确定PAM
利用CRISPR-Cas系统切割细菌中的质粒库,根据功能性PAM质粒比例的变化可确定相应的PAM。此方法利用了CRISPR-Cas系统的特异性切割活性,PAM覆盖度高,是目前被广泛使用的方法之一。Xue等[51]构建了含有3个随机碱基PAM (靶基因为)的质粒库。将库转化到含有CRISPR- Cas系统的中后,质粒被Cas蛋白切割后的细菌绿色荧光丢失。最后,通过流式细胞分选技术分离了功能性PAM(图6)。另外,也有研究者利用细菌质粒含有的抗性作为筛选标记进行功能性PAM的筛选[34]。该质粒清除的方法有其缺点:首先,反向筛选需要制备并使用深度覆盖的质粒库,而细菌内的筛选限制了库大小;其次,需要构建Cas蛋白稳定表达的宿主;另外,这种方法读出的PAM逃逸频率有可能来自靶序列突变或受DNA修复影响。
Abudayyeh等[3]采用了相反的思路,设计了覆盖MS2噬菌体基因组中所有可能的28 nt目标位点的3473个原型间隔区的crRNA库(图7)。将该构建体转化入大肠杆菌后,用MS2噬菌体感染细菌,确定存活细菌携带的间隔序列。分析富集间隔区对应的MS2基因组中原型间隔区的侧翼区域,可寻找到3ʹ端PFS。这种方法提供了一种正向的筛选方式,但其主要的局限在于:crRNA库小于PAM库,并不是所有的PAM都会在噬菌体基因组中展现出来;其次,构建向导RNA库的费用高于PAM库;另外,间隔序列库的设计和制作会消耗大量时间和人力。
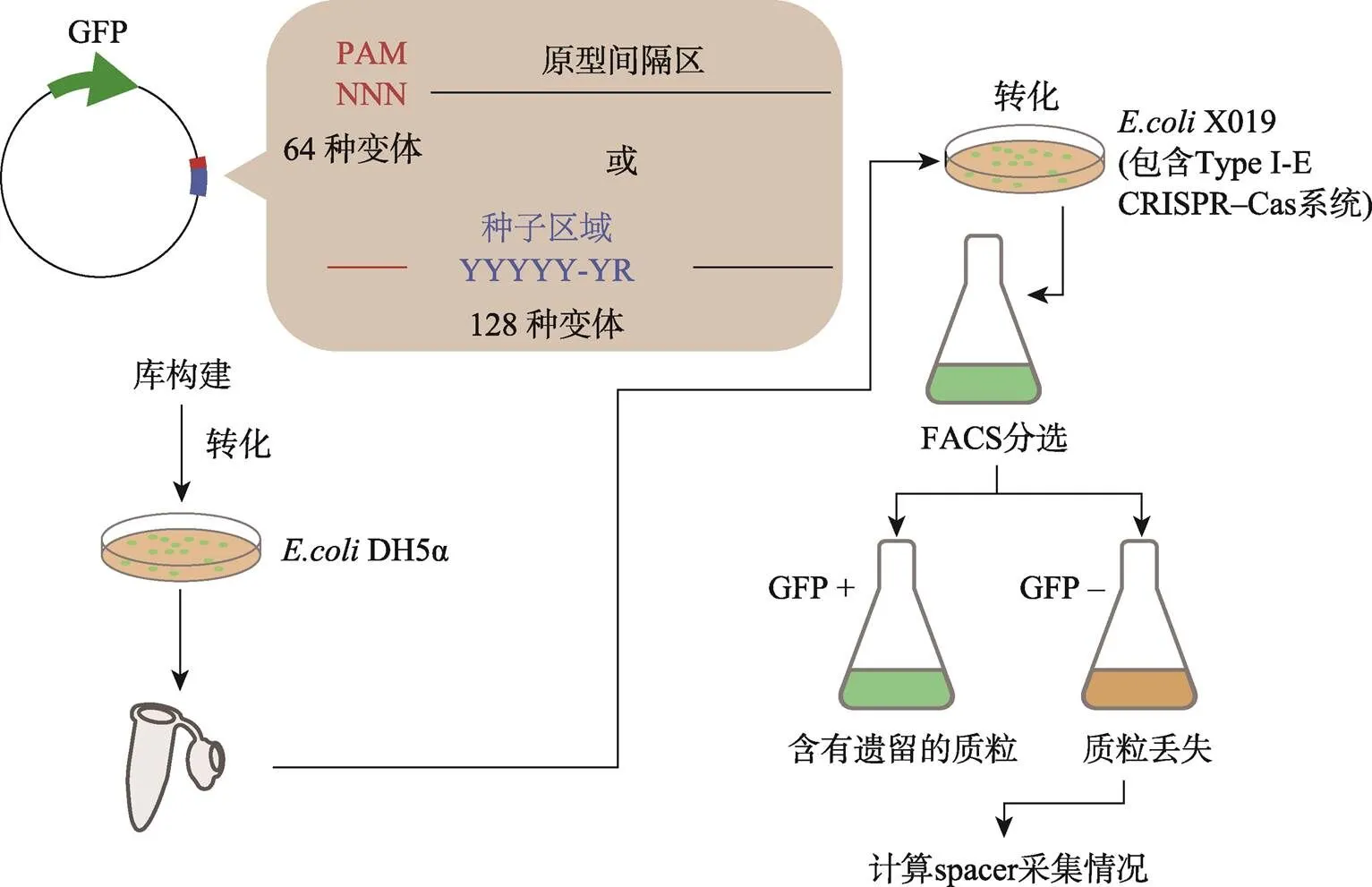
图6 用于PAM的高通量筛选的实验设计
制作不同PAM或种子区域的含有的库质粒,Cas蛋白切割质粒后绿色荧光消失,流式分选后可获得被富集的PAM[51]。
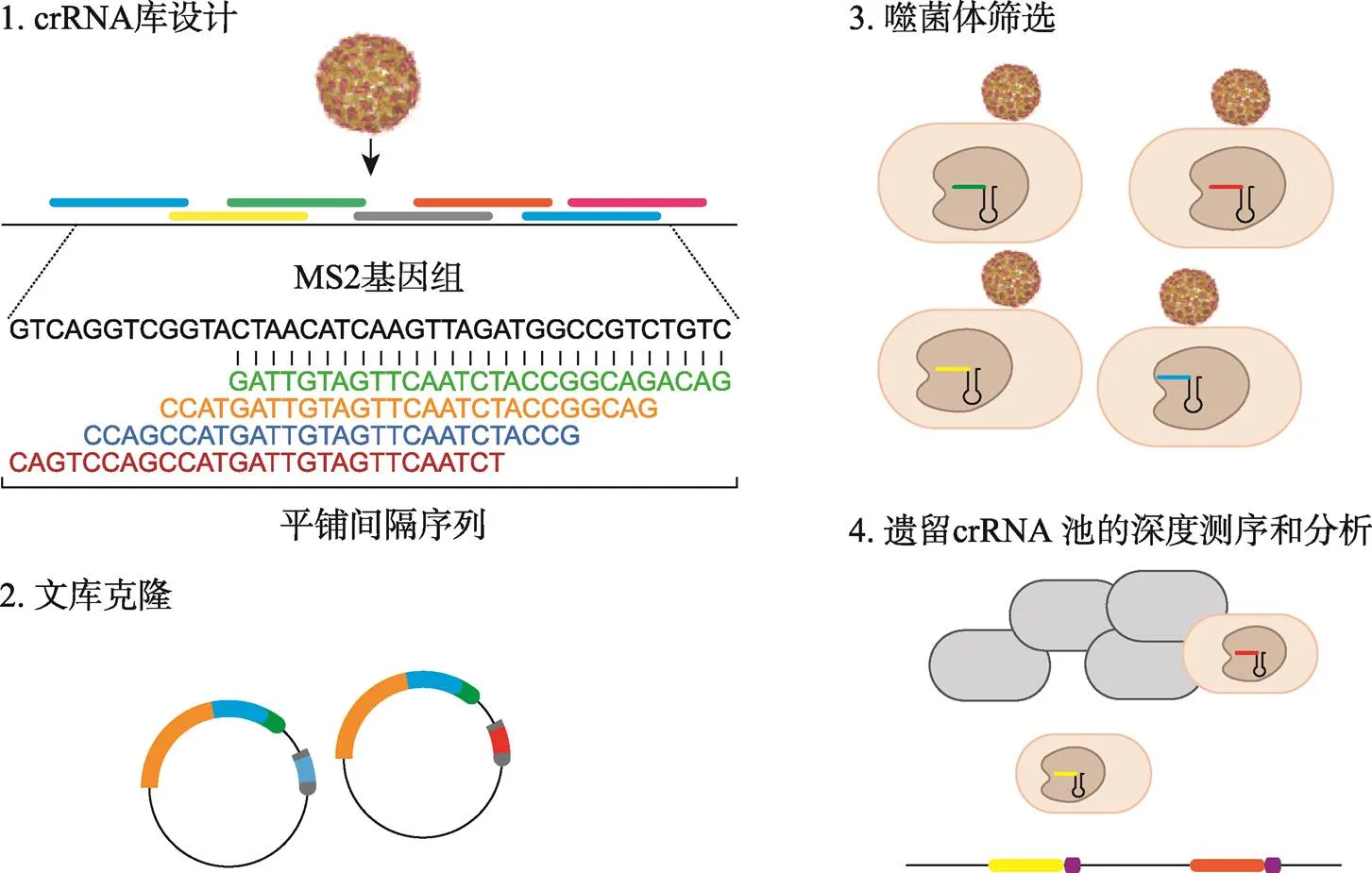
图7 通过噬菌体清除确定PAM (PFS)
研究者设计覆盖MS2病毒基因组的crRNA并转染到细菌中,利用细菌对病毒的抗性筛选出可以赋予细菌抵御病毒感染的质粒,通过测序可获得该spacer对应的PAM[3]。
2.3 利用体外切割产生的序列差异捕获PAM
体外条件下,在含有Cas蛋白、向导RNA和潜在PAM的靶向DNA库的体系中进行切割反应后,进行二代测序,分析PAM比例的变化可描述功能性PAM (典型性PAM)序列。利用这种方法,在体外切割结束后可以直接测序进行反向筛选[29],也可以在切割位点处连接接头进行正向筛选[52,53]。例如,Pattanayak等[52]将其制作的预选库与相应的Cas9及其sgRNA一起孵育,通过非磷酸化双链测序接头的5ʹ磷酸依赖性连接将含有游离5ʹ磷酸的产生切割的库成分与其他库成分分开。然后使用PCR扩增筛选出的库成分并采用高通量测序分析。体外切割寻找PAM的优点是被切割模板量大,可以覆盖更多的潜在PAM,其次是反应在体外,可以精确控制各项切割参数,而且正向筛选结果可以全面鉴定大部分功能性PAM。但此方法需要纯化蛋白,靶向模板容易降解,而且体外条件的变化可能导致PAM识别的差异。总的来说,虽然体外降解得出的PAM不能完全代表体内,却是一种鉴定CRISPR-Cas系统功能性PAM比较快速的方法。
2.4 通过Cas蛋白结合DNA产生的转录抑制筛选PAM
这种方法利用失活的Cas蛋白可以结合却无法切割DNA的特性来筛选未知PAM进行正向筛选。Leenay等[54]利用转录阻遏进行功能性PAM的筛选,开发了PAM-SCANR (PAM screen achieved by NOT- gate repression)。具体方法是在含有PAM库的靶序列下游连接和基因。Cas蛋白结合靶序列后会阻碍阻遏子,解除对GFP表达的阻遏。细菌中若存在PAM可被结合的质粒,细胞就会表达GFP。然后,流式分选GFP阳性细胞并进行二代测序分析,如图8所示。其缺点为采用无切割活性的Cas蛋白仅考虑其结合活性,结果可能与以切割为基础分析获得的PAM有差异;而且将Cas蛋白的结合活性与基因阻遏相关联可能会因为营养条件的变化引起阻遏效应的波动,从而影响PAM筛选的结果;此外,此方法需要利用到流式细胞仪,操作相对复杂。
2.5 通过捕获细胞内Cas蛋白产生的有效切割来鉴定PAM
本研究团队开发了一种在人类细胞中定义PAM的方法——PAM-DOSE (PAM definition by observable sequence excision)[55]。具体方法是利用两种不同的Cas蛋白在人类细胞内对含有PAM库的供体质粒进行双酶切,切割后质粒会通过NHEJ修复并敲除基因。在供体质粒两个切割位点上下游分别设计引物进行PCR扩增。由于基因敲除后,以供体DNA为模板的PCR产物更小,所以切割与未切割为模板的PCR产物在电泳时被分离,这样可以获得产生切割并带有PAM的序列。被双酶切的质粒一端对应靶序列被已知Cas蛋白(如SaCas9)识别并切割,另一端为带有待测Cas蛋白的靶序列和PAM组合。此方法的优点在于:能够直接在人类细胞中进行Cas蛋白对应PAM的确定,为CRISPR- Cas工具的临床应用提供借鉴;采用正向筛选方式,不用制作大容量的PAM库;此方法在筛选PAM的过程中利用绿色荧光的表达和红色荧光的减弱指示了PAM库质粒的切割,使切割过程可视化。迄今为止,本研究团队已经利用该方法重新鉴定了SpCas9、SpCas9-NG、FnCpf1、LbCpf1、AsCpf1和MbCpf1等Cas蛋白的PAM,其原理如图9所示。
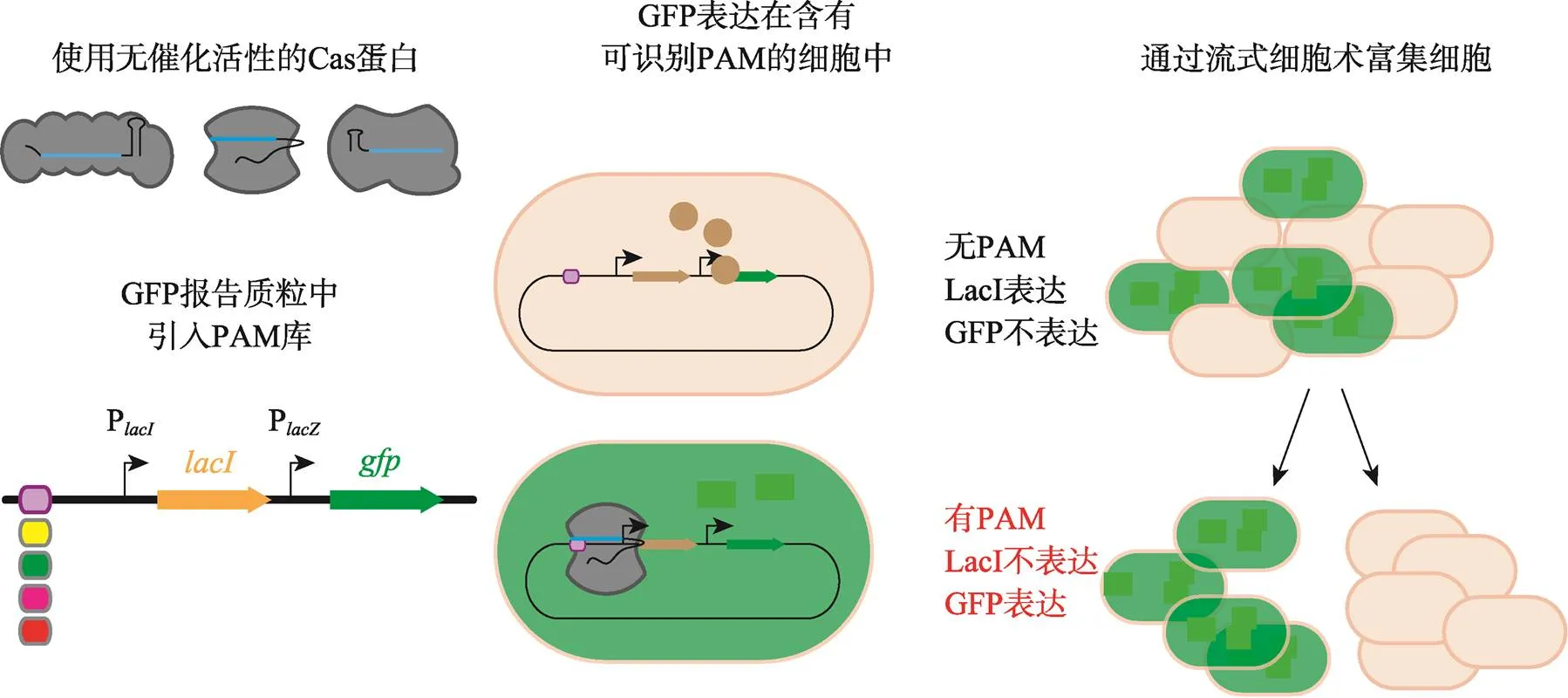
图8 PAM-SCANR法筛选PAM
失去切割活性的Cas蛋白识别的PAM结合靶序列后,导致下游GFP的表达[54]。
2.6 PAM确定方法的比较
Cas蛋白对PAM识别是CRISPR-Cas系统发挥切割功能作用必要的环节之一,表2总结了常见Cas蛋白的PAM鉴定方法。迄今为止,研究者们已经利用多种方法确定了PAM,这些丰富了人们对每种Cas蛋白对应PAM的认识[29,56~58]。不同方法定义PAM时所处的环境,如切割发生的环境是细胞内还是体外、真核细胞中还是原核细胞中,PAM容量等都可能导致PAM结果出现差异。这些不同方法的比较总结见表2。除了鉴定方法外,PAM的差异与靶序列选择也有一定的关系。总之,各种方法对于高活性PAM的定义基本相同,但是对非典型PAM可能有差异。由于PAM定义方法的差异,所以,根据需求选择一个合适的PAM确定方法尤为重要。
3 结语与展望
Cas蛋白的发现、优化和相应PAM的确定是CRISPR-Cas系统开发和升级基因编辑工具最重要的步骤。其大致遵循以下过程:根据已有蛋白质结构域的保守性,利用生物信息学比对以完成一种新的Cas蛋白的确定。采用体外切割方式确定该蛋白的切割活性和对应的PAM,然后在真核细胞中验证对应的PAM;最后根据Cas蛋白晶体结构来进行定向进化,找到改变其PAM识别特性的Cas蛋白或高活性或特异性更强的突变体。这一过程中,生物信息学分析是Cas蛋白研究的起始点,体外切割等方法是鉴定PAM最直接的方法,而晶体结构的解析为CRISPR-Cas系统机理的阐明、定向进化(识别不同PAM的Cas蛋白突变体)奠定了基础。“工欲善其事,必先利其器”,通过生物信息学、晶体结构解析和定向进化,研究者发现了各种各样的Cas蛋白和相关的突变体。不同Cas蛋白通常识别不同的PAM,在靶序列的识别、保真性、活性、切割末端上都有差别。这些不同种类的Cas蛋白及其突变体构成了强大的基因编辑工具箱,为物种改造(“造物”)、疾病诊断和治疗提供了强大的技术支持。目前新型高活性、特异性强的基因组编辑工具仍然是科学界的重大需求。多学科的交叉发展和技术进步将使基因编辑工具不断更新,人类的“造物”能力也将大大增强。在当前新型冠状病毒疫情下,研发临床有效治疗药物和预防用疫苗对于保证人民的健康、恢复国家经济发展意义不言而喻,基因编辑作为生命科学重要技术在寻找抗病毒药物靶点和解析病毒与宿主相关作用、以及快速研发疫苗都将会发挥举足轻重的作用。随着“造物”成果和新型医药产品为人类所用,相信我们终将达到认识自然,敬畏自然,改造自然的理想状态,实现人和自然的和谐、稳健的发展。
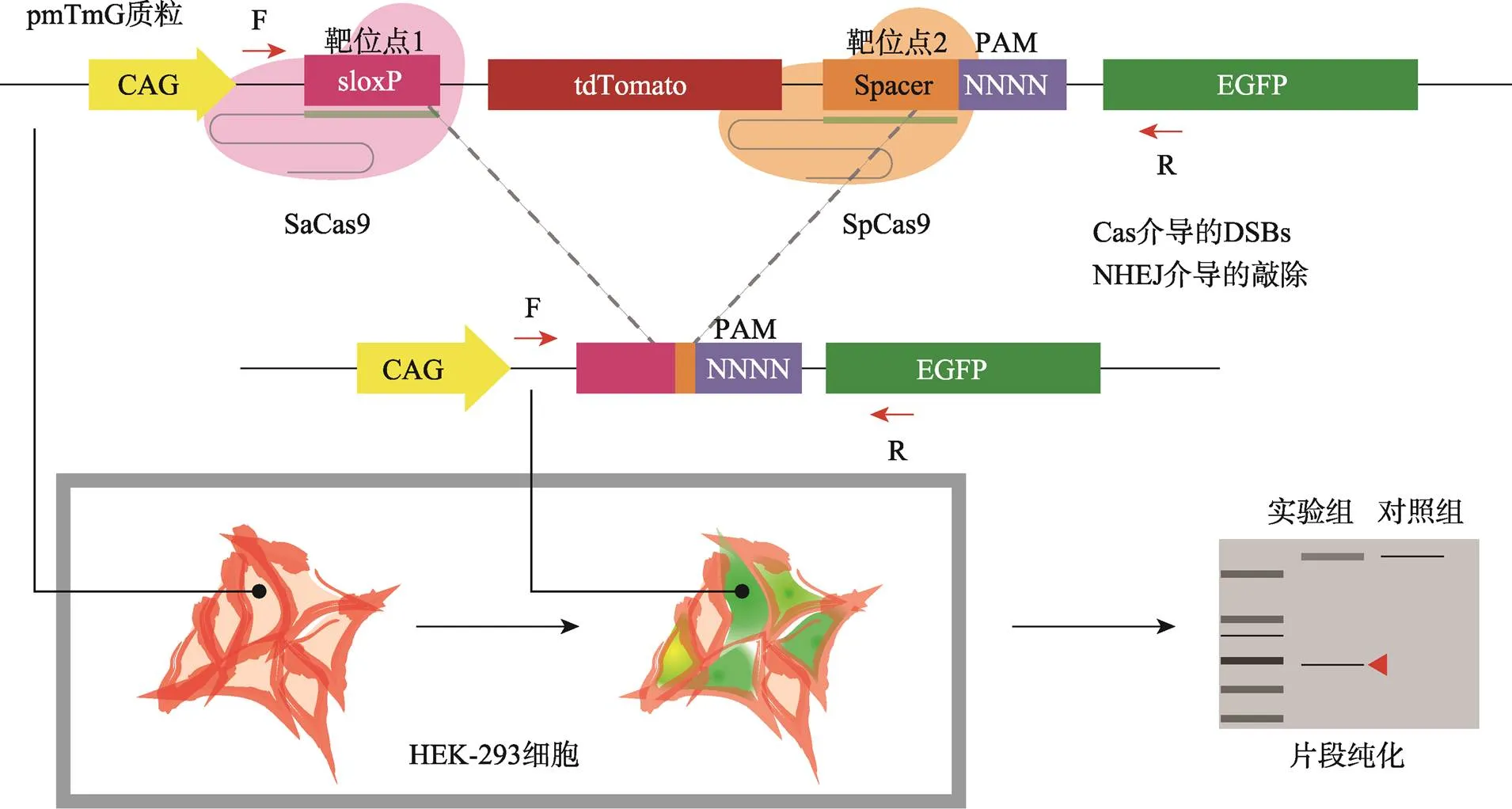
图9 PAM-DOSE方法在人类细胞中鉴定PAM
通过质粒中基因敲除和NHEJ修复将能够被Cas蛋白切割的PAM序列筛选出来[55]。
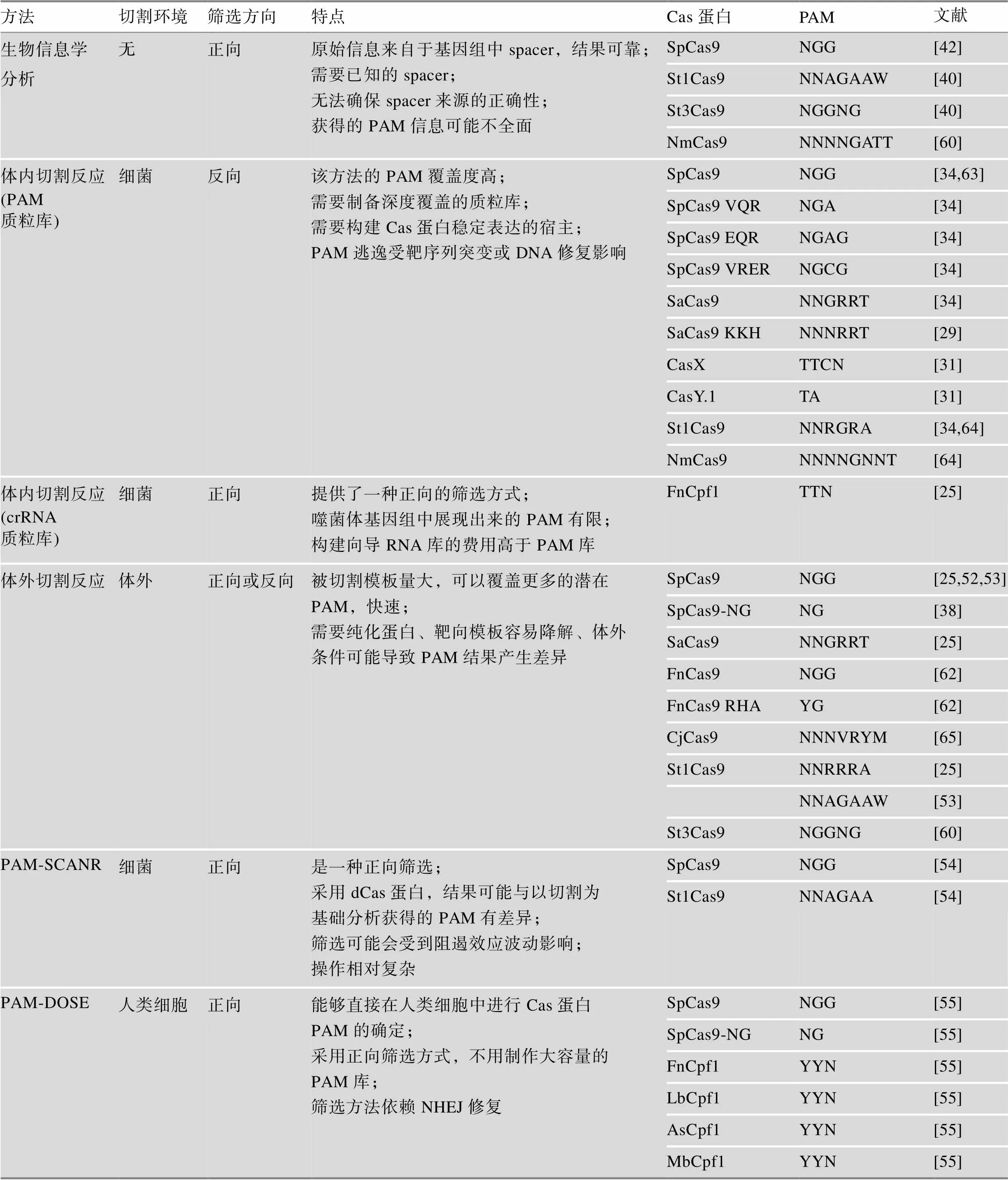
表2 不同Cas蛋白PAM确定方法与比较
[1] Bak RO, Gomez-Ospina N, Porteus MH. Gene editing on center stage., 2018, 34(8): 600–611.
[2] Zhang F, Wen Y, Guo X. CRISPR/Cas9 for genome editing: progress, implications and challenges., 2014, 23(R1): R40–R46.
[3] Abudayyeh OO, Gootenberg JS, Konermann S, Joung J, Slaymaker IM, Cox DB, Shmakov S, Makarova KS, Semenova E, Minakhin L, Severinov K, Regev A, Lander ES, Koonin EV, Zhang F. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector., 2016, 353(6299): aaf5573.
[4] Gootenberg JS, Abudayyeh OO, Lee JW, Essletzbichler P, Dy AJ, Joung J, Verdine V, Donghia N, Daringer NM, Freije CA, Myhrvold C, Bhattacharyya RP, Livny J, Regev A, Koonin EV, Hung DT, Sabeti PC, Collins JJ, Zhang F. Nucleic acid detection with CRISPR-Cas13a/C2c2., 2017, 356(6336): 438–442.
[5] Gootenberg JS, Abudayyeh OO, Kellner MJ, Joung J, Collins JJ, Zhang F. Multiplexed and portable nucleic acid detection platform with Cas13, Cas12a, and Csm6., 2018, 360(6387): 439–444.
[6] Konermann S, Brigham MD, Trevino AE, Joung J, Abudayyeh OO, Barcena C, Hsu PD, Habib N, Gootenberg JS, Nishimasu H, Nureki O, Zhang F. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex., 2015, 517(7536): 583–588.
[7] Komor AC, Kim YB, Packer MS, Zuris JA, Liu DR. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage., 2016, 533(7603): 420–424.
[8] Nishida K, Arazoe T, Yachie N, Banno S, Kakimoto M, Tabata M, Mochizuki M, Miyabe A, Araki M, Hara KY, Shimatani Z, Kondo A. Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems., 2016, 353(6305): aaf8729.
[9] Zong Y, Gao CX. Progress on base editing systems., 2019, 41(9): 777–800.宗媛, 高彩霞. 碱基编辑系统研究进展. 遗传, 2019, 41(9): 777–800.
[10] Anzalone AV, Randolph PB, Davis JR, Sousa AA, Koblan LW, Levy JM, Chen PJ, Wilson C, Newby GA, Raguram A, Liu DR. Search-and-replace genome editing without double-strand breaks or donor DNA., 2019, 576(7785): 149–157.
[11] Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes., 2007, 315(5819): 1709–1712.
[12] Doudna JA, Charpentier E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9., 2014, 346(6213): 1258096.
[13] Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering., 2014, 157(6): 1262–1278.
[14] Van Der Oost J, Westra ER, Jackson RN, Wiedenheft B. Unravelling the structural and mechanistic basis of CRISPR-Cas systems., 2014, 12(7): 479–492.
[15] Barrangou R, Doudna JA. Applications of CRISPR technologies in research and beyond., 2016, 34(9): 933–941.
[16] Jansen R, Embden JD, Gaastra W, Schouls LM. Identificationof genes that are associated with DNA repeats in prokaryotes., 2002, 43(6): 1565–1575.
[17] Mojica FJ, Diez-Villasenor C, Garcia-Martinez J, Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements., 2005, 60(2): 174–182.
[18] Shah SA, Erdmann S, Mojica FJ, Garrett RA. Protospacer recognition motifs: mixed identities and functional diversity., 2013, 10(5): 891–899.
[19] Westra ER, Semenova E, Datsenko KA, Jackson RN, Wiedenheft B, Severinov K, Brouns SJ. Type I-E CRISPR-cas systems discriminate target from non-target DNA through base pairing-independent PAM recognition., 2013, 9(9): e1003742.
[20] Marraffini LA, Sontheimer EJ. Self versus non-self discrimination during CRISPR RNA-directed immunity., 2010, 463(7280): 568–571.
[21] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity., 2012, 337(6096): 816–821.
[22] Mali P, Yang L, Esvelt KM, Aach J, Guell M, Dicarlo JE, Norville JE, Church GM. RNA-guided human genome engineering via Cas9., 2013, 339(6121): 823–826.
[23] Cong L, Ran FA, Cox D, Lin SL, Barretto R, Habib N, Hsu PD, Wu XB, Jiang WY, Marraffini LA, Zhang F. Multiplex genome engineering using CRISPR/Cas systems., 2013, 339(6121): 819–823.
[24] Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F. Genome engineering using the CRISPR-Cas9 system., 2013, 8(11): 2281–2308.
[25] Ran FA, Cong L, Yan WX, Scott DA, Gootenberg JS, Kriz AJ, Zetsche B, Shalem O, Wu X, Makarova KS, Koonin EV, Sharp PA, Zhang F. In vivo genome editing using Staphylococcus aureus Cas9., 2015, 520(7546): 186–191.
[26] Gu F. The new “dark horse” of genome editing tool: FnCpf1 has effective genome editing activity in human cells., 2017, 39(10): 947–949.谷峰. 基因组编辑工具新晋“黑马”:FnCpf1在人类细胞内具备有效基因组编辑活性. 遗传, 2017, 39(10): 947– 949.
[27] He XB, Gu F. Genome-editing: focus on the off-target effects., 2017, 33(10): 1757–1775.何秀斌, 谷峰. 基因组编辑脱靶研究进展. 生物工程学报, 2017, 33(10): 1757–1775.
[28] Schunder E, Rydzewski K, Grunow R, Heuner K. First indication for a functional CRISPR/Cas system in Francisella tularensis., 2013, 303(2): 51–60.
[29] Zetsche B, Gootenberg JS, Abudayyeh OO, Slaymaker IM, Makarova KS, Essletzbichler P, Volz SE, Joung J, Van Der Oost J, Regev A, Koonin EV, Zhang F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system., 2015, 163(3): 759–771.
[30] Shmakov S, Abudayyeh OO, Makarova KS, Wolf YI, Gootenberg JS, Semenova E, Minakhin L, Joung J, Konermann S, Severinov K, Zhang F, Koonin EV. Discovery and functional characterization of diverse class 2 CRISPR-Cas systems., 2015, 60(3): 385–397.
[31] Burstein D, Harrington LB, Strutt SC, Probst AJ, Anantharaman K, Thomas BC, Doudna JA, Banfield JF. New CRISPR-Cas systems from uncultivated microbes., 2016, 542(7640): 237–241.
[32] Liu JJ, Orlova N, Oakes BL, Ma E, Spinner HB, Baney KLM, Chuck J, Tan D, Knott GJ, Harrington LB, Al-Shayeb B, Wagner A, Brötzmann J, Staahl BT, Taylor KL, Desmarais J, Nogales E, Doudna JA. CasX enzymes comprise a distinct family of RNA-guided genome editors., 2019, 566(7743): 218–223.
[33] Gao L, Cox D, Yan WX, Manteiga JC, Schneider MW, Yamano T, Nishimasu H, Nureki O, Crosetto N, Zhang F. Engineered Cpf1 variants with altered PAM specificities., 2017, 35(8): 789–792.
[34] Kleinstiver BP, Prew MS, Tsai SQ, Topkar VV, Nguyen NT, Zheng Z, Gonzales AP, Li Z, Peterson RT, Yeh JR, Aryee MJ, Joung JK. Engineered CRISPR-Cas9 nucleases with altered PAM specificities., 2015, 523(7561): 481–485.
[35] Chen ZL, Zhao HM. A highly sensitive selection method for directed evolution of homing endonucleases., 2005, 33(18): e154.
[36] Esvelt KM, Carlson JC, Liu DR. A system for the continuous directed evolution of biomolecules., 2011, 472(7344): 499–503.
[37] Hu JH, Miller SM, Geurts MH, Tang WX, Chen LW, Sun N, Zeina CM, Gao X, Rees HA, Lin Z, Liu DR. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity., 2018, 556(7699): 57–63.
[38] Nishimasu H, Shi X, Ishiguro S, Gao L, Hirano S, Okazaki S, Noda T, Abudayyeh OO, Gootenberg JS, Mori H, Oura S, Holmes B, Tanaka M, Seki M, Hirano H, Aburatani H, Ishitani R, Ikawa M, Yachie N, Zhang F, Nureki O. Engineered CRISPR-Cas9 nuclease with expanded targeting space., 2018, 361(6408): 1259–1262.
[39] He X, Wang Y, Yang F, Wang B, Xie H, Gu L, Zhao T, Liu X, Zhang D, Ren Q, Liu X, Liu Y, Gao C, Gu F. Boosting activity of high-fidelity CRISPR/Cas9 variants using a tRNAGln-processing system in human cells., 2019, 294(23): 9308–9315.
[40] Horvath P, Romero DA, Coute-Monvoisin AC, Richards M, Deveau H, Moineau S, Boyaval P, Fremaux C, Barrangou R. Diversity, activity, and evolution of CRISPR loci in streptococcus thermophilus., 2008, 190(4): 1401–1412.
[41] Deveau H, Barrangou R, Garneau JE, Labonte J, Fremaux C, Boyaval P, Romero DA, Horvath P, Moineau S. Phage response to CRISPR-encoded resistance in., 2008, 190(4): 1390–1400.
[42] Mojica FJ, Diez-Villasenor C, García-Martinez J, Almendros C. Short motif sequences determine the targets of the prokaryotic CRISPR defence system., 2009, 155(Pt 3): 733–740.
[43] Wiedenheft B, Van DE, Bultema JB, Waghmare SP, Zhou K, Barendregt A, Westphal W, Heck AJ, Boekema EJ, Dickman MJ. RNA-guided complex from a bacterial immune system enhances target recognition through seed sequence interactions., 2011, 108(25): 10092–10097.
[44] Mulepati S, Héroux A, Bailey S. Structural biology. Crystal structure of a CRISPR RNA-guided surveillance complex bound to a ssDNA target., 2014, 345(6203): 1479–1484.
[45] Jinek M, Jiang F, Taylor DW, Sternberg SH, Kaya E, Ma E, Anders C, Hauer M, Zhou K, Lin S, Kaplan M, Iavarone AT, Charpentier E, Nogales E, Doudna JA. Structures of Cas9 endonucleases reveal RNA-mediated conformational activation., 2014, 343(6176): 1247997.
[46] Nishimasu H, Ran FA, Hsu PD, Konermann S, Shehata SI, Dohmae N, Ishitani R, Zhang F, Nureki O. Crystal structure of Cas9 in complex with guide RNA and target DNA., 2014, 156(5): 935–949.
[47] Westra ER, Van Erp PB, Kunne T, Wong SP, Staals RH, Seegers CL, Bollen S, Jore MM, Semenova E, Severinov K, De Vos WM, Dame RT, De Vries R, Brouns SJ, Van Der Oost J. CRISPR immunity relies on the consecutive binding and degradation of negatively supercoiled invader DNA by Cascade and Cas3., 2012, 46(5): 595– 605.
[48] Semenova E, Jore MM, Datsenko KA, Semenova A, Westra ER, Wanner B, Van Der Oost J, Brouns SJ, Severinov K. Interference by clustered regularly interspaced short palindromic repeat (CRISPR) RNA is governed by a seed sequence., 2011, 108(25): 10098–10103.
[49] Elmore J, Deighan T, Westpheling J, Terns RM, Terns MP. DNA targeting by the type I-G and type I-A CRISPR-Cas systems of Pyrococcus furiosus., 2015, 43(21): 10353–10363.
[50] Biswas A, Gagnon JN, Brouns SJ, Fineran PC, Brown CM. CRISPRTarget: bioinformatic prediction and analysis of crRNA targets., 2013, 10(5): 817–827.
[51] Xue C, Seetharam AS, Musharova O, Severinov K, Brouns S JJ, Severin AJ, Sashital DG. CRISPR interference and priming varies with individual spacer sequences., 2015, 43(22): 10831–10847.
[52] Pattanayak V, Lin S, Guilinger JP, Ma E, Doudna JA, Liu DR. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity., 2013, 31(9): 839–843.
[53] Karvelis T, Gasiunas G, Young J, Bigelyte G, Silanskas A, Cigan M, Siksnys V. Rapid characterization of CRISPR- Cas9 protospacer adjacent motif sequence elements., 2015, 16: 253.
[54] Leenay RT, Maksimchuk KR, Slotkowski RA, Agrawal RN, Gomaa AA, Briner AE, Barrangou R, Beisel CL. Identifying and visualizing functional PAM diversity across CRISPR-Cas systems., 2016, 62(1): 137–147.
[55] Tang LC, Yang FY, He XX, Xie HH, Liu XY, Fu JH, Xi HT, Lu XS, Liu CB, Song ZM, Qu J, Zhao JZ, Gu F. Efficient cleavage resolves PAM preferences of CRISPR- Cas in human cells., 2019, 8(2): 44–50.
[56] Hsu PD, Scott DA, Weinstein JA, Ran FA, Konermann S, Agarwala V, Li Y, Fine EJ, Wu X, Shalem O, Cradick TJ, Marraffini LA, Bao G, Zhang F. DNA targeting specificity of RNA-guided Cas9 nucleases., 2013, 31(9): 827–832.
[57] Zhang YL, Ge XL, Yang FY, Zhang LP, Zheng JR, Tan XF, Jin ZB, Qu J, Gu F. Comparison of non-canonical PAMs for CRISPR/Cas9-mediated DNA cleavage in human cells., 2014, 4: 5405.
[58] Tu MJ, Lin L, Cheng YL, He XB, Sun HH, Xie HH, Fu JH, Liu CB, Li J, Chen D, Xi HT, Xue DY, Liu Q, Zhao JZ, Gao CX, Song ZM, Qu J, Gu F. A ‘new lease of life’: FnCpf1 possesses DNA cleavage activity for genome editing in human cells., 2017, 45(19): 11295–11304.
[59] Sampson TR, Saroj SD, Llewellyn AC, Tzeng YL, Weiss DS. A CRISPR/CAS system mediates bacterial innate immune evasion and virulence., 2013, 497(7448): 254–257.
[60] Zhang Y, Heidrich N, Ampattu BJ, Gunderson CW, Seifert HS, Schoen C, Vogel J, Sontheimer EJ. Processing- independent CRISPR RNAs limit natural transformation in neisseria meningitidis., 2013, 50(4): 488–503.
[61] Chylinski K, Le Rhun A, Charpentier E. The tracrRNA and Cas9 families of type II CRISPR-Cas immunity systems., 2013, 10(5): 726–737.
[62] Hirano H, Gootenberg JS, Horii T, Abudayyeh OO, Kimura M, Hsu PD, Nakane T, Ishitani R, Hatada I, Zhang F, Nishimasu H, Nureki O. Structure and engineering of francisella novicida Cas9., 2016, 164(5): 950–961.
[63] Jiang W, Bikard D, Cox D, Zhang F, Marraffini LA. RNA- guided editing of bacterial genomes using CRISPR-Cas systems., 2013, 31(3): 233–239.
[64] Esvelt KM, Mali P, Braff JL, Moosburner M, Yaung SJ, Church GM. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing., 2013, 10(11): 1116–11121.
[65] Yamada M, Watanabe Y, Gootenberg JS, Hirano H, Ran FA, Nakane T, Ishitani R, Zhang F, Nishimasu H, Nureki O. Crystal Structure of the minimal Cas9 from campylobacter jejuni reveals the molecular diversity in the CRISPR-Cas9 systems., 2017, 65(6): 1109–1121.e3.
Next-generation CRISPR-Cas for genome editing: focusing on the Cas protein and PAM
Lianchao Tang, Feng Gu
The emergence of the gene editing technology, especial CRISPR-Cas (clustered regularly intersected short palindromic repeats and CRISPR associated proteins), has greatly promoted the ability of human beings to transform natural species. It has been widely harnessed for the engineering in the medical, industrial, agricultural and other fields. The key component of the CRISPR-Cas system, the Cas protein, possesses its specific features, including self-activity, recognition site, cutting end and guide RNA. PAM (protein assistant motif) is a number of nucleotides adjacent to the target site, which is very important for the Cas protein to recognize the target sequence and is also the key characteristic of CRISPR-Cas. There are several reported methods for identification of PAM. In this review, we summarize the searching for the Cas protein, the identification of Cas mutants with desired traits and the mapping of the PAM (including the extending of PAM spectrum), in order to provide a reference for the development and optimization of next-generation gene editing tools.
gene editing; CRISPR-Cas system; Cas protein; PAM; directed evolution
2019-11-12;
2020-02-26
国家自然科学基金项目(编号81201181)资助[Supported by the National Natural Science Foundation of China (No. 81201181)]
唐连超,硕士研究生,专业方向:基因编辑工具优化及应用。E-mail: 1664089980@qq.com
谷峰,博士,研究员,研究方向:基因编辑基础与转化研究。E-mail: fgu@mail.eye.ac.cn
10.16288/j.yczz.19-297
2020/3/6 16:12:24
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20200306.1044.003.html
(责任编委: 张博)