家兔前体脂肪细胞分化不同时期基因表达谱分析
杜坤,毛初阳,任安勇,吴雪梅,李庆玲,陈婷婷,陈仕毅,赖松家
研究报告
家兔前体脂肪细胞分化不同时期基因表达谱分析
杜坤,毛初阳,任安勇,吴雪梅,李庆玲,陈婷婷,陈仕毅,赖松家
四川农业大学,畜禽遗传资源发掘与创新利用四川省重点实验室,成都 611130
脂肪的过度积累严重危害人类健康。前体脂肪细胞分化是脂肪发育的关键过程,研究前体脂肪细胞分化相关基因的表达有助于认识脂肪沉积的机理。尽管家兔是一种理想的研究脂肪发育的动物模型,但是针对其前体脂肪细胞分化不同时期基因表达谱的研究鲜见报道。本研究通过诱导家兔前体脂肪细胞分化,在分化第0 d、3 d和9 d收集脂肪细胞,利用转录组测序(RNA-seq),在分化第3 d样本与第0 d样本的比较中筛选出1352个差异表达基因(differentially expressed genes, DEGs),在分化第9 d样本与第3 d样本的比较中筛选出888个DEGs。GO (gene ontology)功能富集和KEGG (kyoto encyclopedia of genes and genomes)通路分析发现,0~3 d分化期上调的DEGs显著富集在信号通路和信号通路上,3~9 d分化期上调的DEGs显著富集到与细胞周期调控有关的GO条目和KEGG信号通路,0~3 d和3~9 d阶段特异上调的DEGs可能分别作用于细胞质和细胞核。通过DEGs的蛋白-蛋白互作(protein-protein interaction, PPI)网络分析发现,筛选出的核心节点(hub node)基因可能通过调控细胞周期而影响家兔前体脂肪细胞分化。
脂肪细胞;分化;家兔;前体脂肪细胞;RNA-seq
人体脂肪的过度积累与2型糖尿病和心脑血管疾病的形成密切相关[1,2],严重威胁人类健康。脂肪组织结构复杂,具有高度的活性和内分泌功能,其发育过程在细胞水平上主要体现为前体脂肪细胞分化为成熟脂肪细胞[3],该过程伴随着脂肪细胞体积的增大和脂质的逐渐累积[4],是脂肪发育的关键过程。前体脂肪细胞分化过程十分复杂,受多种激素、信号通路和细胞间通信等因素的综合调控[5~7]。尽管目前对前体脂肪细胞分化过程的具体调控机理尚不完全清楚,但是人们普遍认为(peroxisome proliferator activated receptor gamma)[8,9]、(CCAAT/Enhancer binding protein alpha)[10]和(fatty acid binding protein)[11]等转录因子在前体脂肪细胞分化启动过程中发挥重要作用。脂肪的生成需要大量的基因参与,研究前体脂肪细胞分化相关基因的表达对于认识脂肪沉积具有重要意义。近年来,研究证实了一些基因如、和等可以在特定分化阶段调控脂肪细胞分化[12~14],表明这些参与调控前体脂肪细胞分化基因的表达可能具有时间特异性。
家兔()是一种重要的经济动物,为人类提供肉品和皮毛消费。由于家兔在生长阶段相对于猪、牛、羊等家养动物具有相对较低的脂肪沉积,因此家兔也是一种理想的研究脂肪发育的动物模型[15~17]。然而,针对家兔前体脂肪细胞分化不同时期的基因表达谱的研究却鲜见报道。
为了研究家兔前体脂肪细胞分化不同时期的基因表达谱,本研究通过经典的鸡尾酒法(IBMX- DEX-insulin, MDI)诱导家兔前体脂肪细胞分化为成熟脂肪细胞[18],收集分化第0 d、3 d和9 d的脂肪细胞总RNA,通过高通量转录组测序(RNA-seq)对基因的表达量进行了检测,分别在0~3 d和3~9 d分化阶段筛选出差异表达基因(differentially expressed genes, DEGs),然后利用生物信息学分析预测在对应阶段中表达上调的基因在家兔前体脂肪细胞分化过程中可能的功能和参与的信号通路。通过进一步分析DEGs在0 d,3 d和9 d表达量的变化,分别筛选出在0~3 d分化阶段特异上调表达(0~3 d表达显著上调,而3~9 d表达显著下调)和3~9 d分化阶段特异上调表达(0~3 d表达显著下调,而3~9 d表达显著上调)的DEGs,并预测这些特异上调表达基因可能的功能和参与的信号通路。最后使用在线蛋白分析工具分别构建了家兔前体脂肪细胞在0~3 d和3~9 d分化阶段DEGs的蛋白-蛋白互作网络,预测这两个分化阶段的核心节点(hub node)基因。本研究为更好地了解家兔前体脂肪细胞的分化提供转录数据参考。
1 材料与方法
1.1 家兔前体脂肪细胞的原代培养和诱导分化
本研究所用的新生幼年新西兰白兔来自四川农业大学雅安校区兔场。在无菌条件下,从幼年兔(<3 d)的肾周部位取脂肪组织,剔除其结缔组织和血管后,将脂肪组织剪碎成碎末状,使用0.1%的胶原蛋白酶(美国ThermoFisher公司)消化。待脂肪组织被消化成乳糜状,使用孔径为70 μm的细胞筛过滤,留下液体,离心倒掉上清液后使用PBS清洗脂肪细胞3次,接种于生长培养基(90% DME/F12,10% FBS) (DME/F12购自美国ThermoFisher公司;FBS购自美国ZETA LIFE公司)中并置于含5%CO2、37℃ 的培养箱中培养,2~3 d后对细胞传代。待原代脂 肪细胞生长密度达到80%~90%时,弃掉生长培养基,加入诱导分化培养基(90% DME/F12,10% FBS,500 μmol/L的3-异丁基-1-甲基黄嘌呤,1.7 μmol/L的胰岛素,1 μmol/L的地塞米松) (3-异丁基-1-甲基黄嘌呤、胰岛素和地塞米松均购自北京索莱宝生物公司)诱导分化72 h后,更换维持分化培养基(90% DME/F12,10% FBS,1.7 μmol/L的胰岛素)继续分化培养72 h,最后使用生长培养基培养至细胞完全分化。
1.2 油红O染色、样本收集和甘油三脂测定
使用油红O (美国Sigma公司)染色鉴定脂肪细胞的脂质累积状况,之后使用异丙醇溶解染色后的细胞,在510 nm波长下测定溶解液的吸光度(值),值用于对油红O染色的量化。根据家兔脂肪细胞油红O染色的3种不同表型(几乎没有脂滴,出现花环状的脂滴,以及出现成簇的大脂滴),在诱导分化第0 d (D0组)、3 d (D3组)和9 d (D9组)收获脂肪细胞,每组设置3个生物学重复,共9个样本,提取总RNA用于转录组测序。使用甘油三脂(TG)测定试剂盒(购自南京建成生物工程研究所)并按照其使用说明书,测定家兔前体脂肪细胞分化第0 d、3 d和9 d的细胞TG含量,每组设置3个生物学重复。
1.3 总RNA提取、文库构建及测序
使用Trizol试剂盒(美国ThermoFisher公司)并按照其操作说明书提取样本总RNA。RNA质量检测合格后,在干冰(−80℃)保存下,由江西海普洛斯生物科技公司构建去除核糖体RNA的链特异性文 库。文库质量检测合格后,在Illumina Hiseq X平台上对文库进行上机测序,最终获得双端150 bp的原始读段。
1.4 基因的定量以及差异表达分析
使用Fastp软件[19]对原始读段进行质量过滤,然后使用Hisat2[20]将过滤后剩下的高质量读段比对到家兔参考基因组上。使用Stringtie软件[21]计算每个基因在每个样本中的表达量(fragment per kilobase of transcript per million mapped reads, FPKM)和比对到基因序列上的读段数目。基于每个基因序列比对上的读段数,使用DEseq2软件[22]检测D3. D0和D9. D3的DEGs,同时满足以下两个条件的基因被判定为DEGs:(1)在组与组之间的比较中,log2(fold-change)的绝对值大于2;(2)在组与组之间的比较中,P<0.01。基于FPKM值,使用R软件对样本和差异表达基因进行聚类。
1.5 qRT-PCR验证
本研究利用qRT-PCR对RNA-seq获得的DEGs进行表达量的验证。随机选取6个DEGs,根据Ensemble (http://asia.ensembl.org/index.html)提供的mRNA序列,使用在线的引物设计软件primer- BLAST (https://www.ncbi.nlm.nih.gov/tools/primer-blast/)进行荧光定量引物的设计(附表1)。使用反转录试剂盒(宝生物工程(大连)有限公司)将总RNA反转录为cDNA,以cDNA为模板在Bio-Rad CFX仪器上进行两步法qRT-PCR扩增。反应条件:95℃ 10 s;95℃ 5 s,59℃ 20 s,40个循环。扩增结束后在55~ 95℃区间以0.5℃为上升率绘制溶解曲线。以为内参,使用2–ΔΔCt法计算目的基因的相对表达量。
1.6 功能富集和通路分析
使用DAVID在线数据库[23]进行GO (gene ontology)功能富集和KEGG(kyoto encyclopedia of genes and genomes)通路分析。对于富集到的GO条目和KEGG通路,<0.05被视为具有统计学意义。
1.7 蛋白–蛋白互作分析
将得到的DEGs输入在线的STRING数据库[24],设置蛋白互作置信分数0.7作为阈值(置信分数>0.7),分别构建D3D0和D9D3的DEGs蛋白–蛋白互作(protein-protein interaction, PPI)网络。CytoHubba插件[25]用于筛选在PPI网络中的核心节点基因,使用Cytoscape软件[26]进行图形的可视化展示。
1.8 统计分析
统计分析由R程序实现,两尾检验,*<0.05表示差异显著,**<0.01表示差异极显著。
2 结果与分析
2.1 家兔前体脂肪细胞的诱导分化
在家兔前体脂肪细胞诱导分化第0 d、3 d和9 d进行油红O染色,脂质累积在这3个时期出现明显的表型差异:在分化的第0 d只观察到有少量的脂滴,在分化的第3 d出现花环状的脂滴,在分化的第9 d出现成簇的大脂滴(图1A)。油红O染色的量化结果(图1B)和甘油三脂测定结果(图1C)也显示,脂质累积在家兔前体脂肪细胞诱导分化后的第3 d和第9 d显著升高。
2.2 基因的定量分析及DEGs的筛选
通过诱导家兔前体脂肪细胞分化,在分化的第0 d、3 d和9 d收集脂肪细胞总RNA,每组设置3个生物学重复(样本编号:D0-1、D0-2、D0-3、D3-1、D3-2、D3-3、D9-1、D9-2和D9-3),进行RNA-seq。对RNA-seq原始读段质量过滤后,总共得到155.03 Gb (1 109 630 370条读段)高质量的测序数据,平均每个样本17.22 Gb。对于每个样本的基因总体表达量,log2(FPKM+1)值集中在0~3 (图2A)。在家兔前体脂肪细胞分化0~3 d阶段检测到665个基因表达显著上调,687个基因表达显著下调(图2B)。在分化3~9 d阶段检测到515个基因表达显著上调,373个基因表达显著下调(图2C)。
2.3 DEGs表达量的qRT-PCR验证
为了验证RNA-seq的分析结果,本研究通过qRT-PCR验证了随机选取的6个DEGs的表达量。结果显示,所有的6个DEGs(、、和)的qRT-PCR定量结果与RNA-seq定量结果(FPKM)有相似的表达模式(图3)。因此,本研究通过RNA-seq得到的基因表达量有较高的可靠性。
2.4 上调基因的功能富集和信号通路分析
在0~3 d表达上调的DEGs显著富集到603个GO条目,根据GO条目值分别在生物过程(biological processes, BP)、细胞组分(cellular components, CC)和分子功能(molecular functions, MF)中均筛选出富集显著性最高的10个GO条目(图4A)。在BP分类中显著性最高的3个GO条目为细胞的化学刺激反应、细胞对有机物质的反应和酶联受体蛋白信号通路;在CC分类中显著性最高的3个GO条目为细胞外间隙、细胞外区域和细胞外区部分;在MF分类中显著性最高的3个GO条目为激素结合、硫化合物结合和跨膜受体蛋白激酶激活。KEGG信号通路分析发现在0~3 d表达上调的DEGs显著富集到24个KEGG信号通路。在富集显著性最高的10个KEGG信号通路中(图4B),显著性最高的3个KEGG信号通路为细胞色素P450对外源物的代谢的影响、信号通路和-信号通路。
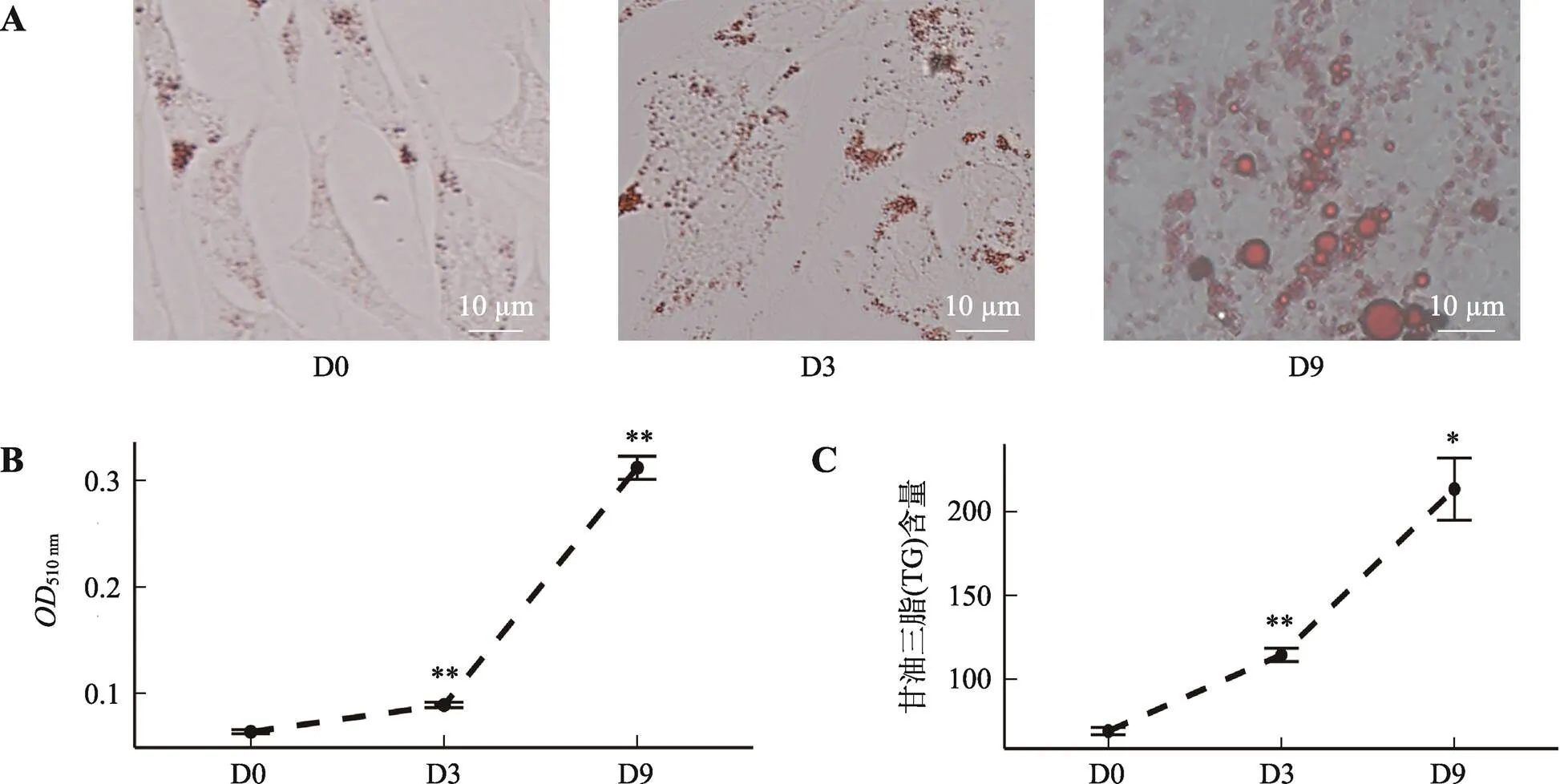
图1 家兔前体脂肪细胞的分化
A:诱导分化第0 d、3 d和9 d细胞油红O染色结果;B:油红O染色量化结果;C:甘油三脂测定结果。D0:分化第0 d样本;D3:分化第3 d样本;D9:分化第9 d样本。*表示与D0比较差异显著(<0.05),**表示与D0比较差异极显著(<0.01)。
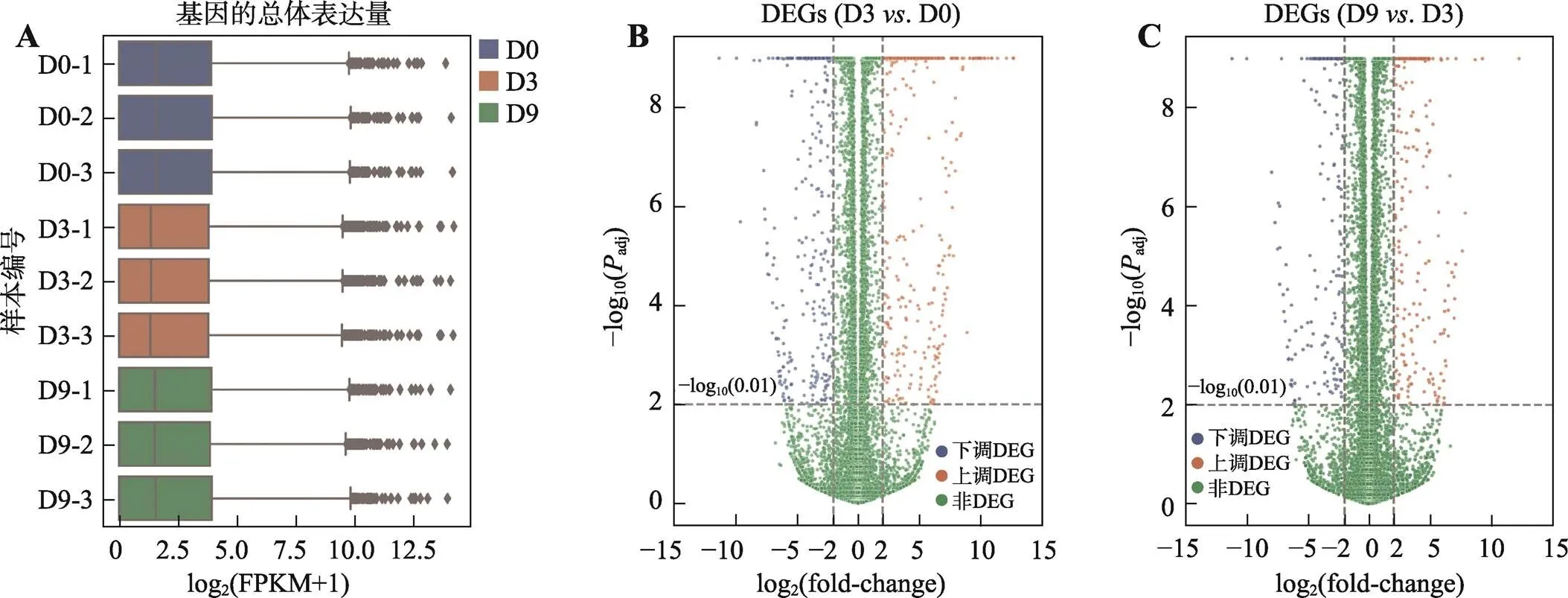
图2 基因的定量和差异表达分析
A:D0、D3、D9各样本基因的总体表达量;B:D3D0基因差异表达分析;C:D9D3基因差异表达分析。D0:分化第0 d样本;D3:分化第3 d样本;D9:分化第9 d样本。
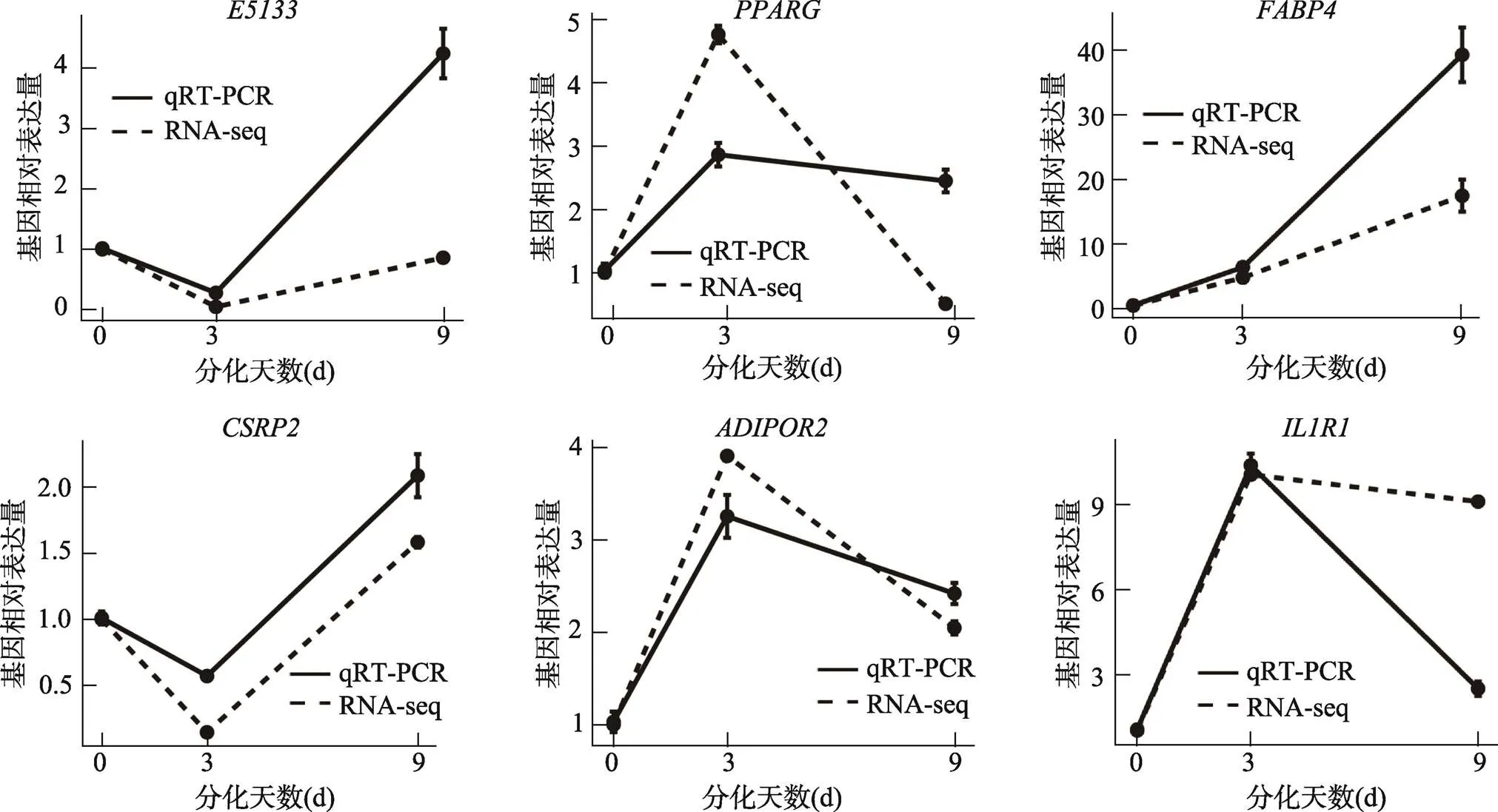
图3 qRT-PCR对随机选取的6个DEGs的表达量验证
:。
在3~9 d表达上调的DEGs显著富集到310个GO条目,根据GO条目值分别在BP、CC和MF中均筛选出富集显著性最高的10个GO条目(图4C)。在BP分类中显著性最高的3个GO条目为细胞周期过程、有丝分裂细胞周期过程和有丝分裂细胞周期;在CC分类中显著性最高的3个GO条目为染色体、染色体区域和染色体部分;在MF分类中显著性最高的3个GO条目为腺苷核糖核苷酸结合、腺嘌呤核苷酸结合和ATP结合。KEGG通路分析发现3~9 d表达显著上调的DEGs显著富集到19个信号通路,在富集显著性最高的10个KEGG信号通路中(图4D),显著性最高的3个KEGG信号通路为细胞周期、DNA复制和卵母细胞减数分裂。
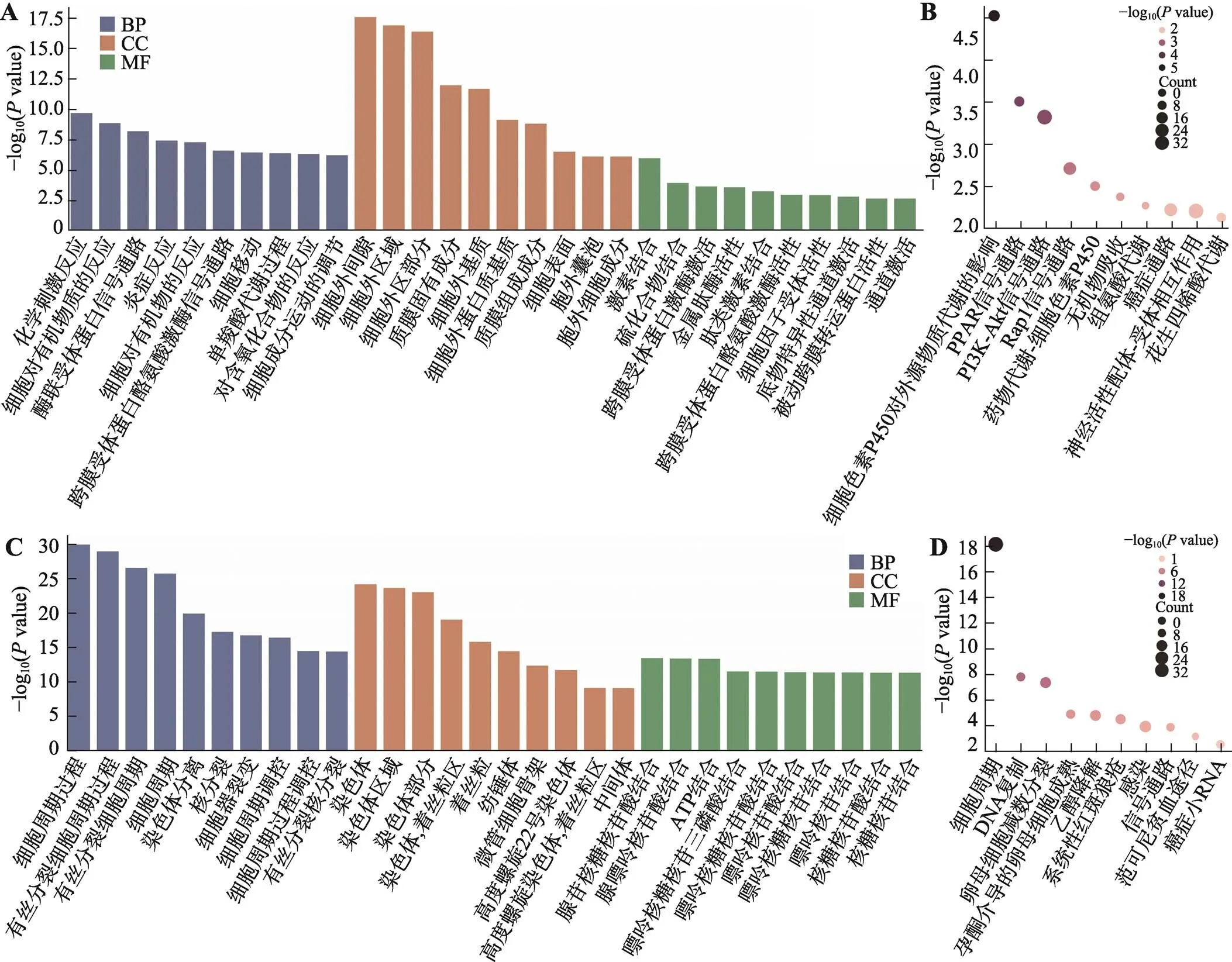
图4 表达上调DEGs的GO功能富集和KEGG通路分析
A:0~3 d上调的DEGs分别在BP、CC和MF中富集显著性最高的GO条目(每个分类各10个);B:0~3 d上调的DEGs富集显著性最高的10个KEGG通路;C:3~9 d上调的DEGs分别在BP、CC和MF中富集显著性最高的GO条目(每个分类各10个);D:3~9 d上调的DEGs富集显著性最高的10个KEGG通路。
2.5 DEGs的表达量分析
对家兔前体脂肪细胞分化过程中所有DEGs进行表达量聚类分析,发现分化第0 d的3个样本D0-1、D0-2和D0-3形成独立的簇,分化第3 d的3个样本D3-1、D3-2和D3-3形成独立的簇,分化第9 d的3个样本D9-1、D9-2和D9-3形成独立的簇(图5A),说明同组样品间的基因表达相关性很高。在1352个0~3 d分化阶段的DEGs中,有601个在3~9 d阶段也呈现差异表达(图5B),统计每个基因在各阶段的表达量,可以将这601个DEGs分为3类:第一类是表达量在0~3 d和3~9 d中都显著上调,或者0~3 d和3~9 d都显著下调的DEGs (8个);第二类是在分化0~3 d表达量显著上调而在分化3~9 d表达量显著下调的DEGs (212个);第三类是在分化0~3 d表达量显著下调而在3~9 d表达量显著上调的DEGs (381个)。
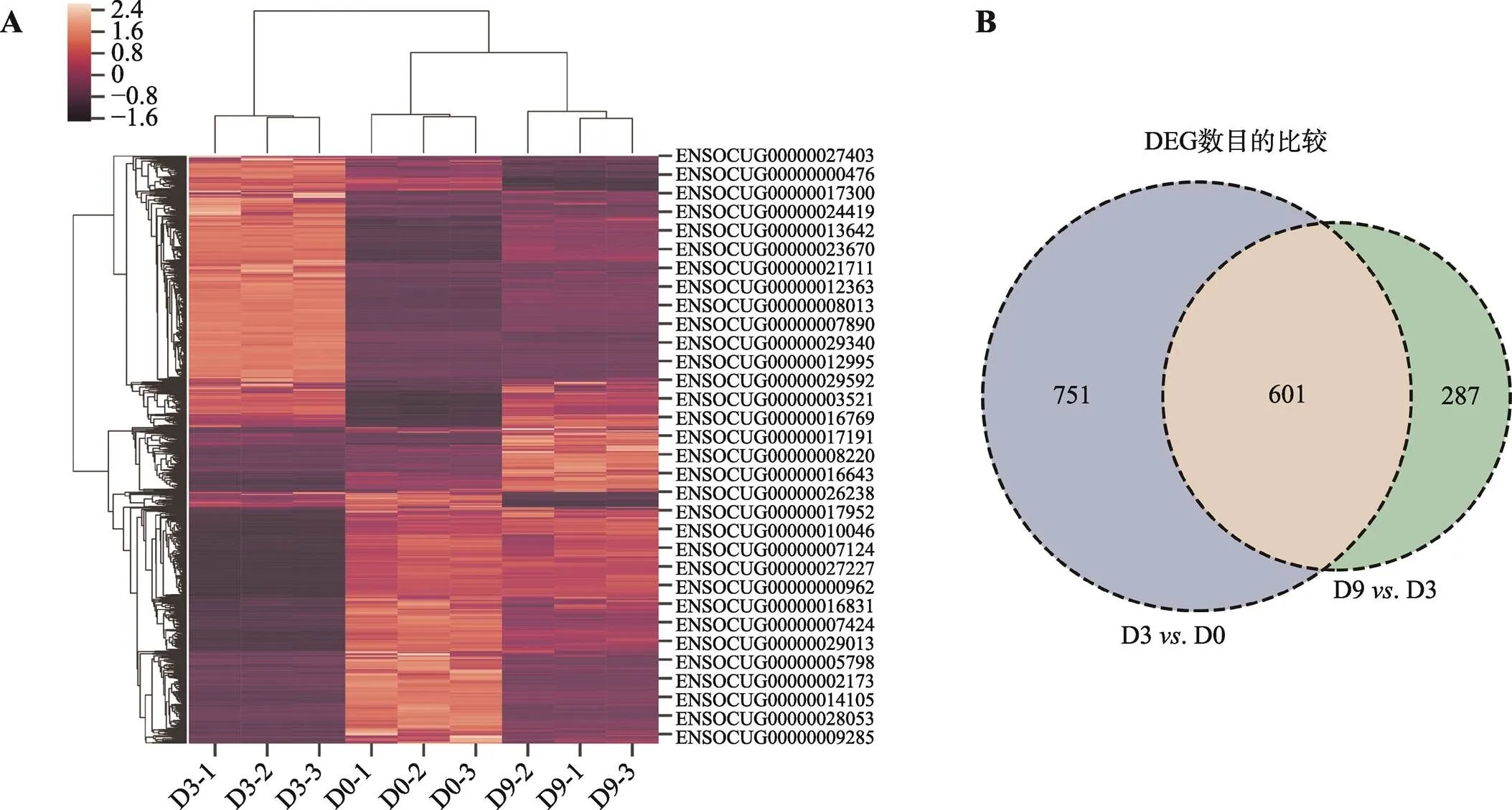
图5 聚类分析和不同阶段DEGs数量比较
A:基于FPKM值对样本和差异表达基因进行聚类分析(颜色由暗到亮表示表达量由低到高);B:D3. D0和D9. D3差异基因数目的比较。
2.6 特异上调表达基因的功能富集和信号通路分析
对0~3 d表达显著上调,而在3~9 d表达显著下调的212个DEGs进行GO富集分析,发现这些基因显著富集到177个GO条目,根据GO条目值分别在BP、CC和MF中均筛选出富集显著性最高的10个GO条目(图6A)。在BP分类中显著性最高的3个GO条目为脂肪生物合成过程、小分子生物合成过程和单羧酸代谢过程;在CC分类中显著性最高的3个GO条目为细胞外基质、细胞外区域部分和细胞外区域;在MF分类中显著性最高的3个GO条目为金属肽酶活性、过渡金属离子结合和GTP结合。KEGG通路分析发现这些基因显著富集到12个信号通路,在富集显著性最高的10个KEGG信号通路中(图6B),显著性最高的3个KEGG信号通路为络氨酸代谢、组氨酸代谢和代谢通路。
对0~3 d表达显著下调,而在3~9 d表达显著上调的381个DEGs进行GO富集分析,发现这些基因显著富集到304个GO条目,根据GO条目值分别在BP、CC和MF中均筛选出富集显著性最高的10个GO条目(图6C)。在BP分类中显著性最高的3个GO条目为细胞周期过程、有丝分裂细胞周期过程和有丝分裂细胞周期;在CC分类中显著性最高的3个GO条目为染色体、染色体区域和染色体部分;在MF分类中显著性最高的3个GO条目为ATP结合、腺苷核糖核苷酸结合和嘌呤核糖核苷三磷酸结合。KEGG信号通路分析发现这些基因显著富集到15个信号通路,在富集显著性最高的10个信号通路中(图6D),显著性最高的3个通路为细胞周期、DNA复制和卵母细胞减数分裂。
2.7 蛋白–蛋白互作分析
将1352个0~3 d分化阶段筛选出的DEGs提交到STRING数据库,得到家兔前体脂肪细胞在分化0~3 d的蛋白互作网络PPI03,PPI03包含1352个节点(node)和4427条边(edge)。使用CytoHubba筛选出在PPI03中排名(degree)前10的核心节点,这些核心节点基因包括、、、、、、、、和(图7A)。
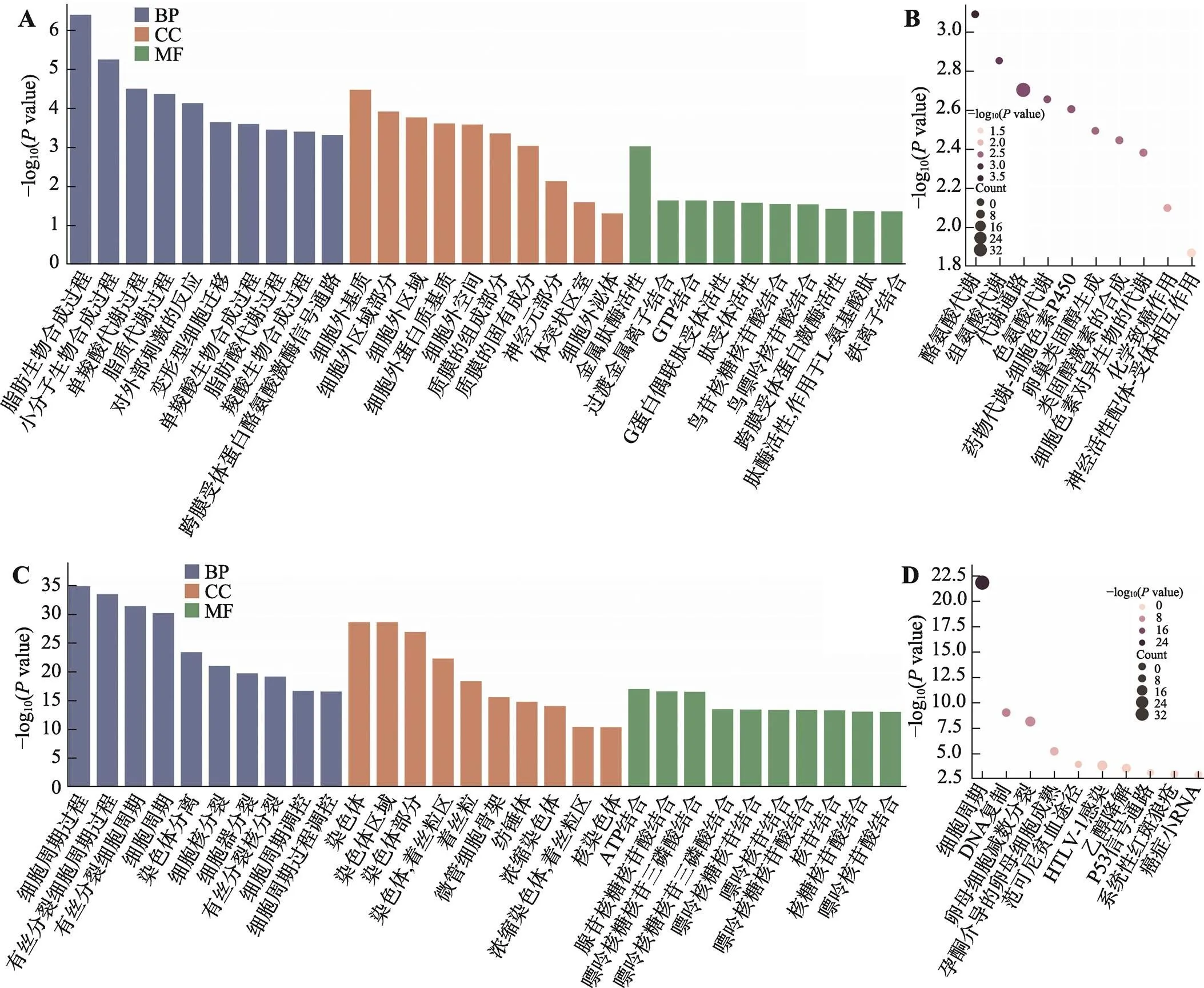
图6 特异上调表达基因的GO功能富集和KEGG通路分析
A:0~3 d特异上调的DEGs分别在BP、CC和MF中富集显著性最高的GO条目(每个类别各10个);B:0~3 d特异上调的DEGs富集显著性最高的10个KEGG通路;C:3~9 d特异上调的DEGs分别在BP、CC和MF中富集显著性最高的10个GO条目(每个类别各10个);D:3~9 d特异上调的DEGs富集显著性最高的10个KEGG通路。
同样,本研究得到3~9 d分化阶段DEGs的蛋白互作网络PPI39,PPI39包含888个节点和3475条边。在PPI39中排名前10的核心节点基因包括、、、、、、、、和(图7B)。
3 讨论
肥胖是一种慢性疾病,严重影响人类的身体健康和生活。研究前体脂肪细胞分化有助于理解肥胖成因。目前,RNA-seq已被广泛用于分析全基因组范围的基因时空表达[27]。本研究使用经典的MDI法诱导家兔前体脂肪细胞成脂分化[18],油红O染色结果和TG测定结果显示随着分化天数增加,脂质累积显著上升。在分化第9 d,能观察到许多成簇的大脂滴,表明得到了成熟脂肪细胞。通过生物信息学分析,在分化第0、3和9 d,获得了家兔前体脂肪细胞的基因表达谱,基于严格的过滤,分别在分化0~3 d和3~9 d筛选出1352个和888个DEGs。
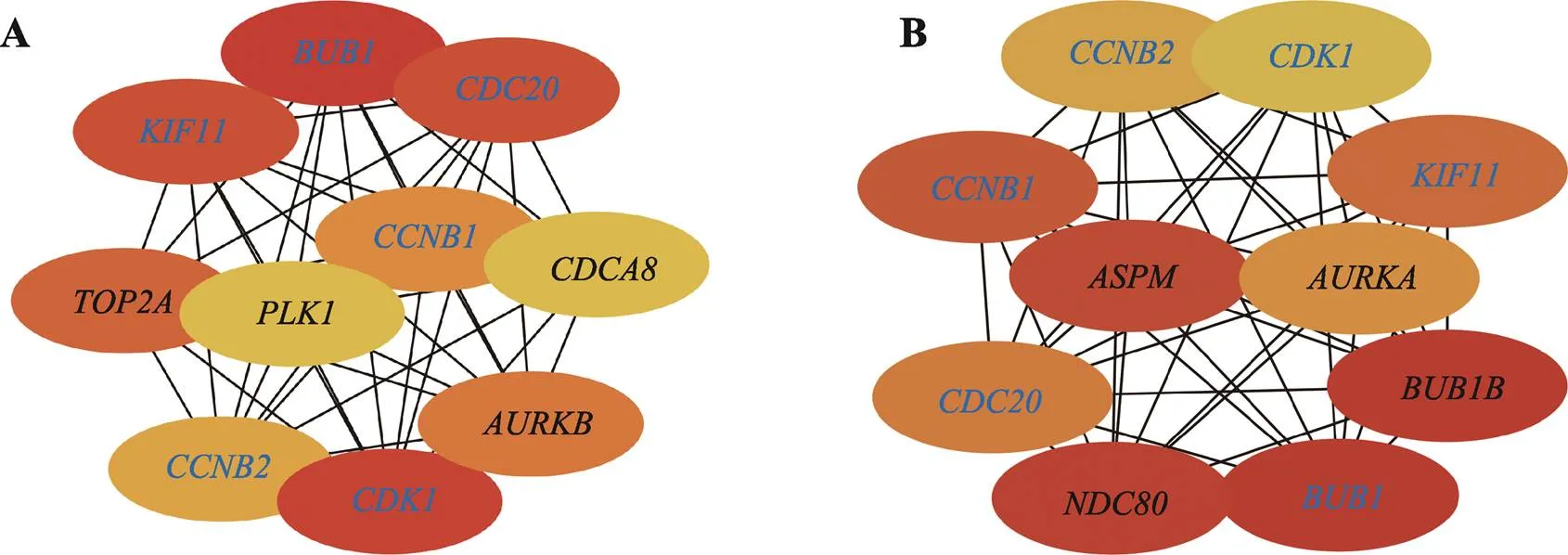
图7 PPI网络核心节点基因的筛选
A:PPI03筛选的核心节点基因;B:PPI39筛选出的核心节点基因。在两个PPI网络中都能筛选到的基因用蓝色字体表示。
本研究中,通过对家兔前体脂肪细胞分化0~3 d显著上调基因的GO富集,发现这些上调基因参与到细胞对外源刺激的响应、分化过程的正调节和与脂质合成相关的多个生物过程。在前体脂肪细胞增殖融合后,通过MDI法诱导前体脂肪细胞成脂分化,MDI主要成分为胰岛素、地塞米松和3-异丁基-1-甲基黄嘌呤,细胞在接收到这些有机物信号后,被诱导进入到分化阶段。在0~3 d富集到分化过程正调节生物过程的DEGs有和在内的34个基因,其中被报道可以通过多种途径调节细胞分化[28~30]。被称为生长分化因子5,被报道不仅可以促进棕色脂肪细胞,而且也是促进3T3-L1脂肪细胞分化的关键基因[31]。和在家兔前体脂肪细胞分化0~3 d表达显著上调,表明和也可能是调控家兔前体脂肪细胞分化的关键基因。脂肪发育伴随着脂质合成、脂肪酸合成和脂肪酸代谢等过程,本研究中,与这些生物过程相关的基因在0~3 d表达显著上调,说明家兔前体脂肪细胞在0~3 d被成功诱导进入到分化期。在分子功能上,0~3 d表达上调的DEGs显著富集到细胞信号传导有关的GO条目,如跨膜受体蛋白激酶激活、跨膜受体蛋白酪氨酸激酶活性、被动跨膜转运蛋白活性和G蛋白偶联肽受体活性。细胞信号转导是膜蛋白的重要功能,膜蛋白的激活可能和家兔前体脂肪细胞分化过程中富集到的一些活跃的信号通路有关。在KEGG信号通路方面,0~3 d表达上调的DEGs显著富集到信号通路和-信号通路,这两条信号通路在人和小鼠上被多次报道与脂肪发育相关[32~34],说明家兔前体脂肪细胞在0~3 d分化期与其他动物的脂肪发育有一定相似性。细胞周期的调控与脂肪沉积关系密切[29],3~9 d分化期上调的DEGs显著富集到的GO条目包括细胞周期过程、有丝分裂细胞周期过程和有丝分裂周期,以及KEGG细胞周期信号通路,富集到这些条目和信号通路的DEGs包括、和等。其中和被报道在高脂饲养的肉鸡的腹脂中表达上调,参与鸡的脂质累积[35]。推测家兔前体脂肪细胞在3~9 d分化期可能通过促进调控细胞周期蛋白的基因表达来维持脂肪细胞分化。
通过表达量的分析,本研究筛选出在0~3 d中特异上调表达的基因为212个,在3~9 d特异上调表达的基因为381个。对0~3 d特异上调表达基因进行GO功能富集,发现这些基因显著富集到脂质合成、脂质代谢过程和脂肪酸代谢过程等GO条目,注释到这些条目的基因如在人前体脂肪细胞中可以促进脂肪的生成[32,36],而是否可以促进家兔前体脂肪细胞的分化需要相关的实验进行验证。在GO富集结果的细胞组分分类中,0~3 d分化期特异上调的基因富集到如细胞外基质、细胞外区域和细胞外的外泌体等GO条目,而在3~9 d分化期特异上调的基因富集到染色体、染色体区域、着丝粒区域和染色质等核区相关的GO条目。因此,推测一些0~3 d分化期特异上调的基因在细胞中作用的位置可能是细胞质,而3~9 d分化期特异上调的基因在细胞中作用的位置可能是细胞核。在3~9 d分化期特异上调的基因富集到信号通路,有研究报道基因的敲除可以促进脂肪生成和脂肪标志基因的表达[37],但信号通路与家兔前体脂肪细胞的后期分化的关联机制需要进一步的实验验证。
通过PPI03网络的构建,筛选出家兔前体脂肪细胞分化0~3 d的10个核心节点基因,包括、、、、、、、、和。其中,被称为细胞周期蛋白依赖性激酶1。被称为DNA拓扑异构酶,在转录过程中控制和改变DNA拓扑状态。是一种丝氨酸/苏氨酸蛋白激酶。被称为Polo样激酶1,是一种广泛存在于真核生物中,具有高度保守性的丝氨酸/苏氨酸激酶。是细胞分裂的必须调节因子。被称为细胞分裂周期蛋白8,在有丝分裂中扮演重要角色。被称为极光激酶B,是一类重要的丝氨酸/苏氨酸激酶。和分别被称为细胞周期蛋白1和细胞周期蛋白2。这些基因都是调控细胞周期的关键基因,而细胞周期的调控与细胞的命运密切相关[38],且可以影响干细胞的多能性和分化潜能[39,40],如Lee等[41]报道基因表达被抑制可以使得MDI诱导的3T3-L1细胞脂肪形成受阻。被报道可以通过的抑制表达而介导癌症细胞的分化[42],本研究中,在0~3 d的表达显著下调,表达显著上调,推测可能被抑制,从而调控家兔前体脂肪细胞分化。在PPI39网络中的10个核心节点基因包括、、、、、、、、和。其中,被称为异常纺锤状小头畸形相关蛋白,其被证实对神经干细胞的增殖和分化至关重要[43],但对前体脂肪细胞的增殖和分化是否具有调控作用仍需要通过相关的研究才能得以被证实。从PPI网络分析来看,家兔前体脂肪细胞分化的0~3 d阶段和3~9 d阶段在PPI网络的核心节点基因上差异不大,都可以通过调控细胞周期来促进家兔前体脂肪细胞的分化进程。然而,目前关于这些核心节点基因的研究大多数建立在非脂肪细胞上,它们调控家兔前体脂肪细胞分化的机理需要更深入地进行探索和验证。
附录:
附表1详见文章电子版www.chinagene.cn。
[1] Saxena A, Tiwari P, Wahi N, Soni A, Bansiwal RC, Kumar A, Sharma B, Punjabi P, Gupta N, Malik B, Medicherla KM, Suravajhala P, Mathur SK. Transcriptome profiling reveals association of peripheral adipose tissue pathology with type-2 diabetes in Asian Indians., 2019, 8(1): 125–136.
[2] Fox CS, Massaro JM, Hoffmann U, Pou KM, Maurovich- Horvat P, Liu CY, Vasan RS, Murabito JM, Meigs JB, Cupples LA, D'Agostino RB Sr, O'Donnell CJ. Abdominal visceral and subcutaneous adipose tissue compartments: Association with metabolic risk factors in the framingham heart study., 2007, 116(1): 39–48.
[3] Kershaw EE, Flier JS. Adipose tissue as an endocrine organ., 2004, 89(6): 2548–2556.
[4] Zhang T, Zhang XQ, Han KP, Zhang GX, Wang JY, Xie KZ, Xue Q. Genome-Wide analysis of lncRNA and mRNA expression during differentiation of abdominal preadipocytes in the chicken., 2017, 7(3): 953–966.
[5] Zhang JW, Luo Y, Wang YH, He LJ, Li MZ, Wang X. MicroRNA regulates animal adipocyte differentiation., 2015, 37(12): 1175–1184.张进威, 罗毅, 王宇豪, 何刘军, 李明洲, 王讯. MicroRNA调控动物脂肪细胞分化研究进展. 遗传, 2015, 37(12): 1175–1184.
[6] Lee MJ. Hormonal regulation of adipogenesis., 2017, 7(4): 1151–1195.
[7] Sarjeant K, Stephens JM. Adipogenesis., 2012, 4(9): a008417.
[8] Tontonoz P, Hu E, Spiegelman BM. Stimulation of adipogenesis in fibroblasts by PPARγ2, a lipid-activated transcription factor., 1994, 79(7): 1147–1156.
[9] Wang L, Na W, Wang YX, Wang YB, Wang N, Wang QG, Li YM, Li H. Characterization of chicken PPARγ expression and its impact on adipocyte proliferation and differentiation., 2012, 34(4): 454–464.王丽, 那威, 王宇祥, 王彦博, 王宁, 王启贵, 李玉茂, 李辉. 鸡PPARγ基因的表达特性及其对脂肪细胞增殖分化的影响. 遗传, 2012, 34(4): 454–464.
[10] Lin FT, Lane MD. CCAAT/enhancer binding protein alpha is sufficient to initiate the 3T3-L1 adipocyte differentiation program., 1994, 91(19): 8757–8761.
[11] Deng TM, Wang YH, Wang CC, Yan H. FABP4 silencing ameliorates hypoxia reoxygenation injury through the attenuation of endoplasmic reticulum stress-mediated apoptosis by activating PI3K/Akt pathway., 2019, 224: 149–156.
[12] Liu Y, Peng WQ, Guo YY, Liu Y, Tang QQ, Guo L. Krüppel-like factor 10 (KLF10) is transactivated by the transcription factor C/EBPβ and involved in early 3T3-L1 preadipocyte differentiation., 2018, 293(36): 14012–14021.
[13] Tang Q, Chen C, Zhang Y, Dai MJ, Jiang YC, Wang H, Yu M, Jing W, Tian WD. Wnt5a regulates the cell proliferation and adipogenesis via MAPK-independent pathway in early stage of obesity., 2018, 42(1): 63–74.
[14] Yuan YY, Xi Y, Chen JY, Zhu P, Kang JY, Zou ZQ, Wang FY, Bu SZ. STAT3 stimulates adipogenic stem cell proliferation and cooperates with HMGA2 during the early stage of differentiation to promote adipogenesis., 2017, 482(4): 1360–1366.
[15] Desando G, Cavallo C, Sartoni F, Martini L, Parrilli A, Veronesi F, Fini M, Giardino R, Facchini A, Grigolo B. Intra-articular delivery of adipose derived stromal cells attenuates osteoarthritis progression in an experimental rabbit model., 2013, 15(1): R22.
[16] Gong LL, Wang C, Li YR, Sun QZ, Li GZ, Wang DR. Effects of human adipose-derived stem cells on the viability of rabbit random pattern flaps., 2014, 16(4): 496–507.
[17] Maneschi E, Vignozzi L, Morelli A, Mello T, Filippi S, Cellai I, Comeglio P, Sarchielli E, Calcagno A, Mazzanti B, Vettor R, Vannelli GB, Adorini L, Maggi M. FXR activation normalizes insulin sensitivity in visceral preadipocytes of a rabbit model of MetS., 2013, 218(2): 215–231.
[18] Zebisch K, Voigt V, Wabitsch M, Brandsch M. Protocol for effective differentiation of 3T3-L1 cells to adipocytes., 2012, 425(1): 88–90.
[19] Chen SF, Zhou YQ, Chen YR, Gu J. Fastp: an ultra-fast all-in-one FASTQ preprocessor., 2018, 34(17): i884–i890.
[20] Kim D, Langmead B, Salzberg SL. HISAT: a fast spliced aligner with low memory requirements., 2015, 12(4): 357–360.
[21] Pertea M, Kim D, Pertea GM, Leek JT, Salzberg SL. Transcript-level expression analysis of RNA-seq experiments with HISAT, stringtie and ballgown., 2016, 11(9): 1650–1667.
[22] Love MI, Huber W, Anders S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2., 2014, 15(12): 550.
[23] Huang Da W, Sherman BT, Lempicki RA. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources., 2009, 4(1): 44–57.
[24] Szklarczyk D, Morris JH, Cook H, Kuhn M, Wyder S, Simonovic M, Santos A, Doncheva NT, Roth A, Bork P, Jensen LJ, von Mering C. The STRING database in 2017: quality-controlled protein-protein association networks, made broadly accessible., 2017, 45(D1): D362–D368.
[25] Chin CH, Chen SH, Wu HH, Ho CW, Ko MT, Lin CY. CytoHubba: identifying hub objects and sub-networks from complex interactome., 2014, 8 (Suppl.4): S11.
[26] Shannon P, Markiel A, Ozier O, Baliga NS, Wang JT, Ramage D, Amin N, Schwikowski B, Ideker T. Cytoscape: a software environment for integrated models of biomolecular interaction networks., 2003, 13(11): 2498–2504.
[27] Huang WL, Zhang XX, Li A, Miao XY. Identification of differentially expressed genes between subcutaneous and intramuscular adipose tissue of large white pig using RNA-seq., 2017, 39(6): 501–511.黄万龙, 张秀秀, 李嫒, 苗向阳. 利用RNA-seq技术筛选大白猪皮下和肌内脂肪组织差异表达基因. 遗传, 2017, 39(6): 501–511.
[28] Chang HR, Kim HJ, Xu X, Ferrante AW Jr. Macrophage and adipocyte IGF1 maintain adipose tissue homeostasis during metabolic stresses., 2016, 24(1): 172–183.
[29] Tang QQ, Lane MD. Adipogenesis: from stem cell to adipocyte., 2012, 81: 715–736.
[30] Hu L, Yang GD, Hägg D, Sun GM, Ahn JM, Jiang N, Ricupero CL, Wu J, Rodhe CH, Ascherman JA, Chen LL, Mao JJ. IGF1 promotes adipogenesis by a lineage bias of endogenous adipose stem/progenitor cells., 2015, 33(8): 2483–2495.
[31] Pei Z, Yang Y, Kiess W, Sun CJ, Luo FH. Dynamic profile and adipogenic role of growth differentiation factor 5 (GDF5) in the differentiation of 3T3-L1 preadipocytes., 2014, 560: 27–35.
[32] Mota de Sá P, Richard AJ, Hang H, Stephens JM. Transcriptional regulation of adipogenesis., 2017, 7(2): 635–674.
[33] Świderska E, Podolska M, Strycharz J, Szwed M, Abramczyk H, Brożek-Pluska B, Wróblewski A, Szemraj J, Majsterek I, Drzewoski J, Śliwińska A. Hyperglycemia changes expression of key adipogenesis markers (C/EBPα and PPARᵞ) and morphology of differentiating Human visceral adipocytes., 2019, 11(8): E1835.
[34] Cai R, Tang GR, Zhang Q, Yong WL, Zhang WR, Xiao JY, Wei CS, He C, Yang GS, Pang WJ. A Novel lnc-RNA, Named lnc-ORA, Is identified by RNA-Seq analysis, and its knockdown inhibits adipogenesis by regulating the PI3K/AKT/mTOR signaling pathway., 2019, 8(5): E477.
[35] Chen JH, Ren XY, Li LM, Lu SY, Cheng T, Tan LT, Liang SD, He DL, Luo QB, Nie QH, Zhang XQ, Luo W. The cell cycle pathway regulates chicken abdominal fat deposition as revealed by transcriptome sequencing., 2019, 41(10): 962–973.陈家辉, 任学义, 李丽敏, 卢诗意, 程湉, 谭量天, 梁少东, 何丹林, 罗庆斌, 聂庆华, 张细权, 罗文. 转录组测序揭示细胞周期通路参与鸡腹脂沉积. 遗传, 2019, 41(10): 962–973.
[36] He J, Chen DL, Samocha-Bonet D, Gillinder KR, Barclay JL, Magor GW, Perkins AC, Greenfield JR, Yang G, Whitehead JP. Fibroblast growth factor-1 (FGF-1) promotes adipogenesis by downregulation of carboxypeptidase A4 (CPA4) - a negative regulator of adipogenesis implicated in the modulation of local and systemic insulin sensitivity., 2016, 34(5–6): 210–216.
[37] Huang Q, Liu ML, Du XL, Zhang RH, Xue Y, Zhang YY, Zhu WD, Li D, Zhao AL, Liu Y. Role of p53 in preadipocyte differentiation., 2014, 38(12): 1384–1393.
[38] Dalton S. Linking the cell cycle to cell fate decisions., 2015, 25(10): 592–600.
[39] Soufi A, Dalton S. Cycling through developmental decisions: how cell cycle dynamics control pluripotency, differentiation and reprogramming., 2016, 143(23): 4301–4311.
[40] Wu RF, Liu YH, Yao YX, Zhao YL, Bi Z, Jiang Q, Liu Q, Cai M, Wang FQ, Wang YZ, Wang XX. FTO regulates adipogenesis by controlling cell cycle progression via m6A-YTHDF2 dependent mechanism., 2018, 1863(10): 1323–1330.
[41] Lee Y, Yang H, Hur G, Yu J, Park S, Kim JH, Yoon Park JH, Shin HS, Kim JE, Lee KW. 5-(3¢,4¢-Dihydroxyphenyl)- γ-valerolactone, a metabolite of procyanidins in cacao, suppresses MDI-induced adipogenesis by regulating cell cycle progression through direct inhibition of CDK2/ cyclin O., 2019, 10(5): 2958–2969.
[42] Dasgupta N, Thakur BK, Ta A, Das S, Banik G, Das S. Polo-like kinase 1 expression is suppressed by CCAAT/ enhancer-binding protein α to mediate colon carcinoma cell differentiation and apoptosis., 2017, 1861(7): 1777–1787.
[43] Fujimori A, Itoh K, Goto S, Hirakawa H, Wang B, Kokubo T, Kito S, Tsukamoto S, Fushiki S. Disruption of Aspm causes microcephaly with abnormal neuronal differentiation., 2014, 36(8): 661–669.
Analysis of gene expression profiles at different stages during preadipocyte differentiation in rabbits
Kun Du, Chuyang Mao, Anyong Ren, Xuemei Wu, Qingling Li, Tingting Chen, Shiyi Chen, Songjia Lai
Excessive accumulation of fat is harmful to human health. The preadipocyte differentiation is a critical process of fat development.Studying the expression profiles of genes related to preadipocyte differentiation contributes to understanding of the mechanism of fat accumulation.Despite being considered an ideal animal model for studying adipogenesis, little is known about the gene expression profiles at different stages during preadipocyte differentiation in rabbits. In the present study, rabbit preadipocytes were culturedand induced for differentiation, and gene expression profiles of adipocytes collected at days 0, 3, and 9 of differentiation were analyzed by RNA-seq. We identified 1352 differentially expressed genes (DEGs) when comparing day 3 with day 0 and identified 888 DEGs when comparing day 9 with day 3. Gene Ontology (GO) enrichment and Kyoto Encyclopedia of Genes and Genomes (KEGG) pathway analysis showed that thesignaling pathway andsignaling pathway were significantly enriched by the DEGs that up-regulated within the period of day 0 – day 3, and the GO terms and KEGG pathways that were associated with cell cycle were enriched by the DEGs that up-regulated within the period of day 3 – day 9. The DEGs that specifically up-regulated within the period of day 0 – day 3 might play roles in the cytoplasm, and the DEGs that specifically up-regulated within the period of day 3 – day 9 might act in the nucleus. The protein-protein interaction (PPI) network constructed by DEGs showed that hub node genes might modulate rabbit preadipocyte differentiation via regulating cell cycle.
adipocytes; differentiation; rabbits; preadipocytes; RNA-seq
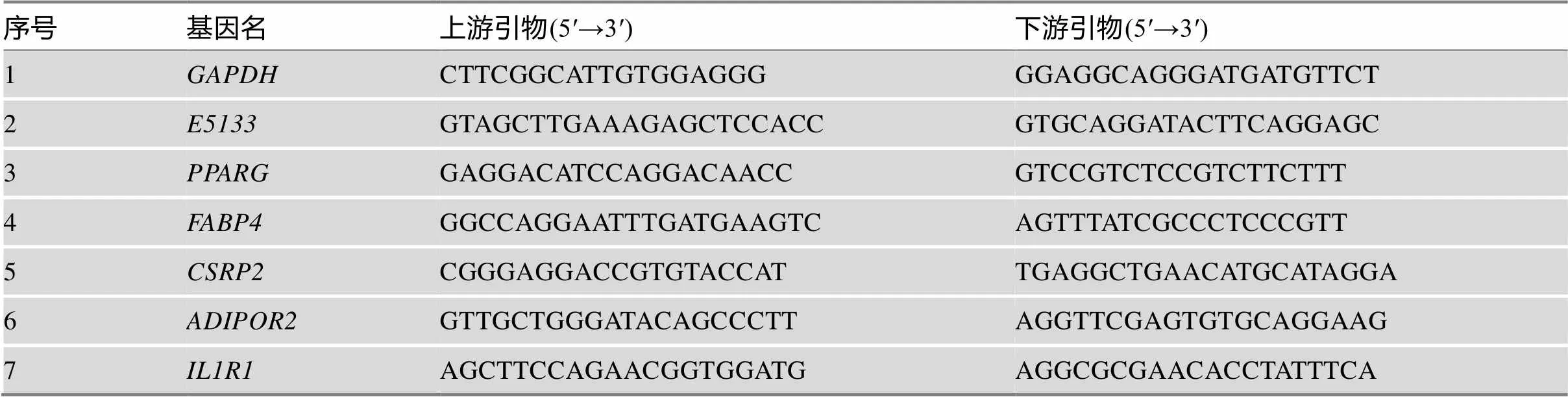
附表1 qRT-PCR引物序列
2019-09-04;
2019-12-11
国家现代农业产业技术体系建设项目(编号:CARS-44-A-2)资助[Supported by the Earmarked Fund for China Agriculture Research System (No. CARS-44-A-2)]
杜坤,硕士研究生,专业方向:动物遗传育种与繁殖。E-mail: 303160294@qq.com
赖松家,博士,教授,研究方向:分子遗传与动物育种。E-mail: laisj5794@163.com
10.16288/j.yczz.19-265
2020/1/14 10:19:28
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20200113.1723.002.html
(责任编委: 李明洲)