胃癌患者伊立替康耐药前后基因表达谱的差异*
赵素芳, 李冬兵, 孙大勇△
1深圳市第二人民医院消化科(广东深圳 518035); 2广州迈景基因医学科技有限公司(广东广州 510300)
胃癌严重威胁到人类的身体健康。胃癌是世上第三大常见的肿瘤,全球每年有约一百万人被诊断患有胃癌。2015年,中国新增癌症病例约429.2万例,癌症死亡人数约281.4万,其中胃癌,在癌症死亡原因中排在前列[1]。研究表明一线和二线化疗对于晚期或转移性胃癌患者在生存率方面有一定的益处。胃癌治疗的手段有很多,包括手术治疗和药物治疗等。大多数胃癌在确诊时,属于部分进展或长远转移的晚期患者。这类患者不宜采用手术治疗,最佳治疗手段是以全身化疗为主的药物综合治疗,可以延长患者的生存时间,同时提高患者生存质量[2]。盐酸伊立替康是一种DNA拓扑异构酶Ⅰ抑制剂,可阻断拓扑异构酶切割复合物的DNA重新连接[3]。伊立替康的体内水解产物为7-乙基-10羟基喜树碱,研究发现,与伊立替康自身相比,该水解产物的抗肿瘤活性更高[4]。与DNA拓扑异构酶结合进而阻断DNA链的组装,引起DNA链断裂,干扰DNA复制最终发挥抗肿瘤作用,这是伊立替康及其水解产物的抗肿瘤机制。伊立替康对增生的肿瘤细胞具有很强的杀伤活性在于肿瘤细胞中DNA拓扑异构酶含量升高,研究发现伊立替康具有广谱抗肿瘤活性,且对多耐药肿瘤依然有效[5]。研究发现单一伊立替康用药相比于伊立替康联合顺铂用药来治疗胃癌,临床疗效无显著性差异,但不良反应发生率明显降低,结果表明单一伊立替康用药方案不良反应更小,安全性较高,值得推广[6]。一些患者在使用伊立替康后产生耐药。耐药性的发展是胃癌治疗中的主要障碍。研究发现伊立替康金属硫蛋白MT1X的诱导上调,可能与胃癌患者的伊立替康耐药有关[7]。因此,对于肿瘤耐药性产生的分子基础的研究显得尤为重要,这为解决胃癌耐药后的进一步有效治疗提供了一定的依据。基于RNA-Seq这一高通量测序方法,能够有效地获得目标肿瘤组织在特定时期的几乎所有基因表达的信息。这些信息包括部分已知热点基因的表达水平的变化和剪接变异,部分稀有基因的表达水平的变化和剪接变异,部分未知基因的表达水平的变化和剪接变异。这些信息有利于我们全面分析不同样本之间基因表达的差异。我们可以采用RNA-Seq来分析鉴定哪些基因的表达水平的差异和剪接变异造成了患者的耐药,筛选这些化疗耐药相关的基因并深入分析相关基因的作用机制,对于肿瘤患者的个体化精准治疗中具有重要的意义。
1 资料与方法
1.1 一般资料 入组患者均被诊断为胃低分化腺癌,用药情况均为伊利替康单药,这些患者在2015年1月至2017年1月期间使用伊利替康后均产生耐药。患者姓名分别为SHD、HDL、TYT、WYM、MQ和LCP。SHD:男性,85岁,病理诊断为胃窦粘膜低分化腺癌;HDL:男性,55岁,病理诊断为(全胃)胃低分化腺癌;TYT: 男性,70岁,病理诊断为(胃)低分化腺癌;WYM:女性,50岁,病理诊断为低分化腺癌;MQ:男性,47岁,病理诊断为低分化腺癌;LCP:女性,65岁,病理诊断为中高分化腺癌。入组患者首先均被履行告知义务,接着签署知情同意书。按照国家和国际伦理学的有关要求,对样本进行收集,包括A组:确诊为胃癌患者,伊立替康用药前样本(SHD-before、HDL-before、TYT-before、WYM-before、MQ-before和LCP-before),及B组:确诊为胃癌患者,伊立替康用药后样本(SHD-after、HDL-after、TYT-after、WYM-after、MQ-after和LCP-after),样本均为穿刺组织样本,用于RNA提取的样品不低于2 g,至于-80℃冰箱保存备用。样本须先进行病理切片诊断。
1.2 方法
1.2.1 文库构建 利用RNA提取试剂盒AllPrep DNA/RNA Mini Kit(QIAGEN,Cat No./ID: 80204)进行组织Total RNA的提取,检测样品核酸的浓度;采用RNA建库试剂盒 mRNA Hyper Prep Kit(KAPA,KK8580)进行高通量测序文库的构建。具体的操作步骤参照试剂盒说明书;采用Qubit和qPCR对文库质量进行检测,判断文库是否符合上机标准;对质检合格的文库进行上机测序。
1.2.2 测序 使用Ⅱlumina测序平台,对基于耐药前后的组织构建的文库进行测序。
1.2.3 生物信息学分析 差异表达基因:通过生物信息学分析软件,获得耐药前后组织样本中基因表达水平的相关信息,同时筛选出耐药前后组织样本中的差异表达基因(FDR<0.05)。肿瘤耐药的特异差异表达基因的定义:基于不同患者差异表达的基因筛选出来的相同基因。
GO功能和KEGG pathway的富集分析(差异表达基因):基于GO FAT 和KEGG pathway进行候选基因的功能注释。
2 结果
2.1 原始下机数据的质控 基于Genome Analyzer Ⅱx 测序仪(Ⅱlumina)进行RNA-Seq双端测序,测得的原始碱基数目范围为62 926 168~109 079 180,见表1。在参考基因组参考序列上,利用软件进行比对,结果表明所获得的碱基和参考基因组的匹配度范围达88.17%~93.27%,见表1。这些Reads在基因组和转录组上的分布比较均匀,覆盖比较完整,见表2、3。这些序列的测序深度接近50*;说明样品的cDNA文件构建比较成功。
2.2 差异表达基因 本研究采用RNA-seq方法,在耐药前后的组织样本中分别检测到22 003和21 127表达基因,所得到的差异基因汇总数据见表4、5。其中差异基因数目为15 970个基因,达到显著差异水平的基因有541个,达到极显著差异水平的基因有148个。本研究中发现的541个突变基因(P值<0.05)与之前的研究结果进行交叉分析,结果发现ABCA3、ADAMTS15、ALMS1、BCL2L11、C1QTNF6、CAPN9、COL12A1、COL5A3、COL7A1、IRF4、KRAS、MPP6、PCDHA1、PIK3CA、POU2AF1、USP13等16个基因也有发现。这些基因的变异可以对患者使用伊立替康药物进行监测。经过分析可以得到2个用于显示两组样本数据的显著性差异的因素,一个因素是样本间基因表达的差异倍数(fold change),以2为底的对数变换;另一个因素是t检验所得到的P值,以10为底的对数变换。基于2个因素共同绘制火山图(图1),其中横坐标为差异倍数,纵坐标为差异的显著性P值,红色及蓝色的点均代表差异表达变化达到显著性水平(fold change>1.2倍且P值<0.05)的基因,黑色点代表无显著差异表达变化的基因。通过进行聚类分析,发现SHD-before、HDL-before、TYT-before、WYM-before、MQ-before和LCP-before为一组,SHD-after、HDL-after、TYT-after、WYM-after、MQ-after和LCP-after为一组,这与这些患者的临床样本分组结果是一致的,证明基于这种算法的分组的后续分析是有意义的。GO功能分析包括生物过程(biological Process,BP)(图2)、分子功能(molecular Function,MF)(图3)和细胞组分(cellular component,nent,CC)(图4)。纵坐标代表每个功能分类下的差异基因数目;条形图颜色代表富集的GO功能分类的显著性(P值基于Fisher′s Exact Test计算得到),P值的大小与颜色梯度有关,以蓝色为背景色渐变,越接近深蓝色P值则越小,相对应的KEGG通路富集的显著性水平越高。通过GO分析,可以发现达到极显著差异水平的基因有148个,将这148个基因按照表达量的上调和下调进行了归类,整理成了表6、7两组数据。我们将得到的差异表达基因与目前已有的数据库进行关联分析,最终明确与胃癌患者产生伊立替康耐药密切相关的基因包括ABCA3、ADAMTS15、ALMS1、BCL2L11、C1QTNF6、CAPN9、COL12A1、COL5A3、COL7A1、IRF4、KRAS、MPP6、PCDHA1、PIK3CA、POU2AF1和USP13等,可以用于胃癌患者产生伊立替康耐药机制的后续研究。

表1 样本转录组测序的质控统计表

表2 样本转录组测序的Genome mapping统计表

表3 组织样本转录组测序的Transcriptome Mapping统计表

图1 B/A组火山图

表4 样本的分组信息及差异基因数目

表5 差异基因的数目

图3 B/A组GO功能富集分析(Cellular Component,CC细胞组分)

图4 B/A组GO功能富集分析(Molecular Function,MF分子功能)
2.3 差异表达的KEGG分析 在生物体内,基因不是独立地行使各自的功能,而是通过相互协调来完成一系列生化反应并最终产生一系列生物学功能。KEGG通路分析有助于全面了解肿瘤的异质化、发生及发展,耐药的产生机制等。在进行KEGG通路分析时,基于筛选出来的差异基因,以KEGG通路为单位,通过Fisher′s Exact Test,来分析计算各个通路基因富集度的显著性水平,最终确定表达水平的变化达到显著性差异的代谢或信号转导通路。其中,横坐标代表可以显著富集的KEGG通路;纵坐标代表每条KEGG通路中表达水平达到显著性差异的基因数目;条形图颜色表示富集的KEGG通路的显著性差异,P值基于Fisher′s Exact Test计算得到,P值的大小与颜色梯度有关,以蓝色为背景色渐变,越接近深蓝色P值则越小,相对应的KEGG通路富集的显著性水平越高;条形图上方的数字代表的是富集因子(rich factor),富集因子的计算方法是特定KEGG通路的差异表达基因数目与参与该条通路的基因数目(所有鉴定到的基因)的比例。表8为差异基因的KEGG分析,我们可以看出差异表达基因的富集通路大多和PI3K-Akt 信号通路、胃酸分泌、黏附连接、蛋白质的消化和吸收、细胞周期和胆汁分泌等相关。
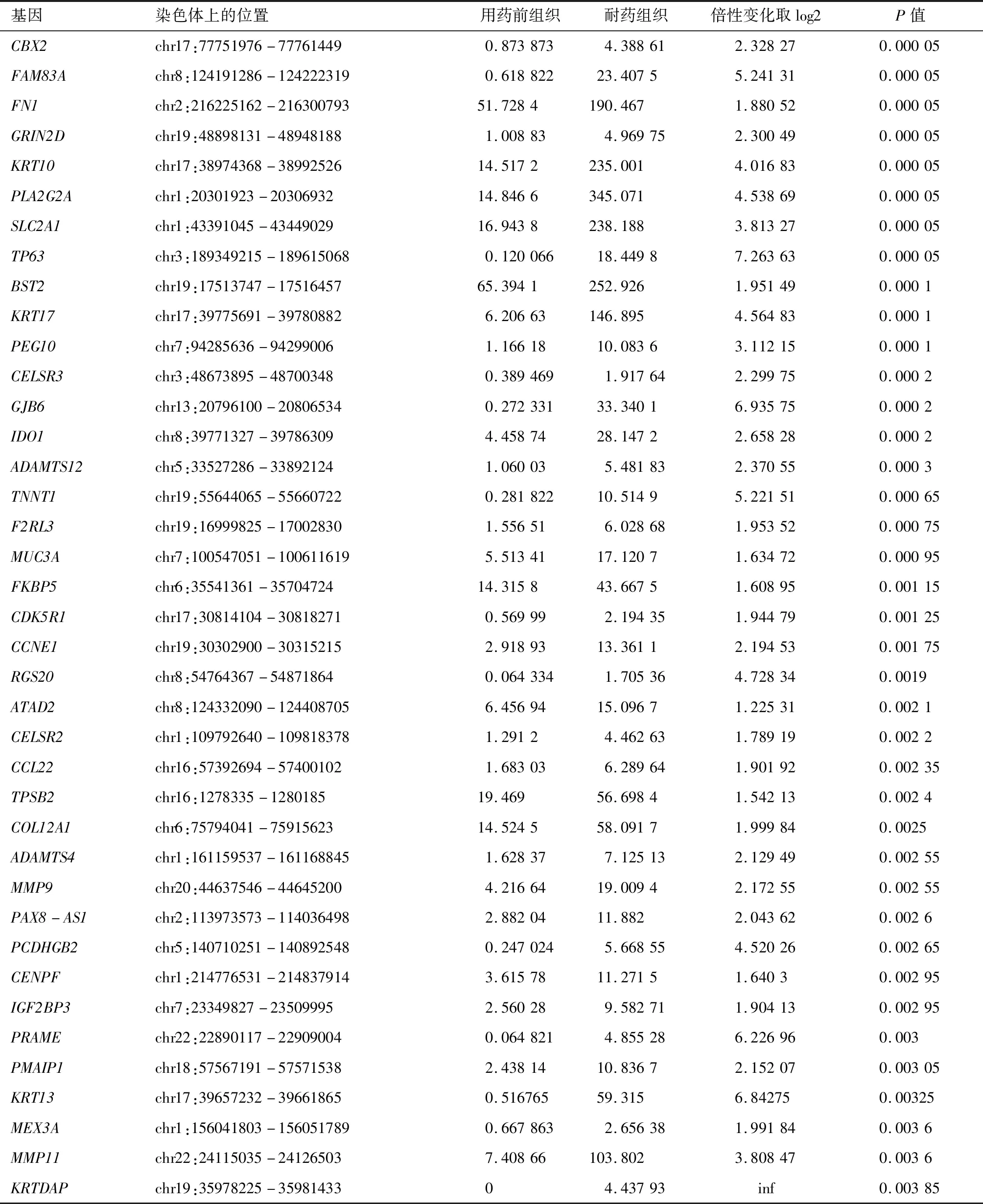
表6 耐药组织及其配对的用药前组织的差异表达基因分析(上调基因)
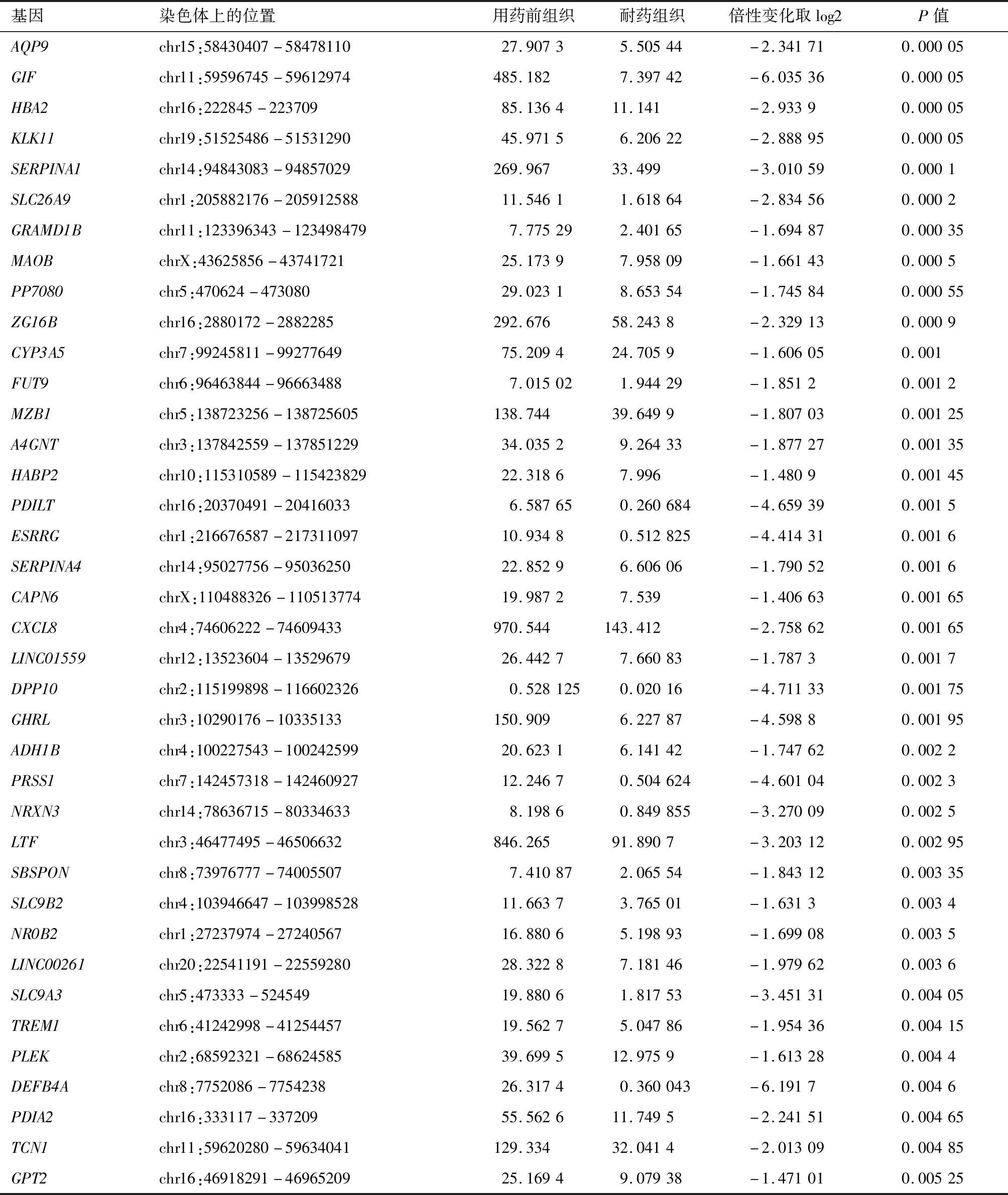
表7 耐药组织及其配对的用药前组织的差异表达基因分析(下调基因)
3 讨论
由于胃癌的发病机制及进展过程十分复杂,单个或者数个基因的研究往往不够全面;系统的检测样本中基因的表达改变有助于同步获取样本中的表达谱,筛选出哪些基因发生了达到显著性差异水平的表达变化,进而为寻找与胃癌耐药相关的基因、开发新的药物作用分子靶点、了解疾病的预后等提供有利的数据信息。基于转录组测序我们能够全面地获取样本中所有基因的表达水平的变化及剪接变异的信息,以RNA-Seq为代表的第2代高通量测序已经成为肿瘤相关研究的重要手段[8-10]。
我们将用药后组织视为实验组,与用药前组织进行比较,差异基因表达的变化与用药有关,这些结果提示在胃癌患者接受化疗的过程中一些基因突变会导致患者产生耐药。
本研究运用转录组测序对胃癌患者耐药组织及其配对的用药前组织进行了深度、全面的转录组测序,从这些差异表达基因中再筛选出与胃癌密切相关的基因,共筛选到16个差异表达的基因,后续将会通过扩大样本检测的方法进行鉴定,有望成为诊断胃癌用药检测的分子标志物。
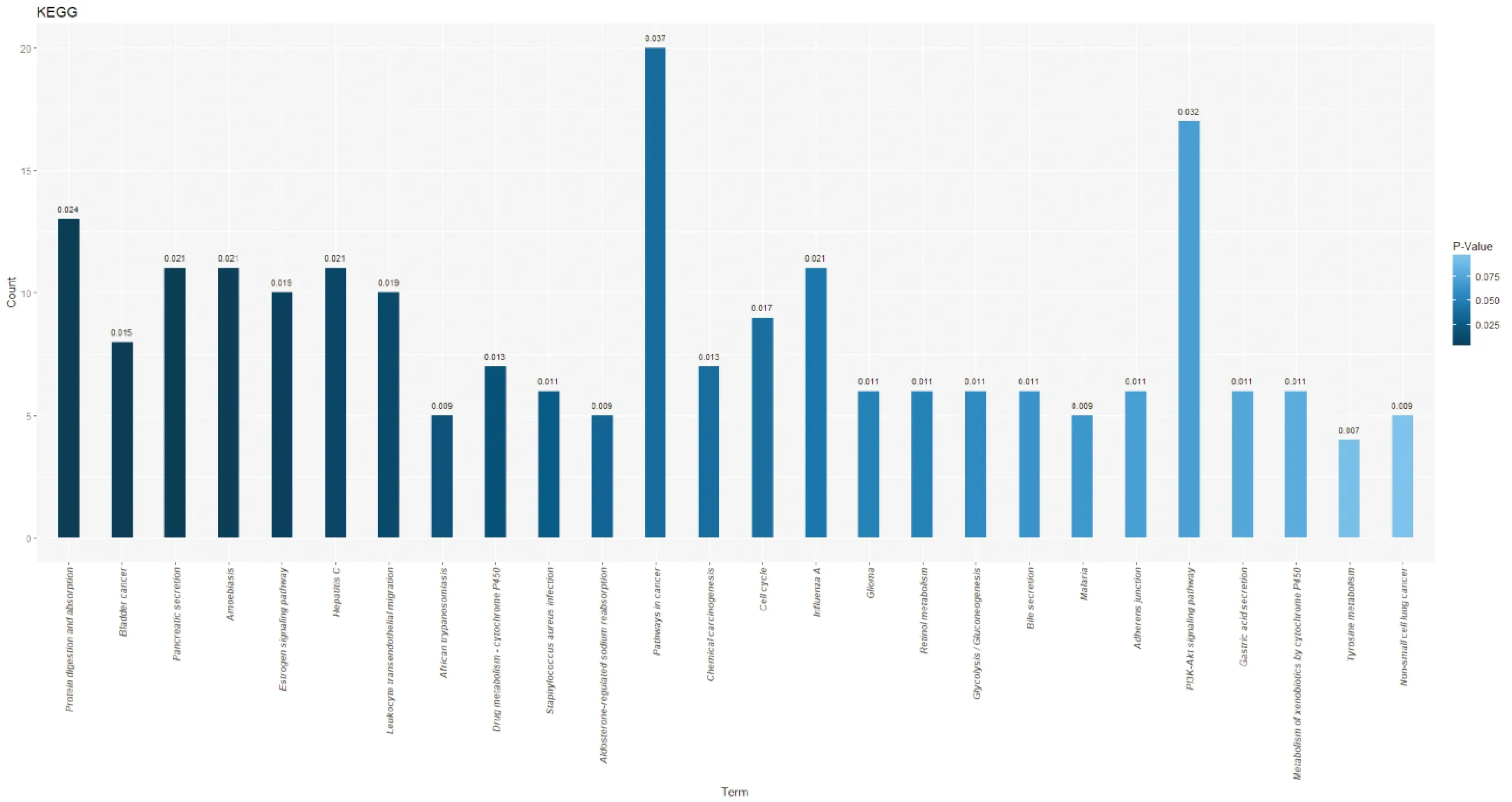
图5 B/A组KEGG通路富集分析

表8 差异表达基因富集的KEGG通路
对差异表达基因进行KEGG通路分析后,结果表明差异表达基因富集在蛋白质消化和吸收、药物代谢-细胞色素P450、胆汁分泌、黏附连接、PI3K-Akt 信号通路、胃酸分泌、细胞色素 P450代谢和酪氨酸代谢等通路中,这些结果也暗示这些通路中的差异基因可能在通路之间有crosstalk,最终导致胃癌患者耐药的产生。
研究发现MRP基因可能在胃癌和结肠癌细胞产生耐药过程中起重要作用[11]。在健康人群中,HER-2/NEU处于非激活状态,一些致癌因素会造成其结构的改变或表达的异常,其表达进而被激活,最终引起细胞的恶性转化[12]。三MMP-9基因(明胶酶B),被激活后形成Ⅳ型胶原酶,会造成肿瘤的侵袭和转移,机制是通过降解基底膜和ECM中的构成蛋白来破坏肿瘤细胞周围的ECM[13]。在肿瘤癌症模型中,伊立替康耐药伴随着EGFR和Src信号传导的上调[14]。一项基于胃癌患者肿瘤样本的研究表明,c-Met受体mRNA表达水平越高,预示肿瘤的分化程度越差,导致肿瘤的深度浸润及淋巴结的转移[15]。癌症干细胞对伊立替康具有化学抗性,c-Met抑制剂可能是基于伊立替康的胃癌患者化疗的有希望的靶分子[16]。SULF2和WRN甲基化患者对伊立替康的敏感性也高于其他患者(P<0.05),SULF2和WRN启动子甲基化检测指示用于鉴定和靶向最敏感的胃癌亚群用于个性化CPT-11疗法的潜在预测生物标志物[17]。GAS通过降解p27Kip1有助于SGC7901细胞MDR的出现[18]。
本研究中筛选出的部分差异基因在其他肿瘤研究中有报道,但这些基因在胃癌研究中尚未见报道。这些基因在胃癌患者产生伊立替康耐药过程中的具体作用机制有待进一步研究。

