改进后的验证SREBP-1c下游调控基因的染色质免疫共沉淀方法*
李建宁, 李旺, 杨玲玲, 李雨涵, 祁慧, 宋辉, 李岩, 杨怡△
宁夏医科大学 1基础医学院生物化学与分子生物学系, 2内分泌研究所(宁夏银川 750004)
染色质免疫共沉淀测序技术(chromatin immunoprecipitation followed by sequencing,ChIP-Seq)创建于2007年,现在已经成为在全基因组范围内分析转录调控和表观遗传学机制的实验标准,该项技术的原理也已被论证无误且被应用到多种研究领域中[1-3]。染色质免疫共沉淀(chromatin co-immunoprecipitation, CHIP)在其中的作用是将待测序DNA提取出来,常规染色质共沉淀方法处理组织或细胞时存在着实验持续时间长、样品DNA回收率低及PCR扩增杂带多等多种欠缺与弊端[2-3]。本课题组长期致力于脂质合成代谢机制的研究,其中固醇调节元件结合蛋白(sterol regulatory element binding proteins, SREBPs)是体内重要的一种核转录因子,其亚类SREBP-1c是我们感兴趣的目标基因之一,该研究通过对CHIP方法的改进,更高效验证SREBP-1c下游的调控基因,此种改进目前尚未见相关报道,为以后研究细胞中DNA与蛋白质相互作用奠定了新的方法学基础。2016年8月至2017年3月,本研究以SREBP-1c下游调控基因为例,对HEK 293T细胞进行调整后的染色质免疫共沉淀实验,通过改进以期获得更好的实验结果,使细胞中基因表达调控研究中DNA提取与鉴定更高效。
1 材料与方法
1.1 材料与试剂 HEK 293T细胞购自ATCC;胎牛血清购自美国Gibco公司(10099-141);DMEM高糖培养基购自Hyclone公司(SH30022);PCR扩增高保真酶HiFi购于北京全式金生物技术有限公司(AP131-02);SREBP-1(H-160)抗体(sc-8984)和Protein A beads (sc-2003)购自于Santa Cruz Biotechnology公司;IgG抗体购自于Proteintech公司(30000-0-AP);基因组DNA提取试剂盒购自于北京康为世纪生物科技有限公司(CW2298S);蛋白酶K购于北京全式金生物技术有限公司(GE201-01)。
1.2 CHIP实验流程 常规CHIP一般包括以下9个步骤:细胞处理、交联、超声裂解、抗体与磁珠耦合、孵育、洗涤、解交联、纯化及PCR。本课题组改进后的CHIP步骤包括:交联后处理、样品-抗体-磁珠孵育、3步洗涤、解交联条件、纯化方式。
1.2.1 细胞处理 将293T细胞以3.8×105平铺于60 mm培养皿中,培养24 h,用预冷的PBS清洗2次。
1.2.2 交联 加入2.0 mL 1%多聚甲醛,室温反应5 min;立即加入200.0 μL 127.0 mmol/L甘氨酸终止反应5 min;用含有1.0 mmol/L PMSF的预冷PBS清洗细胞2次,最后加入2.0 mL含有PMSF的预冷PBS吹打细胞并收集,于4℃ 800×g离心10 min,弃上清。
1.2.3 超声裂解 加入1.0 mL细胞裂解液RIPA buffer(100.0 mmol/L Tris/HCl, 5.0 mmol/L EDTA, 500.0 mmol/L NaCl, 5.0 mmol/L PMSF, 1.25% SDS, pH=7.5),置于冰水浴中超声裂解染色质(功率 25% W,超声 4.5 s,间歇 9.0 s,重复14~16次)。通过核酸凝胶图像评价超声裂解的效果。
1.2.4 样品-抗体-磁珠耦合与孵育 首先对孵育样品分组:不加抗体组,记作Mock;加阴性对照抗体组,记作IgG;加特异性抗体组,记作SREBP-1c。然后分别向50.0 μg染色质中加入50.0 μL Protein A磁珠,于4℃ 旋转3 h,用含有PMSF的预冷PBS清洗细胞2次,于4℃ 800×g离心5 min,弃沉淀;然后加入7.0 μg SREBP-1c抗体和阴性对照IgG抗体,于4℃ 旋转过夜;最后加入50 μL Protein A磁珠,于4℃ 旋转3 h。
1.2.5 洗涤 连续分别加入1.0 mL RIPA buffer(10.0 mmol/L Tris/HCl, 1.0 mmol/L EDTA, 1% Triton X-100, 0.1% SDS, 140.0 mmol/L NaCl, 0.1% 脱氧胆酸钠, 1.0 mmol/L PMSF, pH=7.4),氯化钾溶液 (1.0 mmol/L Tris/HCl, 250.0 mmol/L LiCl, 0.5% 乙基苯基聚乙二醇 P40, 0.5% 脱氧胆酸钠, 1.0 mmol/L EDTA, pH=7.4)和Tris-EDTA buffer (1.0 mmol/L EDTA, 10.0 mmol/L Tris/HCl, pH=7.4),于4℃ 旋转混匀10 min,并于4℃ 800×g离心5 min。
1.2.6 交联 向免疫复合物和作为Input组的超声裂解染色质中加入20.0 μL 蛋白酶K,56℃水浴10 min,于4℃ 800×g离心10 min,弃沉淀。
1.2.7 基因组DNA纯化 使用北京康为世纪生物科技有限公司提供的通用型基因组提取试剂盒,按照相关试剂盒操作流程,纯化DNA,加入100.0 μL灭菌双蒸水溶解DNA。
1.2.8 引物设计 依据Dif等[4]报道,设计SREBP-1c启动子区上下游引物: sense primer: 5′-GCTCAGGGTGCCAGCGAACCAGTG-3′;antisense primer: 5′-GGGTTACTAGCGGACGTCCGCC-3′,由上海生工合成。
1.2.9 PCR扩增体系 5.0 μL含Mg2+buffer,4.0 μL dNTP,10.0 mmol/L引物各1.0 μL,基因组DNA模板1.0 μL,HiFi高保真酶0.5 μL;PCR循环条件:94℃预变性2 min,94℃变性30 s,54℃退火30 s,72℃延伸30 s,循环次数30次,72℃延伸5 min。3%琼脂糖凝胶电泳,使用Bio-Red核酸凝胶成像仪采集PCR结果。
2 结果
2.1 SREBP-1c 启动子区的分析 我们发现SREBP-1c的2个SRE元件分别坐落在-288/218 bp区(5′-TTCACCCCGC-3′)和-127/-117 bp区(5′-CTCACCCCAT-3′),并与5′-TCACNCCAC-3′核心序列高度一致,见图1。下划红线显示的区域为SRE元件区,并能与SREBP-1c蛋白特异性结合,促进自身转录。
2.2 检验染色质超声破碎的效果 3%琼脂糖核酸电泳图显示,多数DNA片段分布在100~200 bp之间,没有其他杂带出现,显示改进后的效果较好,见图2。
2.3 解交联结果 在解交联时,我们采用蛋白酶K在56℃水浴10 min,直接去除结合的蛋白质,见图3。
2.4 DNA纯化及PCR PCR检测结果显示:苯酚/氯仿提纯的DNA在Input组出现PCR扩增杂带多,而且SREBP-1c抗体沉淀组PCR扩增效率极低;反而基因组提取试剂盒法在Input组和特异性SREBP-1c抗体沉淀组PCR扩增效果明显优于苯酚/氯仿提纯法,见图4。
3 讨论
SREBPs是体内重要的一种核转录因子,已经确定的SREBPs家族有3种基因:SREBP-1a、SREBP-1c和SREBP-2,其中SREBP-1a和SREBP-1c倾向于脂肪酸合成的调节,而SREBP-2主要参与调节胆固醇合成[5-7]。SREBP前体在内质网中合成,通过水解蛋白S1P(site 1 protease)和S2P(site 2 protease)裂解为成熟SREBP,随后转运到细胞核中与靶基因固醇调节元件(sterol regulatory element, SRE)结合,从而激活下游调控基因的表达,进而促进脂肪酸合成[8-9],调节胆固醇的合成[10]。已有研究发现通过激活SREBP抑制SELENOP表达达到调控胰岛素敏感性的目的[11]。非固醇介导的SREBP由其他因子激活,比如切应力、多种生长因子、葡萄糖等,而且也是通过相同的途径[12]。不同共刺激因子的募集使得由SREBP调控的基因转录呈现特异性[13]。

图1 SREBP-1c 启动子区-SRE元件结合区(-419/+90 bp)
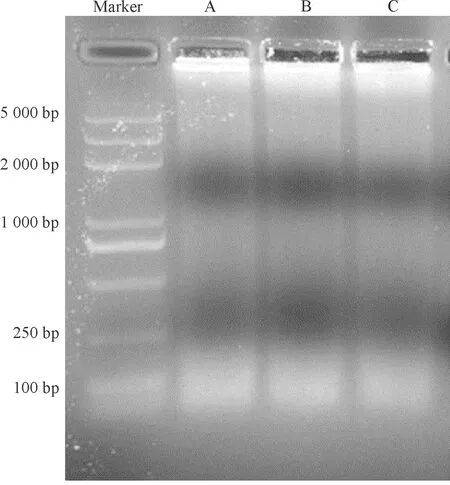
条带A、B、C为HEK 293T 培养细胞裂解超声后染色质结果
ChIP-Seq技术主要利用抗原抗体反应的特异性,可在体内用来确定与某一特定蛋白结合或蛋白定位所在的特异性DNA序列的技术,使我们了解在自然状态下基因是如何被调控的,较真实地反映与DNA直接或间接结合的调控蛋白,是研究体内基因转录调控的主要技术[14-15]。例如高等生物的基因组DNA上结合有组蛋白,组蛋白修饰是可改变的表观遗传机制,通过组蛋白乙酰化、甲基化、磷酸化、泛素化和多聚ADP糖基化等不同的修饰作用,影响基因的转录[16]。ChIP-Seq技术的关键操作步骤即CHIP实验,该实验的目的是将待测序的DNA从样品中提取出来,提供后续实验所需的DNA条带,所以,实验的关键在于如何短时间内提取到高纯度、高稳定性的目标DNA分子,常规CHIP的缺点如前言所述[2-3]。通过SREBP相关实验中的CHIP改进,使得该方法的操作流程更快速、方便,结果更稳定、可靠。改进后的CHIP方法,将细胞染色质通过交联、超声裂解、抗体与磁珠耦合、洗涤、去除蛋白质和纯化共6步处理后,得到了富集有靶序列的DNA片段,且DNA样品质量达到PCR扩增要求;将样品进行PCR实验验证后,PCR实验结果背景干净、重复性高。与常规CHIP流程相比,我们通过采取缩短交联时间,加入含有PMSF的PBS缓冲液和RIPA裂解液等来避免DNA结合后的蛋白质迅速发生降解,确保反应产物的稳定性,减少不必要的损失。在洗涤过程中,我们采用3步洗涤,即分别为RIPA buffer、LiCl buffer和Tris-EDTA buffer进行,极大地缩减了实验时间,减少了操作误差。随后我们应用蛋白酶K,56℃水浴10 min,解除蛋白质与DNA的交联,同样使得实验时间合理缩短,并因此保证了实验结果的可靠性。实验结果应用基因组提取试剂盒法进行,比较于苯酚/氯仿提纯法,在Input组和特异性SREBP-1c抗体沉淀组PCR扩增效果明显优于后者,使得实验结果更加稳定可靠。尽管该改进方法中的部分步骤有文章进行了实际应用,并取得了良好实验结果[4,17],我们首次将整个流程中的部分重要步骤重新进行分析,并合理整合、优化,从而将实验时间缩短,时间误差缩小(例如孵育时间缩短,清洗过程也加入DNA保护试剂等)。在这样的整合与优化后,实验结果能够准确反映因子之间的相互作用,确保了该方法的可信度。改进后的CHIP实验具有快速、操作简便、缩小误差、结果可靠等优点,不仅为特异性因子SREBP实验的应用提供了技术支持,也为其他类似反应原理的相互作用验证实验奠定基础,并具有实践指导意义的基础。本改进仅局限于SREBP在HEK 293T细胞染色质免疫共沉淀中,尚有很多不足:如样本量少,参照设置不严谨,以及具体条件未予以分组对照等。作为一项技术,下一步的研究方向在于扩大样本量,增加对照,选择不同的靶点分子与细胞系进行验证。

图3 改进后CHIP孵育与解交联流程

图4 CHIP检测SREBP-1c调节自身上游启动子区
以上结果说明,改进后的方法更适用于HEK 293T细胞CHIP,使细胞中基因表达调控区DNA提取鉴定方法更高效,更方便,该方法也为研究代谢研究中相关基因表达调控(包括其他多种类型细胞中DNA与蛋白质的相互作用)奠定了技术与方法基础。应用改进后的方法,我们已经获得了相关课题的后续实验结果,并完成了文章撰写,该结果另文发表。

