连续三代减数分裂雌核发育团头鲂的遗传多样性分析和RAPD鉴别方法的建立
唐首杰,毕 详,张飞明,张友良
(1.上海海洋大学 农业农村部淡水水产种质资源重点实验室,上海 201306; 2.上海海洋大学 水产科学国家级实验教学示范中心,上海 201306; 3.上海海洋大学 水产动物遗传育种中心上海市协同创新中心,上海 201306; 4.上海市松江区水产良种场,上海 201616)
人工雌核发育是一种有效的产生纯系的手段,而且在鱼类染色体操作、遗传分析以及性别控制等方面具有潜在的应用价值。迄今为止,各国学者已先后在鲤科、鲑科、鲽科、鳅科和鲷科等近百种重要经济鱼类开展雌核发育研究并获得成功[1-8],在一些重要水生模式动物如青鳉和斑马鱼等也有诸多成功报道[9-10]。
团头鲂浦江1号是养殖性能优良的草食性鱼类首例选育良种[11],为使该良种的优良性状基因进一步纯化、巩固和发展,自1999年以来,上海海洋大学水产种质资源研究室先后对团头鲂浦江1号选育系F5和F6进行了人工雌核发育(抑制第二次成熟分裂)的研究,成功地生产了一定数量的雌核发育鱼(G1)[12];随后,他们又通过抑制一次雌核发育鱼(G1)卵子的第二次成熟分裂成功地诱导出两次雌核发育鱼(G2)[13],在G2的基础上,用同样的方法诱导得到连续3次减数分裂雌核发育鱼(G3)。近几年的养殖实践证明,这些雌核发育团头鲂(G1、G2、G3)及其后代具有很好的生长优势,其生长速度超过了团头鲂选育系。但在实际的育种过程中,不同雌核发育世代团头鲂群体的遗传纯合度的具体指标如何,是否能达到遗传育种和遗传分析所需要的纯合度?此外,在雌核发育过程中可能由于经过辐射处理的异源精子的某些未失活遗传物质、物理隔离的不完善等因素而影响雌核发育鱼群体的遗传纯合性。因此,在将培育的雌核发育群体应用于育种实践前,需要用各种技术在分子水平上对其基因组进行精确的遗传学分析,以确定雌核发育类型鱼类群体的遗传纯合度是否能达到遗传育种的要求。另外,由于雌核发育团头鲂在形态上和普通团头鲂完全一致,从外形上无法鉴别普通团头鲂与雌核发育团头鲂(G1、G2、G3),这导致这几种鱼(团头鲂浦江1号、G1、G2、G3)在养殖生产和选育过程中极易混杂,为下一步的纯系选育工作埋下了隐患。鉴于此,采用分子遗传标记技术分析不同雌核发育团头鲂群体的遗传多样性和遗传纯合度,进而寻找能够有效区分不同团头鲂群体(团头鲂浦江1号、G1、G2、G3)的特异性分子标记已成为迫切需要解决的问题。
随机扩增多态性DNA(randomly amplified polymorphic DNA,RAPD)是建立在聚合酶链式反应(PCR)基础上的多位点DNA指纹技术,具有灵敏、快捷、简便、费用少等特点。该技术自1990年被提出以来[14-15],已被广泛应用于鱼类育种群体的遗传多样性分析[16]和品系鉴别[17]。贾海波等[18]采用RAPD技术分析了两个人工雌核发育红白锦鲤(Cyprinuscarpio)群体的遗传多样性。陈金辉等[19]采用RAPD技术评估了两个不同人工雌核发育草鱼(Ctenopharyngodonidellus)群体的遗传多样性和遗传纯合度。张桂蓉等[20]采用RAPD技术分析了两个人工雌核发育系鲢(Hypophthalmichthysmolitrix)近交F1群体的遗传多样性,验证了雌核发育系后代的遗传稳定性。在鱼类遗传育种实践中,RAPD技术已成为育种群体鉴别的快速有效标记之一。邹曙明等[12]通过RAPD分析发现了团头鲂选育系群体和雌核发育群体各自的特征性条带。贾海波等[18]采用RAPD方法找到了7个引物的扩增谱带可以作为区分两个不同雌核发育锦鲤群体间的遗传标记。陈金辉等[19]采用RAPD技术找到了连续两代人工诱导雌核发育草鱼群体和一代人工诱导雌核发育草鱼群体间的特异性分子标记。司伟等[21]运用RAPD技术成功鉴别了建鲤雌核发育后代。
为评估不同雌核发育团头鲂群体(G1、G2、G3)的遗传多样性和遗传纯合度,鉴别不同团头鲂群体(团头鲂浦江1号、G1、G2、G3),本研究以普通团头鲂(团头鲂浦江1号选育系F9)群体作为对照,利用RAPD技术对连续三代人工雌核发育团头鲂群体(G1、G2、G3)进行基因组扫描,目标有二,其一,确定不同雌核发育团头鲂群体的遗传多样性和遗传纯合度,进而从分子水平评估连续多代人工减数分裂雌核发育的效果;其二,寻找区分不同团头鲂育种群体(团头鲂浦江1号选育系F9、G1、G2、G3)的稳定的分子遗传标记,建立团头鲂雌核发育群体的分子鉴别技术,为团头鲂雌核发育纯系的建立奠定基础。
1 材料与方法
1.1 材料
对照组团头鲂选育系群体样本为浦江1号选育系F9群体,试验组为团头鲂一代雌核发育群体(G1)、团头鲂连续两代雌核发育群体(G2)和团头鲂连续三代雌核发育群体(G3),每群体各采集35尾,每尾剪取少许鳍条于95%乙醇中保存备用,所有样本均采自上海海洋大学鱼类种质研究试验站(上海市浦东新区新场镇)。RAPD随机引物为10碱基引物,引物序号为S1~S100[编号同生工生物工程(上海)股份有限公司引物目录],共100个,均由生工生物工程(上海)股份有限公司合成。
1.2 实验方法
1.2.1 基因组DNA的提取
基因组DNA提取采用常规的酚-氯仿方法进行[22]。
1.2.2 基因组DNA的RAPD分析
PCR扩增反应在Eppendorf Mastercycler Gradient PCR仪上进行,每一样品的反应总体积25 μL,含2.5 μL 10×扩增缓冲液,其中含100 mmol·L-1Tris-HCl (pH 9.0),500 mmol·L-1KCl,15 mmol·L-1MgCl2,1% (质量浓度)明胶,1%(体积分数)Triton X-100,最后调节pH值至8.0。2 μL dNTP混合液(每种dNTP的终浓度 0.2 mmol·L-1),引物2 μL(终浓度0.2 μmol·L-1),2 μL基因组DNA(50~150 ng),0.5 μLTaqDNA聚合酶(1.25单位),16 μL无菌重蒸水,最后加入30 μL石腊油。
PCR的扩增程序为:94 ℃预变性5 min,接下来进行45个循环,每个循环包括94 ℃ 45 s,36 ℃ 45 s,72 ℃ 90 s;最后一次循环结束后在72℃延伸5min。
取10 μL扩增产物经1.5%琼脂糖凝胶电泳,电泳缓冲液为0.5×TBE,电压为5 V·cm-1,采用的分子质量标准为MarkerⅢ[天根生化科技(北京)有限公司产品]。电泳结束后,电泳凝胶经溴化乙锭染色,用Syngene凝胶成像分析系统进行扫描摄像,并用GeneTools软件对每个RAPD随机引物扩增条带的分子质量进行估算。每个RAPD随机引物至少被重扩一次以保证结果的准确性。
1.2.3 数据处理与分析
首先对照扩增图谱分别统计RAPD特异性条带在群体间的出现频率。再进行遗传多样性数据统计,仅记录清晰且重复性好的DNA条带。根据MarkerⅢ各带的迁移率确定扩增产物各带的迁移率,同一引物对不同个体扩增出的RAPD条带,只要迁移率相同都视为同一类型(性状)记为1,缺乏此带的记为0;以此类推,将所有个体的RAPD条带转换成二元数据矩阵。采用POPGENE1.31软件包[23]计算以下遗传变异参数:
多态位点比例(P):P=(多态位点数/位点总数)×100%。
(1)
(2)
式(2)中:pi为某一座位上第i个等位基因在某一群体中出现的频率;lnpi为pi的自然对数;r为等位基因数目。
群体内个体间Nei’s标准遗传距离(Ds)和Nei’s遗传相似系数(I)按照Nei[25]的方法计算:
Ds=-lnI;
(3)
(4)
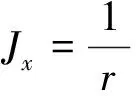
(5)
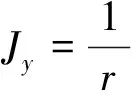
(6)

(7)
式(3)~(7)中,Xij、Yij分别为个体X、Y第j座位第i个等位基因频率;r为基因座数目;m为等位基因数目。
群体间Nei’s标准遗传距离(Ds)和Nei’s遗传相似系数(I)按照Nei[25]的方法计算:
Ds=-lnI;
(8)
(9)
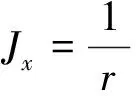
(10)
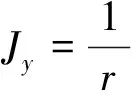
(11)

(12)
式(8)~(12)中:Xij、Yij分别为群体X、Y第j座位第i个等位基因频率;r为群体中的基因座数目;m为等位基因数目。
用Arlequin 3.01软件[26]估测群体间成对FST值[27],并利用置换检验法(permutation test)进行显著性检验(重复次数为1 000)。
采用分子方差分析(analysis of molecular variance,AMOVA)估算各群体间和群体内的遗传变异及分化水平。通过3种分子遗传变量进行分析:FST为群体内个体之间(among individuals within populations)的遗传变异;FSC为组内各群体间(among populations within groups)的变异程度;FCT为不同组别之间(among groups)的遗传变异程度。并用置换检验法(permutation test)进行1 000次重复模拟计算,以上运算在Arlequin 3.01软件[26]中完成。
利用MEGA 4.0软件包[28]根据群体间Nei’s标准遗传距离(DS),用UPGMA法(unweighted pair group method with arithmetic means)和NJ法(neighbour-joining method)构建群体间聚类图。
采用LSD-t检验比较群体间遗传多样性参数(多态位点比例和Shannon信息指数),Bonferroni校正P值[29],检验水准定为α=0.05,在SPSS 19.0 软件上完成运算。
2 结果与分析
2.1 RAPD扩增结果
本研究从100条随机引物中筛选出39条扩增条带清晰、重复性好、扩增效果稳定的随机引物(表1),并对清晰可计数位点进行统计,各引物所产生的条带数见表2。单条引物扩增的条带数为2~10条,扩增片段长度集中在200~2 000 bp。39条引物在F9、G1、G2和G3群体中扩增条带总数分别为213条、202条、200条和190条,呈现出随雌核发育世代数增加而减少的趋势。单条引物在F9、G1、G2和G3群体中扩增条带数的范围分别为2~10、2~9、2~9和2~10。
2.2 群体内遗传变异分析
2.2.1 多态位点比例和Shannon信息指数
39条引物在F9、G1、G2和G3群体中的多态位点数、多态位点比例和Shannon信息指数见表2。39条引物在F9、G1、G2和G3群体中扩增到的多态位点数分别为77、72、54和51,单条引物在F9、G1、G2和G3群体中扩增到的多态位点数范围分别为0~7、0~4、0~5和0~7。F9、G1、G2和G3群体的多态位点比例分别为36.15%、35.64%、27.00%和26.84%,F9群体的多态位点比例最高,G3群体的多态位点比例最低,3个雌核发育群体的多态位点比例均明显低于对照组F9群体;随着雌核发育世代数的增加,多态位点比例呈现逐代降低的趋势,即G1>G2>G3。F9、G1、G2和G3群体的Shannon信息指数为0.138 3~0.207 9,F9群体的Shannon信息指数最高,G3群体的Shannon信息指数最低,3个雌核发育群体的Shannon信息指数均明显低于对照组F9群体;Shannon信息指数随雌核发育世代数的增加而降低,即:G1>G2>G3。
群体间多态位点比例和Shannon信息指数的LSD-t检验结果显示(表3),在α=0.05显著性水平上,群体间多态位点比例差异不显著(P=0.261~1.000),群体间Shannon信息指数差异不显著(P=0.312~1.000)。
总体而言,3个雌核发育群体的遗传多样性水平均明显低于对照组F9群体,随着雌核发育世代数的增加,遗传多样性水平呈现逐代降低的趋势,即G1>G2>G3。
表1 本研究使用的39条RAPD引物序列及编号
Table 1 Sequences and codes of 39 random primers used in the study

引物编号Primer code序列(5′-3′)Sequence(5′-3′)引物编号Primer code序列(5′-3′)Sequence(5′-3′)引物编号Primer code序列(5′-3′)Sequence(5′-3′)S1GTTTCGCTCCS29GGGTAACGCCS66GAACGGACTCS2TGATCCCTGGS30GTGATCGCAGS67GTCCCGACGAS3CATCCCCCTGS32TCGGCGATAGS68TGGACCGGTGS4GGACTGGAGTS33CAGCACCCACS69CTCACCGTCCS7GGTGACGCAGS38AGGTGACCGTS70AGGGCCGTCTS10CTGCTGGGACS40GTTGCGATCCS71AAAGCTGCGGS17AGGGAACGAGS45TGAGCGGACAS72TGTCATCCCCS19ACCCCCGAAGS50GGTCTACACCS73AAGCCTCGTCS21CAGGCCCTTCS51AGCGCCATTGS74GGATGAGACCS22TGCCGAGCTGS56AGGGCGTAAGS75GACGGATCAGS24AATCGGGCTGS58GAGAGCCAACS76CACACTCCAGS25AGGGGTCTTGS60ACCCGGTCACS77TTCCCCCCAGS28GTGACGTAGGS64CCGCATCTACS78TGAGTGGGTG
表2 四个群体的扩增位点数、多态位点数和Shannon信息指数(S)
Table 2 The number of amplified bands, polymorphic loci and Shannon’s information index (S) within four populations

引物Primer扩增位点数(多态位点数)Number of amplified bands (polymorphic loci)F9G1G2G3Shannon信息指数(S)Shannons information index (S)F9G1G2G3S15(2)5(1)5(2)4(1)0.16400.24930.18550.1289S24(3)3(0)3(0)3(0)0.00000.39760.00000.0000S36(1)4(1)4(1)4(0)0.09870.03780.09470.0000S44(1)4(1)4(3)4(2)0.16580.16580.49270.3116S74(1)4(2)4(1)3(0)0.17380.10780.13890.0000S105(2)5(1)6(3)4(0)0.13670.20780.33000.0000S177(1)7(3)7(0)7(0)0.37490.09470.00000.0000S193(0)2(1)2(0)2(0)0.34540.00000.00000.0000S215(0)5(0)6(1)6(3)0.00000.00000.11510.3229S225(0)5(2)5(0)5(0)0.25780.00000.00000.0000S2410(7)9(4)9(4)10(7)0.31570.40120.24110.4266S258(7)4(0)4(0)4(0)0.00000.40170.00000.0000S288(6)7(2)5(0)5(2)0.14660.36980.00000.1558S296(2)6(3)6(0)4(0)0.26660.20000.00000.0000S307(6)6(3)6(1)7(3)0.39470.50320.07940.2508S326(6)8(4)3(0)4(1)0.36270.43520.00000.1159S335(3)5(0)5(4)5(2)0.00000.41450.53050.2652S384(0)5(0)6(5)4(0)0.00000.00000.48610.0000S405(2)5(4)5(3)5(2)0.44110.21510.40340.3292S455(2)5(2)5(0)5(1)0.24000.26710.00000.1326S502(2)2(2)3(2)3(3)0.45130.45130.40630.6211S518(0)8(4)8(1)8(0)0.30470.00000.08290.0000S564(0)4(3)4(0)4(0)0.49500.00000.00000.0000S584(1)4(0)4(1)3(0)0.00000.16580.17270.0000S606(3)5(3)5(1)7(6)0.26620.22720.03780.4491S647(2)6(2)7(2)7(3)0.16580.13680.11590.2440S666(0)7(3)7(4)5(0)0.28740.00000.34040.0000S677(3)6(4)7(3)7(3)0.38420.23510.25500.2828S684(1)4(2)4(1)3(0)0.16400.05290.05290.0000S695(2)5(1)5(2)4(0)0.13910.27630.26710.0000S706(0)6(1)6(0)6(4)0.20000.00000.00000.4421S716(3)6(4)5(2)6(2)0.29510.31370.14370.2257S724(2)4(2)3(1)3(1)0.31160.31160.16110.1078S734(1)4(1)3(0)3(0)0.06610.17270.00000.0000S747(2)6(0)7(1)7(2)0.00000.19080.09210.1603S757(3)7(2)7(3)4(0)0.15360.23820.24840.0000S764(0)4(0)4(0)4(1)0.00000.00000.00000.1727S775(0)5(2)5(1)5(1)0.35330.00000.13260.1326S785(0)5(2)6(1)6(1)0.18520.00000.09260.1151 总计Total213(77)202(72)200(54)190(51) P/%36.1535.6427.0026.84 平均Mean0.20790.18570.14610.1383
2.2.2 群体内个体间的遗传相似系数和遗传距离
F9、G1、G2和G3群体内个体间的遗传相似系数和遗传距离见表4。4个群体的群体内个体间的平均遗传相似系数为0.828 5~0.906 0,G3群体内个体间的平均遗传相似系数最高(0.906 0),F9群体内个体间的平均遗传相似系数最低(0.828 5),3个雌核发育群体的群体内个体间平均遗传相似系数均明显高于对照组F9群体;群体内个体间的平均遗传相似系数呈现随雌核发育世代数的增加而升高的趋势,即G3>G2>G1。
表3 四个团头鲂群体间多态位点比例(对角线上)和Shannon信息指数(对角线下)LSD-t检验P值
Table 3P-values of LSD-ttests from percentage of polymorphic loci (above diagonal) and Shannon’s information index (below diagonal) between four populations ofM.amblycephala

群体PopulationF9G1G2G3F91.0000.9640.402G11.0000.6690.261G20.5061.0001.000G30.3121.0001.000
4个群体的群体内个体间的平均遗传距离为0.099 1~0.190 8,G3群体内个体间的平均遗传距离最低(0.099 1),F9群体内个体间的平均遗传距离最高(0.190 8),3个雌核发育群体的群体内个体间平均遗传距离均明显低于对照组F9群体;群体内个体间的平均遗传距离呈现随雌核发育世代数的增加而降低的趋势,即G1>G2>G3。
总体而言,3个雌核发育群体的遗传纯度(遗传相似系数)均明显高于对照组F9群体,随着雌核发育世代数的增加,雌发育群体内的遗传纯度呈逐代上升的趋势,即G3>G2>G1。
2.3 群体间遗传变异分析
2.3.1 群体间RAPD特异片段分析
在39条扩增稳定、多态性丰富的随机引物中,有5条引物在群体间产生了特异性DNA片段,这5条引物分别是S3、S40、S58、S71和S75(表5)。其中,引物S3扩增出一条长约136 bp片段,该片段在G3群体中出现频率达100%,而在其他3个群体中的出现频率均为0(图1);同时,引物S3扩增出另一条长约797 bp片段,该片段在G3群体中的出现频率为0,而在其他3个群体中的出现频率均为100%(图1)。由此可见,引物S3产生的RAPD图谱及其特异片段(136 bp和797 bp)可以作为区分G3群体和其他3个群体(F9、G1和G2)的分子遗传标记。
如图2所示,引物S40扩增出一条长约780 bp片段,该片段在G3群体中的出现频率为100%,而在其他3个群体中的出现频率仅为5.71%~14.29%,笔者认为,该片段在G3群体中的高出现频率可作为区分G3群体与其他3个群体(F9、G1和G2)的重要依据。
如图3所示,引物S58扩增出一条长约439 bp片段,该片段在G3群体中的出现频率为0,而在其他3个群体(F9、G1和G2)中的出现频率高达71.43%~100%,由此可以认为,引物S58产生的RAPD特异片段(439 bp)可作为区分G3群体和其他3个群体(F9、G1和G2)的分子遗传标记。
如图4所示,引物S71扩增出一条长约504 bp片段,该片段在G2群体中的出现频率为0,而在其他3个群体(F9、G1和G3)中的出现频率高达71.43%~85.71%,因此,引物S71产生的RAPD特异片段(504 bp)可作为区分G2群体和其他3个群体(F9、G1和G3)的分子遗传标记。
如图5所示,引物S75扩增出一条长约313 bp片段,该片段在G3群体中的出现频率为0,而在其他3个群体(F9、G1和G2)中的出现频率高达85.71%~100%,鉴于此,引物S75产生的RAPD特异片段(313 bp)可作为区分G3群体和其他3个群体(F9、G1和G2)的分子遗传标记。
综上所述,4条随机引物(S3、S40、S58和S75)可用于区分G3群体和其他3个群体(F9、G1和G2),其中,使用引物S3进行群体鉴别的可靠性最高。仅1条随机引物(S71)能用于区分G2群体和其他3个群体(F9、G1和G3)。此外,目前尚未找到能有效区分F9群体和G1群体的随机引物。
2.3.2 群体间遗传相似系数、遗传距离和遗传分化
表4 群体内个体间的遗传相似系数和遗传距离
Table 4 Genetic identity and genetic distance between individuals within each population

数值Value遗传相似系数Genetic identityF9G1G2G3遗传距离Genetic distanceF9G1G2G3平均值Mean0.82850.85380.89680.90600.19080.15880.10930.0991最大值Maximum0.91150.92040.93810.94250.36430.22760.15270.1527最小值Minimum0.69470.79650.85840.85840.09270.08300.06390.0592
表5 五个引物在4个群体间扩增出的特异DNA片段及其出现频率
Table 5 Size and frequencies of specific DNA fragments amplified by 5 random primers among four populations
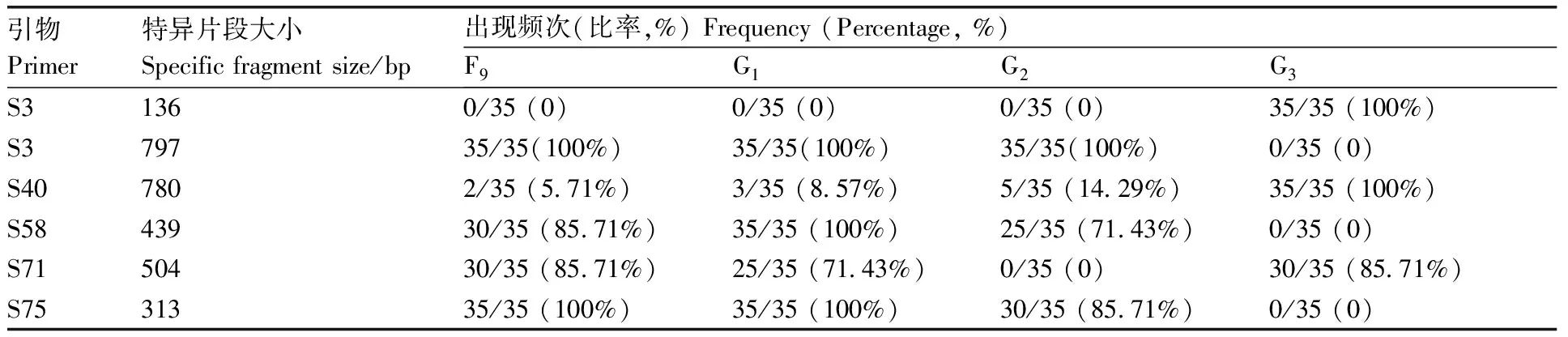
引物Primer特异片段大小Specific fragment size/bp出现频次(比率,%) Frequency (Percentage, %)F9G1G2G3S31360/35 (0)0/35 (0)0/35 (0)35/35 (100%)S379735/35(100%)35/35(100%)35/35(100%)0/35 (0)S407802/35 (5.71%)3/35 (8.57%)5/35 (14.29%)35/35 (100%)S5843930/35 (85.71%)35/35 (100%)25/35 (71.43%)0/35 (0)S7150430/35 (85.71%)25/35 (71.43%)0/35 (0)30/35 (85.71%)S7531335/35 (100%)35/35 (100%)30/35 (85.71%)0/35 (0)

M,MarkerⅢ;箭头示特异DNA片段。下同。M, MarkerⅢ; The arrows showed the specific DNA fragment. The same as below.图1 引物S3在4个群体中的扩增图谱Fig.1 Electrophoretogram of RAPD products obtained from primer S3
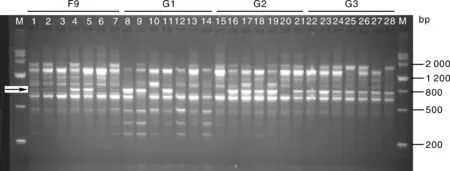
图2 引物S40在4个群体中的扩增图谱Fig.2 Electrophoretogram of RAPD products obtained from primer S40
4个群体间遗传相似系数和遗传距离见表6。群体间遗传相似系数为0.840 4~0.920 4,G1群体和G3群体间遗传相似系数最低(0.840 4),F9群体和G2群体间遗传相似系数最高(0.920 4)。群体间遗传距离为0.082 9~0.173 9,F9群体和G2群体间遗传距离最小(0.082 9),G1群体和G3群体间遗传距离最大(0.173 9)。
4个群体间的遗传分化指数(成对FST值)见表7。群体间成对FST值为0.269 2~0.419 5,F9群体和G2群体间FST值最小(0.269 2),G2群体和G3群体间FST值最大(0.419 5)。经1 000次置换检验得到的FST值的P值为0.000 0~0.009 0,均达到极显著水平(P<0.01),据此可推测,4个群体间存在极显著的遗传分化。
2.3.3 分子方差分析
本研究对4个团头鲂群体进行分组(1个组、2个组、3个组),从不同角度对群体遗传变异进行分子方差分析(AMOVA)。结果显示(表8),在任何一种分组条件下,FST值都达到了极显著水平(P<0.01),说明群体内个体间的遗传分化极显著。当把所有群体分成2个组或3个组时,FSC值均达到极显著水平(P<0.01),表明群体间存在极显著的遗传分化。这证实了群体间遗传分化指数(FST值)的分析结果。当把所有群体分成2个组或3个组时,在任何一种分组条件下,FCT值均未达到显著水平(P>0.05),表明组别之间不存在显著的遗传分化。

图3 引物S58在4个群体中的扩增图谱Fig.3 Electrophoretogram of RAPD products obtained from primer S58

图4 引物S71在4个群体中的扩增图谱Fig.4 Electrophoretogram of RAPD products obtained from primer S71

图5 引物S75在4个群体中的扩增图谱Fig.5 Electrophoretogram of RAPD products obtained from primer S75
2.3.4 聚类分析
基于群体间Nei’s标准遗传距离构建的4个团头鲂群体间NJ聚类图和UPGMA聚类图如图6所示。因聚类方法的差异,2种聚类图显示了各自的特点,很难说哪种聚类图最能反映真实的群体间遗传关系。但2种聚类图(图6-a、b)均显示,F9、G2和G3群体可被聚为一类,G1群体和其他3个群体(F9、G2、G3)间聚类关系较远,这也验证了前文中群体间遗传距离的结果。
表6 四个群体间的遗传相似系数(对角线上)和遗传距离(对角线下)
Table 6 Genetic similarity (above diagonal) and genetic distance (below diagonal) between populations

群体PopulationF9G1G2G3F90.85820.92040.8920G10.15290.87490.8404G20.08290.13360.9037G30.11430.17390.1012
表7 四个群体间的遗传分化指数(成对FST值)(对角线上)及其P值(对角线下)
Table 7 Genetic differentiation index (PairwiseFSTvalues) (above diagonal) and the probabilities of the test of genetic differentiation (below diagonal) between four populations
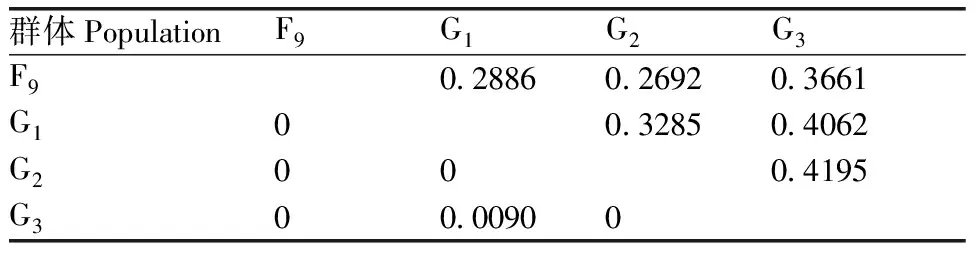
群体PopulationF9G1G2G3F90.28860.26920.3661G100.32850.4062G2000.4195G300.00900
表8 四个团头鲂群体间遗传差异的分子方差分析(AMOVA)
Table 8 Analysis of molecular variance (AMOVA) among four populations ofM.amblycephala

分组Grouping变异百分率Percentage of variation/%群体内Withinpopulations组内群体间Among populationswithin groups组间Amonggroups固定指数Fixation indexFSTFSCFCT未分组One group65.2834.720.3472∗∗2个组Two groups(1.F9;2.G1,G2,G3)67.8239.98-7.800.3218∗∗0.3708∗∗-0.0780ns2个组Two groups(1.F9,G1;2.G2,G3)65.1734.350.480.3483∗∗0.3451∗∗0.0048ns2个组Two groups(1.F9,G2;2.G1,G3)65.3835.10-0.480.3462∗∗0.3493∗∗-0.0048ns2个组Two groups(1.F9,G3;2.G1,G2)65.2834.720.000.3472∗∗0.3472∗∗0.0000ns2个组Two groups(1.F9,G1,G2;2.G3)62.3128.609.090.3769∗∗0.3146∗∗0.0909ns2个组Two groups(1.F9,G1,G3;2.G2)68.7641.91-10.680.3124∗∗0.3787∗∗-0.1068ns2个组Two groups(1.F9,G2,G3;2.G1)62.7329.477.800.3727∗∗0.3196∗∗0.0780ns3个组Three groups(1.F9;2.G1;3.G2,G3)65.1833.890.930.3482∗∗0.3421∗∗0.0093ns3个组Three groups(1.F9;2.G1,G2;3.G3)65.0833.111.810.3492∗∗0.3372∗∗0.0181ns3个组Three groups(1.F9;2.G1,G3;3.G2)67.1049.62-16.720.3290∗∗0.4251∗∗-0.1672ns3个组Three groups(1.F9,G1;2.G2;3.G3)65.3034.87-0.160.3470∗∗0.3481∗∗-0.0017ns3个组Three groups(1.F9,G2;2.G1;3.G3)63.6321.2615.100.3637∗∗0.2504∗∗0.1510ns3个组Three groups(1.F9,G3;2.G1;3.G2)65.4836.34-1.820.3452∗∗0.3569∗∗-0.0182ns
*表示P<0.05,**表示P<0.01,ns表示“无显著性”(P>0.05)。
* denotedP<0.05,**denotedP<0.01,ns denoted non-significant(P>0.05).

图6 基于群体间遗传距离的4个团头鲂群体间NJ聚类图(a)和UPGMA聚类图(b)Fig.6 NJ (a) and UPGMA (b) dendrogram of four populations of M. amblycephala based on genetic distances
3 讨论
3.1 连续多代减数分裂雌核发育效果分析
在传统的育种方法中,建立一个遗传纯系或选育系一般需要通过连续数代的近亲交配来完成。同时还会因近交衰退及繁育条件限制对部分个体的淘汰造成育种材料的丢失,因而这是一项长期、大量而严密的工作[30]。而采用人工诱导雌核发育的方法可大大地加速纯化过程。人工诱导鱼类雌核发育的方式有两种:一种是通过抑制第二极体释放来诱导产生雌核发育二倍体,称为减数分裂雌核发育;另一种是通过抑制第一次卵裂来诱导产生雌核发育二倍体,称为卵裂雌核发育[30]。一次卵裂雌核发育相当于8~10代同胞兄妹交配,一次卵裂雌核发育加一次减数分裂雌核发育就可以获得纯系[10]。据吴清江等[1]报道,鲤(CyprinuscarpioL.)雌核发育第一代近交系数已接近60%,2~3代雌核发育可取代鲤8~10代近交。
理论上,抑制第二极体释放诱导雌核发育二倍体时,如果不发生重组,子代都应成为完全的纯合体,但由于鱼类的染色体一般都发生一次交叉,因此距离着丝粒近的基因位点为纯合体,距离着丝粒远的基因位点,杂合体的比率根据基因位点在染色体上的位置而变化,范围为0~1.0。在实际的雌核发育育种实践中,连续多代诱导减数分裂雌核发育,将快速地提高遗传相似系数。虽然是杂合子,但是亲本的遗传特征已被固定,其子代个体数量性状表现一致,个体之间差异较小,可以作为良种选育的一种有效方法。许多研究显示,人工诱导多代减数雌核发育将大大加速鱼类基因纯合速度[31-33],例如,大黄鱼连续两代减数雌核发育群体15个微卫星座位的平均纯合度高达0.803,群体内个体间平均相似系数达0.867 2[31]。牙鲆连续两代减数分裂雌核发育群体内个体间的平均遗传相似系数为0.923 8,显著高于对照组家系,证明连续多代人工诱导减数分裂雌核发育具有固定母本遗传性状的作用[32]。鲢(Hypophthalmichthysmolitrix)连续两代减数分裂雌核发育群体内个体间平均遗传相似度达0.980 7,达到了快速建立纯系的目的[33]。
团头鲂浦江1号是原农业部公告推广的鱼类良种,目前已成为我国团头鲂养殖业的主导品种之一,鉴于没有永远的良种的理念和推陈出新的时代要求,借助人工雌核发育技术,在浦江1号良种的基础上,快速培育出性状更加优良且遗传稳定的品系具有十分重要的意义。上海海洋大学水产种质资源研究室早在2000年就成功开展了团头鲂减数分裂雌核发育的诱导,并陆续获得了第一代减数分裂雌核发育群体和连续两代减数分裂雌核发育群体[12-13]。为使团头鲂雌核发育家系的遗传纯合度得到更显著的提升,本课题组对连续两代雌核发育团头鲂雌鱼卵子再进行减数分裂雌核发育诱导,成功培育出连续三代减数分裂雌核发育团头鲂群体,并应用于良种选育实践。本研究采用39条随机引物对雌核发育一代群体(G1)、雌核发育二代群体(G2)和雌核发育三代群体(G3)基因组进行RAPD分析,结果显示,G1、G2和G3群体的多态位点比例分别为35.64%、27.00%和26.84%,远低于鲤(C.carpioL.)[20]、双棘黄姑鱼(Nibeadiacanthus)[34]和唐鱼(TanichthysalbonubesLin)[35]等鱼类养殖群体的相应参数。而且,多态位点比例随雌核发育世代数的增加而降低,即:G1>G2>G3,其中,G2比G1降低了24.24%,G3又比G2降低了0.59%,平均每代降低12.42%。G1、G2和G3群体的Shannon信息指数分别为0.1857、0.1461和0.1383,远低于鲤(C.carpioL.)[20]、草鱼(Ctenopharyngodonidellus)[19]和双棘黄姑鱼(Nibeadiacanthus)[34]等鱼类养殖群体的相应参数。而且,Shannon信息指数随雌核发育世代数的增加而降低,即:G1>G2>G3,其中,G2比G1降低了21.32%,G3又比G2降低了5.34%,平均每代降低13.33%。多态位点比例和Shannon信息指数的结果一致表明,连续多代的人工减数分裂雌核发育诱导已使团头鲂育种群体的遗传多样性明显降低,并呈现逐代降低的趋势。G1、G2和G3群体内个体间的平均遗传相似系数分别为0.853 8、0.896 8和0.906 0,呈现随雌核发育世代数的增加而升高的趋势,即:G3>G2>G1,其中,G2比G1升高了5.04%,G3又比G2升高了1.03%,平均每代升高3.04%。表明连续多代的人工减数分裂雌核发育诱导已使团头鲂育种群体的遗传纯度明显升高,并呈现逐代升高的趋势。以上结果表明,连续多代减数分裂雌核发育能显著加快团头鲂基因的纯合速度,使控制优良性状的基因快速纯合化;所培育的雌核发育群体,尤其是雌核发育三代群体已经是一个遗传一致性较高的高纯品系。此外,G3群体与对照组(F9群体) 间成对FST值为0.366 1,经1 000次置换检验得到的FST值的P值为0.000 0,已达到极显著水平(P<0.01),显示G3群体与对照组(F9群体)间已经发生了极显著的遗传分化。这也说明连续的雌核发育是快速建立具有特定遗传特征品系的有效途径,这在以往的研究报道[36-38]中已得到印证。本研究结果为连续多代人工诱导减数分裂雌核发育育种实践提供了新的依据。
3.2 群体间鉴别标记的发现及其成因分析
人工诱导雌核发育是使团头鲂良种重要经济性状基因快速纯化和固定的主要途径。但在实际的雌核发育育种实践中,因雌核发育子代与普通团头鲂在形态上极其相似,故使用传统的形态学方法难以准确区分雌核发育团头鲂与普通团头鲂群体。作为一种高效的分子遗传标记技术,RAPD技术标记位点多,理论上,其检测区几乎覆盖整个基因组,已成为鱼类品种(系)鉴定和遗传多样性分析的有力工具。例如,刘敏等[39]从100条RAPD随机引物的扩增图谱中找到了7条能区分雌核发育草鱼与普通草鱼群体的特异性条带。贾海波等[18]从30个RAPD随机引物中找到7个引物的扩增谱带用于鉴别两个不同的红白锦鲤雌核发育群体。
本研究从100条RAPD随机引物中筛选到5条在群体间产生特异DNA片段的引物,根据这些引物扩增到的特异DNA片段在群体内的出现频率,可以将它们分成两类,第一类特异DNA片段在某个雌核发育群体中出现频率为0,在对照组和其他雌核发育群体中出现频率很高(71.43%~100%)。如引物S3扩增出的一条长约797 bp片段在G3群体中的出现频率为0,而在其他3个群体中的出现频率均为100%(图1)。引物S58扩增出的一条长约439 bp片段在G3群体中的出现频率为0,而在其他3个群体(F9、G1和G2)中的出现频率高达71.43%~100%(图3)。引物S71扩增出的一条长约504 bp片段在G2群体中的出现频率为0,而在其他3个群体(F9、G1和G3)中的出现频率高达71.43%~85.71%(图4)。引物S75扩增出的一条长约313 bp片段在G3群体中的出现频率为0,而在其他3个群体(F9、G1和G2)中的出现频率高达85.71%~100%(图5)。第二类特异DNA片段在某个雌核发育群体中出现频率为100%,而在对照组和其他雌核发育群体中出现频率很低(0~14.29%)。如,引物S3扩增出的一条长约136 bp片段在G3群体中出现频率达100%,而在其他3个群体中的出现频率均为0(图1);引物S40扩增出的一条长约780 bp片段在G3群体中的出现频率为100%,而在其他3个群体中的出现频率仅为5.71%~14.29%(图2)。
在人工诱导雌核发育的过程中,尽管因自然因素和人为因素的作用,也可能诱发突变,但雌核发育诱导本身并不能产生新的基因,而是通过等位基因频率的变化,使一些有利的性状得以积累和加强,一些有害隐性基因控制的性状也会因基因座位的纯合化而表达出来,从而被淘汰。产生第一类特异DNA片段的原因,可能是雌核发育团头鲂群体近交水平和遗传纯度较高,各等位基因在一定程度上被固定,因而频率上升,谱带数目减少。此种现象已在多种鱼类雌核发育群体中被观察到,刘敏等[39]从100条RAPD随机引物的扩增图谱中找到了7条能区分雌核发育草鱼与普通草鱼群体的特异性条带,其中6条仅存在于普通草鱼中,在异精雌核发育草鱼F1中缺失。张虹[40]从180条RAPD随机引物中筛选到一个引物(S68)作为鉴别雌核发育草鱼和普通草鱼的分子标记,该引物在普通草鱼群体中扩增到一条1 311 bp条带,而在雌核发育草鱼群体中未扩增到相应条带。
第二类特异DNA片段的出现违背了雌核发育过程中遗传物质缺失的普遍规律,笔者认为可能有以下两个方面的原因:第一,可能是“异精效应”结果[41],一般认为,雌核发育子代没有出现父本的遗传物质[42],但也有大量研究表明雌核发育子代中整合了部分父本遗传信息。邹桂伟等[43]采用44条RAPD随机引物对连续两代雌核发育鲢、雄鲤和普通鲢进行RAPD分析后发现,有10条引物出现了雌核发育鲢与父本(雄鲤)相同的DNA片段,而野生长江鲢(亲本鲢)则没有,表明雌核发育鲢基因组DNA可能带有父本基因组DNA的特性。张海发等[44]对异精激发的彭泽鲫雌核发育子一代及其双亲进行RAPD分析后发现,异育彭泽鲫子一代中含有少数与父本相同而与母本相异的特异条带,表明彭泽鲫已不再是完全的母性遗传,异育彭泽鲫基因组DNA中可能带有部分异源父本基因组DNA的特性。本研究中第二类特异DNA片段的出现是否为“异精效应”作用的结果还有待通过进一步研究其与父母本的遗传关系来证实。第二,第二类特异DNA片段的出现可能与雌核发育团头鲂的原始母本来源有关,G1群体的母本为团头鲂浦江1号选育系F6,G2群体由G1群体中的雌鱼诱导而来,G3群体由G2群体中的雌鱼诱导产生,因此,雌核发育团头鲂的母本为团头鲂浦江1号选育系F6的部分个体。而F9群体是以团头鲂浦江1号选育系F6为基础,再经连续3代人工选育而来,由此可知,F9群体与3个雌核发育群体(G1、G2、G3)有着相似的亲本来源,但在实际育种过程中,也可能因某些因素(如,繁育群体的非随机交配、遗传漂变等)的影响而导致基因重组进而产生新的遗传变异,但这还有待进一步的研究确认。

