非小细胞肺癌环状RNA表达谱的差异分析
张韶岩 曾小莉 郭琳 区颂雷 马旭晨
肺癌是临床最常见的恶性肿瘤之一,其发病率居常见恶性肿瘤的第2位,病死率居癌症死亡原因首位[1-2],其中非小细胞肺癌(non-small cell lung cancer, NSCLC)约占所有肺癌的80%,主要的病理类型是腺癌和鳞癌。探索NSCLC的生物学特征和发病机制,寻找诊断NSCLC的分子标志物是目前的研究热点[3-4]。环状RNA(circular RNA, circRNA)是广泛存在于哺乳动物细胞中的一种新的内源性非编码RNA分子,呈封闭环状,比线性RNA更稳定,具有一定的保守性、组织和疾病特异性[5]。研究发现circRNA的表达异常与肿瘤的发生发展、侵袭转移及放疗耐受有着密切联系,因此circRNA在肿瘤诊断和治疗方面具有广阔的应用前景[6-11]。但目前尚无NSCLC肿瘤组织中circRNA表达情况的相关研究。本研究应用circRNA芯片技术,对NSCLC组织与对应癌旁组织中差异表达的circRNA进行筛选,为进一步研究circRNA在NSCLC发生发展中的作用提供实验依据。
资料与方法
一、一般资料
收集2016年10至11月于首都医科大学附属北京安贞医院进行手术的NSCLC患者病理组织标本3例,其中男2例,女1例,年龄58~67岁,平均年龄63岁;根据病理诊断,肺腺癌2例,肺鳞癌1例;根据TNM分期,Ⅰ期1例、Ⅱ期1例、Ⅲ期1例。组织标本分别取自NSCLC原发肿瘤组织、距肿瘤边缘≥5 cm的癌旁组织,共6组取标本后立即置于液氮中速冻,然后转移到-80 ℃冻存。本研究符合本院伦理委员会的医学伦理要求。
二、纳入标准及排除标准
纳入标准:①年龄大于18岁;②经病理组织检查确诊为NSCLC者;③手术治疗前血常规、肝肾功能、心电图检查达治疗要求;④手术治疗前无严重合并症。排除标准:①无病理学诊断,TNM分期不详;②手术前接受过化疗或放疗;③存在其他肿瘤;④患者依从性差,不能配合研究者。
三、研究方法
1. 标本总RNA的提取: 按照TRIzol试剂盒(Invitrogen公司,美国)说明,分别提取3例NSCLC患者癌组织与对应癌旁组织中的总RNA,首先测定提取的RNA在260、280、230 nm处的吸光度值,计算浓度并估算纯度。然后采用甲醛变性琼脂糖凝胶电泳方法检测RNA的纯度及完整性。
2. circRNA芯片检测: ①cDNA合成和标记:首先使用RNaseR (Epicentre公司,美国)除去标本总RNA中的线性RNA,然后采用Super RNA Labeling(Arraystar公司,美国)方法对RNA进行cDNA扩增和荧光标记;② 芯片杂交:首先将标记后的样品与Human circRNA Array V2(8×15K)芯片(Arraystar公司,美国)进行排列杂交,然后用清洗液试剂盒(Agilent公司,美国)进行清洗。由上海康成生物公司提供circRNA芯片检测、图像采集、数据分析等。
三、数据分析
应用G2505C基因芯片扫描仪(Agilent公司,美国)扫描各标本Cy3荧光强度,然后采用Agilent Feature Extraction software (version 11.0.1.1)进行数据分析,并且使用R软件包对数据进行归一化处理。根据癌组织样本和癌旁组织样本间circRNA的变化倍数和P值筛选差异表达的circRNA,以变化倍数>2倍,配对t检验P值<0.05为阳性结果。对芯片筛选出的差异表达circRNA作进一步数据分析包括层次聚类分析、绘制散点图和火山图。通过TargetScan[12]和miRanda[13]两个软件对每种差异circRNA分别预测其最可能结合的miRNA,取匹配值较高的5个miRNA,并对这5个miRNA的结合位点进行预测和注释。
结 果
一、总RNA的质量分析
6组标本中提取的RNA的D(260 nm)/D(280 nm)比值在1.8~2.0之间,D(260 nm)/D(230 nm)>1.8。电泳图显示总RNA的28 s、18 s条带清晰,且28 s条带亮度是18 s的2倍以上,5 s 条带模糊。结果表明提取总RNA的质量与纯度符合要求,可用于下一步实验分析,见图1。

图1 组织样本的总RNA 电泳图;注:1~3: 非小细胞肺癌肿瘤组织;4~6: 癌旁组织
二、利用盒型图分析原始样本中circRNA表达情况
所有标本的芯片数据经过归一化处理后,使用盒型图可以直观了解到circRNA表达分布情况在6组标本中几乎是相同的,可进行下一步数据分析,见图2。

图2 3例非小细胞肺癌癌组织(C)与对应癌旁组织(A)中circRNA表达情况;注:C1~C3: 非小细胞肺癌组织;A1~A3: 癌旁组织
三、层次聚类分析
按照各组标本circRNA表达量的高低不同进行监督性层次聚类分析,从而将各组标本进行分类。图中红色表示circRNA表达量相对高,绿色表示circRNA表达量相对低,显示差异表达的circRNA能将癌组织与癌旁组织区分开来,见图3。
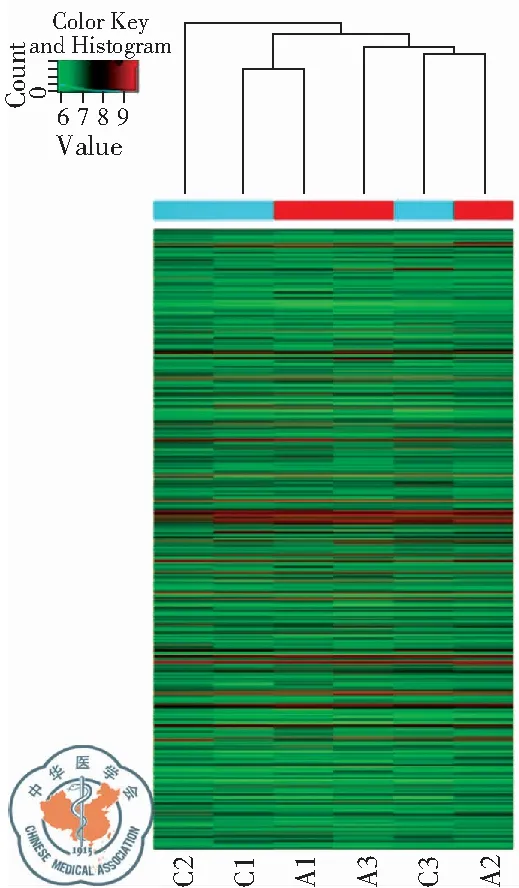
图3 3例非小细胞肺癌癌组织(C)与对应癌旁组织(A)差异表达的circRNA层次聚类分析;注:C1~C3: 非小细胞肺癌组织;A1~A3: 癌旁组织
四、差异表达的circRNA图形分析
对筛选出差异表达的circRNA进行图形分析,绘制散点图和火山图。散点图表示癌组织和癌旁组织circRNA总体分布集中趋势,图中分布于line1上方和line3下方的circRNA差异倍数达2倍以上,且有统计学意义(P<0.05),分别表示circRNA表达上调和表达下调的分布情况,见图4。

图4 非小细胞肺癌癌组织与癌旁组织差异表达的circRNA 绘制的散点图;注:线1(line 1)上方和线3(line 3)下方散点代表的circRNA差异倍数达2倍以上且P<0.05
采用火山图进行差异表达circRNA的筛选,图中红色区域代表circRNA的差异倍数达2倍以上,且有统计学意义(P<0.05),见图5。
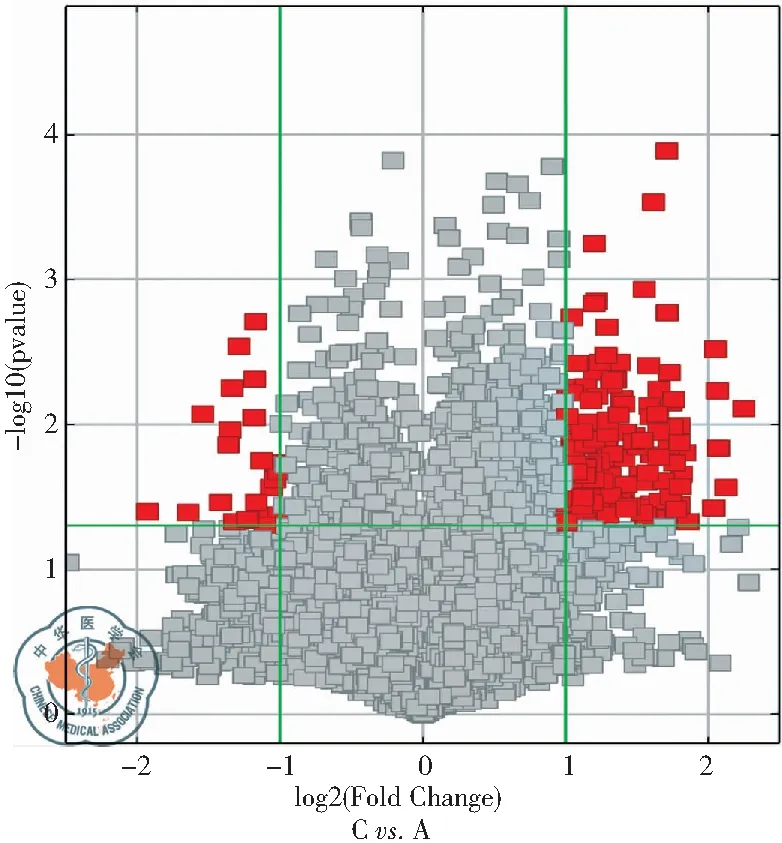
图5 非小细胞肺癌癌组织(C)与癌旁组织(A)差异表达的circRNA 绘制的火山图;注:红色区域代表的circRNA差异倍数达2倍以上且P<0.05
五、差异表达的circRNA
比较癌组织及癌旁组织中circRNA的变化倍数和P值,筛选出差异表达2倍以上且具有统计学意义的circRNA共171种,其中高表达的有148种,表达差异达3倍以上的37种;低表达的有23种,表达差异达3倍以上的有2种,见表1,表2。每种差异circRNA分别预测出其结合匹配值较高的5个miRNA,见表3。

表1 癌组织相对癌旁组织上调表达差异3倍以上的circRNA

表2 癌组织相对癌旁组织下调表达差异3倍以上的circRNA

表3 差异表达的circRNA 的miRNA 结合位点
讨 论
非编码RNA(non-coding RNA, ncRNA)是一类不具备编码蛋白功能的RNA转录本,包括微小RNA(microRNA, miRNA)、长链非编码RNA(long non-coding RNA, lncRNA)和circRNA等[14-16]。ncRNA曾经被认为是基因无用的噪音,然而近年来研究表明ncRNA在表观遗传学、转录调控及转录后调控中发挥重要作用[17],特别是在肿瘤的发生、发展、转移及预后等方面扮演重要角色[18]。Peng等[19]综述了目前与肺癌相关的16种lncRNA的功能及其作用机制,提示lncRNA有可能作为肺癌有效的生物学标志物,并提供新的治疗靶点。
circRNA是一种特殊的内源性ncRNA,随着高通量测序技术和生物信息学的快速发展, circRNA的研究已逐渐从零星到系统化,成为目前RNA研究的热点。circRNA能够作为miRNA海绵,通过与miRNA结合来阻碍miRNA与mRNA靶点结合,进而抑制miRNA功能[20],或者参与调节miRNA,降低miRNA的活性,从而在miRNA调控的疾病中发挥重要作用[21-22]。本研究对NSCLC筛选出的每种差异circRNA分别预测出其结合匹配值较高的5个miRNA,这些circRNA是否能通过结合相关miRNA的结合位点,从而影响NSCLC的发生发展尚有待进一步研究。
近年来国内外研究证实circRNA在人类肿瘤中表达异常,研究报道在喉癌中发现了698种表达量明显变化的circRNA[6],Sand等[23]在基底细胞癌中发现了23个高表达和48个低表达的circRNA。Li等[7]应用两个circRNA公共数据库资源筛选出目标circRNA(hsa-circ-002059),发现其在胃癌中异常低表达,与癌症远处转移情况、TMN分期、性别及年龄呈显著相关。Wang等[8]发现circRNA(hsa-circ-001988)在结肠癌组织中低表达,且表达水平与结肠癌细胞分化和嗜神经侵袭显著相关。Shang等[9]发现circRNA(hsa-circ-0005075)在肝癌组织中呈高表达,并与肿瘤大小显著相关。研究表明circRNA在食管癌中表达量异常[10-11],circRNA ITCH可通过抑制Wnt/β-catenin信号通路而对食管癌和肺癌有抑制效应[24-25]。但目前尚无NSCLC肿瘤组织中circRNA表达情况的研究报道。
本研究采用高通量circRNA芯片对NSCLC癌组织与癌旁组织的表达谱变化进行筛选,结果发现差异表达的circRNA共有171种,高表达的circRNA有148种,低表达的有23种,其中高表达差异3倍以上的37种,低表达差异3倍以上的有2种。初步分析差异表达的circRNA具有miR-21、miR-22、miR-29、miR-129、miR-143、miR-150和miR-200的潜在位点,大量研究证实这些miRNA在NSCLC发生发展中发挥重要调控作用。我们前期研究发现NSCLC患者外周单个核细胞中miR-143表达显著降低,miR-150在肺腺癌中表达量明显高于肺鳞癌,提示miR-143、miR-150与NSCLC相关,有可能作为鉴别诊断NSCLC的分子标志物[26]。本研究发现hsa_circRNA_100352在癌组织(相对于癌旁组织)中显著上调,hsa_circRNA_102062在癌组织(相对于癌旁组织)中显著下调,分别具有miR-143和miR-150的潜在位点,并且这两种circRNA都是外显子circRNA。本研究还对circRNA上的保守miRNA的结合位点进行预测分析,推测hsa_circRNA_100352和hsa_circRNA_102062可能通过与miR-143和miR-150结合来调控靶基因的表达,在NSCLC的发生发展中可能发挥重要作用。但是,由于本筛选研究标本数量较少,有其局限性,还需要大样本的临床验证研究和基础研究加以证实。进一步探讨目标circRNA与NSCLC病理类型、分期和转移的关系,深入研究目标circRNA参与NSCLC发生发展的过程及其作用机制,可为NSCLC的早期诊断、靶向治疗以及预后判断提供新的思路。
参 考 文 献
1 钱桂生. 肺癌不同病理类型发病率的变化情况及其原因[J/CD]. 中华肺部疾病杂志(电子版), 2011, 4(1): 1-5.
2 Siegel RL, Miller KD, Jemal A. Cancer statistics, 2016[J]. CA Cancer J Clin, 2016, 66(1): 7-30.
3 吕艳玲, 袁冬梅, 金淑贤, 等. 非小细胞肺癌患者癌胚抗原、C反应蛋白表达水平与预后的关系[J/CD]. 中华肺部疾病杂志(电子版), 2016, 9(2): 140-144.
4 张邵岩, 曾小莉, 区颂雷, 等. 肺癌患者血清多效蛋白表达水平及其临床意义[J/CD]. 中华肺部疾病杂志: 电子版, 2017, 10(3): 292-295.
5 Guo JU, Agarwal V, Guo H, et al. Expanded identification and characterization of mammalian circular RNAs[J]. Genome Biol, 2014, 15(7): 409.
6 Xuan L, Qu L, Zhou H, et al. Circular RNA: a novel biomarker for progressive laryngeal cancer[J]. Am J Transl Res, 2016, 8(2): 932-939.
7 Li P, Chen S, Chen H, et al. Using circular RNA as a novel type of biomarker in the screening of gastric cancer[J]. Clinica Chimica Acta, 2015, 444:132-136.
8 Wang X, Zhang Y, Huang L. et al. Decreased expression of hsa_circ_001988 in colorectal cancer and its clinical significances[J]. Int J Clin Exp Pathol, 2015, 8(12): 16020-16025.
9 Shang X, Li G, Liu H, et al. Comprehensive circular RNA profiling reveals that hsa_circ_0005075, new circular RNA biomarker, is involved in hepatocellular carcinoma development[J]. Medicine (Baltimore), 2016, 95(22): e3811.
10 Xia W, Qiu M, Chen R, et al. Circular RNA has_circ_0067934 is upregulated in esophageal squamous cellcarcinoma and promoted proliferation[J]. Sci Rep, 2016, 6: 35576.
11 Su H, Lin F, Deng X, et al. Profiling and bioinformatics analyses reveal differential circular RNA expression in radioresistant esophageal cancer cells[J]. J Transl Med, 2016, 14(1): 225.
12 Enright A, John B, Gaul U, et al. MicroRNA targets in drosophila[J]. Genome Biology, 2003, 5(1): R1.
13 Pasquinelli AE. MicroRNAs and their targets: recognition, regulation and an emerging reciprocal relationship[J]. Nat Rev Genet, 2012, 13(4): 271-282.
14 夏世金, 高文, 胡明冬, 等. 环状RNA的研究现状及展望[J/CD]. 中华肺部疾病杂志: 电子版, 2014, 7(6): 670-2673.
15 田芳, 王云, 肖哲, 等. 环状RNA CircHIPK3通过miR-379调控IGF1表达促进非小细胞肺癌细胞系 NCI-H1299与NCI-H2170的细胞增殖[J]. 中国肺癌杂志, 2017, 20(7): 459-467.
16 Memczak S, Jens M, Elefsinioti A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495(7441): 333-338.
17 Guttman M, Rinn JL. Modular regulatory principles of large non-coding RNAs[J]. Nature, 2012, 482(7385): 339-346.
18 Sugihara H, Ishimoto T, Miyake K, et al. Noncoding RNA expression aberration is associated with cancer progression and is a potential biomarker in esophageal squamous cell carcinoma[J]. Int J Mol Sci, 2015, 16(11): 27824-27834.
19 Peng Z, Zhang C, Duan C. Functions and mechanisms of long noncoding RNAs in lung cancer[J]. Onco Targets Ther, 2016, 9: 4411-4424.
20 Hansen TB, Jensen TI, Clausen BH, et al. Natural RNA circles function as efficient microRNA sponges[J]. Nature, 2013, 495(7441): 384-388.
21 Thomas LF, Strom P. Circular RNAs are depleted of polymorphisms at microRNA binding sites[J]. Bioinformatics, 2014, 30(16): 2243-2246.
22 Li J, Yang J, Zhou P, et al. Circular RNAs in cancer: novel insights into origins, properties, functions and implications[J]. Am J Cancer Res, 2015, 5(2): 472-480.
23 Sand M, Bechara FG, Sand D, et al. Circular RNA expression in basal cell carcinoma[J]. Epigenomics, 2016, 8(5): 619-632.
24 Li F, Zhang L, Li W, et al. Circular RNA ITCH has inhibitory effect on ESCC by suppressing the Wnt/β-catenin pathway[J]. Oncotarget, 2015, 6(8): 6001-6013.
25 Wan L, Zhang L, Fan K, et al. Circular RNA-ITCH suppresses lung cancer proliferation via inhibiting the Wnt/β-Catenin pathway[J]. Biomed Res Int, 2016, 2016: 1579490.
26 Zeng XL, Zhang SY, Zheng JF, et al. Altered miR-143 and miR-150 expressions in peripheral blood mononuclear cells for diagnosis of non-small cell lung cancer[J]. Chin Med J, 2013, 126(23): 4510-4516.

