云南白莺山地区茶树遗传多样性研究
毛娟,江鸿键,李崇兴,马建强*,陈亮*
云南白莺山地区茶树遗传多样性研究
毛娟1,江鸿键2,李崇兴3,马建强1*,陈亮1*
1. 中国农业科学院茶叶研究所,农业部茶树生物学与资源利用重点实验室,浙江 杭州 310008;2. 云南省临沧市人民政府茶叶办公室,云南 临沧 677000;3. 云南省临沧市茶叶科学研究所,云南 临沧 677000
云南省云县白莺山地区是大理茶()、阿萨姆茶(var.)及中间过渡形态茶树广泛分布的区域。本研究利用30个SSR核心标记分析了130份白莺山地区茶树种质资源的遗传多样性,共检测到202个等位基因,平均每个位点的等位基因数()为6.73,期望杂合度(E)为0.6135,近交系数()为-0.1745,多态信息含量(PIC)为0.5652,基因多样性()为0.6112。通过模拟不同样本数量,计算遗传多样性参数与样本量变化的回归曲线,发现样本量在40个时,能较好地反映白莺山茶树资源的遗传多样性。研究白莺山地区茶树的遗传多样性及取样策略,对茶树种质资源的保护与利用具有积极意义。
白莺山;茶树;SSR;遗传多样性
茶树()是最重要的饮料经济作物之一,广泛种植于全球52个国家和地区[1]。云南被认为是茶树种质资源类型和数量分布最多的地区[2-5],其临沧市北部的亚热带常绿阔叶林可能是大理茶()的起源中心[6]。白莺山位于云南省临沧市北部,毗邻澜沧江,古茶树数量多,利用历史悠久。经过长期的自然及人工选择,白莺山形成了大理茶、阿萨姆茶(var.)及中间过渡形态茶树混合分布的格局。
根据形态特征,当地居民将白莺山茶树资源划分为7个主要类群,包括本山茶、勐库种、白芽子茶、贺庆茶、藤子茶、二嘎子茶及黑条子茶[7]。目前,针对白莺山茶树资源的研究,大多是基于上述形态学分类,如陈文雄等[8]利用RAPD标记分析茶树类群间的遗传关系,推测黑条子茶和二嘎子茶可能是本山茶发生较大变异的产物,亦可能是本山茶与勐库种的F1代又经混交后繁衍数代的产物。赵东伟等[7]发现黑条子茶和二嘎子茶的形态特征与大苞茶一致,并且这两种茶树类群没有独立的分布区,总是与大理茶和茶相伴出现,且形态特征处于大理茶与茶的过渡状态,推测它们可能是大理茶与茶在长期栽培条件下自然杂交形成。吴华玲等[9]通过形态及叶片解剖结构研究,将12个茶树类群归为二大类四小类。由于生境差异和长期异交,完全依靠形态特征的茶树类群划分具有一定的局限性。
本研究利用30个SSR核心标记对白莺山地区茶树资源进行遗传多样性分析,并结合群体遗传结构分析,探讨了白莺山茶树资源遗传特性及其类群的划分,同时通过遗传多样性参数和样本量的回归分析,研究了最佳的采样策略,为以白莺山为代表的野生和地方品种茶树种质资源的保护和利用提供参考。
1 材料与方法
1.1 试验材料
于云南省云县白莺山村采集130份茶树资源:本山茶(在植物分类上属于典型的大理茶)53份,编号Y1-Y53;白芽子茶11份,编号Y54-Y64;二嘎子茶14份,编号Y65-Y78;贺庆茶5份,编号Y79-Y83;黑条子茶34份,编号Y84-Y117;勐库种(在植物分类上属于典型的阿萨姆茶)10份,编号Y118-Y127;藤子茶3份,编号Y128-Y130。采集幼嫩叶片,经液氮处理后放至-80℃备用。
1.2 DNA提取及SSR基因分型
采用改良的CTAB法[10]提取基因组DNA。30个SSR核心标记选自本实验室构建的遗传图谱,每个连锁群上各2个标记[11]。使用Applied Biosystems 9700 PCR System进行PCR扩增,扩增反应在25 μL反应体系中进行:12.5 μL KAPA 2G Robust Mix,上下游引物(10 mmol·L-1)各1 μL,1 μL DNA模板,9.5 μL ddH2O。扩增反应程序为:94℃预变性5 min;94℃变性30 s,各种Tm退火30 s,72°C延伸1 min,共30个循环,最后72℃延伸5 min。PCR产物使用3730xl DNA分析仪进行毛细管凝胶电泳检测和基因分型。
1.3 数据分析
分别使用PopGene 1.32[12]计算观测杂合度(e)、期望杂合度(o)和近交系数(),GenALEx 6.5[13]进行主坐标分析(PCoA),PowerMarker 3.25[14]计算等位基因数()、多态信息含量(PIC)、基因多样性指数()、样本间Nei′s遗传距离,并用邻接法(Neighbor-joining Tree, NJ)构建系统进化树;使用Strcture 2.3.4[15]进行遗传结构分析,Structure Harvester[16]确定最佳K值,CLUMPP[17]进行不同K值重复抽样分析,Distrcut[18]绘制图形。
参考Zhao等[19]对野生大豆的研究,分析遗传多样性参数与样本量的关系,分别从130份材料中随机抽取5、10、15、20、30、40、60、90个样本,每组随机抽样20次,计算每组抽样样本的遗传多样性参数平均值,及其占总样本量遗传多样性参数的百分比,并用SPSS20.0[20]对抽样样本遗传多样性参数百分比与样本量进行曲线拟合分析。
2 结果与分析
2.1 遗传多样性分析
30个SSR核心标记对130份茶树资源的遗传多样性分析结果如表1所示,其中共检测到202个等位基因(),每个位点的等位基因数为3~15个,平均等位基因数为6.73。观测杂合度(O)为0.1385~0.9154,平均值为0.5468;期望杂合度(E)为0.2459~0.8847,平均期望杂合度为0.6135,观测杂合度小于期望杂合度,说明在群体内存在非随机交配现象。近交系数()为-0.6436~0.6772,平均值为-0.1745,近交系数为负,表明白莺山茶树资源存在杂合子过剩的现象。多态信息含量为0.2259~0.8695,平均值为0.5652,其中大于0.5的高多态性位点有20个。基因多样性指数()为0.2449~0.8813,平均值为0.6112。
2.2 遗传聚类与遗传结构分析
基于Nei′s遗传距离构建系统进化树,结果如图1-a所示,130份材料可划分为类群Ⅰ和Ⅱ两个大类群,其中类群Ⅱ又可分为Ⅱ-1和Ⅱ-2两个小类群。使用不同颜色标注基于形态特征划分的7种茶树类群,发现勐库种(Y118-Y127)全部聚在类群Ⅰ,白芽子茶(Y54-Y64)和藤子茶(Y128-Y130)全部聚在类群Ⅱ-1,本山茶(Y1-Y53)全部聚在类群Ⅱ-2,二嘎子茶(Y65-Y78)、贺庆茶(Y79-Y83)和黑条子茶(Y84-Y117)主要聚在类群Ⅱ-1,并在其他类群中有零星分布。
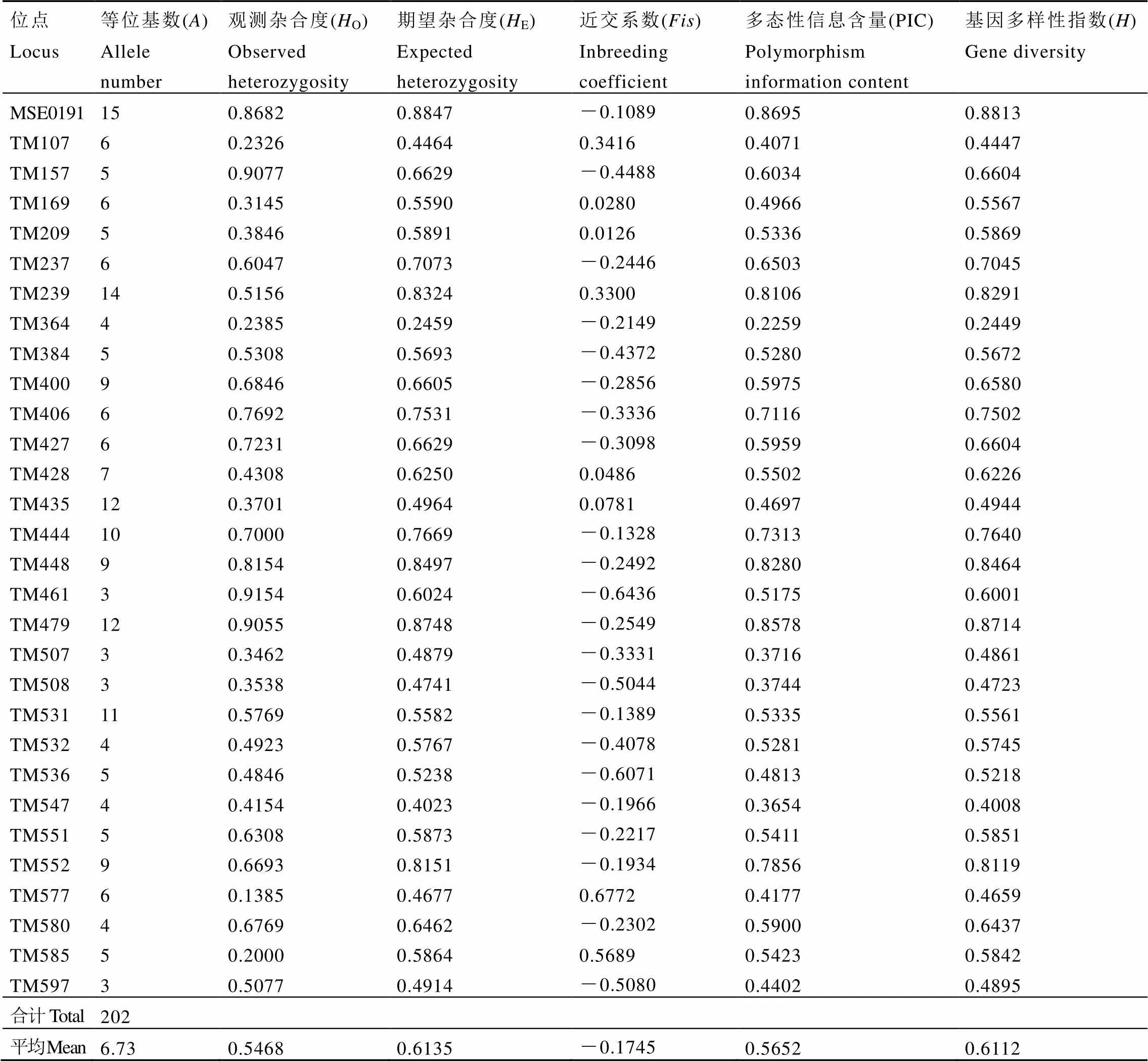
表1 基于30个SSR核心标记的白莺山茶树种质资源遗传多样性参数
主坐标分析显示,130份材料可划分为4个类群(图2),聚类结果与系统进化树基本一致,其中勐库种全部聚在类群A,白芽子茶全部聚在类群B,本山茶全部聚在类群D,黑条子茶和二嘎子茶主要聚在类群C,其次为类群D,此外黑条子茶在类群A和B中有零星分布,而藤子茶和贺庆茶较为分散,聚集在不同的类群中。
利用Structure进行群体遗传结构分析,结果表明当K=2时,ΔK具有最大峰值,130份材料被分为两个亚群(图1-b);当K=3时,ΔK也具有1个明显峰值,划分为3个亚群(图1-c)。无论K=2或K=3,本山茶均单独成为1个亚群,而藤子茶、二嘎子茶、黑条子茶和贺庆茶均表现为混合遗传结构;在K=3时,勐库种与白芽子茶被进一步细分,其中勐库种成为独立的亚群。
2.3 取样策略分析
对130份茶树样品进行随机抽样,抽样个数分别为5、10、15、20、30、40、60和90个,抽样样本的遗传多样性参数平均值见表2,遗传多样性参数百分比与样本量的回归拟合曲线如图3所示,当样本量达到40个时,全部曲线进入平台期。以等位基因总数(a)进行衡量,样本量为60个时,抽样样本遗传参数达到总样本的95%左右;以有效等位基因数(e)、香农指数()和期望杂合度(E)进行衡量,样本量为30个时,抽样样本遗传参数就可达到总样本的95%。综合分析,当取样量达到40个时,能较好地反映白莺山地区茶树资源的遗传多样性水平。
3 讨论
3.1 白莺山茶树群体遗传多样性总体评价
微卫星(SSR)被广泛应用于茶树[21-23]及其他作物[24-26]的遗传多样性研究,Taniguchi等[21]利用SSR标记分析来自14个国家和地区的788份茶树资源,发现来源于中国的茶树资源遗传多样性最高。Yao等[22]研究了450份野生茶树、地方品种和改良品种的遗传多样性,发现距离茶树起源中心越远,等位基因数、基因多样性和PIC等遗传参数逐渐变小,其中广西资源基因多样性最高(=0.636),其次为云南(=0.609)。王丽鸳等[23]对41份茶组植物进行分析,其中37份云南样本的基因多样性达到0.5319。刘本英等[27-28]、周萌等[29]对云南不同类型的茶树资源进行分析,结果显示PIC平均值最高达0.527。与这些建立在多个国家和地区的研究结果相比,一个县域范围内白莺山地区的茶树资源,具有相对较高的遗传多样性(=0.6112,PIC=0.5652)。
遗传聚类和主坐标分析结果表明,本山茶、勐库种和白芽子茶分别单独成为一个类群,其中白芽子茶类群具有最高的杂合度,以及最低的等位基因数、基因多样性指数、PIC和近交系数,其近交系数为-0.9296,本山茶为0.0280,勐库种为0.1117。近交系数为负,表明群内杂合子过剩,近交系数越大,表明群体内非随机交配程度越低[30]。张颖君等[31]比较分析了白莺山茶树资源的生化成分差异,发现白芽子茶含有大理茶的特征成分大理茶素,其含量仅次于本山茶(大理茶),是最接近野生大理茶的类群;同时白芽子茶的儿茶素组分、咖啡碱和茶氨酸含量介于本山茶与阿萨姆茶之间,且更接近于本山茶。吴华玲等[9]基于形态鉴定及叶片结构解剖,将白芽子茶、勐库大叶茶、勐库小叶茶、豆蔑茶归为一类,但白芽子茶的叶肉石细胞与勐库种有明显区别。因此,我们推测白芽子茶可能是本山茶与勐库种杂交后代分化出的一个类群,且分离时间较短,由于奠基者效应,表现出近交系数为负值的情况;另一种可能是白芽子茶是由本山茶与勐库种通过同倍体杂交物种形成机制而产生的新种[32-34]。

注:a:基于Nei’s 遗传距离构建的邻接系统进化树,b:基于Structure 的群体遗传结构(K=2),c:基于Structure 的群体遗传结构(K=3);BSC:本山茶,BYZ:白芽子茶,EGZ:二嘎子茶,HQC:贺庆茶,HTZ:黑条子茶,MKZ:勐库种,TZC:藤子茶。
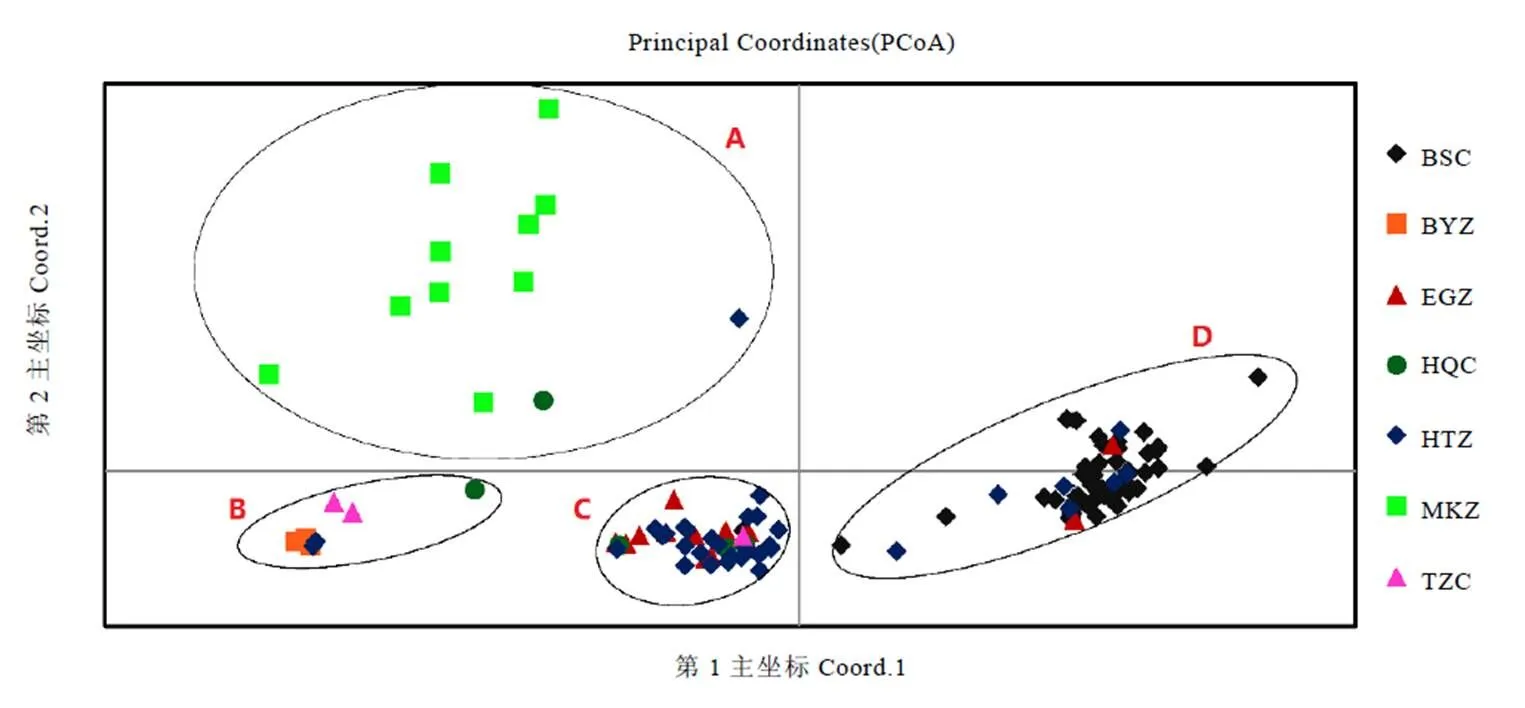
注:BSC:本山茶,BYZ:白芽子茶,EGZ:二嘎子茶,HQC:贺庆茶,HTZ:黑条子茶,MKZ:勐库种,TZC:藤子茶。
3.2 白莺山茶树根据形态特征划分类群的合理性
目前,茶树分类主要依据形态学特征[2-3,5]。白莺山当地居民根据综合形态特征对当地茶树资源进行了类群划分。本研究中基于SSR的遗传聚类分析发现,本山茶、勐库种和白芽子茶能很好地与其他茶树类群区分开,与依据形态特征划分的类群基本一致;二嘎子茶和黑条子茶大部分聚集在一个类群,并有少量个体分散到其他类群;而贺庆茶和藤子茶分布在不同的类群中,没有形成较为集中的聚集类群,其可能的原因是样本量相较其他类群偏少,也可能是其形态特征与其他类群相似难以区分。吴华玲等[9]在对白莺山茶树资源的表型性状调查中,将勐库种和白芽子茶都描述为小乔木、半开张、主干不明显、分枝低;而本研究通过遗传结构分析发现,它们分属于不同的亚群,因此仅依据形态学的描述,在对外形相似的茶树资源进行划分时可能会有误差。综上所述,白莺山当地居民对茶树资源的类群划分具有一定合理性,大部分能够被分子标记证据支持,但是仍有部分茶树的类群划分存在争议,需要进一步综合形态学、细胞生物学、基因组学等技术手段深入解析。
3.3 茶树遗传多样性保护与研究的合适样本量
遗传多样性被世界自然保护组织列为三大生物多样性之一[35]。对茶树而言,了解并保护其遗传多样性能够用来避免遗传育种中的近亲繁殖及遗传背景变窄等问题[36]。在遗传多样性研究与保护过程中,选择适合的样本量至关重要。目前,在收集和保护茶树地方品种等资源时,样本量一般为10~30个[6,37]。本研究结果表明,样本量对遗传多样性参数有不同程度的影响,当样本量达到40个,各遗传参数趋于稳定。王丽鸳等[38]对龙井群体的研究发现,当样本量达到15个,各遗传参数就能趋于稳定。相较白莺山茶树资源,龙井群体的遗传多样性较低,在样本量较少时就能较好地反映其遗传多样性。因此,在今后的茶树资源保护和研究中,针对遗传多样性较高的群体时,需要考虑扩大样本数量。
致谢:
临沧市人民政府茶叶办公室李文雄主任、卢锦春科长,临沧市云县茶叶办公室左成琳主任在资源收集过程中给予了莫大帮助,谨此致谢!
.表2不同抽样样本的遗传多样性参数比较
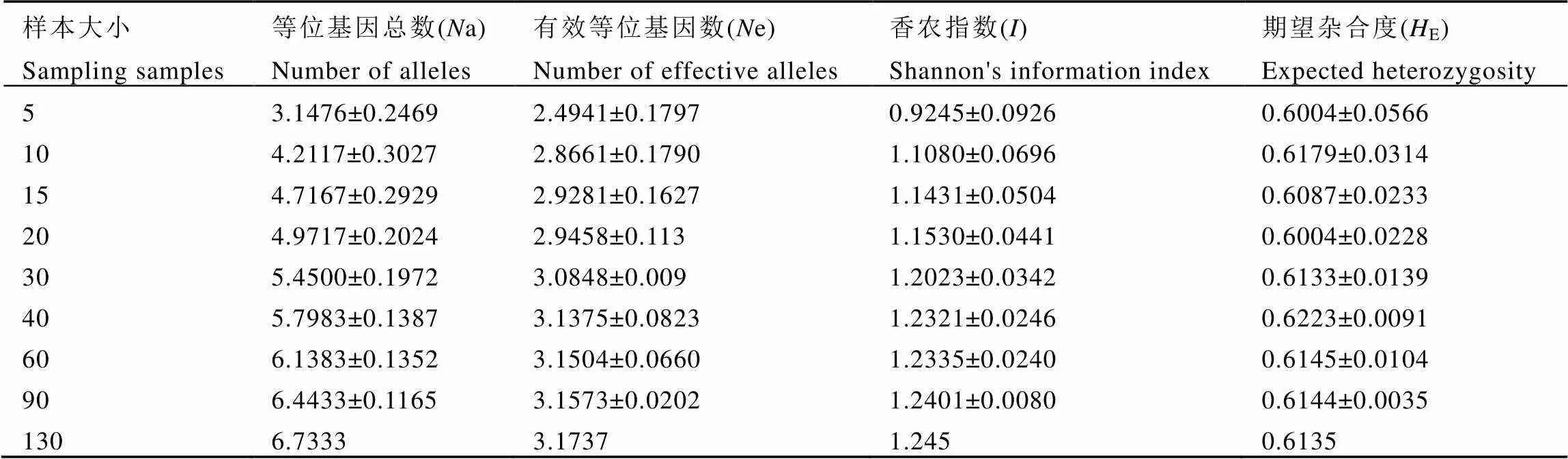
Table 2 Comparison of genetic parameters of tea populations with different sample sizes
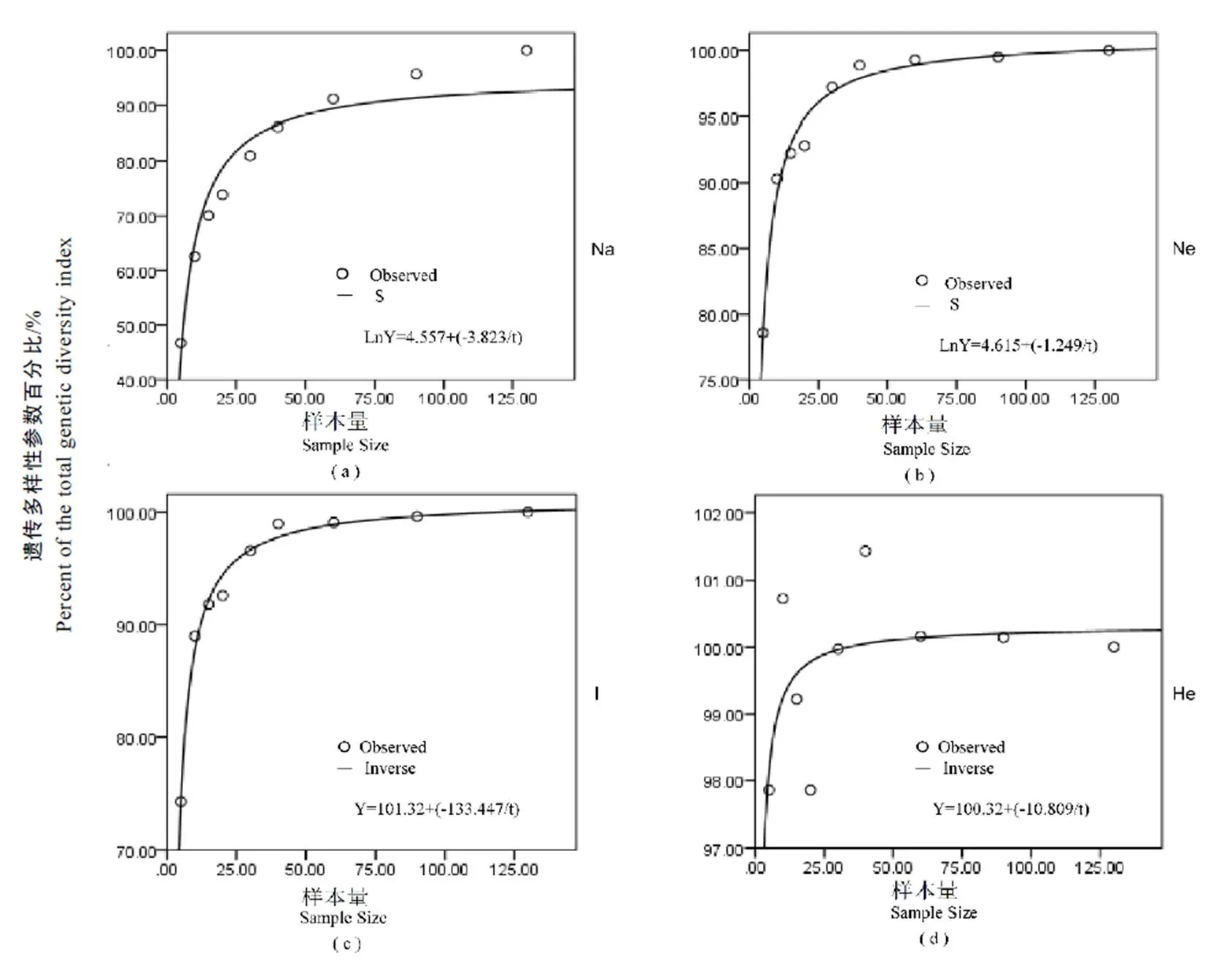
图3 遗传多样性参数百分比值随样本量变化的拟合回归曲线
[1] Chen L, Apostolides Z, Chen Z M. Global tea breeding: achievements, challenges and perspectives [M]. Springer-Zhejiang University Press, 2012: 1.
[2] 张宏达. 茶树的系统分类[J]. 中山大学学报(自然科学版), 1981(1): 87-99.
[3] 陈亮, 虞富莲, 童启庆. 关于茶组植物分类与演化的讨论[J]. 茶叶科学, 2000, 20(2): 89-94.
[4] 虞富莲. 论茶树原产地和起源中心[J]. 茶叶科学, 1986, 6(1): 1-8.
[5] 闵天禄, 张文驹. 山茶属植物的进化与分布[J]. 云南植物研究, 1996, 18(1): 1-13.
[6] Zhao D W, Yang J B, Yang S X, et al. Genetic diversity and domestication origin of tea plant(Theaceae) as revealed by microsatellite marker [J]. BMC Plant Biology, 2014, 14(1): 14.
[7] 赵东伟, 杨世雄. 山茶科大苞茶的再发现及形态特征修订[J].热带亚热带植物学报, 2012, 20(4): 399-402.
[8] 陈文雄, 季鹏章, 黄兴奇, 等. 本山茶与勐库茶疑似杂交后代的RAPD鉴定[J]. 北方园艺, 2008, 10: 153-155.
[9] 吴华玲, 李崇兴, 陈栋, 等. 基于形态鉴定及叶片解剖结构对云南白莺山古茶树资源的聚类分析[J]. 中国农学通报, 2014, 30(31): 114-121.
[10] Porebski S, Bailey L G, Baum B R. Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components [J]. Plant Molecular Biology Reporter, 1997, 15(1): 8-15.
[11] 黄丹娟. 我国茶树优良品种遗传多样性分析及指纹图谱构建[D]. 北京: 中国农业科学院, 2016: 10-28.
[12] Yeh F C, Boylet J B. Population genetic analysis of codominant and dominant markers and quantitative traits [J]. Belgian Journal of Botany, 1997, 129: 157-163.
[13] Peakall R, Smouse P E. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research [J]. Molecular Ecology Notes, 2006, 6(1): 288-295.
[14] Liu K, Muse S V. PowerMarker: an integrated analysis environment for genetic marker analysis [J]. Bioinformatics, 2005, 21(9): 2128-2129.
[15] Hubisz M J, Falush D S, Stephens M, et al. Inferring weak population structure with the assistance of sample group information [J]. Molecular Ecology Resources, 2009, 9(5): 1322-1332.
[16] Earl D A, VonHoldt B M. STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method [J]. Conservation Genetics Resources, 2012, 4(2): 359-361.
[17] Jakobsson M, Rosenberg N A. CLUMPP: a cluster matching and permutation program for dealing with label switching and multimodality in analysis of population structure [J]. Bioinformatics, 2007, 23(14): 1801-1806.
[18] Rosenberg N A. DISTRUCT: a program for the graphical display of population structure [J]. Molecular Ecology Resources, 2004, 4(1): 137-138.
[19] Zhao R, Cheng Z, Lu W F, et al. Estimating genetic diversity and sampling strategy for a wild soybean () population based on different molecular markers [J]. Chinese Science Bulletin, 2006, 51(10): 1219-1227.
[20] IBM Corp. Statistics for windows, version 20.0. Armonk, NY: IBM Corp, 2012.
[21] Taniguchi F, Kimura K, Saba T, et al. Worldwide core collections of tea () based on SSR markers [J]. Tree Genetics & Genomes, 2014, 6(10): 1555-1565.
[22] Yao M Z, Ma C L, Qiao T T, et al. Diversity distribution and population structure of tea germplasms in China revealed by EST-SSR markers [J]. Tree Genetics & Genomes, 2012, 8(1): 205-220.
[23] 王丽鸳, 刘本英, 姜燕华, 等. 用SSR分子标记研究茶组植物种间亲缘进化关系[J]. 茶叶科学, 2009, 29(5): 341-346.
[24] Emanuelli F, Lorenzi S, Grzeskowiak L, et al. Genetic diversity and population structure assessed by SSR and SNP markers in a large germplasm collection of grape [J]. BMC Plant Biology, 2013, 13(1): 39.
[25] Garris A J, Tai T H, Coburn J, et al. Genetic structure and diversity inL [J]. Genetics, 2005, 169(3): 1631-1638.
[26] Würschum T, Langer S M, Longin C F H, et al. Population structure, genetic diversity and linkage disequilibrium in elite winter wheat assessed with SNP and SSR markers [J]. Theoretical and Applied Genetics, 2013, 126(6): 1477-1486.
[27] 刘本英, 李友勇, 孙雪梅, 等. EST-SSR分析云南茶树资源的遗传多样性和亲缘关系[J]. 核农学报, 2010, 5: 956-967.
[28] 刘本英, 孙雪梅, 李友勇, 等. 基于EST-SSR标记的云南无性系茶树良种遗传多样性分析及指纹图谱构建[J]. 茶叶科学, 2012, 32(3): 261-268.
[29] 周萌, 李友勇, 孙雪梅, 等. 基于EST-SSR标记的云南大茶树遗传多样性分析[J]. 华北农学报, 2013, 28(S1): 91-96.
[30] 熊敏, 田双, 张志荣, 等. 华木莲居群遗传结构与保护单元[J]. 生物多样性, 2014, 22(4): 476-484.
[31] 张颖君, 杨崇仁, 曾恕芬, 等. 白莺山古茶的化学成分分析与栽培茶树的起源[J]. 云南植物研究, 2010, 32(1): 77-82.
[32] Schumer M, Rosenthal G G, Andolfatto P. How common is homoploid hybrid speciation? [J]. Evolution, 2014, 68(6): 1553-1560.
[33] Rieseberg L H, Raymond O, Rosenthal D M, et al. Major ecological transitions in wild sunflowers facilitated by hybridization [J]. Science, 2003, 301(5637): 1211-1216.
[34] Schumer M, Cui R, Rosenthal, G G, et al. Reproductive isolation of hybrid populations driven by genetic incompatibilities [J]. PLoS Genetics, 2015, 11(3): e1005041.
[35] McNeely J A, Miller K R, Reid W V, et al. Conserving the world's biological diversity [M]. Switzerland: International Union for Conservation of Nature and Natural Resources, 1990: 17.
[36] Yao M Z, Chen L, Liang Y R. Genetic diversity among tea cultivars from China, Japan and Kenya revealed by ISSR markers and its implication for parental selection in tea breeding programmes [J]. Plant Breeding, 2008, 127(2): 166-172.
[37] 季鹏章, 汪云刚, 蒋会兵, 等. 云南大理茶资源遗传多样性的AFLP分析[J]. 茶叶科学, 2009, 29(5): 329-335.
[38] 王丽鸳, 姜燕华, 段云裳, 等. 利用SSR分子标记分析茶树地方品种的遗传多样性[J]. 作物学报, 2010, 36(12): 2191-2195.
Genetic Diversity Analysis of Tea Plant in Baiyingshan Mountain of Yunnan
MAO Juan1, JIANG Hongjian2, LI Chongxing3, MA Jianqiang1*, CHEN Liang1*
1. Key Laboratory of Tea Plant Biology and Resources Utilization, Ministry of Agriculture, Tea Research Institute of the Chinese Academy of Agricultural Sciences, Hangzhou 310008, China; 2. Tea Administration Office of Lincang, Lincang 677000, China; 3. Tea Research Institute of Lincang, Lincang 677000, China
Tea germplasms includingvar., wild and semi-wild, and interspecific hybrid populations of the aforesaid species, are widely distributed in Baiyingshan Mountain of Yunnan Province. In this study, a core set of SSR markers were employed to assess the genetic diversity of tea germplasms derived from Baiyingshan Mountain. The results showed that a total of 202 alleles were detected, and the average number of allele () per SSR was 6.73, and 0.6135 for the average of expected heterozygosity (E), -0.1745 for the mean of inbreed coefficient (). The polymorphic information content (PIC) and gene diversity () were estimated to be 0.5652 and 0.6112 on average, respectively. The regression curves of genetic parameters influenced by population size showed that it could reach an optimized coverage of genetic diversity as the sample size was higher than 40. Investigation of the genetic diversity of tea germplasms in Baiyingshan Mountain was of vital importance for protection and utilization of these resources in the future.
Baiyingshan, tea plant, SSR, genetic diversity
S571.1;Q52
A
1000-369X(2018)01-069-09
2017-04-05
2017-08-15
国家自然科学基金(No.31500568)、国家茶叶产业技术体系(CARS-023)、中国农业科学院科技创新工程(CAAS-ASTIP-2017-TRICAAS)
毛娟,女,硕士研究生,主要从事茶树资源育种研究。*通讯作者

