基于SSR标记的白化和黄化茶树品种遗传多样性分析及指纹图谱构建
王松琳,马春雷,黄丹娟,马建强,金基强,姚明哲*,陈亮*
基于SSR标记的白化和黄化茶树品种遗传多样性分析及指纹图谱构建
王松琳1,2,马春雷1,2,黄丹娟1,马建强1,金基强1,姚明哲1*,陈亮1*
1. 中国农业科学院茶叶研究所国家茶树改良中心/农业部茶树生物学与资源利用重点实验室,浙江 杭州 310008;2. 中国农业科学院研究生院,北京 100081
利用62对SSR引物对16个白化、黄化茶树品种资源的遗传多样性进行了分析,初步明确了不同白化、黄化品种的遗传结构,以及SSR标记在白化、黄化品种鉴定上的适用性,为此类茶树品种资源的鉴定评价提供了理论依据。经过对引物筛选和扩增条带的分析,结果显示:具有多态性的55对引物中,共检测出169个等位基因,每对引物检测出的等位基因数为2~5个,平均为3.07个;多态信息含量(PIC)和Shannon信息指数(I)的平均值分别是0.40和0.79;169个等位基因出现频率在3.12%~96.88%之间;16个参试品种的遗传距离在0.086~0.532,品种间遗传结构差异明显;当D≈0.18时,可将16个品种划分为3类,其中大部分亲缘关系相近或地理位置一致的品种聚为一类。此外,笔者根据不同引物的等位基因带型构建了16个白化和黄化茶树品种的分子指纹图谱,并筛选出3对核心引物(TM156、TM508、MSG0380)用于不同白化、黄化茶树品种的鉴定。
白化;黄化茶树;SSR标记;指纹图谱
白化、黄化茶树是一类在特定条件下产生的具有不同程度叶绿素缺失的叶色突变体,属于茶树中的珍稀资源,目前在全国各地均有发现,如浙江的安吉白茶、景宁白茶、黄金芽,福建的白鸡冠、金冠茶,江西的黄金菊等。已有研究表明,这类茶树资源由于代谢机制的特异性,新梢芽叶的叶绿素合成减少,碳代谢受抑制,使其芽叶呈现出不同程度的白化或黄化现象,同时由于氮代谢增强,显著提高了游离氨基酸的生成量,使得有些品种资源的游离氨基酸含量能达到6%以上,茶氨酸含量达3%以上[1-4],以这类叶色突变体为原料制作的茶样,因具有更高的氨基酸含量和更优异的外形,使其经济价值更高,已成为各地茶农增收的重要途径[5-6]。但随着新育成白化、黄化茶树品种的增多,以及新品种较高的苗木及成品茶价格,很多不法商贩开始利用茶叶和茶苗品种不易鉴定的特点,随意对已有品种进行更名销售,导致市面上的此类茶树品种鱼龙混杂,“同物异名”和“同名异物”现象时有发生,严重损害了茶农和消费者的权益。因此,迫切需要建立一套快速、准确的技术体系用于不同白化、黄化茶树品种资源的鉴别,从而保证我国“白化和黄化茶”产业的健康发展。
基于形态特征的品种鉴定方法易受环境条件的影响,且要求鉴定者具有丰富的品种形态学鉴定经验,同时在实际应用中,往往因受到物候期的影响而无法及时准确地鉴定大量品种。随着分子生物学技术的不断发展,从DNA水平上对品种的遗传特异性进行快速、准确、不受环境影响的鉴定已成为可能。作为当今最先进的指纹鉴定技术,分子指纹图谱已发展出多种如RAPD(Random amplified polymorphic DNA)、AFLP(Amplified fragment length polymorphism)、SSR(Simple sequence repeat)和SNP(Single nucleotide polymorphism)等标记技术。其中SSR标记以其多态性好、扩增位点数量多、共显性遗传及操作简单等特点,在构建DNA指纹图谱中应用最为广泛[7-9]。2007年SSR标记和SNP标记一起被国际植物新品种保护组织(Union for the Protection of New Varieties of Plants,UPOV)推荐为构建DNA指纹数据库的标记方法。目前这一方法已广泛应用于小麦[10-11]、玉米[12-13]、水稻[14-15]等多种农作物的品种鉴定,同时在杨树、樱桃、银杏等木本植物[16-18]中也有大量应用。李莉等[19]利用36对SSR引物建立了大穗稻1126的DNA指纹数据库,可以有效解决杂交水稻亲本1126的真伪性及与其他品种的区分问题;王立新等[20]利用SSR标记建立了苹果栽培品种的DNA指纹图谱库,并进行了品种鉴定。同时SSR分子标记在茶树遗传多样性和构建指纹图谱上的研究亦有大量的报道,如乔小燕等[21]利用105对核心EST-SSR标记分析了广西、广东的105份茶树资源,剖析遗传结构并据此绘制了遗传结构图;黄丹娟等[22]从30对SSR引物中筛选出4对核心引物,构建了39个PVP申请茶树品种及近似品种的指纹图谱,并为每个参试品种建立了24位数的图谱二维码。
本研究利用62对SSR引物对16种不同白化、黄化茶树品种资源进行分子鉴定,通过选取最少的核心引物组合,构建SSR分子指纹图谱;同时通过聚类分析初步揭示不同参试茶树品种之间的遗传结构,以期为今后鉴定区别此类茶树品种提供支撑。
1 材料与方法
1.1 材料
供试的16种白化、黄化茶树品种,均采自国家种质杭州茶树圃(表1)。采摘各供试样品完整、无病虫害感染的一芽二叶新梢,液氮迅速冷冻后,于-80℃环境中保存备用。
1.2 方法
1.2.1 基因组DNA提取方法
将所有样品在液氮中磨成粉末,采用改良的CTAB法[23]提取基因组DNA,利用ND-1000微量紫外分光光度计测定DNA浓度和纯度,1%的琼脂糖凝胶电泳检测DNA的质量,后将DNA浓度稀释至工作浓度20 ng·μL-1,用于PCR扩增,剩余原液于-20℃保存备用。
1.2.2 PCR扩增及产物检测
从本实验室设计、筛选的SSR引物中选取62对多态性高、扩增条带清晰、重复性好的引物,用于供试材料的SSR-PCR分析。PCR反应在ABI公司的PCR扩增仪上进行,采用10 μL的PCR反应体系:ddH2O 6.2 μL、DNA 2 μL、Tap酶(2 U·μL-1)0.2 μL、dNTP(10 nmol·L-1)0.2 μL、10×PCR Buffer(Mg2+)1 μL,以及上下游Primer(10 μmol·L-1)各0.2 μL,PCR扩增程序为94℃预变性4 min;94℃变性30 s,不同温度下退火30 s,72℃延伸30 s,共35个循环,循环结束后72℃延伸7 min,4℃保存。扩增产物用10%的聚丙烯酰胺凝胶电泳检测,150 V电压下电泳80 min,银染法显色[24],之后用凝胶成像仪拍照并记录。

表1 供试茶树品种的名称及产地
1.2.3 数据处理及分析
数据处理采用人工读带的方法,将电泳图上目的片段范围内清晰的条带,按照分子量大小,从大到小按照A、B、C、D、E等依次记录,缺失条带以“XX”表示,记录每个参试品种的基因型。利用软件POPGENE[25]分析62对引物在16个参试品种中的扩增等位基因数(Observed number of alleles, Na)、有效等位基因(Effective number of alleles, Ne)、观测杂合度(Observed heterozygosity, HO)、期望杂合度(Expected heterozygosity, HE)、Shannon信息指数(Shannon′s information index, I)及等位基因出现频率(Allele frequency);利用软件PowerMarker[26]计算基因型数(Genotype)和多态性信息量(Polymorphism information content, PIC),之后计算16个品种间的Nei′s遗传距离[27],并构建Neighbor-Joining(NJ)系统进化树,进行聚类分析。最后从中挑选多态性含量高、扩增条带清晰的引物作为核心引物,根据其基因型转化为0和1代码,用于构建16个参试品种的DNA指纹图谱。
2 结果与分析
2.1 16个白化和黄化茶树品种的SSR多态性
观测62对引物的谱带,发现除7对引物(TM347、TM368、TM414、TM568、TM577、TM585、TM614)之外,剩余的55对引物扩增出的谱带均表现出多态性。分析这55对引物的扩增结果(表2),发现共扩增出169个等位基因(Na),平均每对引物扩增出3.07个,最多有5个,最少的只有2个;有效等位基因(Ne)的变异范围在1.06~3.61之间,平均值为2.06;共检测到基因型234个,范围在2~9个之间;引物的观测杂合度(HO)最大值为0.87(TM154),最小值为0(TM209);期望杂合度(HE)最大值为0.75(TM508),最小值为0.12(TM512、TM590、TM598、TM609);Shannon信息指数(I)的范围在0.14~1.39之间,平均值为0.79;而多态性信息含量(PIC)变化范围较大,在0.06~0.68之间,平均值为0.40,其中引物TM508的PIC值最高,而大于等于平均值的引物有31个,超过具有多态性引物总数的一半,这说明这些引物总体上多态性情况尚好,而多态性的高低往往与引物鉴别力有关,多态性越高,则鉴别力就越强[28]。
2.2 SSR标记的各等位基因出现频率
55对具多态性的引物共扩增得到169个等位基因,其中TM584等位基因B的出现频率最高,达到96.88%;而等位基因出现频率最低为3.12%,分别为TM237的等位基因C、TM377的等位基因C、TM384的等位基因A等15个等位基因上(图1)。由此可以看出16个参试茶树品种各等位基因出现频率差异很大。
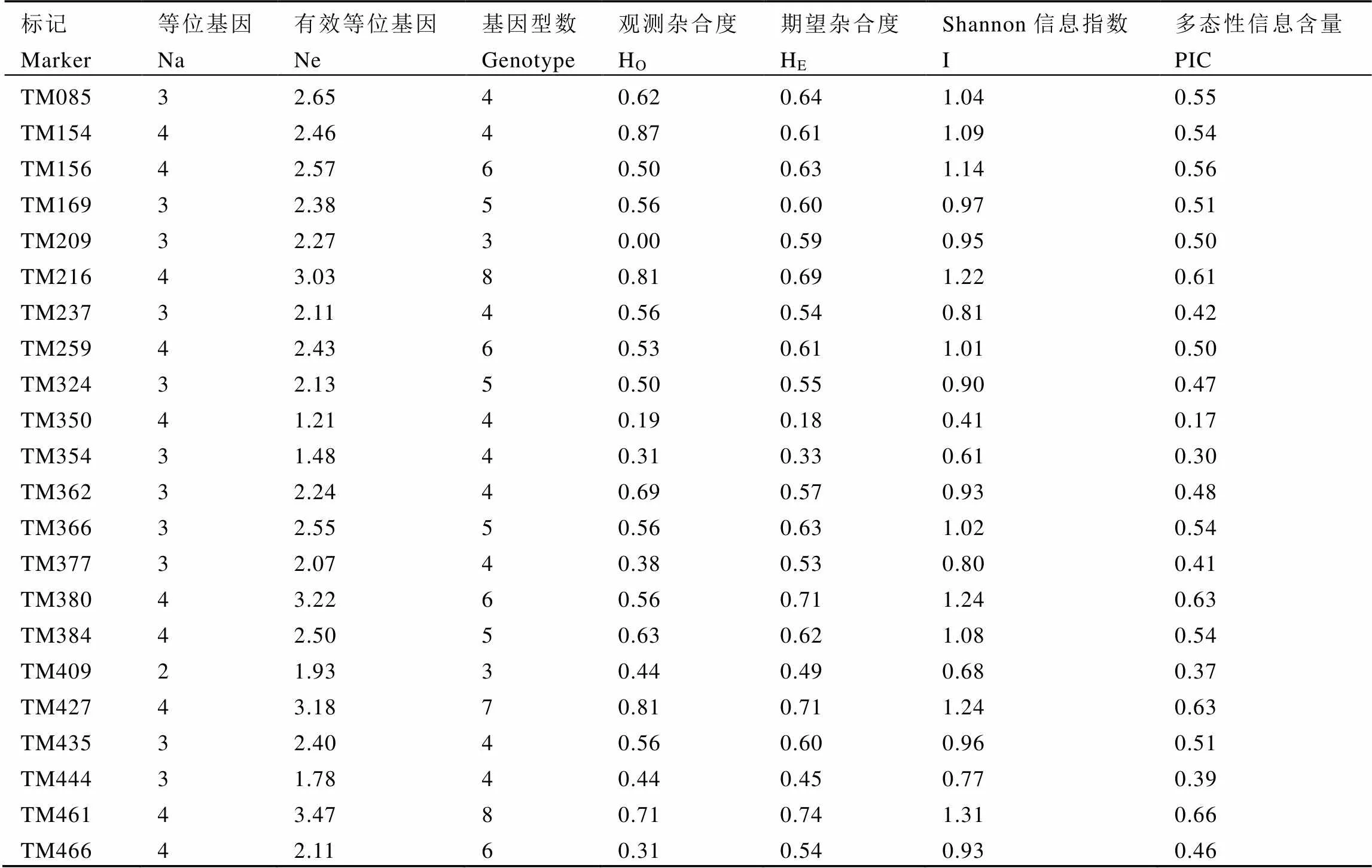
表2 16个参试茶树品种SSR标记的扩增结果
续表2
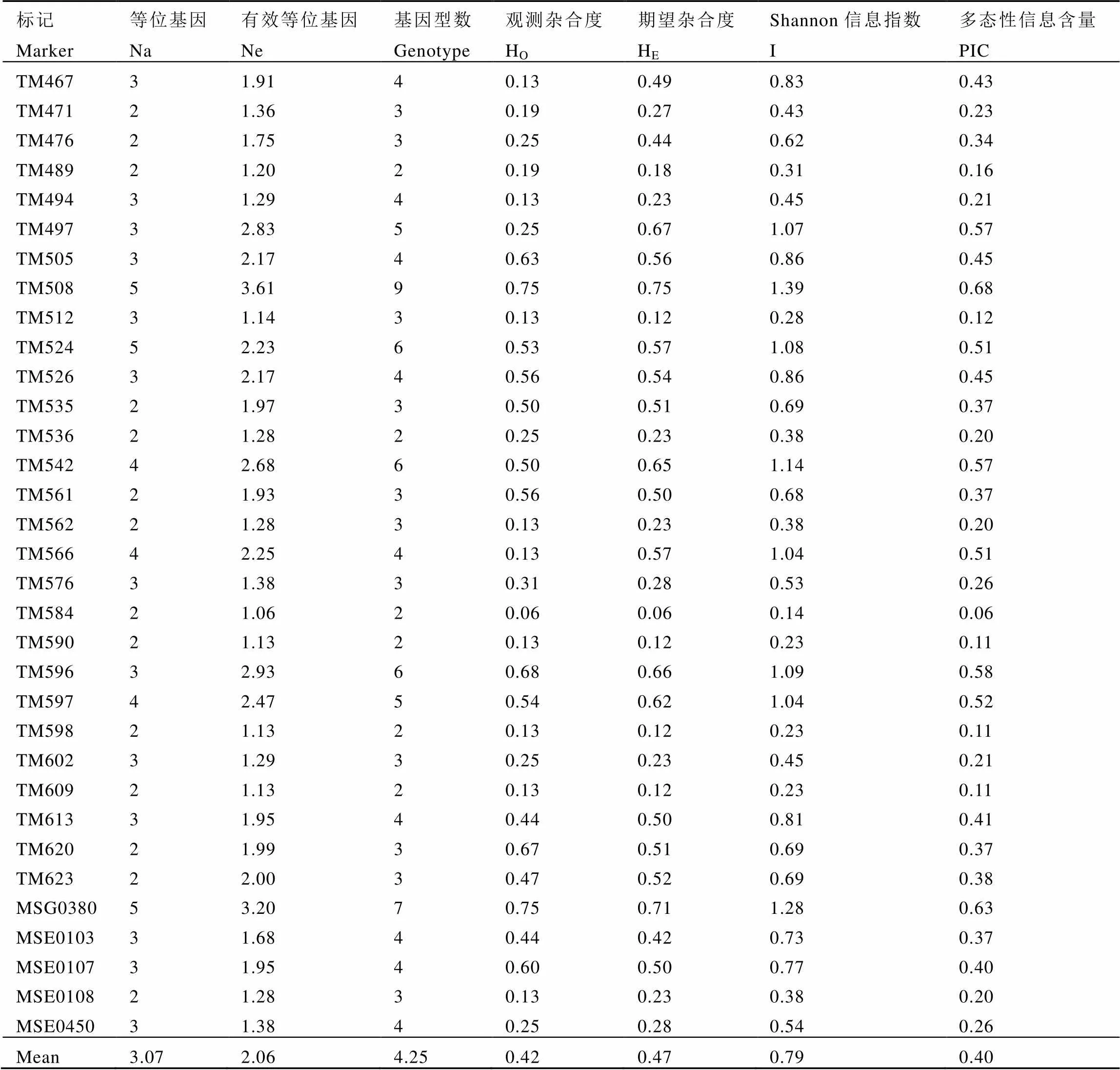
标记等位基因有效等位基因基因型数观测杂合度期望杂合度Shannon信息指数多态性信息含量 MarkerNaNeGenotypeHOHEIPIC TM467TM471TM476TM489TM494TM497TM505TM508TM512TM524TM526TM535TM536TM542TM561TM562TM566TM576TM584TM590TM596TM597TM598TM602TM609TM613TM620TM623MSG0380MSE0103MSE0107MSE0108MSE04503222333535322422432234232322533231.911.361.751.201.292.832.173.611.142.232.171.971.282.681.931.282.251.381.061.132.932.471.131.291.131.951.992.003.201.681.951.281.384332454936432633432265232433744340.130.190.250.190.130.250.630.750.130.530.560.500.250.500.560.130.130.310.060.130.680.540.130.250.130.440.670.470.750.440.600.130.250.490.270.440.180.230.670.560.750.120.570.540.510.230.650.500.230.570.280.060.120.660.620.120.230.120.500.510.520.710.420.500.230.280.830.430.620.310.451.070.861.390.281.080.860.690.381.140.680.381.040.530.140.231.091.040.230.450.230.810.690.691.280.730.770.380.540.430.230.340.160.210.570.450.680.120.510.450.370.200.570.370.200.510.260.060.110.580.520.110.210.110.410.370.380.630.370.400.200.26 Mean3.072.064.250.420.470.790.40
2.3 16个白化和黄化茶树品种亲缘关系分析
根据选取的55对SSR标记的等位基因出现频率,计算其Nei′s遗传距离(D),结果发现16个茶树品种的遗传距离在0.086~0.532之间,其中黄金芽与黄叶宝之间的遗传距离最大,四明雪芽和千年雪的遗传距离则最小(表3)。
基于Nei′s遗传距离,采用NJ法绘制16个品种的聚类图(图2)。从聚类图中可以看出,当D≈0.18时,16个品种基本聚为三大类,第Ⅰ类仅有2个品种,分别是来自江西的黄金菊和本课题组选育的株系花叶,它们属于黄化茶树资源,其黄化期较长,且春季新梢均表现出浅紫红色;第Ⅱ类包括御金香、天台白茶等10个品种,其中来自景宁的白茶品种景宁白茶1号和景宁白茶2号,以及来自浙江余姚的品种四明雪芽和千年雪都聚到了一起,但同样来自余姚的御金香和黄金芽则与它们表现出较远的遗传距离,说明地理来源相同的品种可能会因为具有相似的遗传背景聚集到一起,也可能因为亲本来源不同而表现出较大的遗传差异;第Ⅲ类则包括中黄2号、黄金芽、中黄1号和越乡白茶4个品种,它们分别是从浙江缙云、余姚、天台和嵊州当地群体种中发现的自然突变单株选育而来,聚类结果说明它们的遗传基础更相近。
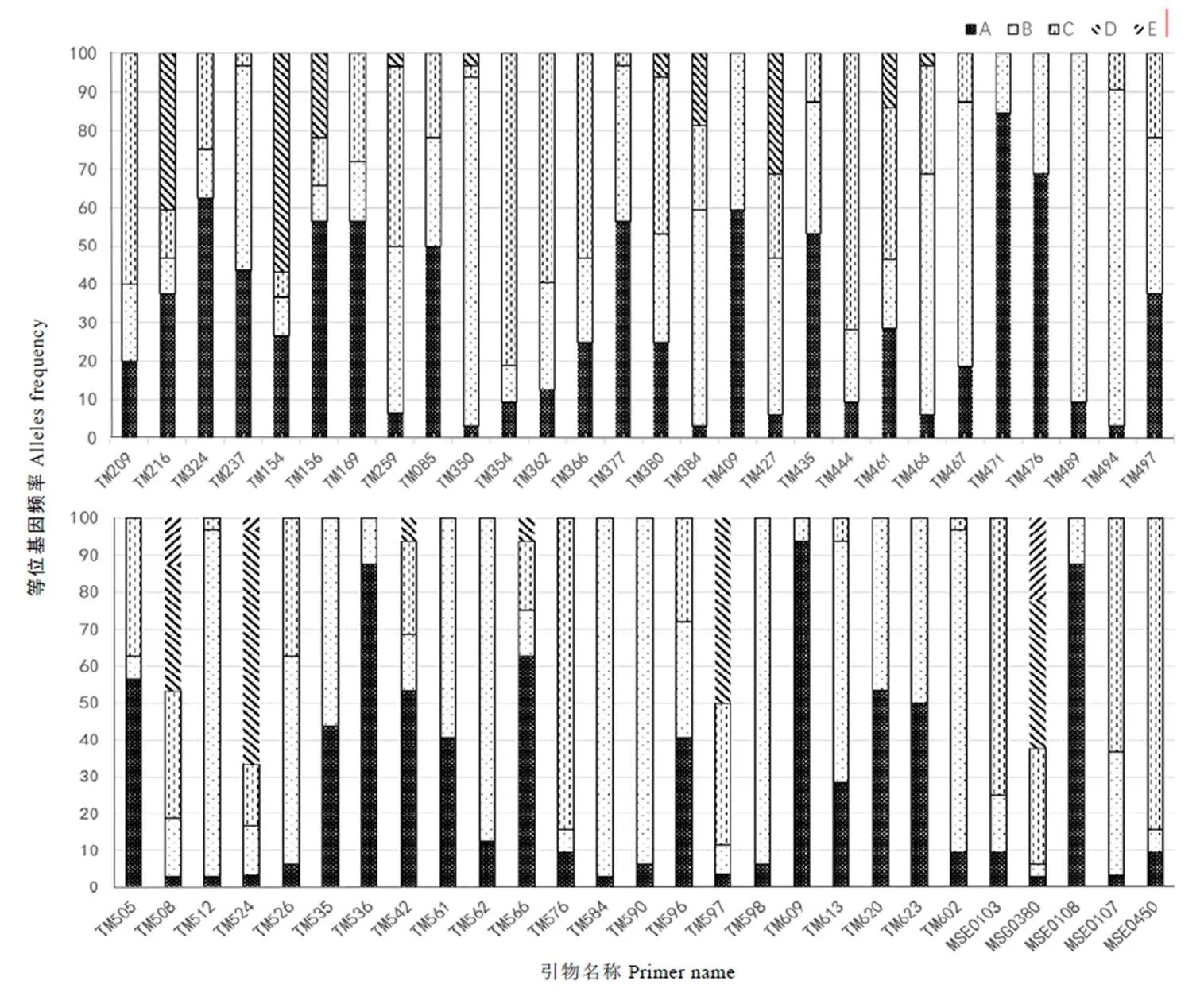
注:A~E 表示不同等位基因。Note: A-E present different alleles.
2.4 DNA指纹图谱的构建
分析筛选出的55对SSR引物扩增结果,参试的16个品种均具有特征谱带(表4)。从表中可以看出,具有特征引物的16个品种中,每个品种都至少有1对引物可以独立鉴别,最多的可达9对,利用特征引物可以对相应品种完成快速鉴定,但没有任何一对引物能够将这16个白化、黄化茶树品种全部区分开,这就需要通过引物相互组合的方式,利用2对或2对以上的引物进行区分。
综合考虑55对引物扩增条带清晰度、基因型数目及PIC值等因素,遵循以最少的引物鉴定全部品种的原则,筛选引物通过排列组合进行区分。本研究选取TM156、TM508、MSG0380 3对引物(图3),便可将参试的16个品种全部区分开,3对引物基因型数均≥6,PIC值≥0.54。之后3对引物按TM156、TM508、MSG0380的固定顺序,将其等位基因型转化0、1代码串联下来,作为16个茶树品种的指纹图谱,每个品种均得到1个14位数字的指纹图谱编码(表5)。
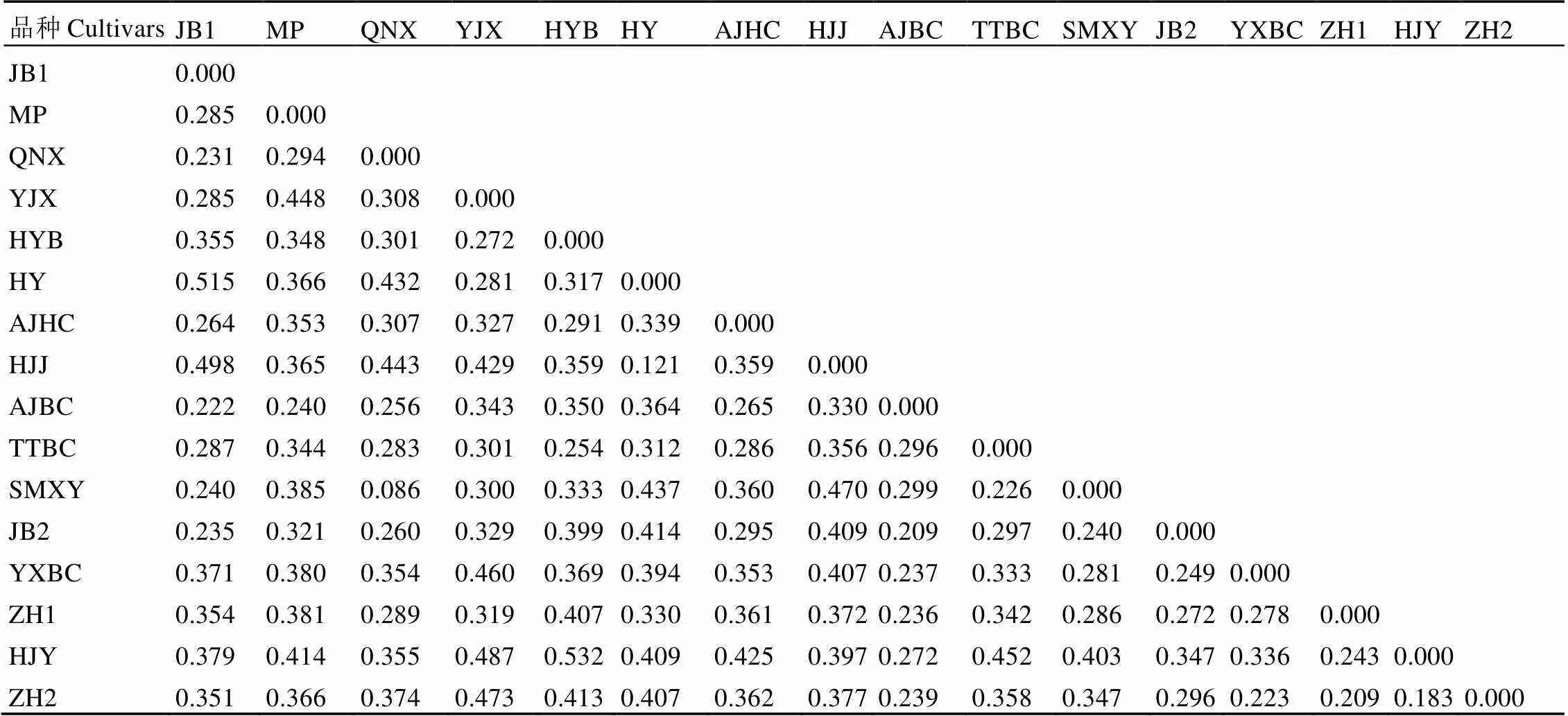
表3 16个参试茶树品种之间的遗传距离
注:JB1:景宁白茶1号,MP:MP1001,QNX:千年雪,YJX:御金香,HYB:黄叶宝,HY:花叶,AJHC:安吉黄茶,HJJ:黄金菊,AJBC:安吉白茶,TTBC:天台白茶,SMXY:四明雪芽,JB2:景宁白茶2号,YXBC:越乡白茶,ZH1:中黄1号,HJY:黄金芽,ZH2:中黄2号。
Note: JB2: Jingning Baich1, MP: MP1001, QNX: Qiannianxue, YJX: Yujinxiang, HYB: Huangyebao, HY: Huaye, AJHC: Anji Huangcha, HJJ: Huangjinju, AJBC: Anji Baicha, TTBC: Tiantai Baicha, SMXY: Siming Xueya, JB2: Jingning Baich2, YXBC: Yuexiang Baicha, ZH1: Zhonghuang1, HJY: Huangjinya, ZH2: Zhonghuang2.
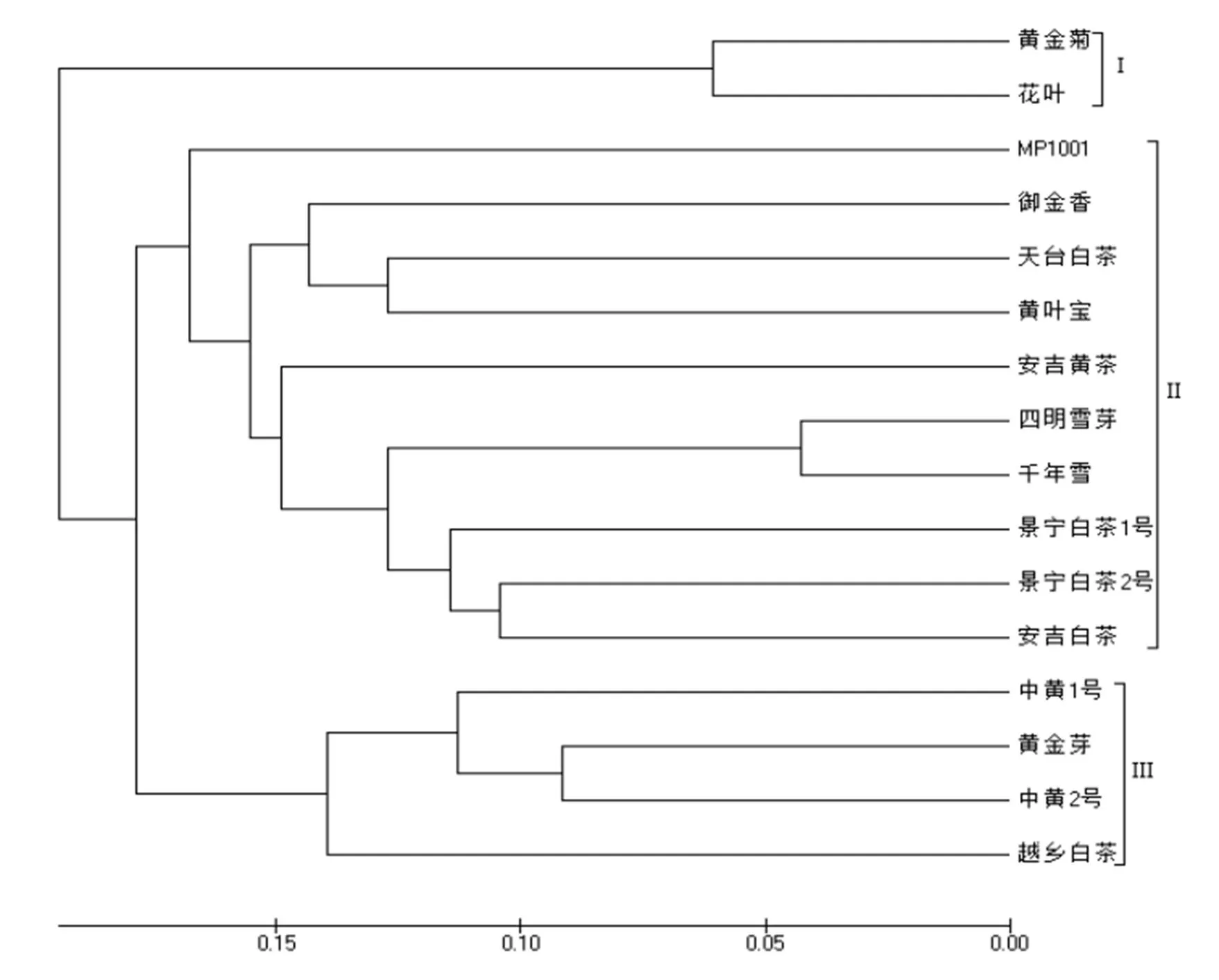
图2 16 个参试茶树品种Neighbor-Joining 聚类图
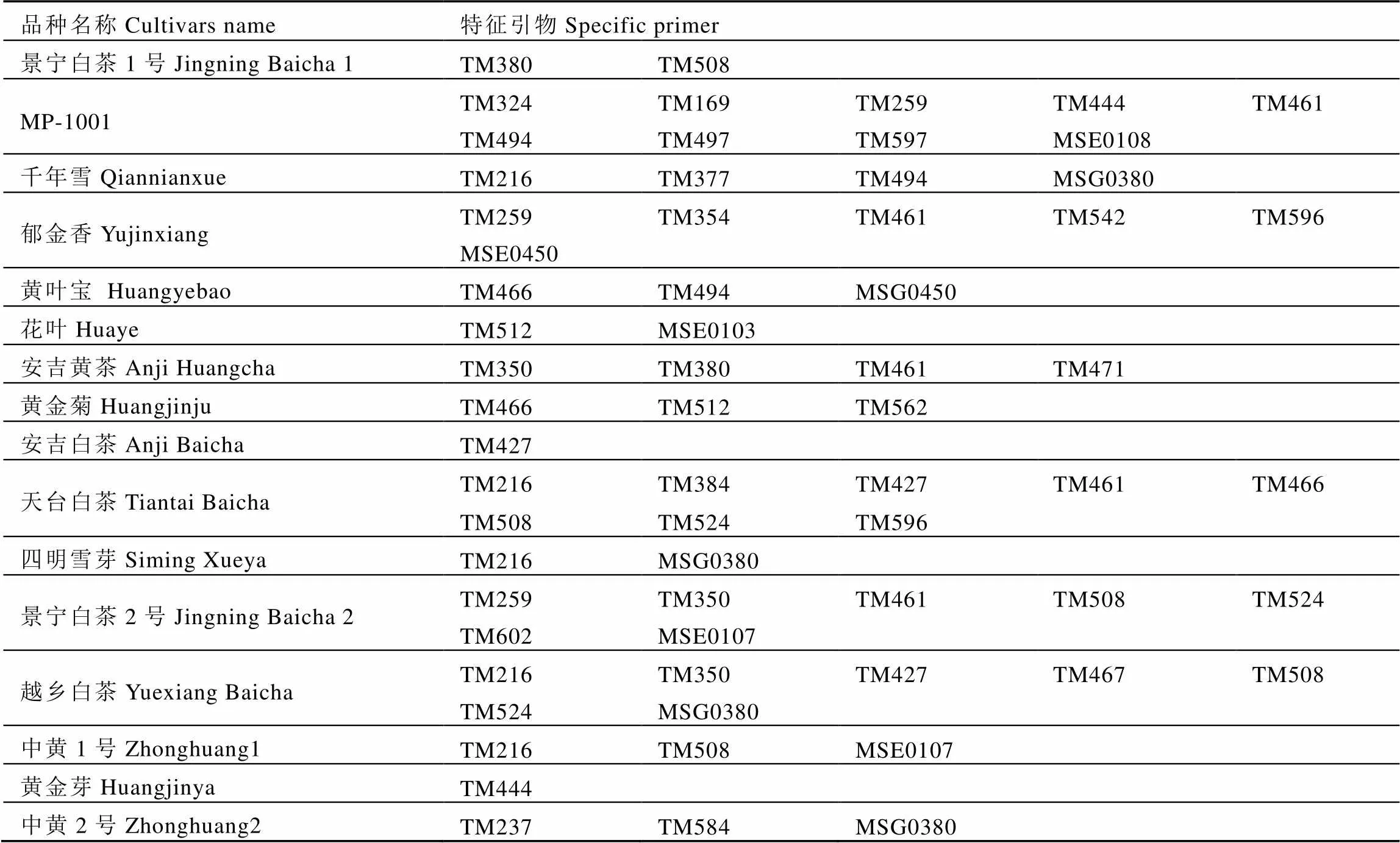
表4 16个参试茶树品种的特征引物
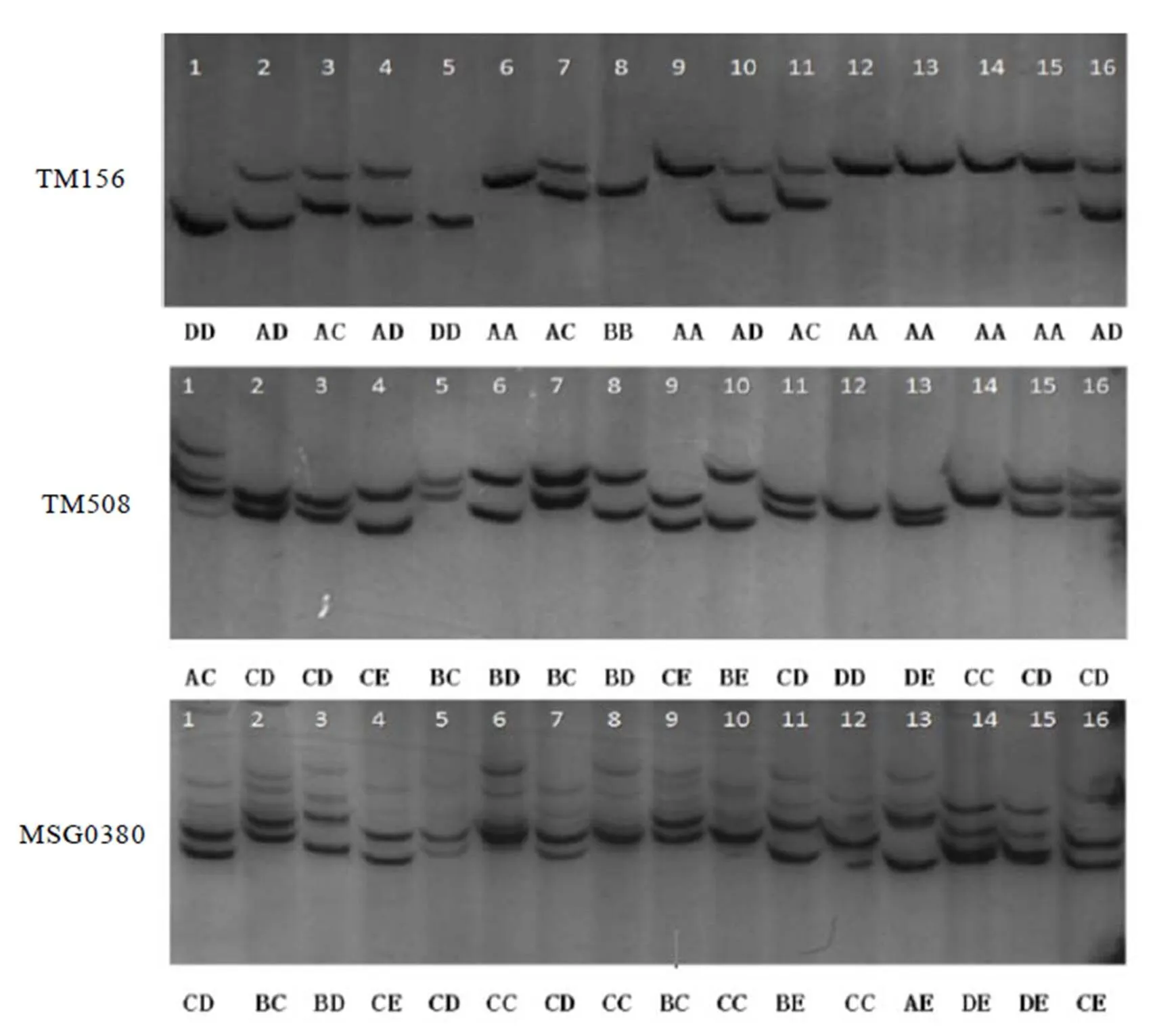
图3 3 对核心引物的扩增条带图
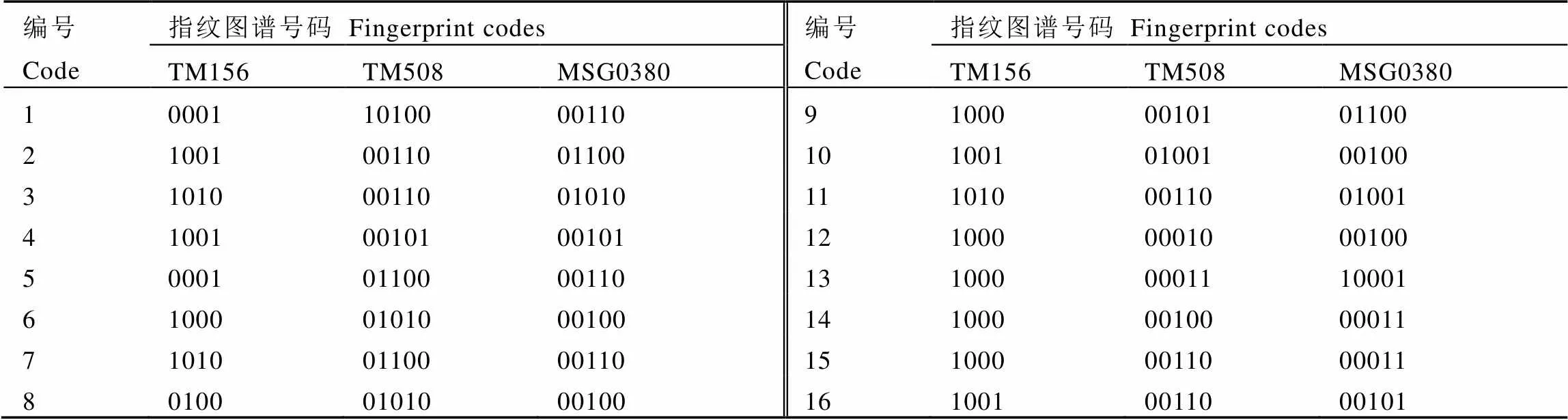
表5 16个茶树品种的分子指纹图谱号码
3 讨论
3.1 不同白化品种的遗传特性分析
茶产业的发展与新品种的培育和推广有着密不可分的关系,而茶树种质资源是培育优质茶树品种的基础,在茶树育种研究中具有非常重要的地位。我国作为茶树的原产地,茶树种质资源非常丰富,而其中紫化、黄化、白化等叶色变异资源因其较高含量的特征成分而备受关注。近十几年在全国各地陆续发现了十几种白化、黄化茶树资源,而浙江是目前白化茶树种质资源出现最为集中的区域,如目前推广面积较大的白叶1号(原安吉白茶)、黄金芽及中黄系列等[29-33]。这些资源大多源自群体种中的自然突变,因对其遗传背景不了解,导致很多资源无法作为骨干亲本快速地用于白化茶树品种的遗传改良[34-36]。
因此,本实验室从2013年开始,对保存在国家种质杭州茶树圃的十几份白化茶树资源的遗传特性进行了初步分析,发现目前这些资源的白化表型均不属于细胞质遗传,其中具有叶色遗传特性的品种有7个,分别为黄金芽、白鸡冠、安吉黄茶、中黄1号、中黄2号和安吉白茶,其中白鸡冠的遗传力最强,但因目前获得的实生种子和成活植株数量都有限,其他还有未被发现的具有遗传特性的黄化、白化资源也还有待进一步观察确定。
3.2 SSR引物的筛选
目前,构建指纹图谱往往遵循以尽可能少的引物区分全部参试品种这一原则,以求降低实验成本,减少繁重的实验工作,提高检测效率[37]。引物的筛选是利用SSR标记构建指纹图谱的重要组成部分,往往要求引物多态性PIC值高,稳定性好。
本研究最初选用62对引物对参试品种进行扩增,通过扩增谱带进行初步筛选,每个样品均能获得1~2条谱带,其中55对引物的扩增效果较为稳定,均具有多态性,且平均多态信息含量为0.4,其中有21对引物多态信息含量较高(PIC>0.5),多态性适中(0.2<PIC<0.5)的引物有26个,而多态性较差(PIC<0.2)的仅有8个。多态信息含量是选取核心引物构建指纹图谱的参考条件,本研究所选用的55对SSR引物整体水平良好,符合构建指纹图谱进而进行品种鉴定的要求。
3.3 不同白化和黄化茶树品种DNA指纹图谱的构建
目前建立DNA指纹图谱,主要采用的方法是核心引物组合法,这一方法并不需要进行大量的引物筛选工作,只需通过核心引物组合的方式就能将多个亲缘关系很近的品种区分开[38-40],既保证鉴别的准确度,又能提高鉴别效率,在其他作物研究中已有大量应用。目前茶树中利用核心引物组合法构建指纹图谱已有一定研究基础,章志芳等[41]采用EST-SSR标记对14个茶树品种进行遗传多样性分析和分子指纹鉴定,并从中选取4个核心标记构建各品种的指纹图谱;郭燕等[42]从20对SSR标记中筛选出4对核心标记,构建了40份贵州古茶树资源的指纹图谱,每份材料都获得1个18位的分子指纹图谱号码。本研究从55对引物中筛选出了3对核心引物,构建了16个白化茶树品种的分子指纹图谱,利用该图谱可对目前市场上主栽的白化茶树品种进行鉴别,研究结果对今后白化新品种的选育和保护具有重要的意义。
[1] Xu YX, Shen CJ, Ma JQ, et al. Quantitative succinyl-proteome profiling ofcv. ‘Anji Baicha’ during periodic albinism [J]. Scientific Reports, 2017, 7(1): 1873.
[2] Li CF, Yao MZ, Ma CL, et al. Differential metabolic profiles during the albescent stages of 'Anji Baicha' () [J]. PLoS One, 2015, 10(10): e0139996.
[3] Ma CL, Chen L, Wang XC, et al. Differential expression analysis of different albescent stages of ‘Anji Baicha’ ((L.) O. Kuntze) using cDNA microarray [J]. Scientia Horticulturae, 2012, 148(4): 246-254.
[4] Feng L, Gao MJ, Hou RY, et al. Determination of quality constituents in the young leaves of albino tea cultivars [J]. Food Chemistry, 2014, 155(11): 98-104.
[5] 卢翠, 沈程文. 茶树白化变异研究进展[J]. 茶叶科学, 2016, 36(5): 445-451.
[6] 马春雷, 姚明哲, 王新超, 等. 茶树叶绿素合成相关基因克隆及在白叶1号不同白化阶段的表达分析[J]. 作物学报, 2015, 41(2): 240-250.
[7] 梁明山, 曾宇, 周翔, 等. 遗传标记及其在作物品种鉴定中的作用[J]. 植物学报, 2001, 18(3): 257-265.
[8] 马建强, 姚明哲, 陈亮. 茶树种质资源研究进展[J]. 茶叶科学, 2015, 35(1): 11-16.
[9] 黄丹娟, 马建强, 陈亮. 茶树DNA分子指纹图谱研究进展[J]. 茶叶科学, 2015, 35(6): 513-519.
[10] 陈新民, 何中虎, 史建荣, 等. 利用SSR标记进行优质东小麦品种(系)的遗传多样性研究[J]. 作物学报, 2003, 29(1): 13-19.
[11] 程保山, 徐海风, 顾正中, 等. 淮安地区主栽小麦品种指纹图谱的构建及遗传多样性分析[J]. 浙江农业学报, 2011, 23(1): 20-24.
[12] 谭君, 丁仲芳, 孙仕贤, 等. 西南常用玉米自交系SSR指纹图谱构建[J]. 西南农业学报, 2003, 16(2): 1-6.
[13] 李丽华, 魏昕, 潘光堂. 20个骨干玉自交系的SSR指纹图谱构建[J]. 西南农业学报, 2007, 20(6): 1172-1175.
[14] 肖小余, 王玉平, 张建勇, 等. 四川省主要杂交稻亲本的SSR多态性分析和指纹图谱的构建与应用[J]. 中国水稻科学, 2006, 20(1): 1-7.
[15] 陆徐忠, 从夕汉, 刘海珍, 等. 杂交水稻亲本分子身份证及SSR指纹数据库的建立[J]. 核农学报, 2012, 26(6): 853-861.
[16] 藕丹, 樊军锋, 周永学, 等. 美洲黑杨×青杨派杂种无性系SSR指纹图谱的构建与遗传差异性分析[J]. 西北林学院学报, 2017(2): 112-116.
[17] 艾呈祥, 张力思, 魏海蓉, 等. 甜樱桃品种SSR指纹图谱数据库的建立[J]. 中国农学通报, 2007, 23(5): 55-58.
[18] 王星星, 周琦, 陶圆圆, 等. 48个果用银杏品种SSR指纹图谱构建与遗传多样性分析[J]. 分子植物育种, 2017, 15(5): 1963-1970.
[19] 李莉, 宋书锋, 刘全胜, 等. 利用SSR分子标记建立杂交水稻指纹图谱[J]. 长江大学学报(自然科学版):农学卷, 2008, 5(4): 67-70.
[20] 王立新, 张小军, 史星雲, 等. 苹果栽培品种SSR指纹图谱的构建[J]. 果树学报, 2012(6): 971-977.
[21] 乔小燕, 乔婷婷, 周艳华, 等. 基于EST-SSR的广东御广西茶树资源遗传结构和遗传分化比较分析[J]. 中国农业科学, 2011, 44(16): 3297-3311.
[22] 黄丹娟, 马建强, 陈亮. 茶树PVP申请品种的SSR分子标记鉴定和系谱关系分析[J]. 茶叶科学, 2016, 36(1): 68-76.
[23] Dellaporta S L, Wood J, Hicks J B. A plant DNA mini preparation: version II [J]. Plant Molecular Biology Reporter, 1983(1): 19-21.
[24] Charters Y M, Wilkinson M J. The use of self-pollinated progenies as ‘in-groups’ for the genetic characterization of cocoa germplasm [J]. Theoretical and Applied Genetics, 2000, 100(1): 160-166.
[25] Yeh F C, Yang R C, Boyle T B, et al. POPGENE, the user-friendly shareware for population genetic analysis [R]. Edmonton: Molecular Biology and Biotechnology Centre, University of Alberta, 1997: 112-118.
[26] Liu K, Muse S V. PowerMarker an integrated analysis environment for genetic marker analysis [J]. Bioinformatics, 2005, 21(9): 2128-2129.
[27] Takezaki H, Nei M. Genetic distances and reconstruction of phylogenetic trees from microsatellite DNA [J]. Genetics, 1996, 144(1): 389-399.
[28] 杨阳, 刘振, 赵洋, 等. 湖南省主要茶树品种分子指纹图谱的构建[J]. 茶叶科学, 2010, 30(5): 367-373.
[29] 张兰, 李鑫, 魏吉鹏, 等. 白叶一号的低温抗逆特性分析[J]. 茶叶科学, 2017, 37(1): 71-77.
[30] 王开荣, 李明, 梁月荣, 等. 茶树新品种黄金芽选育研究[J]. 中国茶叶, 2008, 30(4): 21-23.
[31] 夏功敏, 唐茜, 张淑娟, 等. 黄金芽等五个引进特色茶树品种春梢生化品质特性初探[J]. 西南农业学报, 2014, 27(3): 978-983.
[32] 黄茶育种协作组. 特异优质茶树新品种“中黄1号”选育研究[J]. 中国茶叶, 2014, 36(9): 8-9.
[33] 虞富莲. 中黄1号、中黄2号的特异性、一致性和稳定性[J]. 中国茶叶, 2016, 38(3): 14-16.
[34] 王开荣, 梁月荣, 李明, 等. 白化和紫花茶种质资源开发进展[J]. 中国茶叶加工, 2015(3): 5-8.
[35] 王开荣, 梁月荣, 张龙杰, 等. 白化茶种质资源的分类及特性[J]. 中国茶叶, 2008, 30(8): 9-11.
[36] 王开荣, 梁月荣, 李明, 等. 白化茶骨干亲本及其家系种质性状分析[J]. 茶叶, 2015, 41(3): 130-132.
[37] 刘新龙, 马丽, 陈学宽, 等. 云南甘蔗自育品种DNA指纹身份证构建[J]. 作物学报, 2010, 36(2): 202-210.
[38] 王让剑, 杨军, 孔祥瑞, 等. 福建15个茶树品种SSR遗传差异分析与指纹图谱建立[J]. 福建农业学报, 2014, 29(10): 970-975.
[39] Ujihara T, Ohta R, Hayashi N, et al. Identification of Japanese and Chinese green tea cultivars by using simple sequence repeat markers to encourage proper labeling [J] . Bioscience Biotechnology and Biochemistry, 2009, 73(1): 15-20.
[40] 张成才, 刘园, 姜燕华, 等. SSR标记鉴定浙江省主要无性系茶树品种的研究[J]. 植物遗传资源学报, 2014, 15(5): 926-931.
[41] 章志芳, 马建强. 基于SSR标记的茶树新品种遗传多样性分析及指纹图谱构建[J]. 湖南农业科学, 2012(19): 1-4.
[42] 郭燕, 刘声传, 曹雨, 等. 基于SSR标记贵州古茶树资源的遗传多样性分析及指纹图谱构建[J]. 西南农业学报, 2016, 29(3): 491-497.
Analysis of Genetic Diversity and Construction of DNA Fingerprints of Chlorophyll-deficient Tea Cultivars by SSR Markers
WANG Songlin1,2, MA Chunlei1,2, HUANG Danjuan1, MA Jianqiang1, JIN Jiqiang1,YAO Mingzhe1*, CHEN Liang1*
1. Tea Research Institute of the Chinese Academy of Agricultural Sciences, National Center for Tea Improvement, Key Laboratory of Tea Biology and Resource Utilization, Ministry of Agriculture, Hangzhou 310008, China; 2. Graduate School of Chinese Academy of Agriculture Science, Beijing 100081, China
To differentiate and identify different albino tea cultivars, sixty two SSR primers were used to analyze the genetic diversity of 16 tea cultivars that exhibit the chlorina phenotype. The result showed that a total of 169 alleles were amplified by 55 SSR primers with good polymorphism, and the number of alleles per primer ranged from 2 to 5, with an average of 3.07. The average value of polymorphism information content (PIC) and Shannon’s information index (I) was 0.40 and 0.79 respectively, which indicate these albino and yellow-leaf tea cultivars having a moderate level of diversity. And the occurrence frequency of 169 alleles ranged from 3.12% to 96.88%. It suggested the difference of genetic structure among tested varieties is obvious. The Nei′s genetic distance (D) of sixteen tested cultivars ranged from 0.086 to 0.532. These cultivars could be divided into three categories when D was 0.18, and the cultivars with close relatives or similar geographical origin were clustered into one group. Lastly, three primers (TM156, TM508, MSG0380) with easy identification, good stability and high polymorphism, were finally chosen to establish the fingerprint. The 16 albino and yellow-leaf tea cultivars could be effectively distinguished by the primers. This study provided an important basis for the identification of albino tea cultivars, and the evaluation and utilization of these germplasm resources.
albino tea cultivars, SSR marker, fingerprinting
S571.1;Q52
A
1000-369X(2018)01-058-11
2017-07-07
2017-08-21
中国农业科学院科技创新工程(CAAS-ASTIP-TRICAAS)、国家茶叶产业技术体系(CARS-23)、国家自然科学基金(31100504、31500568)、浙江省公益技术研究农业项目(2016C32024)、浙江省农业(茶树)新品种选育重大科技专项子课题(2016C02053-5)
王松琳,男,硕士研究生,主要从事茶树资源育种研究。*通讯作者

