Fe(II)对反硝化过程及其功能微生物群落的影响
李 爽,李晓敏,李芳柏* (1.中国科学院广州地球化学研究所,广东 广州 510640;.广东省生态环境技术研究所,广东 广州 510650;3.中国科学院大学,北京 100049)
氮元素是生物圈中最主要的基本组成元素之一,其中硝酸盐(NO3-)可以作为营养物质被植物和微生物吸收利用,还可以作为电子受体参与一些细菌、古菌甚至真核微生物的能量代谢和生长过程[1].在氮元素生物地球化学循环过程中,厌氧条件下微生物驱动的反硝化过程是其中一条比较普遍且重要的分支[2-3].微生物驱动的反硝化过程为:NO3-→NO2-→NO→N2O→N2.其中 nar基因编码的细胞膜硝酸盐还原酶和 nap基因编码的周质硝酸盐还原酶在 NO3-还原成亚硝酸盐(NO2-)的过程中起着关键作用;在 NO2-还原成为氧化氮(NO)的过程中,铜亚硝酸盐还原酶(nirK)和含细胞色素 cd1的亚硝酸还原酶(nirS)起着重要作用;norBC基因和nosZ基因编码的NO还原酶和N2O还原酶在NO和N2O的还原中起着重要作用[1,4].目前已有很多文献报道不同生境中反硝化微生物群落的研究,比如:酸性泥炭土中narG、nirK/nirS、nosZ-反硝化微生物丰度及群落组成[5];草地土壤环境中narG-硝酸盐还原微生物群落的组成[6];河流沉积物中nirS-亚硝酸盐还原微生物的群落特征[7].
铁(Fe)是地壳中含量第 4高的元素,也是红壤中重要的氧化还原元素[8-9].Fe的氧化还原转化对环境中许多元素转化起着至关重要的作用,比如:N 的生物地球化学循环[8,10],重金属和类金属钝化固定[11-12],有机污染物的脱毒和降解[13].由于人为活动在稻田环境中施加大量的化肥,导致稻田土壤中NO3-浓度增高,进而使稻田环境中微生物介导的NO3-还原过程活跃[14-15].目前关于厌氧稻田环境中 NO3-与亚铁(Fe(II))耦合过程的研究也有报道,主要集中在参与该过程的微生物群落组成,且研究表明Geothrix、Sunxiuqinia、Vulcanibacillus、 Azospira、 Zoogloea和Dechloromonas等是该过程的优势微生物[16-17],然而对于厌氧稻田中硝酸盐还原过程中的功能基因的含量以及功能微生物的研究较为缺乏.深入了解Fe(II)的存在对稻田环境中反硝化功能微生物丰度及其功能群落多样性组成的影响,有利于揭示Fe(II)对稻田环境中反硝化过程的影响机制,对调节稻田环境中 Fe-N循环具有重要意义.本研究以华南地区的水稻土作为研究对象,在实验室模拟中性厌氧培养条件下,设置了添加或者未添加Fe(II)/NO3-的不同处理,研究了其NO3-还原以及 Fe(II)氧化的动力学变化,并通过分子生物学技术分析了参与反硝化过程的功能基因的含量以及功能优势微生物群落结构与相对丰度的变化情况,进而探讨了 Fe(II)对稻田土壤中反硝化功能基因及其微生物群落的影响.
1 材料与方法
1.1 样品采集与理化分析
用于本实验的稻田土壤采集于广州市的中国科学院华南植物园水稻实验田(23°10'38.26"N, 113°21'10.12"E).采集时的水稻田处于淹水状态,土壤样品的采集采用四分法.采集的土壤样品除去其中的动植物残体等杂物,一部分放置于厌氧袋中于 4℃的冰箱中保存待用,一部分置于干燥通风阴凉处自然风干.风干后的样品通过研磨过筛(100目)收集,并利用同位素钴60Co释放出来的高能γ射线对土壤进行灭菌,灭菌后的土壤用于实验中灭菌对照实验.土壤理化特性的测定参考《土壤农业化学分析方法》[18],其中供试土壤pH值约为6.0,有机碳含量为 41.0g/kg,总铁含量约 27.9g/kg,总氮含量约258mg/kg.
1.2 实验方法
实验共设计了 5个处理组,包括灭菌非生物对照(Sterilized soil+Fe(II)+NO3-),生物对照组(Soil、Soil+NO3-、Soil+Fe(II))以及实验组(Soil+Fe(II)+NO3-).本实验采用 100mL西林瓶作为反应器,培养基包括:30mmol/L NaHCO3溶液作为缓冲溶液(通过通入 CO2气体后缓冲液的 pH值为 7.0),5mmol/L乙酸钠作为碳源,10mmol/L NaNO3,5mmol/L FeCl2,微量元素(1mL/L)和维生素溶液(1mL/L)为微生物提供生长所必须的营养物质.所有处理水土比为100:1(mL/g),通入 N2:CO2混合气(80:20)30min使体系中保持充分厌氧状态,然后快速加盖橡胶塞和铝盖,置于 30℃恒温培养箱中避光培养,在不同时间点进行取样.本实验采取破坏取样,每个处理的每个时间点设置 3个重复,西林瓶中的液体摇匀取出一定量的悬浊液用于 Fe的测定,然后再取西林瓶中的悬浊液用0.22μm 的水系滤膜过滤,滤液保存至-40℃用于NO3-、NO2-和 NH4+的测定.剩余的悬液经过离心,弃去上清液,将固体保存至-40℃用于DNA的提取.
1.3 分析方法
NO3-、NO2-和NH4+的测定采用流动分析仪(Skalar SAN++,荷兰)进行定量测定[19].由于还原所生成的 NO2-可以和 Fe(II)发生化学反应,因此取样后要将样品充分暴露于空气中,使体系中的 Fe(II)充分氧化,避免对 NO2-含量测定的影响.N2O 采用气相色谱(GC-7900,上海,天美)进行定量测定,用1mL的取样针抽取1mL西林瓶中的顶空气体注入进样口进行测定,采用的是ECD检测器,柱箱温度 50℃;ECD 检测器温度 250℃;进样口温度 200℃.溶解态和提取态亚铁的测定使用邻菲罗啉比色法[20].其中提取态亚铁用40mmol/L氨基磺酸(用HCl将pH值调至1.8)作为提取液[21].
1.4 微生物群落组成和多样性分析
土壤样品(湿样 0.25g)总 DNA 的提取采用MO BIO公司生产 PowerSoilTMDNA Isolation Kit试剂盒.提取的DNA保存-40℃待用.
1.4.1 微生物和功能基因的定量 微生物和功能基因的定量采用荧光染料 SYBR Green,用MyiQTM 2Optics Module荧光定量仪(BIORAD,USA)对总细菌(16S rRNA)、硝酸盐还原基因(narG、napA)、亚硝酸盐还原基因(nirS)、N2O还原基因(nosZ)进行定量测定.总细菌的定量分析用引物Eub338F/Eub518R进行扩增,反硝化功能基因的定量分别采用引物 NarG1960m2F/NarG2050m2R、NapAV17m/NapA4r、NirSCd3aF/NirSR3cd、NosZ2F/NosZ2R 进行扩增.PCR 扩增需要的引物以及扩增条件参照表1.

表1 定量PCR扩增引物序列及相应的扩增程序Table 1 Primers and programs for the PCR amplification of the real-time qPCR
1.4.2 高通量和克隆文库分析 高通量和克隆文库分析采用细菌通用引物 515F/806R对 16S rRNA基因的V4高变区进行扩增.亚硝酸盐还原基因和氧化亚氮还原基因的扩增分别采用引物NirSCd3aF/NirSR3d 和 NosZF/NosZ1622R.反向引物 806R、NirSR3d、NosZ1622R都带有识别不同样品的标签序列(barcode).扩增的PCR产物用纯化试剂盒(QIAquick Gel Extraction Kit)进行纯 化,纯 化 后 的 PCR 产 物 用 Qubit®3.0Fluorometer测定浓度后以等摩尔混匀,样品合格后在 Illumina MiSeq平台进行测序. PCR扩增的具体条件如表2所示.
完成高通量测序后,所有的原始序列都要质量控制,过滤去除低质量的序列.将高质量的序列通过特有的barcode标签分配到每个样品中,16S rRNA基因、nirS基因、nosZ基因分别按照97%、80%、82%的序列相似性进行OUT的分配挑选.使用QIIME和RDP将每个样品中的所有序列划分到不同的物种分类等级(门、纲、目、科、属、种),统计每个样品的群落结构组成和相对丰度.测试序列提交至 NCBI 数据库,分别获得 16S rRNA、nirS、nosZ基因序列登陆号为SRP109611、SRP108801、SRP108746.
由于硝酸盐还原基因(narG、napA)的基因片段较长,无法采高通量测序分析,所以采用克隆文库来分析硝酸盐还原功能微生物的组成.其所用引物和 PCR扩增的具体条件如表 2所示.其中Soil和 Soil+Fe(II)实验处理没有成功扩增出napA基因.通过蓝白斑筛选出阳性克隆子进行测序,并将测试序列提交至NCBI数据库,分别获取narG和napA序列登陆号为 MF626620-MF627322、MF626402-MF626619.目标序列采用 Mothur软件进行OTU聚类,并建立相应的克隆文库.

表2 高通量和克隆文库PCR扩增引物序列及相应的扩增程序Table 2 Primers and programs for the PCR amplification of the High-throughput sequencing and clone libraries
2 结果与讨论
2.1 硝酸盐还原及其产物生成
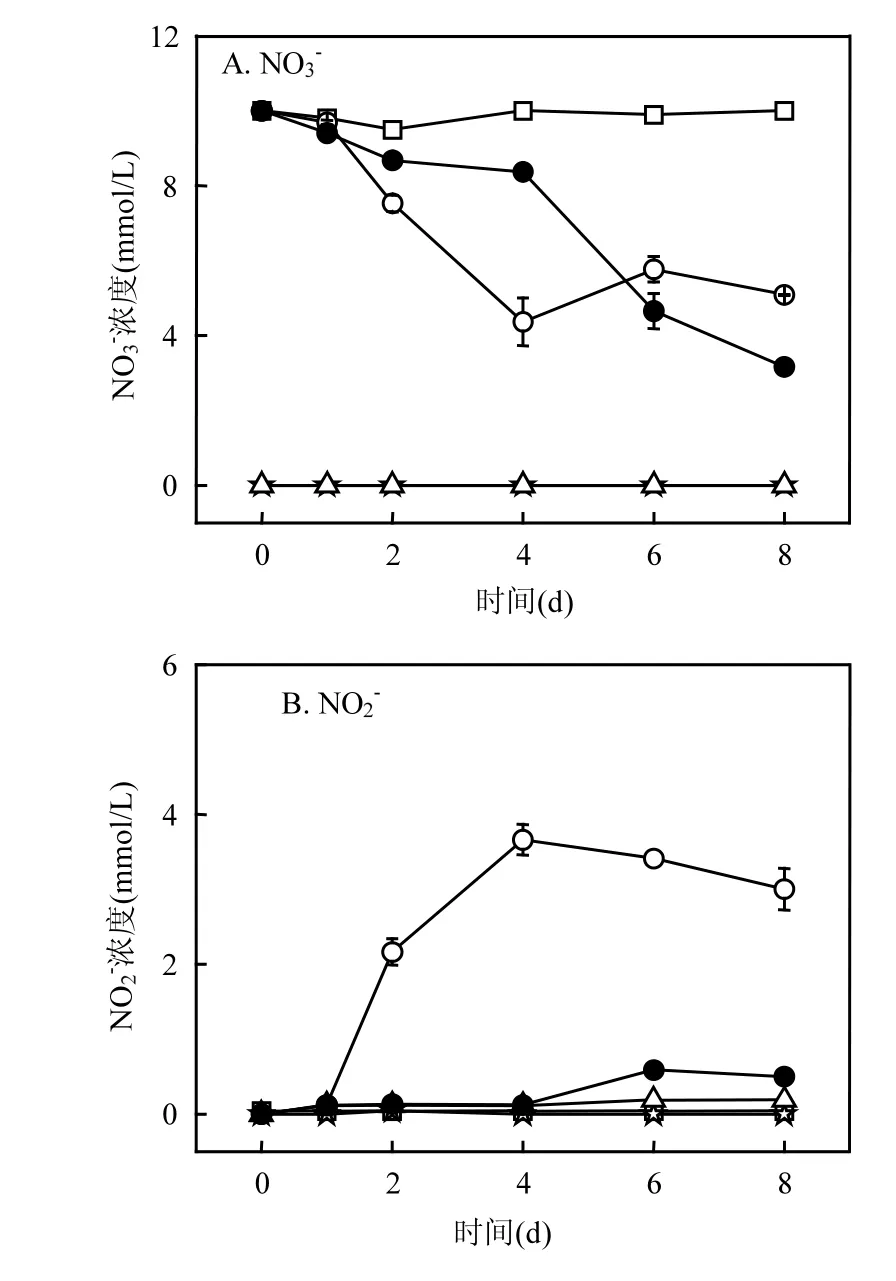
图1 不同处理中NO3-, NO2-和N2O浓度随反应时间的变化Fig.1 Changes of NO3-, NO2- and N2O concentrations with time dependence in different treatments
从NO3-的浓度变化(图1A)可见,在Soil+ NO3-和Soil+Fe(II)+NO3-处理中,NO3-的浓度随着时间变化逐渐降低;其中在 Fe(II)存在的条件下,的还原出现了延滞,在第 4d后才出现大量的还原.这可能是 Fe(II)的加入对土壤中的微生物有一定的毒害作用[30,32].而在Sterilized soil+Fe(II)+NO3-处理中,NO3-没有发生明显的还原,说明在 Soil+Fe(II)+NO3-处理中 NO3-的还原主要是由微生物介导的.微生物介导的完整的硝酸盐还原过程包括反硝化作用(NO3-→NO2-→ NO→N2O→N2)和异化硝酸盐还原成铵(NO3-→ NO2-→NH4+)[4].在所有实验处理中都未检测到 NH4+的存在,所以在本文所研究的稻田土壤中微生物驱动的 NO3-还原过程主要以反硝化过程为主.前人研究发现在稻田环境中即使存在异化硝酸盐还原成铵这个过程,与反硝化过程相比该过程也是较为缓慢的,并不起主导作用[17,32].在Soil+NO3-和Soil+Fe(II)+NO3-处理中,随着时间的变化均有NO2-生成,分别在第1d和第4d后NO2-才有明显的积累,经检测在NO3-还原过程中生成 NO2-的浓度分别高达 3.7,0.6mmol/L(图1B).在Soil+Fe(II)+NO3-处理中,NO2-浓度明显低于Soil+NO3-处理.造成这种现象的主要原因可能是在Fe(II)存在时,NO2-可以与Fe(II)发生快速的化学反应(其反应式为:2NO2-+4Fe2++5H2O→4FeOOH+N2O+6H+)[21],从而致使NO2-不能在体系中大量积累,这与之前的研究结果比较相似[16].培养2d后,在Soil+NO3-和 Soil+Fe(II)+ NO3-处理中都有N2O生成,并且Soil+Fe(II)+NO3-处理中N2O的增长幅度比Soil+NO3-处理要大,在第8d时它们的浓度分别为 0.3,0.6mmol/L(图 1C).而在对照处理 Soil和 Soil+Fe(II)中,均未测到 NO3-、NO2-和N2O的存在.
2.2 亚铁的氧化
图2显示,在Soil+Fe(II)处理中,溶解态Fe(II)和氨基磺酸提取态Fe(II)的浓度均没有发生明显变化,说明在厌氧条件下土壤中的 Fe(II)不容易发生氧化[33].在 Soil+Fe(II)+NO3-处理中,溶解态Fe(II)和氨基磺酸提取态Fe(II)的浓度在第4d后明显降低,在第8d时80%以上的Fe(II)被氧化;而在Sterilized soil+Fe(II)+NO3-处理中 Fe(II)也不发生氧化,说明微生物以及 NO3-对 Fe(II)的氧化起着重要的驱动作用[33].

图2 不同处理中溶解态Fe(II)和氨基磺酸提取态Fe(II)浓度随反应时间的变化Fig.2 Changes of dissolved Fe(II) and sulfamic acid-ext Fe(II) concentrations with time dependence in different treatments
2.3 16S rRNA和功能基因的丰度变化
从图 3A可知,在 Soil和 Soil+Fe(II)处理中,细菌16S rRNA基因拷贝数随时间变化并没有出现明显的增长;在 Soil+NO3-处理中,细菌 16S rRNA基因拷贝数从第1d后开始出现明显的增长;在Soil+Fe(II)+NO3-处理中,细菌16S rRNA基因拷贝数的增长延滞到第4d后才开始;在Soil+NO3-和Soil+Fe(II)+NO3-处理中,细菌 16S rRNA基因拷贝数在第 8d时增长了约一个数量级,从1010copies/g soil增长到1011copies/g soil.
如图 3B 所示,在 Soil、Soil+Fe(II)和 Soil+Fe(II)+NO3-处理中,细胞膜硝酸盐还原基因narG的拷贝数随时间变化没有明显的变化;而在Soil+NO3-处理中narG基因的拷贝数在第一天后随时间变化出现明显增长,从第 1d的106copies/g soil增长到第8d的107copies/g soil.如图3C所示,在Soil和Soil+Fe(II)处理中,周质硝酸盐还原基因napA拷贝数随时间变化没有明显的变化;在Soil+NO3-处理中,napA拷贝数从第1d后开始显著增加;在Soil+Fe(II)+NO3-处理中,napA拷贝数从第4d后才开始显著增加;第8d时,napA拷贝数在Soil+和Soil+Fe(II)+处理中均达到 107copies/g soil.由此可见,在Soil+NO3-处理中, NO3-还原主要发生在细胞膜和周质中;而Fe(II)存在的条件下,NO3-还原主要发生在周质中.
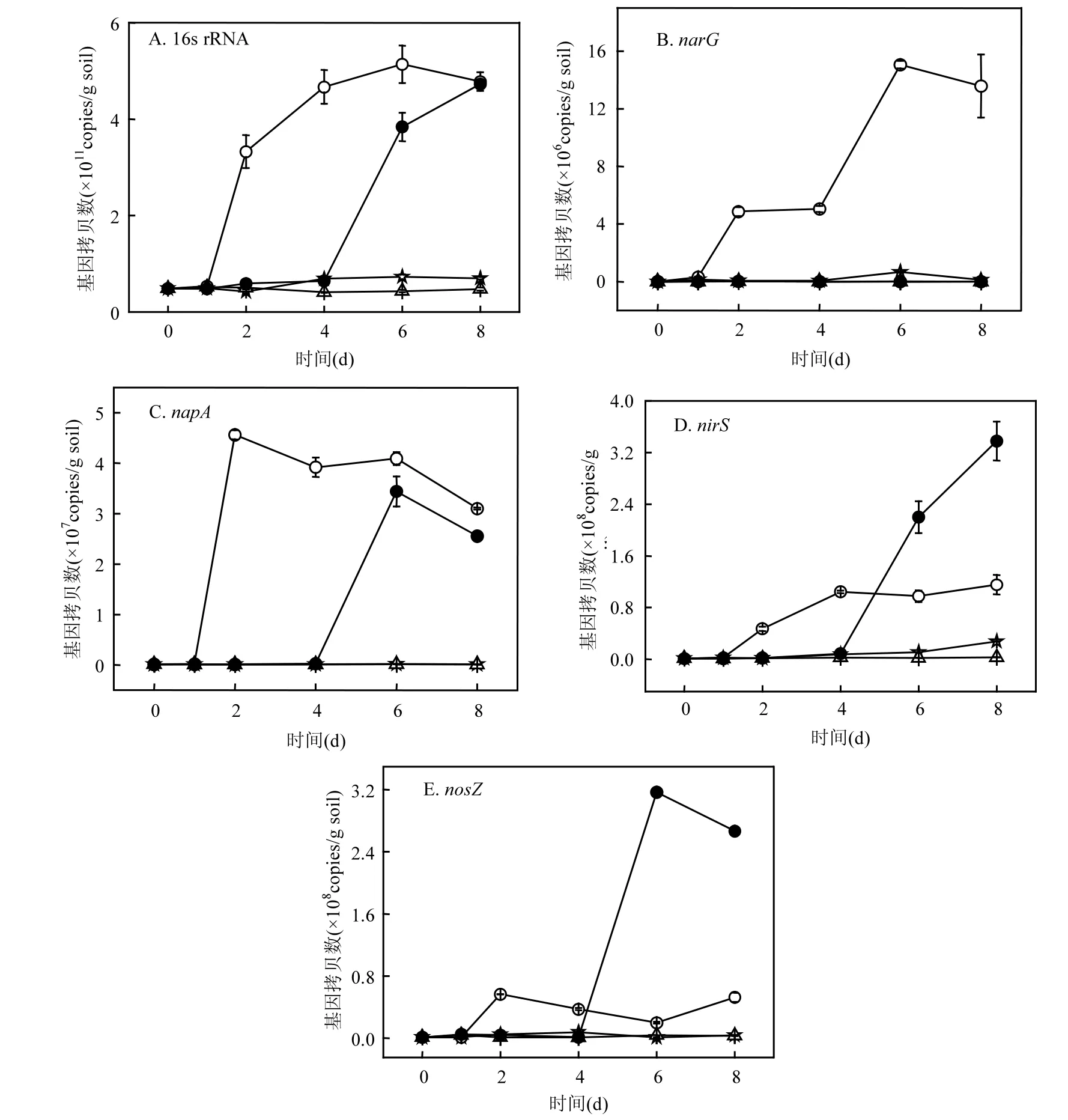
图3 不同处理中16S rRNA、narG、napA、nirS和(nosZ基因拷贝数随反应时间的变化(copies/g soil)Fig.3 Copy numbers of (A)16S rRNA, (B) narG, (C) napA, (D) nirS and (E) nosZ genes with time dependence in different treatments
对于nirS和nosZ基因拷贝数(图3D和图3E),在 Soil和 Soil+Fe(II)处理中,两者随着时间的变化基因拷贝数没有出现明显的增长.在 Soil+NO3-处理中,nirS和nosZ基因拷贝数均从第 1d后开始出现明显的增长.在Soil+Fe(II)+NO3-处理中,nirS和nosZ基因拷贝数均从第4d后才开始出现明显的增长.与 Soil+NO3-处理相比,Soil+Fe(II)+NO3-处理中nirS和nosZ基因拷贝数增长较为显著,在第 8d时均达到 108copies/g soil,同时Soil+NO3-处理中的拷贝数只有 107copies/g soil.由此表明,Fe(II)的加入提高了亚硝酸盐还原基因和氧化亚氮还原基因的丰度,进而促进了NO2-和 N2O的进一步还原,这与图 1B中 Soil+Fe(II)+处理的浓度显著低于Soil+处理的结果一致.
2.4 细菌群落结构组成及相对丰度变化
从微生物群落在门水平的组成(图 1A)得知,作为接种物的原始土壤在门水平主要以Betaproteobacteria(20%)为主.随着时间变化其微生物群落组成发生明显变化,Gammaproteobacteria成为Soil处理中的优势菌群,其丰度在第4d达到最高(52%),造成这种结果的原因可能在该处理中添加了乙酸盐作为碳源,影响了微生物群落结构组成.前人报道在厌氧条件 下,向 被 hexahydro-1,3,5-trinitro-1,3,5-triazine污染的地下水中加入碳源乙酸盐,会造成群落结构改变,促使高达 90%以上的Betaproteobacteria的富集[34].在 Soil+Fe(II)处理中,微生物群落组成随时间的变化呈现较稳定水平,说明 Fe(II)的加入并没有明显改变微生物在门水平的群落结构组成[17].在 Soil+NO3-和Soil+Fe(II)+NO3-处理中, Betaproteobacteria在群落中的相对丰度随着时间变化呈现明显的增长趋势:其中在 Soil+NO3-处理中,在第 2d Betaproteobacteria的相对丰度就达到 82%;在Soil+Fe(II)+NO3-处理中,Betaproteobacteria的相对丰度在第4d开始增长,并在第8d时达到最高的 93%.上述结果与前人研究相一致,即 NO3--N的存在(不管 Fe(II)存在与否)会促进Betaproteobacteria的富集[17,32].研究表明,大部分的反硝化微生物都属于 Proteobacteria的 alpha和 beta纲[1],而本文所研究的体系中则主要促进了Betaproteobacteria反硝化菌的富集.
Soil和 Soil+Fe(II)处理在属水平中均以Belliinea、Thermodefovibrio、Geobacter和vogesella的相对丰度较高,且随着时间的变化它们的相对丰度保持较为稳定水平(≤7%)(图 4B).这说明Fe(II)的加入对原始土壤的微生物群落组成没有明显的影响.在 Soil+NO3-处理中,主要的优势菌属为Pseudogulbenkiania(1.7%~34%)、Vogesella(2.8%~25%)和Dechloromonas(1.3%~12%)(图 4B).这 3种菌属均具有硝酸盐还原功能[35-37].在Soil+Fe(II)+NO3-处理中,主要的优势菌属为Rhodocyclus(0.1%~52%)和Dechloromonas(1.3%~39%)(图 4B).据报道,Rhodocyclus在微氧条件下具有 Fe(II)氧化功能, 而Dechloromonas则具有硝酸盐还原型亚铁氧化功能[16,38].由此可见,在 Soil+NO3-和Soil+ Fe(II)+NO3-处理中的优势微生物及其相对丰度有着明显的差异,说明Fe(II)的加入对稻田土壤中微生物群落的组成有明显的影响.
2.5 反硝化功能微生物群落组成
图5A中narG-硝酸盐还原功能微生物群落组成可知,富集培养到第 8d时,Dechlorosomas agitata(11%)和Pseudogulbenkiania ferrooxidans(6.4%)是Soil+NO3-处理中主要的narG-细胞膜硝酸盐还原优势微生物,说明上述 2种菌属是本文所研究的稻田土壤中通过细胞膜酶还原硝酸盐的功能优势微生物.由于本实验未能扩增出Soil和Soil+Fe(II)处理中napA基因用于克隆文库分析,因此图 5B中只展示了 Soil+NO3-和Soil+Fe(II)+NO3-处理在第8d时napA基因的克隆文库结果. 在Soil+NO3-处理中,主要的 napA-周质硝酸盐还原功能微生物包括Dechloromonas denitrificans-1(44%)、Dechloromonas agitata-1(20%)和Dechloromonas denitrificans-2(16%),三者的相对丰度总和高达 80%,表明Dechloromonas是本实验稻田土壤中具有周质硝酸盐还原基因的优势菌属.在 Soil+NO3-处理中,不管是发生在细胞膜还是周质中的硝酸盐还原过程,其主要功能微生物菌属均为Dechloromonas,由此可见该菌在稻田土壤中对NO3-的还原起着重要作用.尽管对于单个微生物来说一个基因组可能携带高达3个narG基因拷贝而仅拥有一个napA基因拷贝[39],然而本文结果表明,在Fe(II)存在的条件下,napA基因拷贝数明显高于narG基因拷贝数,说明在加入 Fe(II)后,napA在厌氧稻田环境中起主导作用.

图4 不同处理中细菌群落在门水平和属水平随反应时间的变化Fig.4 Changes in bacterial communities with time dependence at the (A) phyla level and (B) genus level detected in different treatments仅显示相对丰度大于5%的微生物群落
在Soil+Fe(II)+NO3-处理中,相对丰度较高的napA-周质硝酸盐还原功能微生物为Azonexus hydrophilus(34%)、Dechloromonas denitrificans-1(27%)和Azospira oryzae(17%).与上述Soil+NO3-处理的结果相比,Fe(II)的加入显著影响厌氧稻田土壤中napA-硝酸盐还原功能微生物群落的结构组成.目前关于 Fe(II)对硝酸盐还原功能微生物群落组成的影响尚未有报道.根据narG和napA基因定量结果,与 Soil+NO3-处理相比较,Fe(II)的存在降低了 Soil+Fe(II)+NO3-处理中narG基因的拷贝数(图3B),而提高了napA基因的拷贝数(图 3C),也就是说Azonexus、Dechloromonas和Azospira是Fe(II)存在的情况下NO3-还原的主要功能微生物.Azonexus、Dechloromonas和Azospira均属于Proteobacteria,其中Dechloromonas和Azospira都属于硝酸盐还原型亚铁氧化菌,在还原 NO3-的同时还可以氧化Fe(II)[35,40].Nar和Nap这两种异化硝酸盐还原酶的主要区别是它们在微生物细胞中所处的位置不同,分别位于细胞膜和周质中[4].Nar通常存在于 Proteobacteria、Frmicutes、Actinobacteria、甚至于Archaea中,而Nap仅存在于 Proteobacteria[41-42].与 Nar相比,目前关于Nap在微生物中的生理地位尚不清楚.根据现有的报道,Nap在不同微生物中的功能是不同的,比如:在光合细菌Rhodobacter capsulatus和Rhodobacter sphaeroides中Nap的主要作用就是催化NO3-的还原来重新平衡电子循环传输[44];而对于厌氧微生物在反硝化过程中利用 Nap会减少能量的消耗[42].还有文献报道提出,在厌氧条件下Nap可以取代Nar作为一种可供选择的代谢方式[44-45].
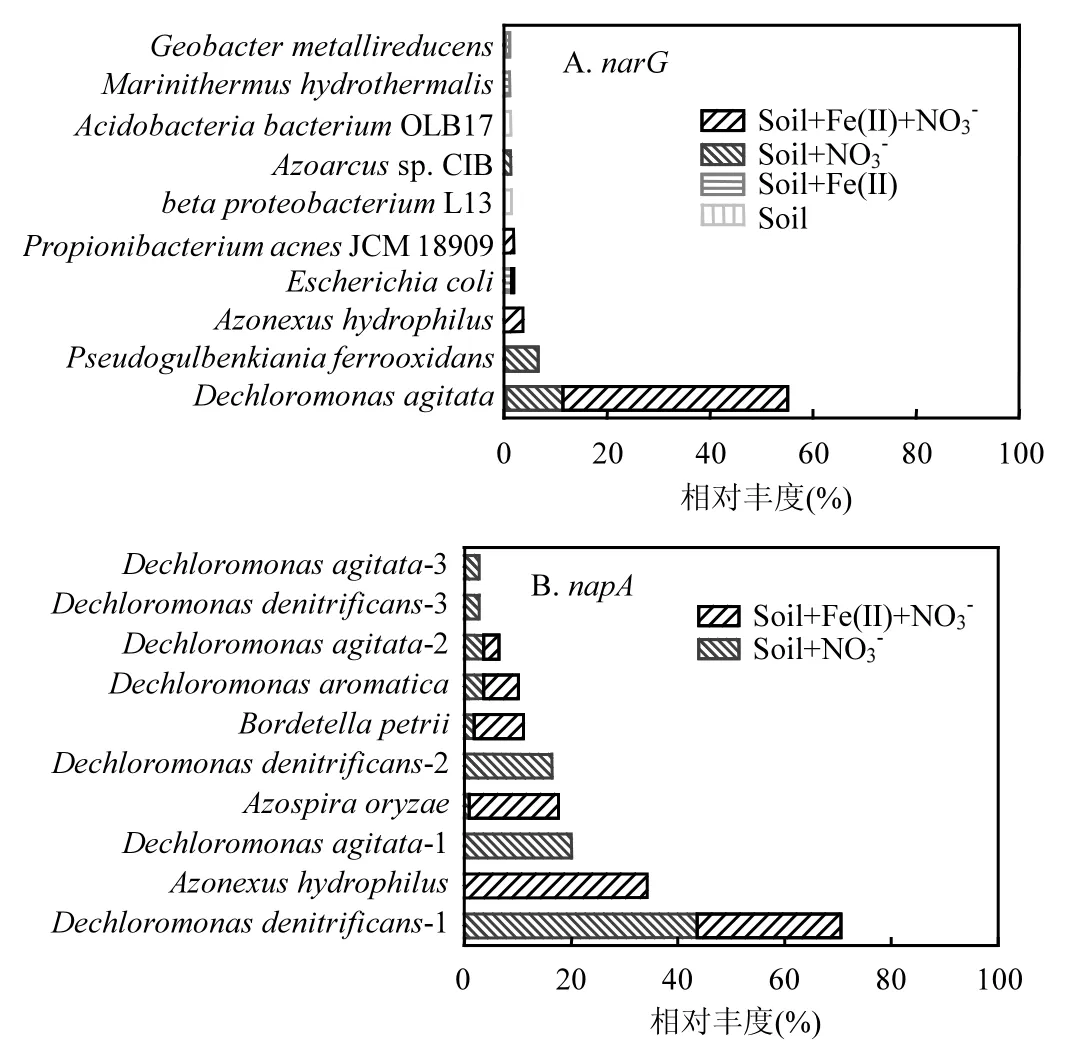
图5 反应结束时(第8d)基于硝酸盐还原基因narG和napA构建的克隆文库中每个OUT在不同处理中的物种相对丰度Fig.5 Relative abundance of OTUs identified to be nitrate-reducing bacteria based on narG (A) and napA (B) gene DNA clone library sequencing in different treatments at the end of incubation (Day 8)仅显示相对丰度大于1%的OTU
图 6亚硝酸盐还原功能微生物群落组成表明,在Soil和Soil+Fe(II)处理中,主要的亚硝酸盐还原优势微生物是Dechloromonas、Sideroxydans、Chloroflexi和Oceanithermus,它们的相对丰度低于9%.在Soil+NO3-处理中,优势的亚硝酸盐还原微生物包括Dechloromonas(33%)、Vogesella(6.6%)和Pseudogulbenkiania(6.0%).在Soil+Fe(II)+NO3-处理中,Dechloromonas(29%)和Dechlorospirillum(13%)是主要的亚硝酸盐还原优势微生物.Dechloromonas和Dechlorospirillum都具有Fe(II)氧化功能,Dechloromonas是硝酸盐依赖亚铁氧化菌,Dechlorospirillum在微好氧条件下能够氧化 Fe(II)[35,46].因此,Fe(II)的存在能够改变稻田境中的微生物群落结构并促使稻田环境中亚铁氧化菌的富集.
图 7中氧化亚氮还原功能微生物群落组成显示,Bradyrhizobium和Thiobacillus是 Soil、Soil+Fe(II)、Soil+NO3-和Soil+Fe(II)+NO3-处理中的 N2O还原优势微生物,Fe(II)的添加对稻田环境中的氧化亚氮还原微生物并没有明显的影响.文献报道Bradyrhizobium和Thiobacillus在氮循环中起着重要作用,分别具有固氮和脱氮功能[47-48].
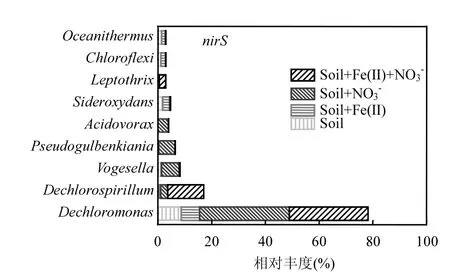
图6 反应结束时(第8d)不同处理中nirS基因编码的亚硝酸盐还原微生物在属水平的分布Fig.6 Relative abundance (%) of nirS-encoding nitritereducing bacteria at the genus level detected in different treatments at the end of incubation (Day 8)仅显示相对丰度高于1%的属

图7 反应结束时(第8d)不同处理中nosZ基因编码的氧化亚氮还原微生物在属水平的分布Fig.7 Relative abundance (%) of nosZ encoding nitrous oxide-reducing bacteria at the genus level detected in different treatments at the end of incubation (Day 8)仅显示相对丰度高于1%的属
3 结论
3.1 在中性厌氧稻田中, Fe(II)的存在延滞了NO3-的还原,但促进了NO2-的进一步还原和N2O的生成.
3.2 Fe(II)存在的情况下,降低了细胞膜硝酸盐还原基因narG的拷贝数,但提高了亚硝酸盐还原基因nirS和N2O还原基因nosZ的拷贝数,其硝酸盐还原过程主要发生在基于napA基因的周质上.
3.3 Fe(II)影响了基于16S rRNA的微生物群落组成,其中 Soil+NO3-处理的优势菌属为Pseudogulbenkiania、Vogesella和Dechloromonas,而 Soil+Fe(II)+NO3-处理的优势菌属为Dechloromonas和Rhodocyclus.
3.4 Fe(II)的加入改变了napA-周质硝酸盐还原微生物群落的组成结构,降低了Dechloromonas的相对丰度,提高了Azonexus和Azospira的相对丰度;并提高了nirS-亚硝酸盐还原优势菌Dechlorospirillum的相对丰度;对narG-细胞膜硝酸盐还原菌和nosZ-氧化亚氮还原菌的群落组成没有明显影响.
[1]Zumft W G. Cell biology and molecular basis of denitrification [J].Microbiology & Molecular Biology Reviews, 1997,61(4):533-616.
[2]Castresana J, Saraste M. Evolution of energetic metabolism: the respiration-early hypothesis [J]. Trends in Biochemical Sciences,1995, 20(11):443-448.
[3]Ducluzeau A L, Lis R V, Duval S, et al. Was nitric oxide the first deep electron sink? [J]. Trends in Biochemical Sciences, 2009,34(1):9-15.
[4]Kraft B, Strous M, Tegetmeyer H E. Microbial nitrate respirationgenes, enzymes and environmental distribution [J]. Journal of Biotechnology, 2011,155(1):104-117.
[5]Palmer K, Biasi C, Horn M A. Contrasting denitrifier communities relate to contrasting N2O emission patterns from acidic peat soils in arctic tundra [J]. ISME Journal, 2012,6(5):1058-1077.
[6]Deiglmayr K, Philippot L, Kandeler E. Functional stability of the nitrate-reducing community in grassland soils towards high nitrate supply [J]. Soil Biology & Biochemistry, 2006, 38(9):2980-2984.
[7]鲍林林,王晓燕,陈永娟,等.北运河沉积物中主要脱氮功能微生物的群落特征 [J]. 中国环境科学, 2016,36(54):1520-1529.
[8]Nordhoff M, Tominski C, Halama M, et al. Insights into nitrate-reducing Fe(II) oxidation mechanisms by analyzing cellmineral associations, cell encrustation and mineralogy in the chemolithoautotrophic enrichment culture KS [J]. Applied &Environmental Microbiology, 2017,doi:10.1128/AEM.00752-17.
[9]赵其国.红壤物质循环及其调控 [M]. 北京:科学出版社, 2002.
[10]谢小兰,余光伟,种云霄,等.污染河流底泥亚铁氧化硝酸盐还原菌分离及代谢特性 [J]. 环境科学, 2015,35(5):1224-1562.
[11]Smith R L, Kent D B, Repert D A, et al. Anoxic nitrate reduction coupled with iron oxidation and attenuation of dissolved arsenic and phosphate in a sand and gravel aquifer [J]. Geochimica Et Cosmochimica Acta, 2017,196:102-120.
[12]Zhang J, Zhao S, Xu Y, et al. Nitrate stimulates anaerobic microbial arsenite oxidation in paddy soils [J]. Environmental Science & Technology, 2017,51(8):4377-4386.
[13]TongH, Hu M, Li F, et al.Burkholderialesparticipating in pentachlorophenol biodegradation in iron-reducing paddy soil as identified by stable isotope probing [J]. Environmental Science Processes & Impacts, 2015,17(7):1282-1289.
[14]Chen X P, Zhu Y G, Hong M N, et al. Effects of different forms of nitrogen fertilizers on arsenic uptake by rice plants [J].Environmental Toxicology and Chemistry, 2008,27(4):881-887.
[15]罗希茜,陈 哲,胡荣桂,等.长期施用氮肥对水稻土亚硝酸还原酶基因多样性的影响 [J]. 环境科学, 2010,31(2):423-430.
[16]Li X, Zhang W, Liu T, et al. Changes in the composition and diversity of microbial communities during anaerobic nitrate reduction and Fe(II) oxidation at circumneutral pH in paddy soil[J]. Soil Biology & Biochemistry, 2016,94:70-79.
[17]陈鹏程,李晓敏,李芳柏.水稻土Fe(II)氧化耦合NO3-还原的微生物变化 [J]. 中国环境科学, 2017,37(1):358-366.
[18]鲁如坤.土壤农业化学分析方法 [M]. 北京:中国农业科技出版社, 2000.
[19]Zhu G, Wang S, Wang Y, et al. Anaerobic ammonia oxidation in a fertilized paddy soil [J]. ISME Journal, 2011,5(12):1905-1912.
[20]Lovley D R, Phillips E J. Rapid assay for microbially reducible ferric iron in aquatic sediments [J]. Applied & Environmental Microbiology, 1987,53(7):1536—1540.
[21]Klueglein N, Kappler A. Abiotic oxidation of Fe(II) by reactive nitrogen species in cultures of the nitrate-reducing Fe(II) oxidizer Acidovorax sp. BoFeN1- questioning the existence of enzymatic Fe(II) oxidation [J]. Geobiology, 2013,11(2):180-190.
[22]Petersen D J, Blazewicz S J, Firestone M, et al. Abundance of microbial genes associated with nitrogen cycling as indices of biogeochemical process rates across a vegetation gradient in Alaska [J]. Environmental Microbiology, 2012,14(4):993—1008.
[23]Lópezgutiérrez J C, Henry S, Hallet S, et al. Quantification of a novel group of nitrate-reducing bacteria in the environment by real-time PCR [J]. Journal of Microbiological Methods, 2004,57(57):399-407.
[24]Bru D, Sarr A, Philippot L. Relative abundances of proteobacterial membrane-bound and periplasmic nitrate reductases in selected environments [J]. Applied &Environmental Microbiology, 2007,73(18):5971-5974.
[25]Palmer K, Biasi C, Horn M A. Contrasting denitrifier communities relate to contrasting N2O emission patterns from acidic peat soils in arctic tundra [J]. ISME Journal, 2011,6(5):1058-1077.
[26]Vilar-Sanz A, Puig S, García-Lledó A, et al. Denitrifying bacterial communities affect current production and nitrous oxide accumulation in a microbial fuel cell [J]. PloS One, 2013,8(5):e63460.
[27]Caporaso J G, Lauber C L, Walters W A, et al. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample [J]. Proceedings of the National Academy of Sciences of the United States of America, 2011,108(Suppl 1):4516-4522.
[28]Throbäck I N, Enwall K, Jarvis A, et al. Reassessing PCR primers targeting nirS, nirK and nosZ genes for community surveys of denitrifying bacteria with DGGE [J]. FEMS Microbiology Ecology, 2004,49(3):401—417.
[29]Philippot L, Piutti S, Martin-Laurent F, et al. Molecular analysis of the nitrate-reducing community from unplanted and maizeplanted soils [J]. Applied & Environmental Microbiology,2002,68(12):6121-6128.
[30]Walter P B, Knutson M D, Paler-Martinez A, et al. Iron deficiency and iron excess damage mitochondria and mitochondrial DNA in rats [J]. Proceedings of the National Academy of Sciences, 2002,99(4):2264-2269.
[31]Klausner R D, Rouault T A, Harford J B. Regulating the fate of mRNA: the control of cellular iron metabolism [J]. Cell,1993,72(1):19-28.
[32]Ishii S, Yamamoto M, Kikuchi M, et al. Microbial populations responsive to denitrification-inducing conditions in rice paddy soil, as revealed by comparative 16S rRNA gene analysis [J].Applied & Environmental Microbiology, 2009,75(22):7070-7078.
[33]Straub K L, Benz M, Schink B, et al. Anaerobic, nitratedependent microbial oxidation of ferrous iron [J]. Applied &Environmental Microbiology, 1996,62(4):1458-1460.
[34]Livermore J A, Jin Y O, Arnseth R W, et al. Microbial community dynamics during acetate biostimulation of RDX-contaminated groundwater [J]. Environmental Science & Technology, 2013,47(14):7672-7678.
[35]Chakraborty A, Picardal F. Neutrophilic, nitrate-dependent, Fe(II)oxidation by aDechloromonasspecies [J]. World Journal of Microbiology and Biotechnology, 2013,29(4):617-623.
[36]Grimes D J, Woese C R, Macdonell M T, et al. Systematic study of the genusVogesellagen. nov. and its type species,Vogesella indigoferacomb. Nov [J]. International Journal of Systematic Bacteriology, 1997,47(1):19-27.
[37]Ishii S, Joikai K, Otsuka S, et al. Denitrification and nitratedependent Fe(II) oxidation in variousPseudogulbenkianiastrains[J]. Microbes & Environments, 2016,31(3):293-298.
[38]Sobolev D, Roden, E. E. Characterization of a neutrophilic,chemolithoautotrophic Fe(II)-oxidizing β-proteobacterium from freshwater wetland sediments [J]. Geomicrobiology Journal, 2004,21(1):1-10.
[39]Philippot L. Denitrifying genes in bacterial and Archaeal genomes[J]. Biochimica Et Biophysica Acta, 2002,1577(3):355-376.
[40]Chaudhuri S K, Lack J G, Coates J D. Biogenic magnetite formation through anaerobic biooxidation of Fe(II) [J]. Applied &Environmental Microbiology, 2001,67(6):2844-8.
[41]Philippot L, Højberg O. Dissimilatory nitrate reductases in bacteria [J]. Biochimica et Biophysica Acta - Gene Structure and Expression, 1999,1446(1/2):1-23.
[42]Richardson D J, Berks B C, Russell D A, et al Functional,biochemical and genetic diversity of prokaryotic nitrate reductases [J]. Cellular & Molecular Life Sciences: CMLS,2001,58(2):165-178.
[43]Richardson D J, King G F, Kelly D J. The role of auxiliary oxidants in maintaining redox balance during phototrophic growth ofRhodobacter capsulatuson propionate or butyrate [J].Archives of Microbiology, 1988,150(6):602-602.
[44]González P J, Correia C, Moura I, et al. Bacterial nitrate reductases: Molecular and biological aspects of nitrate reduction[J]. Journal of Inorganic Biochemistry, 2006,100(6):1015-1023.
[45]Delgado M J, Tresierra-Ayala A, Bedmar E J, et al. Thebradyrhizobium japonicum napEDABCgenes encoding the periplasmic nitrate reductase are essential for nitrate respiration[J]. Microbiology, 2003,149(Pt 12):3395-3403.
[46]Picardal F W, Zaybak Z, Chakraborty A, et al. Microaerophilic,Fe(II)-dependent growth and Fe(II) oxidation by aDechlorospirillumspecies [J]. FEMS Microbiology Letters,2011,319(1):51-57.
[47]Baldani J I, Baldani V L D, Seldin L, et al. Characterization ofHerbaspirillum seropedicaegen. nov., sp. nov., a root-associated nitrogen-fixing bacterium [J]. International Journal of Systematic Bacteriology, 1986,36(1):86-93.
[48]Zhang Z, Lei Z, He X, et al. Nitrate removal byThiobacillusdenitrificans immobilized on poly(vinyl alcohol) carriers [J].Journal of Hazardous Materials, 2009,163(2/3):1090-1095.

