piRNA在胃癌与正常胃黏膜组织中表达的比较
蒋佩佩,蔡一奇,王鹏飞,陈孝冬,金劲激,胡畅远,陈文静,薛向阳,张丽芳,朱冠保
(1.温州医科大学附属第一医院 胃肠外科,浙江 温州 325015;2.温州医科大学 分子病毒与免疫研究所 微生物学与免疫学教研室,浙江 温州 325035)
piRNA在胃癌与正常胃黏膜组织中表达的比较
蒋佩佩1,蔡一奇1,王鹏飞1,陈孝冬1,金劲激1,胡畅远1,陈文静1,薛向阳2,张丽芳2,朱冠保1
(1.温州医科大学附属第一医院 胃肠外科,浙江 温州 325015;2.温州医科大学 分子病毒与免疫研究所 微生物学与免疫学教研室,浙江 温州 325035)
目的:分析piRNA在胃癌组织与正常胃黏膜组织中表达的差异性.方法:通过新一代高通量测序技术Solexa对胃癌组织和正常胃黏膜组织进行小分子RNA(sRNA)深度测序,并通过生物信息学分析胃癌组织与正常胃黏膜组织中piRNA的表达差异.根据分析结果选取4种piRNA(登录号分别为DQ570956、DQ575659、DQ594126和DQ597128),通过茎环反转录实时定量PCR(简称茎环RT-qPCR)技术验证其存在及其在胃癌组织和正常胃黏膜组织中的表达差异.结果:Solexa深度测序结果显示,正常胃黏膜与胃癌组织中测得的piRNA种类在各标本所测得所有sRNA种类中所占的比例差异无统计学意义(Z=0.835,P=0.678);正常胃黏膜组织中测得的piRNA数量在各标本所测得sRNA总量中所占的比例高于胃癌组织,差异有统计学意义(Z= 2.167,P=0.042);根据分析结果选取4种piRNA,经茎环RT-qPCR法检测显示,这4种piRNA在胃癌组织中的表达量显著低于正常胃黏膜组织,差异有统计学意义(P<0.05).结论:胃癌组织存在多种差异表达的piRNA,且在表达量上与正常胃黏膜组织存在显著性差异,本研究选取的4种piRNA可能参与胃癌的发生发展进程.
胃肿瘤;piRNA;小分子RNA;聚合酶链反应
胃癌是全球发病率最高的五大恶性肿瘤之一[1].目前,我国胃癌每年的发病率持续上升,且呈现显著的年轻化趋势,发病率和病死率均居我国恶性肿瘤第三位[2-3].寻求新的胃癌标记物及有效的生物治疗靶点是进一步改善胃癌预后的关键.Piwiinteracting RNA(piRNA)是近年来在动物生殖细胞中发现的一类新小分子非编码RNA,长度为26~32个核苷酸,主要集中在29~30 nt,能特异性地同Argonuat(AGO)蛋白家族中的Piwi亚家族蛋白相互结合而产生作用,因此被命名为piwi-interacting RNA,简称piRNA[4-7].自piRNA被发现以来,研究人员对其研究进展迅速,现已发现其在生殖干细胞分化、胚胎发育、维持DNA完整性、表观遗传学调控和物种的性别决定等方面起着重要作用[8-12].迄今许多与肿瘤相关的piRNA仍不为人所知,有关其表达调节的研究也不多[13].本研究通过小RNA测序及后续的生物信息学技术分析胃癌中的piRNA表达情况,并选取4个piRNA采用茎环RT-qPCR技术验证其在胃癌组织与正常组织中表达的差异性,为今后研究piRNA在胃癌发病中的作用提供一定的研究基础.
1 材料和方法
1.1 实验材料 Trizol试剂购自美国Invitrogen公司;M-MLV反转录酶、RNA酶抑制剂、dNTPs、SYBR Green I Mastermix等试剂购自中国台湾Toyobo公司;引物由上海英骏生物技术有限公司合成.
1.2 方法
1.2.1 标本采集:选取2010年至2011年间在温州医科大学附属第一医院胃肠外科手术治疗的31例胃癌患者的胃癌组织及距癌组织边缘5 cm以上的配对癌旁正常胃黏膜组织(经病理确定不含癌细胞),全部病例术前均未经过放疗、化疗等治疗.31例胃癌患者中,男26例,女5例,年龄48~83岁,中位年龄62岁,其中≥60岁19例,<60岁12例;胃癌临床分期I期3例,II期0例,III期19例,IV期9例;有淋巴结转移27例,无淋巴结转移4例.所有组织标本取出后10 min内置于液氮中保存,于液氮中放置24 h后再放入-80 ℃冰箱保存.本研究经温州医科大学附属第一医院伦理委员会审核通过,所有患者均知情同意.
1.2.2 RNA的提取:按Trizol试剂说明书提取总RNA,异丙醇沉淀时间为-20 ℃放置2 h以上以增加小RNA提取率.用80%乙醇冲洗,DEPC处理过的蒸馏水溶解并保存于-80 ℃冰箱中.采用德国EPPENDORF公司的分光光度仪检测RNA溶液,即OD260和OD280,OD260/OD280比值>1.8为符合实验要求的RNA浓度和纯度.1%琼脂糖变性凝胶电泳检测RNA完整性.
1.2.3 胃癌相关piRNA筛选:选取RNA完整性佳的3例标本(均包括胃癌组织和正常胃黏膜组织)各1份送华大基因研究中心进行小分子RNA(small RNA,sRNA)深度测序,对测序所得的35 nt原始reads做去接头、去低质量reads、去污染等处理,得到高质量的测序reads.选取美国国立生物技术信息中心(national center for biotechnology,NCBI)中的piRNA序列对测序所得的sRNA序列进行注释,经过数据统计,挑选出匹配上piRNA的sRNA序列.
1.2.4 茎环RT-qPCR检测piRNA:采用茎环RT-qPCR方法[14],以U6为内参,分析各piRNA的表达.相关引物序列见表1.4 μg总RNA分别以4个piRNA及U6茎环RT-qPCR引物进行反转录,反应条件为:16 ℃30 min,42 ℃ 30 min,70 ℃ 15 min,反应产物于-20 ℃保存.以20 μL体系进行RT-qPCR.piRNA的检测反应体系包括:1 μL RT产物、1XSYBR Green I Mastermix、10 μmol/L piRNA特异前向引物、10 μmol/L通用的反向引物.RT-qPCR的反应条件:95 ℃ 2 min后,95 ℃ 15 s,60 ℃ 1 min,40个循环.RT-qPCR使用Applied Biosystems 7500仪器进行.所有样品做3复孔,记录每个反应孔中荧光信号到达所设定的阈值时所经历的循环数(即Ct值).以U6为内参,采用RT-qPCR中的相对定量法,以N=2-ΔΔCt表示胃癌组织相对配对正常组织表达的倍数
1.3 统计学处理方法 采用SPSS16.0统计软件进行统计分析.正常胃黏膜组织和胃癌组织piRNA与NCBI比对的结果采用Mann-Whitney U检验.piRNA的RT-qPCR检测结果以M(P25,P75)表示,2组间比较采用Mann-Whitney U检验.P<0.05为差异有统计学意义.
2 结果
2.1 胃癌组织piRNA差异表达分析 Solexa深度测序所得序列长度分布见图1.与正常胃黏膜组织比较,长度约30 nt的sRNA在胃癌组织中呈减少趋势.采用生物信息学对测序获得的sRNA进行分类注释,确定piRNA的种类及数量(见表2).从表2可以看出正常胃黏膜与胃癌组织中测得的piRNA种类在各标本所测得的所有sRNA种类中所占的比例差异无统计学意义(Z=0.835,P=0.678);而正常胃黏膜组织中测得的piRNA数量与各标本所测得所有sRNA序列总读数的比例显著高于胃癌组织,差异有统计学意义(Z=2.167,P=0.042).在此基础上,选取4个reads丰度大于10 000,在胃癌组织与相应正常胃黏膜组织中reads数相差3倍以上的piRNA(登录号分别为DQ570956、DQ575659、DQ594126、DQ597128)进一步验证.

表1 茎环RT-qPCR引物序列

图1 sRNA片段测序长度分布图
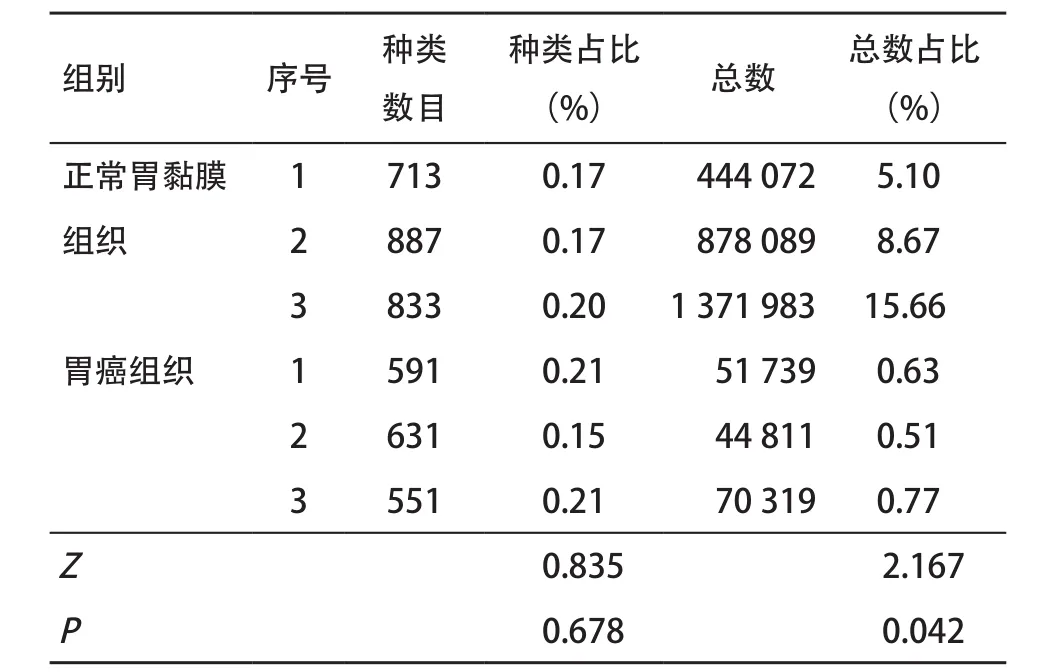
表2 正常胃黏膜组织和胃癌组织piRNA与NCBI比对的检测结果
2.2 胃癌组织差异表达piRNA验证 我们采用TANG等[15]建立的茎环RT-qPCR分析piRNA.所选的4种piRNA(DQ570956、DQ575659、DQ594126、DQ597128)及U6 RT-qPCR扩增产物的溶解曲线均显示为单峰(见图2).4种piRNA在胃癌组织中的相对表达量分别为0.28(0.12,0.85)、0.28(0.02,0.70)、0.56(0.29,1.11)、0.30(0.13,0.84),表明其在胃癌组织中的相对表达量均显著低于正常胃黏膜组织,差异均有统计学意义(P<0.05).
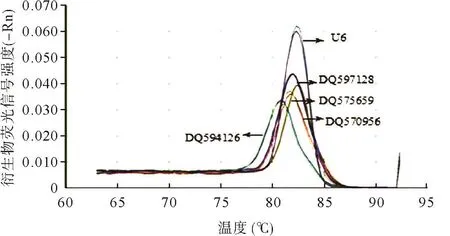
图2 茎环RT-qPCR扩增产物的溶解曲线
3 讨论
近年来,小分子非编码RNA因其在转录及转录后水平对基因表达的有力调节而被广泛研究[16-18].特别是非编码小RNA中的siRNA和miRNA已经被证明参与了肿瘤的发生和进展过程[19-21].研究表明,非编码小RNA在多种肿瘤中具有潜在的诊断价值,并且可以作为备选的治疗靶点[10,22].
piRNA是一类新型小分子非编码RNA,自2006年7月被发现以来,相继有4个独立的实验室从鼠睾丸里面分离得到piRNA[1-4].目前发现piRNA主要在生殖细胞中表达,在基因表达的调控和干细胞的分化中充当着重要的作用[23].其生物学功能主要是维持基因组中转座子的正常沉默状态,以防止基因组中转座子爆发而引起相应基因的改变[6,13].近来各研究显示piRNA可能在更大范围的生物学过程中发挥重要影响.如WONG等[23]与CARMELL等[24]研究发现蝇类piRNA突变引起的反转录转座子的过表达和作用可以导致生殖系DNA的损伤;ESPOSITO等[25]研究证实piR-015520在人组织中表达并能够抑制褪黑素受体1A(MTNR1A)基因的表达;ASHRAF等[26]研究发现参与piRNA生产的蛋白与体细胞基因表达的控制及学习记忆相关.此外,piRNA的生物学起源与作用机制并未完全明确,比如尚不明确piRNA主要通过控制染色质组织、基因转录、RNA稳定还是RNA翻译来发挥作用.
在人类中,与piRNA相互作用的蛋白质是PiWi蛋白.PiWi蛋白是最早发现的调控干细胞增殖的蛋白之一,其过高表达将引起干细胞过度增长及分裂加速,并在多个物种中通过与PiWi介导的piRNA相互作用以调控基因表达[27].先前研究人员认为piRNA仅仅存在于生殖细胞中[5].最近的研究发现,piRNA在人类肿瘤细胞中也存在,并且通过大规模平行测序技术在人类宫颈癌细胞中得到了鉴定[8].另外,已经有研究证实piRNA水平的改变能够明显地增加男性胰腺癌患者的死亡风险[29].这一系列的发现说明,探寻piRNA在人类肿瘤发生中可能的机制是有十分有必要的.
肿瘤已经被广泛地认为是一类干细胞疾病[30].肿瘤细胞和干细胞一样拥有自我更新的能力[31].PiWi基因家族是多种生物体中干细胞自我更新的关键基因.已有研究证明,HiWi基因作为人类PiWi基因家族的成员之一,参与了生殖细胞的增殖过程,HiWi基因的异常表达与软组织肉瘤的发生相关[31].人类PiWi基因家族的另外一个成员Hili则发挥着癌基因的作用,明显地抑制了逆转座子line-1的表达[28].有研究证实,Hili基因参与了胃癌的发生过程并且可以作为生物治疗的潜在作用靶点[32].目前,与肿瘤相关的piRNAs少有研究,其在胃癌中的研究更是罕见.为分析piRNA在人胃癌中的表达情况,本研究采用Solexa测序技术对胃癌和正常胃黏膜组织进行siRNA深度测序,通过生物信息学分析了解piRNA表达情况.结果显示,胃癌组织中piRNA的种类占所测组织全部sRNA种类的比例较正常组织无明显差异;但在表达的总体数量上明显少于正常组织,提示piRNA表达量的减少可能与胃癌的发生密切相关.之后,我们采用茎环RT-qPCR分析piRNA,选取4种piRNA(DQ570956、DQ575659、DQ594126、DQ597128)经茎环RT-qPCR技术检测其在31例标本中的表达情况,RT-qPCR扩增产物的溶解曲线均显示为单峰,说明茎环RT-qPCR能扩增相应的目的基因,与对应正常胃黏膜组织比较,各piRNA在31例胃癌组织和对应正常胃黏膜组织中的表达量存在显著性差异,进一步证实piRNA在胃癌组织中低表达.与正常胃黏膜比较,piRNA在胃癌组织中存在明显差异表达.4种piRNA经验证表达量在胃癌组织中呈下调趋势,考虑到piRNA控制转座子的正常沉默状态这一功能,提示了其可能作为一种抑癌因素存在.其在胃癌发生、发展中的具体作用及确切机制有待深入研究.相信随着今后对有关研究的深入,可为进一步阐明piRNA在胃癌发生、发展(包括肿瘤的药物治疗和分子靶向疗法)提供新的着眼点.
[1] TORRE L A, BRAY F, SIEGEL R L, et al. Global cancer statistics, 2012[J]. CA Cancer J Clin, 2015, 65(2): 87-108.
[2] 孙秀娣, 牧人, 周有尚, 等. 中国胃癌死亡率20年变化情况分析及其发展趋势预测[J]. 中华肿瘤杂志, 2004, 26(1):4-9.
[3] 王永川, 魏丽娟, 刘俊田, 等. 发达与发展中国家癌症发病率与死亡率的比较与分析[J]. 中国肿瘤临床, 2012, 39(10):679-682.
[4] GIRARD A, SACHIDANANDAM R, HANNON G J, et al.A germline-specific class of small RNAs binds mammalian Piwi proteins[J]. Nature, 2006, 442(7099): 199-202.
[5] ARAVIN A, GAIDATZIS D, PFEFFER S, et al. A novel class of small RNAs bind to MILI protein in mouse testes[J].Nature, 2006, 442(7099): 203-207.
[6] LAU N C, SETO A G, KIM J, et al. Characterization of the piRNA complex from rat testes[J]. Science, 2006, 313(5785): 363-367.
[7] GRIVNA S T, BEYRET E, WANG Z, et al. A novel class of small RNAs in mouse spermatogenic cells[J]. Genes Dev,2006, 20(13): 1709-1714.
[8] 孟雅楠, 孟丽军, 宋亚娟, 等. 小RNA分子与精子发生调控[J]. 遗传, 2011, 33(1): 9-16.
[9] CHEN Y, PANE A, SCHÜPBACH T. Cutoff and aubergine mutations result in retrotransposon upregulation and checkpoint activation in Drosophila[J]. Curr Biol, 2007, 17(7):637-642.
[10] KLATTENHOFF C, THEURKAUF W. Biogenesis and germline functions of piRNAs[J]. Development, 2008, 135(1): 3-9.
[11] LIU G, LEI B, LI Y, et al. Discovery of potential piRNAs from next generation sequences of the sexually mature porcine testes[J]. PLoS One, 2012, 7(4): e34770.
[12] KAWAOKA S, ARAI Y, KADOTA K, et al. Zygotic amplification of secondary piRNAs during silkworm embryogenesis[J]. RNA, 2011, 17(7): 1401-1407.
[13] HOUWING S, KAMMINGA L M, BEREZIKOV E, et al. A role for Piwi and piRNAs in germ cell maintenance and transposon silencing in Zebrafish [J]. Cell, 2007, 129(1): 69-82.
[14] XUE X, SUN J, ZHANG Q, et al. Identification and characterization of novel microRNAs from Schistosoma japonicum[J]. PLoS One, 2008, 3(12): e4034.
[15] TANG F, HAJKOVA P, BARTON S C, et al. MicroRNA expression profiling of single whole embryonic stem cells[J].Nucleic Acids Res, 2006, 34(2): e9.
[16] LAGOS-QUINTANA M, RAUHUT R, LENDECKEL W,et al. Identification of novel genes coding for small expressed RNAs[J]. Science, 2001, 294(5543): 853-858.
[17] LAU N C, LIM L P, WEINSTEIN E G, et al. An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans[J]. Science, 2001, 294(5543): 858-862.
[18] BARTEL D P. MicroRNAs: genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116(2): 281-297.
[19] VOLINIA S, CALIN G A, LIU C G, et al. A microRNA expression signature of human solid tumors defines cancer gene targets[J]. Proc Natl Acad Sci U S A, 2006, 103(7):2257-2261.
[20] TAFT R J, PANG K C, MERCER T R, et al. Non-coding RNAs: regulators of disease[J]. J Pathol, 2010, 220(2): 126-139.
[21] ZHU S, WU H, WU F, et al. MicroRNA-21 targets tumor suppressor genes in invasion and metastasis[J]. Cell Res,2008, 18(3): 350-359.
[22] BARBAROTTO E, SCHMITTGEN T D, CALIN G A.MicroRNAs and cancer: profile, profile, profile[J]. Int J Cancer, 2008, 122(5): 969-977.
[23] WONG E W, MRUK D D, LEE W M, et al. Par3/Par6 polarity complex coordinates apical ectoplasmic specialization and blood-testis barrier restructuring during spermatogenesis[J]. Proc Natl Acad Sci U S A, 2008, 105(28): 9657-9662.
[24] CARMELL M A, GIRARD A, van de KANT H J, et al. MIWI2 is essential for spermatogenesis and repression of transposons in the mouse male germline[J]. Dev Cell, 2007, 12(4): 503-514.
[25] ESPOSITO T, MAGLIOCCA S, FORMICOLA D, et al.piR_015520 belongs to Piwi-associated RNAs regulates expression of the human melatonin receptor 1A gene[J]. PLoS One, 2011, 6(7): e22727.
[26] ASHRAF S I, MCLOON A L, SCLARSIC S M, et al. Synaptic protein synthesis associated with memory is regulated by the RISC pathway in Drosophila[J]. Cell, 2006, 124(1):191-205.
[27] WU Q, MA Q, SHEHADEH L A, et al. Expression of the Argonaute protein PiwiL2 and DiRNAs in adult lnouse mesenchymal stem cells[J]. Biochem Biophys Res Conmmn,2010, 396(4): 915-920.
[28] LU Y, LI C, ZHANG K, et al. Identification of piRNAs in Hela cells by massive parallel sequencing[J]. BMB Rep,2010, 43(9): 635-641.
[29] GROCHOLA L F, GREITHER T, TAUBERT H, et al. The stem cell-associated Hiwi gene in human adenocarcinoma of the pancreas: expression and risk of tumour-related death[J].Br J Cancer, 2008, 99(7): 1083-1088.
[30] DALERBA P, DYLLA S J, PARK I K, et al. Phenotypic characterization of human colorectal cancer stem cells[J].Proc Natl Acad Sci U S A, 2007, 104(24): 10158-10163.
[31] DALERBA P, CHO R W, CLARKE M F. Cancer stem cells:models and concepts[J]. Annu Rev Med, 2007, 58: 267-284.
[32] LIU X, SUN Y, GUO J, et al. Expression of Hiwi gene in human gastric cancer was associated with proliferation of cancer cells[J]. Int J Cancer, 2006, 118(8): 1922-1929.
(本文编辑:贾建敏)
Comparison of the expression of piRNA in gastric carcinoma and normal gastric mucosa tissues
JIANG Peipei1, CAI Yiqi1, WANG Pengfei1, CHEN Xiaodong1, JIN Jinji1, HU Changyuan1, CHEN Wenjing1,XUE Xiangyang2, ZHANG Lifang2, ZHU Guanbao1.
1.Department of Gastroenterological Surgery, the First Affiliated Hospital of Wenzhou Medical University, Wenzhou, 325015; 2.Instiute of Molecular Virology and Immunology, Department of Medical Microbiology and Immunology, Wenzhou Medical University, Wenzhou, 325035
Objective:To analyze the differences of expression of piRNA in gastric carcinoma and normal gastric mucosa.Methods:The small RNA (sRNA) deep sequencing of gastric cancer tissue and normal gastric mucosa was performed by a new generation of high throughput sequencing technology, and the differential expression of piRNA was detected in gastric cancer tissue and normal gastric mucosa by follow-up bioinformatics.Four kinds of piRNAs (login ID: DQ570956, DQ575659, DQ594126 and DQ597128) were selected according to the results of the analysis, and their presence and their differences in gastric cancer tissues and normal gastric mucosa were verified by real-time quantitative PCR.Results:Solexa depth sequencing results showed that there was no significant difference in the proportion of sRNA measured in normal gastric mucosa and gastric cancer tissues (Z=0.835, P=0.678). The number of piRNAs measured in normal tissues was significantly higher than that in gastric cancer tissues (Z=2.167, P=0.042). Four piRNAs were selected according to the analysis results. The expression of that 4 piRNA in gastric cancer tissues was significantly lower in normal gastric mucosa (P<0.05).Conclusion:There are many differentially expressed piRNAs in gastric cancer tissues, and there is significant difference in the normal gastric mucosa. In this study, four piRNAs may be involved in the development and progression of gastric cancer, and the subsequent development of piRNA and gastric cancer of the pathogenesis of the study to provide experimental basis.
stomach neoplasms; piRNA; small RNA; polymerase chain reaction
R392.7
A
10.3969/j.issn.2095-9400.2017.10.002
2017-03-18
国家自然科学基金资助项目(81372447);浙江省自然科学基金资助项目(Y2100660);温州市科技局科研基金资助项目(H20100028).
蒋佩佩(1986-),女,浙江永嘉人,住院医师,硕士.
朱冠保,教授,Email:zgbwmc@126.com.

