飞蝗内表皮蛋白基因LmAbd-5的表达与功能分析
赵小明,贾盼,2,勾昕,2,刘卫敏,马恩波,张建珍
(1山西大学应用生物学研究所,太原 030006;2山西大学生命科学学院,太原 030006)
飞蝗内表皮蛋白基因LmAbd-5的表达与功能分析
赵小明1,贾盼1,2,勾昕1,2,刘卫敏1,马恩波1,张建珍1
(1山西大学应用生物学研究所,太原 030006;2山西大学生命科学学院,太原 030006)
【目的】基于飞蝗(Locusta migratoria)转录组数据库获得内表皮蛋白(endocuticle structural glycoprotein)基因LmAbd-5的cDNA序列,分析该基因的序列特征和mRNA表达特性,采用RNAi方法分析其生物学功能,探讨其在飞蝗表皮形成中的作用,为害虫防治提供新的分子靶标。【方法】采用生物信息学方法搜索飞蝗转录组数据库,获得LmAbd-5 cDNA全长序列并克隆验证;采用SignalP在线软件分析蛋白的信号肽,利用SMART网站预测其功能域,使用MEGA 7.0软件中neighbor-joining(NJ)方法,与其他昆虫同源序列进行聚类分析;采用reverse-transcription quantitative PCR(RT-qPCR)方法检测LmAbd-5在飞蝗5龄第2天若虫不同组织部位和5龄不同发育时期体壁组织中的表达情况,揭示其组织和时期表达模式;采用RNA干扰(RNAi)技术及透射电镜技术(TEM)观察沉默LmAbd-5后对飞蝗生长发育和表皮结构的影响。【结果】通过搜索得到LmAbd-5 cDNA全长序列并进行了克隆和测序验证,获得520 bp全长cDNA序列,其中ORF为303 bp;基因结构分析显示该基因含有3个外显子;功能域分析发现其含有1个信号肽和1个几丁质结合域(chitin binding domain 4,ChtBD4),与沙漠蝗、白蚁、赤拟谷盗等的Abd-5结构类似;BLAST分析结果表明Abd-5在昆虫中高度保守,飞蝗与沙漠蝗Abd-5序列一致度高达81%;对其保守基序进行WebLogo分析发现LmAbd-5属于表皮蛋白CPR家族中的RR-1亚类;聚类分析结果显示,LmAbd-5与沙漠蝗和白蚁的Abd-5显示出较近的亲缘关系;RT-qPCR结果显示LmAbd-5在前肠、后肠、气管和体壁等由外胚层形成的组织中高表达,而在胃盲囊、中肠、马氏管、脂肪体和翅芽中低表达或不表达;不同时期表达分析发现,LmAbd-5在4龄若虫蜕皮后0—72 h(5龄早期)具有高表达,其中蜕皮后72 h时达到最大表达量,随后表达量急剧降低(96—168 h),其表达时期与内表皮形成时间一致;采用RNAi技术分析该基因的生物学功能,对5龄第2天若虫分别注射等量的dsLmAbd-5和dsGFP(对照),发现注射dsLmAbd-5的5龄若虫和对照组相同,均可正常蜕皮,发育至成虫第2天目的基因表达量显著降低,但未出现肉眼可见的异常表型。分别取成虫第2天处理组和对照组成虫表皮进行超微结构观察,发现与对照组相比,处理组成虫内表皮片层结构较为疏松,导致内表皮片层变厚,最终表现为整个内表皮变厚。【结论】根据转录组数据库分析获得1个CPR家族表皮蛋白LmAbd-5,该蛋白含有1个信号肽和1个几丁质结合域ChtBD4,属于RR-1亚类;LmAbd-5主要在外胚层起源的组织中高表达,沉默LmAbd-5后飞蝗没有肉眼可见的表型,但超微结构分析发现其参与飞蝗内表皮片层结构的形成。
飞蝗;表皮蛋白;LmAbd-5;RNAi;透射电镜
0 引言
【研究意义】中国是一个农业生物灾害频发、生态环境脆弱的农业大国,毁灭性的农作物病虫害频繁暴发,导致农业经济损失与环境破坏。飞蝗(Locusta migratoria)作为世界性农业害虫,具有暴发性、群集性和迁飞性等特点,蝗灾发生面积广,致灾严重。因此,对飞蝗的研究和蝗灾的综合治理对经济和民生发展具有重要意义。昆虫体壁具有重要的保护作用和生理功能,能够保护虫体内部结构,是抵御外界环境的第一道防线,在抵御水分蒸发和应对外界不良环境的影响等方面起着重要作用。表皮蛋白是昆虫体壁的重要组成成分,其成分的缺失或含量变化都可引起表皮功能异常,从而影响昆虫的生长发育。因此,从飞蝗表皮蛋白分子特性和生理功能入手,探索表皮蛋白在飞蝗表皮结构中的作用,可筛选蝗虫防治新分子靶标,从而为蝗灾有效治理提供新的思路和方法。【前人研究进展】昆虫体壁具有保护和防御等功能,在生命过程中发挥着重要作用。昆虫体壁由上表皮、原表皮(内表皮和外表皮)和真皮细胞构成,昆虫蜕皮包括皮层溶离和蜕去旧表皮两个连续的过程,昆虫表皮在蜕皮液作用下首先发生皮层溶离,最先形成表皮质层,然后形成上表皮和外表皮,在上表皮开始沉积的同时,旧表皮逐渐被消化,在旧表皮蜕去后开始合成内表皮[1-3]。原表皮主要成分是几丁质和蛋白质,它们随蜕皮过程而周期性的合成和降解。几丁质是一种多糖类物质,是由N-乙酰胺基葡糖以β-l,4糖苷键组成的线性生物聚合物,从真菌界到节肢动物门都大量存在[4]。在自然界中,几丁质与结构蛋白、酶和抗菌蛋白序列中的几丁质结合域(chitin-binding domains,CBDs)以非共价键形式结合形成糖蛋白,糖蛋白在外表皮中经鞣化后形成坚固稳定的结构。昆虫表皮蛋白(insect cuticular proteins,ICP)是结构蛋白,根据几丁质结合域的不同可分为两类:第一类几丁质结合域为R&R consensus(Rebers and Riddiford consensus),具有RR-1、RR-2、RR-3 等 3种形式[5-6],此类表皮蛋白富含组氨酸,缺失半胱氨酸一段68个氨基酸的序列,也称为CPR家族。随着果蝇(Drosophila melanogaster)、家蚕(Bombyx mori)和赤拟谷盗(Tribolium castaneum)等模式昆虫全基因组测序相继完成,ICP的鉴定也随之展开。学者已在黑腹果蝇[7]、赤拟谷盗[8]、家蚕[9]和烟草天蛾(Manduca sexta)[10]等昆虫基因组中分别鉴定获得101、102、148和207个属于CPR家族的表皮蛋白。目前含有R&R consensus结构域的表皮蛋白与几丁质的结合能力已得到实验证实[5,11]。另一类几丁质结合域是含有6个半胱氨酸残基,形成3个二硫键的保守序列,称为the type 2 chitin-binding domain (ChtBD2 domain),此类表皮蛋白主要存在于昆虫表皮和围食膜中[12-13]。目前,对昆虫CPR家族表皮蛋白的研究中,以家蚕、果蝇和赤拟谷盗等模式昆虫的研究较为深入,如日本学者从翅原基化蛹前cDNA库中随机选出cDNA进行序列测定,鉴定了10种不同的表皮蛋白基因,命名为BmWCPs[14],韩国全南国立大学Yasuyuki Arakane课题组以赤拟谷盗为对象,研究鞘翅中高丰度的表皮蛋白功能[15-18],这些研究为表皮蛋白在昆虫变态发育和表皮形成过程的作用机制研究奠定了重要基础。然而,尽管国内外多个课题组正在开展表皮蛋白在昆虫表皮重塑过程中的作用及其机制研究,但尚未在超微结构水平开展昆虫内外表皮蛋白的作用机制研究工作。【本研究切入点】20世纪90年代,JESPERSEN等通过质谱分析方法从飞蝗和沙漠蝗(Schistocerca gregaria)的腹部表皮中分别鉴定出了1个内表皮结构糖蛋白(endocuticle structural glycoprotein),分别命名为LmAbd-5和SgAbd-5,并对其氨基酸序列进行了初步分析[19],然而其生物学功能尚不清楚。【拟解决的关键问题】了解飞蝗蜕皮过程中内外表皮的形成规律,合理设计取样点研究LmAbd-5在不同发育时期的表达特性,明确该基因的表达与内表皮形成的相关性。进一步采用 RNAi技术和透射电镜技术分析其对飞蝗内表皮发育的影响,为筛选新型分子靶标提供理论和实践依据。
1 材料与方法
试验于 2015—2016年在山西大学应用生物学研究所完成。
1.1 试验材料
供试昆虫:将笔者实验室保存的飞蝗虫卵置于人工气候箱内孵化,待其孵化为1龄若虫后转移至纱笼内置于人工气候箱中,控制人工气候箱内环境:温度为(30±2)℃,相对湿度为(40±10)%,光周期为14 h﹕10 h(L﹕D),以新鲜小麦苗饲喂,3龄后开始辅以麦麸饲喂。
试验试剂:RNAisoTMPlus,cDNA反转录试剂购自TaKaRa公司;pMD18 Cloning Kit购自TransGen Biotech公司;SYBR®Premix EX TaqTM购自TaKaRa公司;PCR Master Mix购自天根公司;E.Z.N.A.® Gel Extraction Kit试剂盒购于Omega公司;T7 RiboMAXTMExpress RNAi System试剂盒购于Promega公司。
1.2 LmAbd-5序列搜索及cDNA全长序列获得
在笔者实验室构建的飞蝗转录组数据库中进行搜索,通过NCBI进行BLAST比对、分析,确定LmAbd-5的cDNA部分序列。根据已报道的飞蝗翅转录组数据库对LmAbd-5 cDNA序列进行比对分析[20],获得cDNA全长序列并设计引物验证(引物见表1,由上海生工生物公司合成),序列已上传至 NCBI (GenBank登录号:KX503039)。LmAbd-5的cDNA序列用ExPASy网站translate tool翻译后(http://web. expasy.org/translate/),SignaIp4.1Server对其信号肽进行分析(http://www.cbs.dtu.dk/services/ SignalP/),利用SMART分析其结构域(http://smart. embl-heidelberg. de/),利用WebLogo进行保守基序分析(http://weblogo. berkeley.edu/logo.cgi)。
1.3 昆虫Abd-5系统进化树的构建
从NCBI网站下载昆虫Abd-5氨基酸序列,使用MEGA 7.0软件中neighbor-joining(NJ)方法,将飞蝗LmAbd-5的氨基酸序列与其他昆虫Abd-5进行聚类分析,进行1 000次独立分析,数值代表bootstrap估算值。用于构建系统进化树的物种和 Abd-5的GenBank登录号见表2。
1.4 LmAbd-5 组织部位和发育时期表达特性
1.4.1 LmAbd-5的组织特异性表达 选用发育整齐的飞蝗5龄第2天若虫,雌、雄各半,设置3个生物学重复,每个重复4头虫体,解剖体壁、前肠、胃盲囊、中肠、后肠、马氏管、气管、脂肪体和翅芽等9个组织部位,冻存于液氮中。利用RNAisoTMPlus提取试剂(TaKaRa公司)提取上述不同组织的总RNA,1.5%的琼脂糖凝胶电泳检测所提取 RNA的质量,Nanodrop 2000对总RNA进行定量,然后以1 µg总RNA为模板,依据TaKaRa公司反转录试剂说明书合成cDNA模板。采用RT-qPCR检测LmAbd-5在飞蝗不同组织部位的表达情况,RT-qPCR反应体系为20 μL:2×SYBR®Premix EX TaqTM10 μL,50×ROX Reference Dye 0.4 μL,cDNA模板(20×)2 μL,上下游引物2 μL(2 μmol·L-1),补dH2O 至20 μL。扩增程序为95℃ 30 s,95℃ 5 s,60℃ 31 s,40个循环,熔解曲线程序为ABI Prism 7300 SDS 1.1软件自动添加。采用ABI Prism 7300 SDS 1.1软件对数据进行记录,以β-actin作为内参(KX276642),引物见表1(由上海生工生物公司合成)。
1.4.2 LmAbd-5在飞蝗5龄若虫体壁不同发育时期的表达特性 解剖5龄刚蜕皮(0 h)、蜕皮后24、48、72、96、120、144 和168 h若虫的第2、3腹节处体壁,并冻存于液氮中,4头虫体为1个生物学重复,共设3个重复。总RNA提取和cDNA模板合成、反应体系及方法同1.4.1。采用RT-qPCR检测LmAbd-5在飞蝗5龄若虫体壁不同发育时期的表达特性,引物见表1(由上海生工生物公司合成)。

表1 本研究中所用引物Table 1 Primer sequences used in this study

表2 本研究中用于构建系统进化树的物种和Abd-5的GenBank登录号Table 2 Species and GenBank accession number for phylogenetic tree used in this study
1.5 基于RNAi和透射电镜技术的生物学功能分析
根据LmAbd-5和绿色荧光蛋白(GFP)的cDNA序列,设计合成双链引物。使用PCR Master Mix(天根公司)进行PCR扩增,反应体系:12.5 μL PCR Master Mix,5 μL引物(上游/下游,2 μmol·L-1,引物见表1),2 μL表皮cDNA,加去离子水至25 μL,PCR程序:95℃预变性5 min;95℃ 15 s,60℃ 15 s,72℃ 30 s,72℃延伸10 min。产物用E.Z.N.A.®Gel Extraction Kit按照说明书进行回收,然后参照 T7 RiboMAXTMExpress RNAi System试剂盒说明书体外合成dsRNA。随机挑选发育整齐的飞蝗5龄第2天若虫进行 dsRNA注射,每头虫体注射 10 μg dsRNA,对照组注射等量dsGFP,每组注射30头虫体。正常饲喂至成虫第2天,观察表型并剪取腹部第2、3腹节表皮,同一虫体的表皮一部分用于提取总RNA后,反转录为cDNA模板,RT-qPCR检测沉默效率,另一部分(每头虫体均取相同部位)经3%戊二醛固定,用于透射电镜观察,透射电镜样品切片制备和染色等工作委托青岛大学医学院电镜室完成。
1.6 数据分析
LmAbd-5的相对表达量用2-ΔΔCt方法进行分析[21],采用student t-test方法进行表达差异分析,*表示差异显著(P<0.05),**表示差异极显著(P<0.01)。
2 结果
2.1 LmAbd-5的cDNA序列及基因结构分析
基于飞蝗转录组搜索获得 1条 LmAbd-5基因Unigene序列。根据已报道的飞蝗翅转录组数据库对LmAbd-5 cDNA序列进行比对分析,获得cDNA全长序列并进行克隆验证。结果显示,该基因 cDNA序列全长为520 bp,ORF长303 bp,编码100个氨基酸,预测等电点为4.58,分子量为10.7 kD,是一种偏酸性小分子量的表皮蛋白。结合飞蝗基因组序列对其基因结构进行分析[22],结果显示该基因含有3个外显子,与大多数物种表皮蛋白基因结构类似[9],第1外显子阻断信号肽(图1-A);将得到的LmAbd-5基因编码的氨基酸进行功能域分析,发现其含有1个信号肽和1个几丁质结合域ChtBD4(图1-B)。
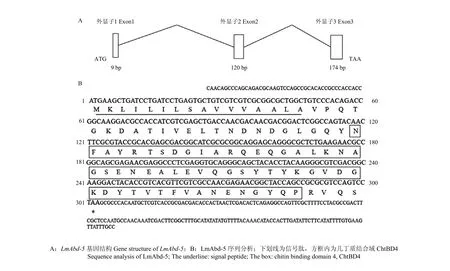
图1 LmAbd-5基因结构和序列分析Fig. 1 Gene structure and sequence analysis of LmAbd-5
2.2 LmAbd-5蛋白结构域分析、序列比对及系统进化分析
LmAbd-5的全长氨基酸序列信息及功能域分析见图 2,结果显示飞蝗与沙漠蝗、白蚁和赤拟谷盗等Abd-5类似,不同的是沙漠蝗SgAbd-5仅为部分序列,缺少信号肽(图2-A);通过NCBI进行BLAST分析显示飞蝗 LmAbd-5与沙漠蝗 SgAbd-5高度同源,氨基酸序列一致度高达81%,其中几丁质结合域序列完全一致(39—94 aa),与白蚁Abd-5一致度次之(70%),而与赤拟谷盗 Abd-5序列一致度较差(52%)(图2-B)。将其保守基序进行WebLogo分析发现,LmAbd-5属于表皮蛋白 CPR家族中的RR-1亚类,该基因的RR-1基序与前人在家蚕、按蚊和赤拟谷盗等昆虫中研究的 RR基序在关键氨基酸的位点上是保守的,但 RR基序之外的氨基酸位点变化较大(图2-C)。
从NCBI网站下载不同物种Abd-5氨基酸序列,物种名和登录号如表2所示,主要包括双翅目、鳞翅目、鞘翅目、蜚蠊目和直翅目昆虫。将得到的飞蝗LmAbd-5全长序列与其他物种Abd-5氨基酸全长序列构建系统发育树,聚类分析结果显示,双翅目和鳞翅目的Abd-5蛋白分别聚为一支,鞘翅目仅有赤拟谷盗一个Abd-5蛋白,直翅目飞蝗LmAbd-5与沙漠蝗、蜚蠊目白蚁的Abd-5聚为一支,并且与沙漠蝗Abd-5有最近的亲缘关系(图3)。
2.3 LmAbd-5的组织部位和发育时期表达特性
为了探讨LmAbd-5在飞蝗不同组织部位和发育时期的表达特性,首先利用 RT-qPCR方法对LmAbd-5在飞蝗5龄第2天若虫不同组织部位表达进行了分析(图4)。结果显示LmAbd-5在飞蝗前肠、后肠、气管和体壁等由外胚层形成的组织中高表达,而在胃盲囊、中肠、马氏管、脂肪体和翅芽中低表达或不表达(图4-A),表明LmAbd-5可能参与表皮的形成;其次,选用5龄不同发育时期体壁组织进行表达分析,发现LmAbd-5在4龄若虫蜕皮后0—72 h(5龄早期)具有高表达,其中蜕皮后72 h达到最大表达量,随后表达量急剧降低(蜕皮后96—168 h)(图4-B),LmAbd-5的表达时期与内表皮形成时间一致,推测其可能参与飞蝗内表皮结构的形成。

图2 LmAbd-5蛋白结构、序列比对和RR-1基序分析Fig.2 Domain structure and alignment of LmAbd-5 and analysis of RR-1 motifs
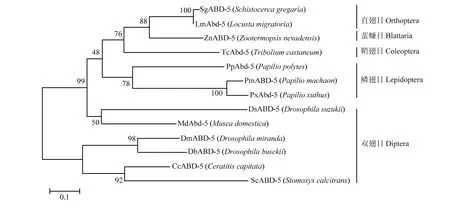
图3 LmAbd-5与其他昆虫Abd-5的系统聚类分析Fig. 3 The phylogenetic analysis of LmAbd-5 and Abd-5 from different insect species

图4 LmAbd-5在飞蝗 5 龄若虫期不同组织部位和不同时期的表达Fig. 4 The relative expression of LmAbd-5 in different tissues and different stages of the 5th instar nymphs
2.4 基于RNAi的LmAbd-5生物学功能分析
为了探讨LmAbd-5在飞蝗表皮形成中的作用,体外合成dsRNA并向5龄第2天若虫分别注射等量的dsLmAbd-5和dsGFP(对照),正常饲养至蜕皮后,观察成虫表型并检测沉默效率(图5-A),发现注射dsLmAbd-5的5龄若虫和对照组相同,均能正常蜕皮,尽管目的基因在成虫第2天表达量显著降低(处理组LmAbd-5表达量显著降低了85.5%),但未出现肉眼可见的异常表型。分别取处理组和对照组蜕皮至成虫第2天表皮进行超微结构观察。结果表明与对照组相比,注射 dsLmAbd-5的成虫内表皮片层结构疏松,导致内表皮片层变厚,使得整个内表皮变厚(图5-B)。
3 讨论
昆虫表皮具有保护和防御等功能,在昆虫生命过程中发挥着重要作用。昆虫表皮由上表皮、外表皮、内表皮和细胞层构成,各层表皮的成分和机械性能不同[23],其中上表皮和外表皮主要在蜕皮前合成和沉积,内表皮是在蜕皮后合成和沉积。昆虫表皮的主要成分是由几丁质和大量表皮蛋白构成,其中CPR家族是最大的表皮蛋白家族,包括RR-1、RR-2 和RR-3等3种类型,在双翅目、鳞翅目、鞘翅目、膜翅目、半翅目和直翅目等昆虫中均有发现[6]。早在1988年,REBERS等[24]最先鉴定了CPR家族保守基序(R&R motif)G-x(8)-G-x(6)-Y-x-A-x-E-x-GY-x(7)-P-x(2)-P(x表示氨基酸,括弧内的数字为该处x的数目)。

图5 注射dsLmAbd-5后LmAbd-5的相对表达量和蜕皮至成虫第2天的表皮超微结构观察Fig. 5 The relative expression of LmAbd-5 and ultrastructure observation of day 2 adult cuticle after injected with dsRNA in 5th instar nymphs
20世纪90年代,NOHR等[25]利用双向电泳技术,发现飞蝗内外表皮蛋白组成具有明显的差异性。随后,ANDERSEN[26]利用MALDI-MS技术,从沙漠蝗中分析鉴定出 8个内表皮蛋白,分别命名为 SgAbd-1、SgAbd-2、SgAbd-3、SgAbd-4、SgAbd-5、SgAbd-6、SgAbd-8和 SgAbd-9。随着昆虫基因组学的发展,越来越多昆虫的Abd-5被鉴定,如双翅目、鳞翅目、鞘翅目、蜚蠊目和直翅目等昆虫。然而,在这些昆虫中,Abd-5仅仅是序列被报道,具体功能尚未见深入研究。
本研究克隆获得飞蝗内表皮蛋白基因LmAbd-5,其编码的蛋白含有一个RR-1保守基序,属于CPR家族中 RR-1亚类。组织分布研究发现,其主要在外胚层形成的组织中高表达,如前肠、后肠、气管和体壁,而在胃盲囊、中肠、马氏管、脂肪体和翅芽中低表达或不表达,表明 LmAbd-5可能参与表皮的形成;发育研究显示其主要在蜕皮后72 h内具有高表达,而在96 h后表达急剧降低。飞蝗蜕皮后0—72 h是内表皮完全形成的时期,之后皮层出现溶离,新表皮开始合成。因此,LmAbd-5的表达与飞蝗内表皮合成和沉积时期一致,表明LmAbd-5可能是一种内表皮蛋白,参与昆虫内表皮的形成。有研究发现,RR-1亚类的表皮蛋白主要存在于柔软和具有韧性的水化角质层中,如双翅目和鳞翅目昆虫幼虫表皮和节间膜以及飞蝗的内表皮[27],昆虫内表皮中的几丁质和表皮蛋白通过共价键与氢键结合,不被鞣化。CPR家族的保守RR基序具有几丁质结合位点,能够结合几丁质,已在部分昆虫研究中得到证实[11-13]。而飞蝗表皮蛋白LmAbd-5是否能结合几丁质仍有待于进一步研究。
目前,RNAi技术是研究基因生物学功能的有效工具,笔者课题组利用RNAi在飞蝗几丁质合成、降解和表皮蛋白家族等方面开展了大量工作[28-30]。飞蝗对RNAi极为敏感,本研究采用RNAi技术对飞蝗5龄第2天若虫分别注射等量的dsLmAbd-5和dsGFP(对照),正常饲养观察飞蝗表皮发育情况,发现注射dsLmAbd-5的5龄若虫和对照组相同,均能正常蜕皮,尽管目的基因表达量显著降低,但未发现可见的异常表型。分别取dsLmAbd-5注射组蜕皮后2 d的成虫表皮(LmAbd-5表达量显著降低)及对照组成虫表皮进行透射电镜观察,发现与对照组相比,注射dsLmAbd-5的成虫内表皮片层结构较为疏松,导致内表皮片层变厚,表明LmAbd-5在飞蝗内表皮的形成中具有重要作用。在飞蝗和其他昆虫中,Abd是一个家族基因。根据节肢动物表皮蛋白数据库[31]和飞蝗转录组数据库,共发现包括LmAbd-5在内的8个Abd类型的表皮蛋白基因,然而其表达特性和功能以及它们相互之间是否具有协同或互补作用尚有待进一步的研究。
4 结论
经转录组数据库分析获得飞蝗LmAbd-5,其编码的蛋白含有1个信号肽和1个几丁质结合域ChtBD4,属于CPR家族中的RR-1亚类;LmAbd-5主要在外胚层起源的组织中高表达,发育表达特性表明该基因主要在内表皮形成时期表达;沉默LmAbd-5后飞蝗未出现肉眼可见的表型,但超微结构观察发现其参与飞蝗内表皮片层结构的形成。
[1] CHARLES J P. The regulation of expression of insect cuticle protein genes. Insect Biochemistry and Molecular Biology, 2010, 40(3): 205-213.
[2] CHAPMAN R F, SIMPSON S J, DOUGLAS A E. The Insects Structure and Function. 5th ed. Cambridge University Press, 2013.
[3] 王荫长. 昆虫生理学. 北京: 中国农业出版社, 2004. WANG Y C. Insect Physiology. Beijing: China Agriculture Press, 2004. (in Chinese)
[4] TETREAU G, DITTMER N T, CAO X, AGRAWAL S, CHEN Y R, MUTHUKRISHNAN S, HAOBO J, BLISSARD G W, KANOST M R, WANG P. Analysis of chitin-binding proteins from Manduca sexta provides new insights into evolution of peritrophin A-type chitinbinding domains in insects. Insect Biochemistry and Molecular Biology, 2015, 62: 127-141.
[5] REBERS J E, WILLIS J H. A conserved domain in arthropod cuticular proteins binds chitin. Insect Biochemistry and Molecular Biology, 2001, 31: 1083-1093.
[6] WILLIS J H. Structural cuticular proteins from arthropods: annotation, nomenclature, and sequence characteristics in the genomics era. Insect Biochemistry and Molecular Biology, 2010, 40(3): 189-204.
[7] KAROUZOU M V, SPYROPOULOS Y, ICONOMIDOU V A, CORNMAN R S, HAMODRAKAS S J, WILLIS J H. Drosophila cuticular proteins with the R&R consensus: Annotation and classification with a new tool for discriminating RR-1 and RR-2 sequences. Insect Biochemistry and Molecular Biology, 2007, 37(8): 754-760.
[8] GIBBS R A, BROWN S J, BEEMAN R W, WEINSTOCK1 G M. The genome of the model beetle and pest Tribolium castaneum. Nature, 2008, 452(7190): 949-955.
[9] FUTAHASHI R, OKAMOTO S, KAWASAKI H, ZHONG Y S, IWANAGA M, MITA K, FUJIWARA H. Genome-wide identification of cuticular protein genes in the silkworm, Bombyx mori. Insect Biochemistry and Molecular Biology, 2008, 38(12): 1138-1146.
[10] DITTMER N T, TETREAU G, CAO X, JIANG H, WANG P, KANOST M R. Annotation and expression analysis of cuticular proteins from the tobacco hornworm, Manduca sexta. Insect Biochemistry and Molecular Biology, 2015, 62: 100-113.
[11] DENG H M, LI Y, ZHANG J L, LIU L, FENG Q L. Analysis of expression and chitin-binding activity of the wing disc cuticle protein BmWCP4 in the silkworm, Bombyx mori. Insect Science, 2016, 23(6): 782-790.
[12] ARAKANE Y, ZHU Q, MUTHUKRISHNAN S, MATSUMIY M, KRAMER K J. Properties of catalytic, linker and chitin-binding domains of insect chitinase. Insect Biochemistry and Molecular Biology, 2003, 33: 631-648.
[13] WANG P, LI G, GRANADOS R R. Identification of two new peritrophic membrane proteins from larval Trichoplusia ni: structural characteristics and their functions in the protease rich insect gut. Insect Biochemistry and Molecular Biology, 2004, 34(3): 215-227.
[14] TAKEDA M, MITA K, QUAN G X, SHIMADA T, OKANO K, KANKE E, KAWASAKI H. Mass isolation of cuticle protein cDNAs from wing discs of Bombyx mori and their characterizations. Insect Biochemistry and Molecular Biology, 2001, 31(10): 1019-1028.
[15] NOH M Y, KRAMER K J, MUTHUKRISHNAN S, KANOST M R, BEEMAN R W, ARAKANE Y. Two major cuticular proteins are required for assembly of horizontal laminae and vertical pore canals in rigid cuticle of Tribolium castaneum. Insect Biochemistry and Molecular Biology, 2014, 53: 22-29.
[16] MUN S, YOUNG NOH M, DITTMER N T, MUTHUKRISHNAN S, KRAMER K J, KANOST M R, ARAKANE Y. Cuticular protein with a low complexity sequence becomes cross-linked during insect cuticle sclerotization and is required for the adult molt. Scientific Reports, 2015, 5: 10484.
[17] NOH M Y, MUTHUKRISHNAN S, KRAMER K J, ARAKANE Y. Tribolium castaneum RR-1 cuticular protein TcCPR4 is required for formation of pore canals in rigid cuticle. PLoS Genetics, 2015, 11(2): e1004963.
[18] ARAKANE Y, LOMAKIN J, GEHRKE S H, HIROMASA Y, TOMICH J M, MUTHUKRISHNAN S, BEEMAN R W, KRAMER K J, KANOST M R. Formation of rigid, non-flight forewings (Elytra) of a beetle requires two major cuticular proteins. PLoS Genetics, 2012, 8(4): e1002682.
[19] JESPERSEN S, HOJRUP P, ANDERSENT S O, ROEPSTORFF P. The primary structure of an endocuticular protein from two locust species, Locusta migratoria and Schistocerca gregaria, determined by a combination of mass spectrometry and automatic Edman degradation. Comparative Biochemistry and Physiology - Part B: Biochemistry & Molecular Biology, 1994, 109(1): 125-138.
[20] LIU S, WEI W, CHU Y, ZHANG L, SHEN J, AN C. De novo transcriptome analysis of wing development related signaling pathways in Locusta migratoria Manilensis and Ostrinia furnacalis (Guenée). PLoS ONE, 2014, 9(9): e106770.
[21] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔ Ctmethod. Methods, 2001, 25(4): 402-408.
[22] WANG X, FANG X, YANG P, JIANG X, JIANG F, ZHAO D, LI B, CUI F, WEI J, MA C, WANG Y, HE J, LUO Y, WANG Z, GUO X, GUO W, WANG X, ZHANG Y, YANG M, HAO S, CHEN B, MA Z, YU D, XIONG Z, ZHU Y, FAN D, HAN L, WANG B, CHEN Y, WANG J, YANG L, ZHAO W, FENG Y, CHEN G, LIAN J, LI Q, HUANG Z, YAO X, LV N, ZHANG G, LI Y, WANG J, WANG J, ZHU B, KANG L. The locust genome provides insight into swarm formation and long-distance flight. Nature Communications, 2014, 5: 2957.
[23] MOUSSIAN B, SEIFARTH C, MULLER U, BERGER J, SCHWARZ H. Cuticle differentiation during Drosophila embryogenesis. Arthropod Structure & Development, 2006, 35(3): 137-152.
[24] REBERS J E, RIDDIFORD L M. Structure and expression of a Manduca sexta larval cuticle gene homologous to Drosophila cuticle genes. Journal of Molecular Biology, 1988, 203(2): 411-423.
[25] NOHR C, ANDERSEN S O. Cuticular proteins from fifth instar nymphs of the migratory locust, Locusta migratoria. Insect Biochemistry and Molecular Biology, 1993, 23(4): 521-531.
[26] ANDERSEN S O. Amino acid sequence studies on endocuticular proteins from the desert locust, Schistocerca gregaria. Insect Biochemistry and Molecular Biology, 1998, 28: 421-434.
[27] NOH M Y, MUTHUKRISHNAN S, KRAMER K J, ARAKANE Y. Cuticle formation and pigmentation in beetles. Current Opinion Insect Science, 2016, 17: 1-9.
[28] 刘晓健, 崔淼, 李大琪, 张欢欢, 杨美玲, 张建珍. 飞蝗几丁质合成酶2基因的表达特性、功能及调控. 中国农业科学, 2014, 47(7): 1330-1340. LIU X J, CUI M, LI D Q, ZHANG H H, YANG M L, ZHANG J Z. Expression, function and regulation of chitin synthase 2 gene in Locusta migratoria. Scientia Agricultura Sinica, 2014, 47(7): 1330-1340. (in Chinese)
[29] LI D, ZHANG J, WANG Y, LIU X, MA E, SUN Y, LI S, ZHU K Y, ZHANG J. Two chitinase 5 genes from Locusta migratoria: Molecular characteristics and functional differentiation. Insect Biochemistry and Molecular Biology, 2015, 58: 46-54.
[30] 王燕, 李大琪, 刘晓健, 李涛, 马恩波, 范仁俊, 张建珍. 飞蝗表皮蛋白Obstructor家族基因的分子特性及基于RNAi的功能分析. 中国农业科学, 2015, 48(1): 73-82. WANG Y, LI D Q, LIU X J, LI T, MA E B, FAN R J, ZHANG J Z. Molecular characterization and RNAi-based functional analysis of Obstructor family genes in Locusta migratoria. Scientia Agricultura Sinica, 2015, 48(1): 73-82. (in Chinese)
[31] MAGKRIOTI C K, SPYROPOULOS I C, ICONOMIDOU V A, WILLIS J H, HAMODRAKAS S J. cuticleDB: a relational database of Arthropod cuticular proteins. BMC Bioinformatics, 2004, 5: 138.
(责任编辑 岳梅)
Expression and Functional Analysis of Endocuticle Structural Glycoprotein Gene LmAbd-5 in Locusta migratoria
ZHAO XiaoMing1, JIA Pan1,2, GOU Xin1,2, LIU WeiMin1, MA EnBo1, ZHANG JianZhen1
(1Research Institute of Applied Biology, Shanxi University, Taiyuan 030006;2College of Life Science, Shanxi University, Taiyuan 030006)
Locusta migratoria; cuticular proteins; LmAbd-5; RNAi; transmission electron microscopy
2016-11-28;接受日期:2017-03-24
国家自然科学基金(31640075,31672364)、山西省青年科学基金(201601D021102)、山西省高校科技创新基金(2016113)
联系方式:赵小明,Tel:0351-7018871;E-mail:zxming@sxu.edu.cn。通信作者张建珍,Tel:0351-7018871;E-mail:zjz@sxu.edu.cn
Abstract:【Objective】The objective of this study is to obtain a cDNA sequence of endocuticle structural glycoprotein LmAbd-5 based on Locusta migratoria transcriptome, clarify its molecular characteristics and biological function, reveal its role in the formation of cuticle in L. migratoria, and provide a new molecular target for pest control. 【Method】The full length cDNA of LmAbd-5 was searched from transcriptome database of L. migratoria using bioinformatics method. The cDNA was cloned and sequenced. The signal peptide and function domain of deduced amino acid were analyzed by SignalP and SMART, respectively. Phylogenetic tree was constructed using the sequences of amino acid from different insect species by the MEGA 7.0 software with the neighbor-joining (NJ) method. Reverse transcription quantitative PCR (RT-qPCR) was applied to reveal the expression patterns of LmAbd-5 in different tissues on day 2 of 5th instar nymph and different developmental stages of integument. The effects of LmAbd-5 on locust growth development and the structure of cuticle were investigated by using RNA interference (RNAi) and transmission electron microscopy (TEM).【Result】The full length cDNA of LmAbd-5 was got from transcriptome database, which had 520 bp including ORF 303 bp. The gene structure analysis showed that LmAbd-5 has three exons. The deduced protein contains a signal peptide and one chitin binding domain 4 (ChtBD4) through the BLAST analysis, Abd-5 was highly conserved among insect species, and the sequence identity is as high as 81% between LmAbd-5 and SgAbd-5. Abd-5 belongs to the RR-1 class of CPR family by WebLogo analysis. The result of phylogenetic tree showed that LmAbd-5 has a close genetic relationship with SgAbd-5. RT-qPCR results showed that LmAbd-5 was predominately expressed in the tissues originated from ectoderm, such as the foregut, hindgut, trachea and integument, and lower expressed or not detected in the gastric caecum, midgut, Malpighian tube, fat body and wing pads. The expression at different stages showed that LmAbd-5 mainly expressed at early of 5th instar (0-72 h after ecdysis from 4th instar nymph), and up to the peak at 72 h after molting, then markedly decreased at 96-168 h. The expression pattern is related with the formation of endocuticle. Compared with dsGFP injected control, the nymphs with the injection of dsLmAbd-5 could normally molt, and no visible abnormal phenotypes was found although the expression of LmAbd-5 was decreased significantly after dsLmAbd-5 injection. However, compared to the control group, the lamellar structure from adult cuticle with injection of dsLmAbd-5 was loose, and lamellar became thicker, finally led to the endocuticle thickening.【Conclusion】LmAbd-5 was obtained from locust transcriptome database, which contains a signal peptide and ChtBD4, belonging to the RR-1 class of CPR family. LmAbd-5 mainly expressed in the tissues derived from ectoderm and in integument at early of 5th instar. Although there was no visible phenotypes after silencing LmAbd-5, but it was found that the lamellar structure of endocuticle is loose and endocuticle becomes thicken from ultrastructure by TEM, suggesting it may be participated in the formation of endocuticle in L. migratoria.

