秸秆蓝藻混合发酵产沼气过程中微生物群落分析
穆维娜, 李玉成, 武 超, 张学胜, 王 宁, 高兆慧
(安徽大学 资源与环境工程学院,安徽 合肥 230601)
秸秆蓝藻混合发酵产沼气过程中微生物群落分析
穆维娜, 李玉成*, 武 超, 张学胜, 王 宁, 高兆慧
(安徽大学 资源与环境工程学院,安徽 合肥 230601)
采用实验室自制秸秆蓝藻混合厌氧反应装置进行沼气发酵实验,利用16S rRNA基因克隆文库的方法,对不同产气阶段的细菌和古菌的优势菌群进行多样性研究。结果表明:①不同产气阶段细菌优势种类存在差异,供试秸秆沼气反应器阶段细菌种类较为丰富,分属于6个门:产气初始阶段优势菌群为变形菌门(Proteobacteria),相对丰度为51.76%;产气高峰阶段优势菌群为厚壁菌门(Firmicutes),相对丰度为47.13%;产气结束阶段优势菌群为厚壁菌门(Firmicutes),相对丰度为28.89%;此外,还包括绿弯菌门、螺旋体门、绿菌门的细菌。②古菌种类明显少于细菌,均属于甲烷微菌纲(Methanomicrobia)、甲烷杆菌纲(Methanobacteria)和热原体纲(Thermoplasmata)。秸秆蓝藻沼气系统微生物群落结构的阐明具有一定的意义,可为秸秆沼气工程调控提供科学依据。
秸秆;混合发酵;微生物群落;克隆文库
安徽省作为农业大省,每年都有大量的秸秆产生,主要农作物秸秆年理论资源量达4 487.99万t,年可收集资源量达3 878.84万t[1]。秸秆作为农业生产中的副产品是一种容易获取的生物能源,然而大量的秸秆被随意丢弃和焚烧,造成了极大的资源浪费和环境污染[2]。巢湖是中国第五大淡水湖,连通长江,巢湖的水质不仅会影响到其周边环境,还会影响到整个长江流域。巢湖水体近年来富营养化严重,蓝藻暴发成为环境问题。蓝藻含丰富的有机质,是一种生物质资源[3]。厌氧发酵可以使秸秆和蓝藻转化为清洁能源,既保护了环境也使资源得到了利用[4]。秸秆是一种富含木质纤维素与其他多糖的有机质,而蓝藻中含丰富的蛋白质和藻多糖,利用含氮量比较丰富的蓝藻调节底物的碳氮比,可以提高发酵的效率[5-6]。彭书传等利用玉米秸秆和巢湖蓝藻进行混合厌氧发酵,与单一的玉米秸秆或蓝藻发酵过程相比,玉米秸秆和蓝藻混合发酵能够有效促进沼气的生成[7]。有机物厌氧降解过程主要分为水解发酵、产氢产乙酸及产甲烷3个阶段。其中水解发酵与产氢产乙酸阶段主要由细菌参与,涉及一系列复杂的化学代谢过程,生成乙酸等挥发性有机酸;而产甲烷阶段主要由甲烷古菌参与,将乙酸转化为甲烷[8]。沼气发酵过程需要不同阶段的微生物相互协作完成,因此沼气发酵过程中的微生物群落结构和功能的解析已经成为研究的热点[9]。目前国内外关于利用秸秆进行发酵的微生物群落研究主要集中在秸秆和禽畜粪便等常规原料的混合上[10],而以秸秆和蓝藻混合发酵为对象的研究鲜有报道。分子生物学技术在微生物鉴定方面的应用,可以研究微生物在厌氧发酵过程中变化情况[11-13]。本文在秸秆蓝藻混合厌氧产气的基础上,通过构建16S rRNA基因克隆文库,了解混合发酵不同阶段的细菌和古菌的优势菌群及其变化规律,为调控混合发酵过程提供微生物基本信息。结合不同产气阶段的产气率,解析沼气发酵各阶段优势微生物的功能,为优化秸秆沼气发酵工艺提供理论依据。
1 材料与方法
1.1 材料
1.1.1 实验材料 实验所用水稻秸秆采自安徽省周边某稻田,总固体(TS)含量为91.1%,挥发性固体(VS)含量为85.9%,晒干后剪至1 cm左右待用;新鲜蓝藻采自巢湖塘西河口,总固体(TS)含量为1.96%,挥发性固体(VS)含量为91.58%,经滤网滤水后装入自封袋置于冰箱冷冻备用,实验时常温解冻待用;接种污泥采自实验室35 ℃中温厌氧发酵反应器剩余污泥,TS=3.9%,VS=72.51%,pH=7。
1.1.2 实验装置 实验装置主要由发酵瓶、集气瓶、集水瓶组成(见图1)。
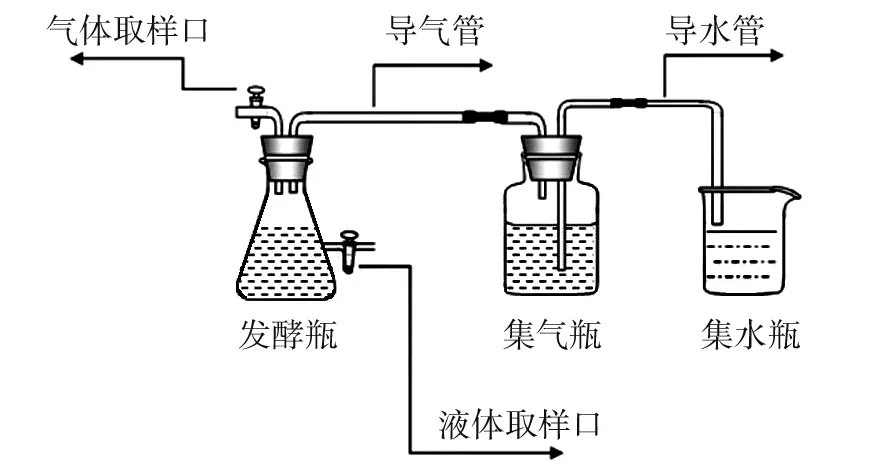
图1 厌氧发酵装置图Fig.1 Schematic diagram of bio-gas fermentation system
1.2 方法
1.2.1 发酵与取样 将处理后秸秆与解冻新鲜蓝藻按碳氮比为25∶1混合成发酵原料。将原料分别置于4 L厌氧发酵罐中,加入500 mL厌氧污泥,配水至2.5 L,调节反应总底物的pH至(7.0±0.05)。混匀后装入反应器内,用氮气对整个装置进行吹氮除氧0.5 h以上。密封后进行厌氧发酵,发酵温度35 ℃,每天保证人为手动振荡摇晃发酵瓶2~3次,防止颗粒污泥沉淀或秸秆蓝藻浮于液面。共采样3次,分别在第1、7、29天取样,将样品依次命名为d1、d7、d29。样品采集前,先排空采样管道中残余的厌氧污泥,然后再将厌氧污泥向外流动几分钟后从反应器中取固液混合样30 mL装入无菌聚丙烯离心管中,冻存于-80 ℃超低温冰箱。
1.2.2 常规测试方法 TS:105 ℃烘至恒重;VS:于550 ℃马弗炉中烧至恒重;沼气产量:采用水压集气法,每日测量排出的饱和食盐水总量;甲烷含量:采用安捷伦7890A气相色谱测定。
1.2.3 基因组DNA提取 基因组DNA的提取采用QIAamp DNA Stool mini kit基因组提取试剂盒。为了保证提取样品的基因组DNA的代表性,每个厌氧污泥样品平行提取3次,通过1%琼脂糖凝胶电泳对基因组DNA的提取结果进行检测。
1.2.4 克隆文库构建 用16S rRNA基因通用引物分别对3个样品中的细菌和古菌的16S rRNA基因片段进行PCR扩增。其中细菌引物序列为27F(5′-AGAGTTTGATCCTGGCTCAG-3′),1492R(5′-TACG GCTACCTTGTTACGACTT-3′);古菌引物序列为AR109F(5′-ACKGCTCAGTAACACGT-3′),AR-912r(5′-CTCCCCCGCCAATTCCTTTA-3′)。PCR反应体系与反应条件:预变性94 ℃预变性4 min,94 ℃变性1 min,56 ℃退火1 min,72 ℃延伸2 min(古菌16S rRNA基因延伸时间1 min),共35个循环;最后72 ℃延伸10 min;4 ℃保存。扩增产物进行1%琼脂糖凝胶电泳,切取目的片段并用胶回收试剂盒(TAKARA,大连)进行纯化。纯化的PCR扩增产物用pUC-mT载体(生工,上海)依操作说明进行连接,采用热激法将连接产物转化至Trans 1大肠埃希菌感受态细胞(全式金,北京)中。通过蓝白斑筛选阳性克隆对于每个克隆文库,随机挑取96个白色克隆子进行PCR鉴定,引物采用M13F/M13R。
1.2.5 酶切分型与测序 将鉴定为阳性PCR产物的片段用HhaI和Hind III限制性内切酶消化(37 ℃),酶切产物用1.5%琼脂糖凝胶电泳检测,据酶切电泳图,将酶切图谱完全一致的克隆子划分为1个操作分类单元(operational taxonomic unit,OTU)[14],统计各OTU所含阳性克隆的数量,每个酶切分型选取一个代表克隆送至生工生物工程(上海)有限公司进行测序。获得的16S rRNA基因序列与测定序列在GenBank中进行BLAST同源性检索,使用MEGA 6.06软件进行系统发育分析。
2 结果与分析
2.1 秸秆蓝藻混合发酵过程曲线
发酵期间日产气量及每日甲烷含量如图2所示。反应开始1 d快速产气,7 d达到产气高峰(397 mL)。然后随着原料的减少而逐渐下降,在12 d达到第2次产气高峰(300 mL)。甲烷浓度在9 d达到63%,随后小幅回落后上升,并保持50%以上;前12 d为单日产气量较高,12 d后进入缓慢增长期;推测在发酵12 d后,系统中易产沼气的物质基本利用完毕,后期主要靠分解难降解物质来产沼气。29 d产气基本停止。

图2 秸秆和蓝藻混合发酵产气量及甲烷含量Fig.2 Bio-gas production and methane percentage composition in the fermentation mixed straw with algae
2.2 总DNA提取和PCR扩增
图3为蓝藻秸秆厌氧产气装置中样品总DNA提取电泳图,所用Marker为全式金的Trans15k,图中泳道1~3、4~6、7~9依次为样品d1、d7、d29平行样。由图3可知,提取的基因组较完整,大小在20 kb左右,可进行下一步的16S rRNA基因片段扩增。图4为样品d7总基因组细菌16S rRNA基因PCR扩增电泳图,所用Marker为全式金的Trans2K Plus,图中泳道1、2为电泳试验平行样。电泳结果表明PCR扩增所产生的DNA片段大小约为1.6 kb,说明得到的PCR扩增产物适合下一步的16S rRNA基因克隆文库的构建。
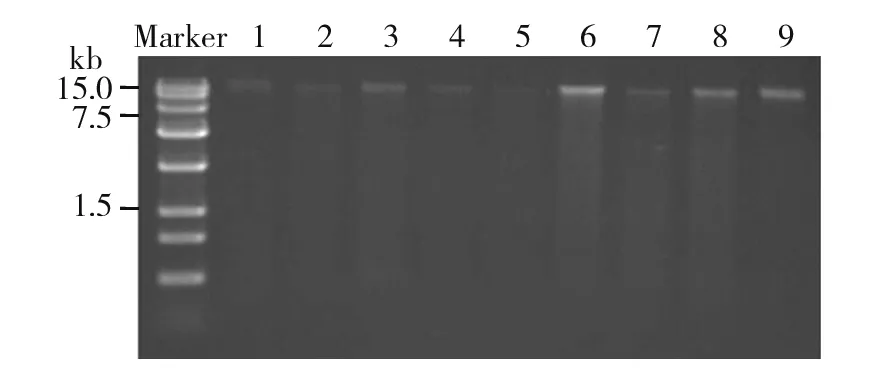
图3 样品总基因组提取电泳图Fig.3 Electrophoretogram of total genome
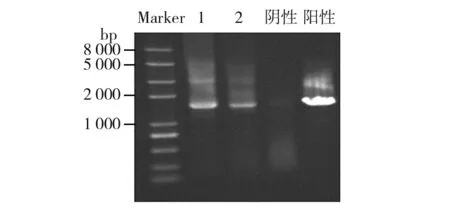
图4 样品d7总基因组细菌16S rRNA基因扩增产物电泳图Fig.4 Electrophoretogram of d7 genome bacterial 16S rRNA genes amplification products
2.3 细菌种群群落分析
样品d196个克隆子阳性克隆85个,共有9个操作分类单元(OTU);样品d796个克隆子阳性克隆87个,共有15个操作分类单元(OTU);样品d2996个克隆子阳性克隆90个,共有10个操作分类单元(OTU)。测得有效克隆子16S rRNA基因序列在GenBank数 据 库 中 进 行BLAST比对结果见表1。
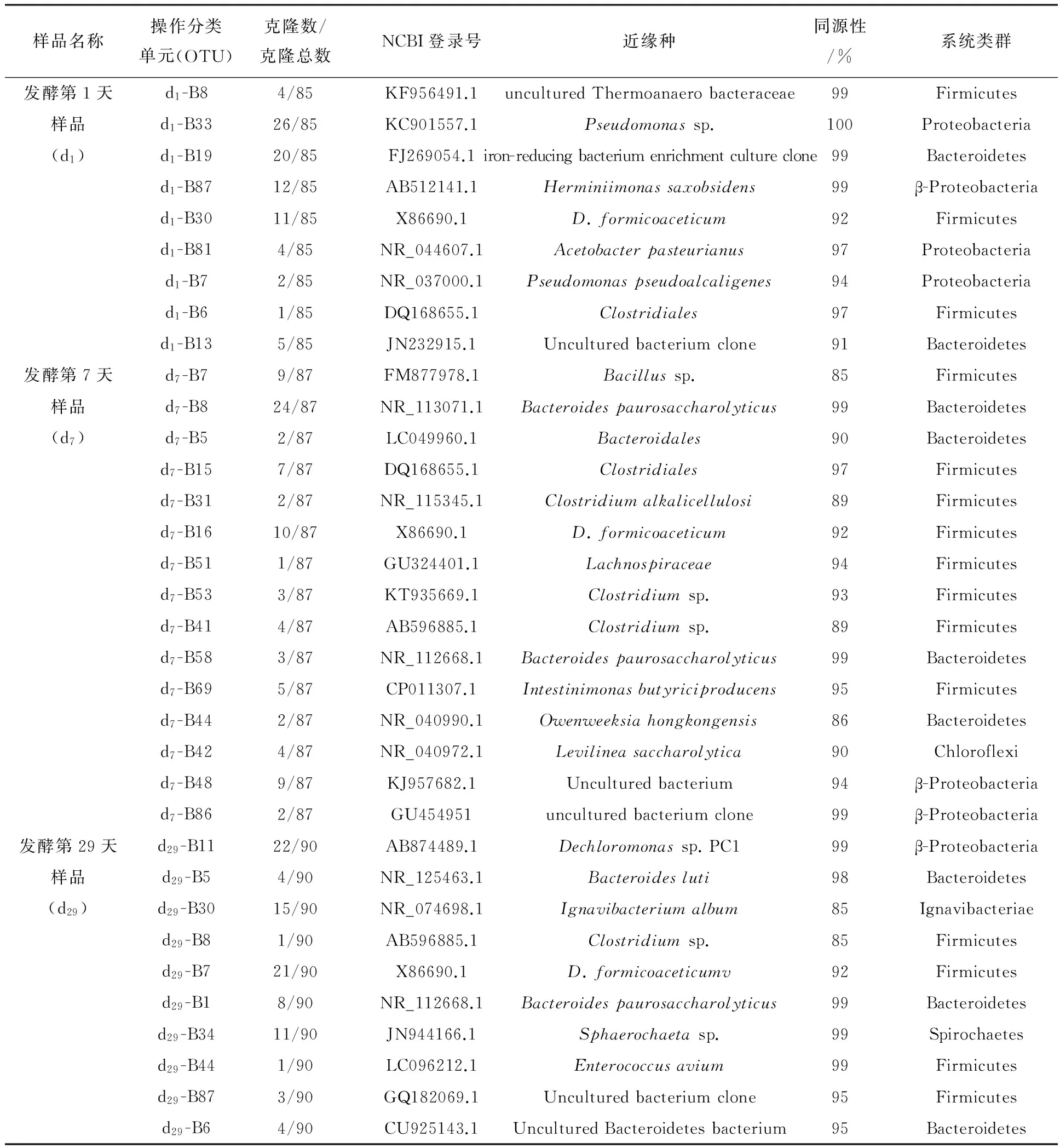
表1 细菌16S rRNA基因文库克隆子在数据库中的相似性比对结果
由表1可知,随机挑选的克隆子与GenBank数据库中已知细菌的序列同源性最高为100%,最低为85%。由比对结果发现,厌氧颗粒污泥中微生物种群丰富,共分6个类群(见表2),分别为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、绿弯菌门(Chloroflexi)、螺旋体门(Spirochaetes)、绿菌门(Ignavibacteriae)。其中厚壁菌门、变形菌门与拟杆菌门为主要优势菌群,分别占3个样品细菌克隆子总数18.82%~47.13%、12.64%~51.76%、13.33%~35.63%。系统中微生物以变形菌和不可培养细菌为主。
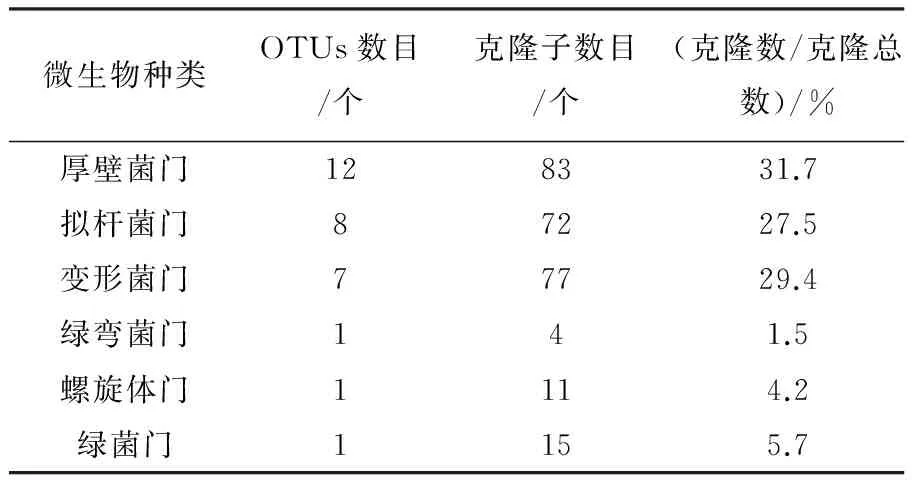
表2 反应器中所有细菌所属门类分布情况
通过分析不同发酵时间样品细菌群中丰度最大的OTU,可以了解不同发酵时间的重要细菌。3个不同发酵时间细菌群中丰度最大的OTU是不同的(见图5)。d1细菌群中丰度最大的OTU为Pseudomonassp.,属于变形菌门,占样品d1总克隆子数的30.6%。Pseudomonassp.分离自土壤,为短杆状,革兰阴性,好氧菌,生长温度为4~65 ℃,可利用葡萄糖,不能水解淀粉[15]。d7细菌群中丰度最大的OTU为Bacteroidespaurosaccharolyticus,属于拟杆菌门,占样品d7总克隆子数的27.6%。Bacteroidespaurosaccharolyticus分离自稻草残渣,革兰阴性,严格厌氧菌,生长温度为10~40 ℃,最优35 ℃,可优先利用阿拉伯糖、木糖、葡萄糖等,麦芽糖、糊精、糖原、淀粉和果胶也可利用,代谢产生乙酸、丙酸和琥珀酸[16]。d29细菌群中丰度最大的OTU为Dechloromonassp. PC1和D.formicoaceticum,D.formicoaceticum属于厚壁菌门,占样品d29总克隆子数的23.3%,是一种严格厌氧的革兰阳性细菌,可利用甲酸、乙酸和少量的甲醇[17];Dechloromonassp. PC1属于变形菌门,占样品d29总克隆子数的24.4%,为革兰阴性短杆菌,微好氧,最适条件为25 ℃,pH 7。
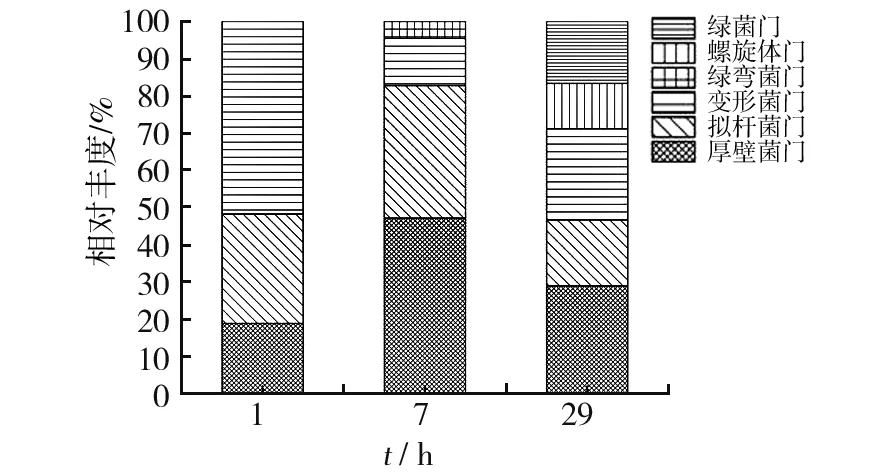
图5 不同发酵时间样品细菌组成Fig.5 Bacterial compositions in samples collected at different fermentation time
变形菌门是产气初始阶段的优势菌群。该门菌的纯培养主要分离自土壤、粪便、厌氧活性污泥等,可利用淀粉、长链脂肪酸及氨基酸等,具有水解作用,部分细菌有脱氮作用[18]。结合发酵装置中的物料及接种物,推测该菌很可能来自厌氧污泥或蓝藻;拟杆菌门是产气高峰阶段的优势菌群。该门菌主要分离自海底、肠道、厌氧反应器等厌氧环境,有降解大分子碳水化合物产酸的功能[19];厚壁菌门是产气终止阶段的优势细菌种群,该门细菌已在厌氧消化污泥、废水处理反应器[20]、玉米秸秆厌氧反应器[21]等环境中被大量发现,主要进行纤维素降解、有机物水解、长链脂肪酸降解,生成小分子物质。
2.4 古菌种群群落分析
样品d196个克隆子阳性克隆85个,共有8个操作分类单元(OTU);样品d796个克隆子阳性克隆有84个,共有6个操作分类单元(OTU);样品d2996个克隆子阳性克隆80个,共有4个操作分类单元(OTU)。测得有效克隆子16S rRNA基因序列在GenBank数 据 库 中 进 行BLAST比对,其微生物所属古菌类别较为丰富(见表3)。在科和属分类水平上(见表4),11个古菌OTUs中可分为Methanoregula、甲烷杆菌属(Methanobacterium)、甲烷微菌属(Methanosphaerula)、甲烷八叠球菌属(Methanosarcina)、甲烷鬃菌属(Methanosaeta)、甲烷绳菌属(Methanolinea)、马氏甲烷球菌科(Methanomassiliicoccus)与甲烷螺菌属(Methanospirillum)、甲烷杆菌科(Methanobacteriaceae)。其中,Methanoregula和甲烷八叠球菌属为文库中的优势克隆,分别占克隆总数的15.1%和13.2%。
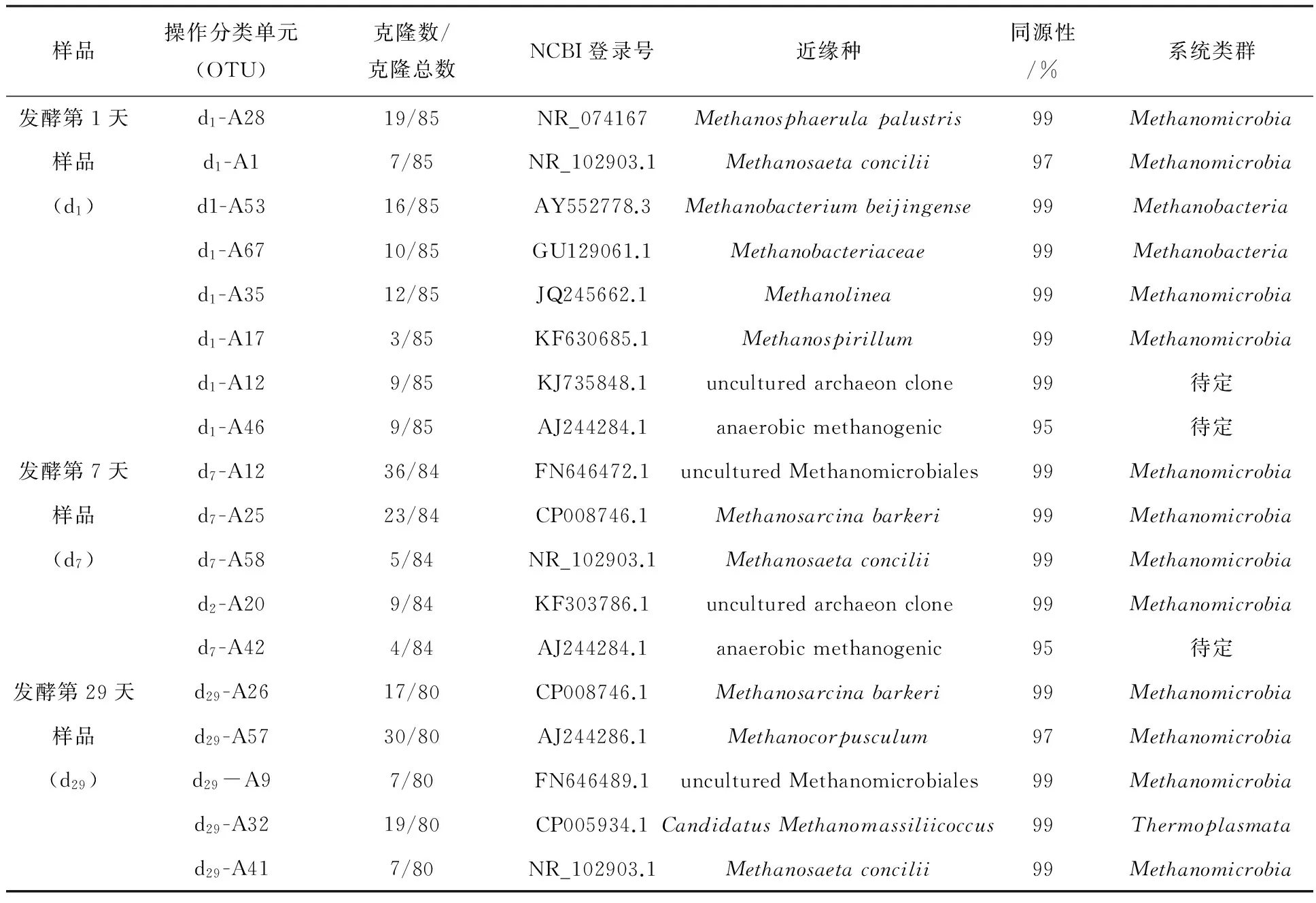
表3 古菌16S rRNA基因文库克隆子在数据库中的相似性比对结果
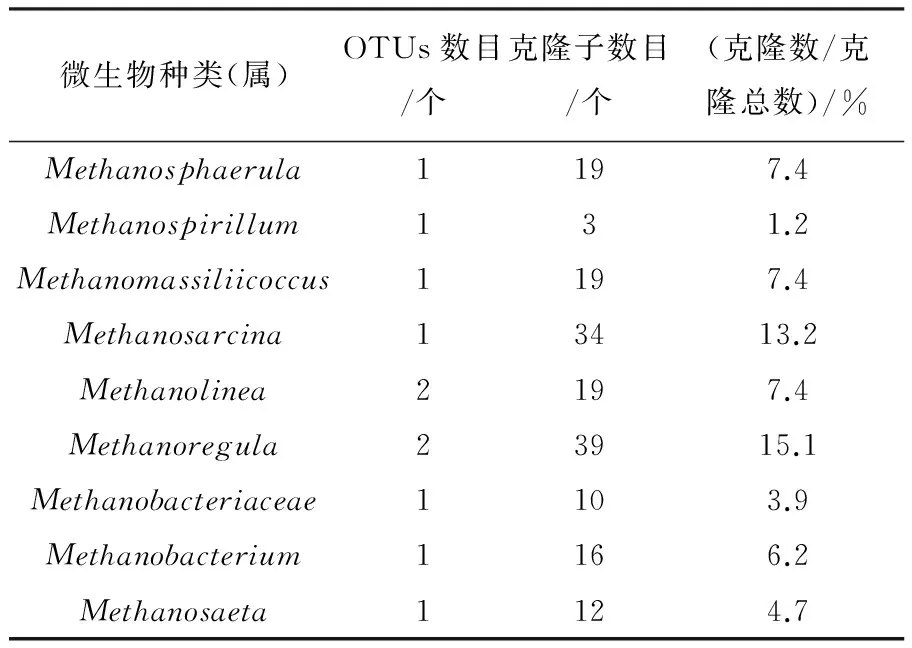
表4 古菌的16S rRNA基因文库中克隆子的分布情况
通过分析不同发酵时间样品古菌群中丰度最大的OTU,可以了解不同发酵时间的重要古菌。各样品在纲(Class)水平上的组成情况是不同的(见图6)。甲烷微菌在d1系统中占48.2%,到d7迅速升高至95.2%,d29后又有所下降,但仍占76.3%。甲烷微菌呈短、弯杆,不产芽胞,革兰阴性,极端严格厌氧。最适生长温度40 ℃,最适pH 6.1~6.9。以H2或甲酸盐为电子供体还原CO2为CH4,不利用乙酸、甲基胺和甲醇、H2,可在瘤胃液中分离到。甲烷微菌纲含量变化规律与沼气中的甲烷含量变化规律相似,都是从d1后迅速上升并维持在高位,说明甲烷微菌纲古菌在该混合发酵系统中起主要作用。甲烷杆菌丰度则从d1的30.6%迅速下降到d7的0,随后d29也未检测出。甲烷杆菌极端严格厌氧,最适生长温度是37 ℃,可还原CO2为CH4,不能代谢甲基胺和乙酸,不能固氮,可在水田、沉积物或其他缺氧的环境分离到,推测装置中甲烷杆菌来自厌氧污泥。热原体纲在d1和d7未检测出,而在d29丰度较高,占23.8%。
在属(Genus)分类水平上,d1古菌群中丰度最大的2个OTU依次属于甲烷微菌属(22.3%)、甲烷杆菌(18.8%);d7古菌群中丰度最大的2个OTU为Methanoregula(42.9%)、甲烷八叠球菌属(27.4%);d29古菌群中丰度最大的2个OTU为甲烷粒菌属(37.5%)、马氏甲烷球菌科(23.8%)。
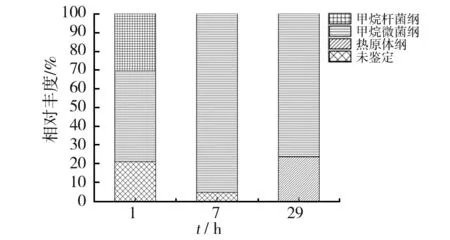
图6 不同发酵时间样品古菌组成Fig.6 Archaeal compositions in samples collected at different fermentation time
3 讨 论
本研究首先采用实验室自制秸秆蓝藻混合厌氧反应装置进行沼气发酵实验,再分别采集产气初始期(d1)、产气高峰期(d7)和产气终止期(d29)的厌氧发酵液,利用16S rRNA基因克隆文库的方法,对不同产气阶段的细菌和古菌进行多样性研究。尽管利用该技术不能反映样品中所有的微生物类群,但可以反映样品中主要的优势细菌、古菌类群。
系统发育分析表明,整个发酵过程中共检测到6个门类的细菌,即厚壁菌门(31.7%)、拟杆菌门(27.5%)、变形菌门(29.4%)、绿弯菌门(1.5%)、螺旋体门(4.2%)、绿菌门(5.7%)。这与其他秸秆产气装置典型类群一致。例如,张蕾等[19]应用16S rRNA基因克隆文库技术对四种规模化秸秆产气装置中微生物的群落分析中发现,厚壁菌门、变形菌门与拟杆菌门为优势种群。Yan等[22]发现稻草沼气系统细菌主要归属于拟杆菌门、变形菌门、脱铁杆菌门(8.9%)和厚壁菌门。Klocke等[23]发现青贮甜菜沼气系统中优势细菌主要归属于厚壁菌门、变形菌门和拟杆菌门。乔江涛等[9]对玉米秸秆厌氧发酵过程的细菌群落结构进行研究,结果发现厚壁菌门、拟杆菌门、互养菌门及热袍菌门为优势菌群。d1细菌群中丰度最大的OTU属于变形菌门(30.6%),d7细菌群中丰度最大的OTU属于拟杆菌门(27.6%),d29细菌群中丰度最大的OTU属于变形菌门(24.4%),这与袁月祥等[21]对玉米秸秆产生物燃气及其微生物群落解析结果相比发现,产气初始和高峰优势菌群一致,而产气终止的优势菌群不同,推测是由于物料中添加蓝藻,发酵装置含氮量升高导致变形菌门丰度随之升高。这些沼气系统中丰度最大的几个门基本上都是拟杆菌门、厚壁菌门和变形菌门,但含量有些变化,且不同细菌在发酵过程中的丰度变化趋势不同。以上结果表明,拟杆菌门和厚壁菌门在富含纤维素的有机废弃物厌氧发酵过程中发挥着重要作用,需要结合反应器的运行参数与运行状况,进一步研究这些优势微生物在沼气发酵过程中群落演替与代谢功能。
通过构建古菌克隆文库,得知各样品所测得的OTU都归为广古菌门。分别属于甲烷微菌纲、甲烷杆菌纲和热原体纲。其丰度变化与袁月祥等[21]对玉米秸秆产生物燃气及其微生物群落解析中的古菌含量随发酵时间变化情况基本吻合。张蕾等[19]在规模化秸秆沼气发酵反应器中微生物群落特征时,古菌序列均属于甲烷杆菌纲和甲烷微菌纲。杨承剑等[24]应用16S rRNA基因克隆文库技术分析广西富钟水牛瘤胃产甲烷菌组成及多样性中发现,所有古菌群落均属于甲烷杆菌目、热原体目、除硫球菌目。以上实验均是只研究了一个时间点,本实验研究了混合发酵系统中古菌的变化情况。通过比较推测,蓝藻的添加对厌氧发酵产沼气装置中古菌群落影响不大,不同发酵时间古菌变化趋势与以秸秆为单一物料产沼气的趋势基本相同。不同发酵原料对沼气系统古菌的多样性的影响有待进一步研究。
[1] 鲍恩财,田争光,刘伟伟,等.主要农作物秸秆资源调查及能源化利用评价——以安徽省为例[J].中国农学通报,2014,30(29):222-228.
[2] 孙晓艳.秸秆焚烧的危害及综合利用研究[J].绿色科技,2015, (3):222-224.
[3] 李媛,张家卫,魏杰,等.我国蓝藻水华的发生机理、危害及防控利用研究进展[J].微生物学杂志,2015, 35(8):93-97.
[4] 徐双锁,刘爱民,蔡欣,等.稻秸与蓝藻混合厌氧发酵产沼气试验研究[J].安徽师范大学学报,2011,34(3):260-264.
[5] Westerholm M, Hansson M,Schnürer A. Improved bio-gas production from whole still-age by co-digestion with cattle manure[J]. Bioresour Technol, 2012, 114:314-319.
[6] 刘战广,朱洪光,王彪,等.粪草比对干式厌氧发酵产沼气效果的影响[J].农业工程学报,2009, 12(4):196-200.
[7] 彭书传,侯成虎,王进,等.玉米秸秆与巢湖蓝藻混合厌氧发酵的产沼气性能[J].农业工程学报,2012,(15):173-178.
[8] Carina S, Waleed A, Madeleine L, et al. 454 pyrosequencing analyses of bacterial and archaeal richness in 21 full-scale biogas digesters[J]. FEMS Microbial Ecol, 2013, 85(12):612-626.
[9] 乔江涛,郭荣波,袁宪正,等.玉米秸秆厌氧降解复合菌系的微生物群落结构[J].环境科学,2013, 34(4):1531-1539.
[10]郎会花,杨洪江,张永刚.鸡粪沼气池产甲烷菌多样性[J].微生物学通报,2010, 37(4):508-512.
[11]Kaeberlein T, Lewis K, Epstein S S. Isolating"uncultivable"microorganisms in pure culture in a simulated natural environment[J].Science, 2002, 296(5570):1127-1129.
[12]Traversi D, Villa S, Lorenzi E, et al. Application of a real-time qPCR method to measure the methanogen concentration during anaerobic digestion as an indicator of biogas production capacity[J]. J Environ Manage, 2012, 111(7):173-177.
[13]叶凝芳,何品晶,吕凡,等.厌氧发酵过程pH对微生物多样性和产物分布的影响(英文)[J].应用与环境生物学报,2007, 13(2):238-242.
[14]Wu L, Ge G, Zhu G F, et al. Diversity and composition of the bacterial community of Poyang Lake(China) as determined by 16S rRNA gene sequence analysis[J].World Journal of Microbiology and Biotechnology, 2012, 28(1):233-244.
[15]丁延芹,黄伟红,姚良同,等.PseudomonaspseudoalcaligenesB50铁载体合成相关基因pyrD的克隆与功能分析[J].微生物学通报,2010, 37(7):981-985.
[16]Ueki A, Abe K, Ohtaki Y, et al.Bacteroidespaurosaccharolyticussp. nov., isolated from a methanogenic reactor treating waste from cattle farms[J]. Int J Syst Evol Microbiol, 2011, 61(Pt 2):448-53.
[17]Frank C, Schwarz U, Matthies C, et al. Metabolism of aromatic aldehydes as cosubstrates by the acetogen Clostridium formicoaceticum[J]. Arch Microbiol, 1998, 170(6):427-34.
[18]Lai Q,Shao Z.Pseudomonasxiamenensissp.nov.,a denitrifying bacterium isolated from activated sludge[J].Int J Syat Evol Microbiol, 2008, 58(8):1911-1915.
[19]张蕾,梁军锋,崔文文,等.规模化秸秆沼气发酵反应器中微生物群落特征[J].农业环境科学学报,2014, 33(3):584-592.
[20]朱文秀,黄振兴,任洪艳,等.IC反应器处理啤酒废水的效能及其微生物群落动态分析[J].环境科学,2012, 33(8):2715-2722.
[21]袁月祥,文昊深,黄显波,等.玉米秸秆产生物燃气及其微生物群落解析[J].化工学报,2014, 65(5):1784-1791.
[22]Yan L, Gao Y F, Wang Y J, et al. Diversity of a mesophilic lignocellulolytic microbial consortium which is useful for enhancement of biogas production[J]. Bioresour. Technol, 2012, 111:49-54.
[24]杨承剑,梁辛,韦升菊,等.基于16S rRNA基因克隆文库技术分析广西富钟水牛瘤胃产甲烷菌组成及多样性[J].动物营养学报,2014, 16(12):3635-3642.
Analysis of Microbial Community in Methane Co-Fermentation of Straw & Blue Algae
MU Wei-na, LI Yu-cheng, WU Chao, ZHANG Xue-sheng, WANG Ning, GAO Zhao-hui
(Schl.ofRes. &Environ’lEngin.AnhuiUniversity,Hefei230601)
The diversity study on different stages of gas production by dominant communities of bacteria and archabacteria was carried out adopting self-made anaerobic reaction device filled with mixed straw and blue algae for methane fermentation experiment, using methods of 16S rRNA gene cloning library. The results indicated that the dominant bacterial communities are different in different methane producing stages. At the stage of the tested straw methane reactor the categories of bacteria were more abundant, respectively belonging to six phyla: At the beginning of methane producing stage, the dominant bacterial community was Proteobacteria, with relative abundance at 51.76%, and at the peak stage of gas production the dominant bacterial community was Firmicutes, with relative abundance at 47.13%, while at the completion stage of gas production, the dominant bacterial community was Firmicutes with relative abundance at 28.89%. The category of archabacterial communities were apparently less than bacterial communities, all belonging to Methanomicrobia, Methanobacteria and Thermoplasmata. The interpretation of the structure of microbial communities of straw-blue algae methane system possessed certain significance, it provide a scientific foundation for the regulation of straw bio-gas project.
straw; mixed fermentation; microbial community; cloning library
国家自然科学基金项目(41172121)
穆维娜 女,硕士研究生。主要从事环境微生物方面的研究。Tel:0551-63861783,E-mail:mu_weina@sina.com
* 通讯作者。男,博士,教授,博士生导师。研究方向为地质与古生物。Tel:0551-63861783,E-mail:li-yucheng@163.com
2016-02-17;
2016-05-05
Q938;X712
A
1005-7021(2017)01-0070-08
10.3969/j.issn.1005-7021.2017.01.011

