人巨细胞病毒US31基因序列特征分析及B细胞表位预测
郭刚强,谢尙丹,叶思思,孙祥威,张良,张丽芳,薛向阳
(1.温州医科大学 微生物学与免疫学教研室 分子病毒与免疫研究所 热带医学研究所,浙江 温州325035;2.温州医科大学 第二临床医学院,浙江 温州 325035;3.温州医科大学附属第一医院 胃肠外科,浙江 温州 325015)
·论 著·
人巨细胞病毒US31基因序列特征分析及B细胞表位预测
郭刚强1,谢尙丹2,叶思思1,孙祥威3,张良3,张丽芳1,薛向阳1
(1.温州医科大学 微生物学与免疫学教研室 分子病毒与免疫研究所 热带医学研究所,浙江 温州325035;2.温州医科大学 第二临床医学院,浙江 温州 325035;3.温州医科大学附属第一医院 胃肠外科,浙江 温州 325015)
目的:分析人巨细胞病毒(HCMV)US31基因序列,预测US31基因编码蛋白的B细胞优势表位。方法:基于US31基因编码蛋白的氨基酸序列,结合亲水性参数、可及性参数、抗原性参数、柔韧性参数及二级结构方案对HCMV US31基因编码各型蛋白的B细胞表位进行预测,参照已建立的预测方法综合评价US31基因B细胞优势表位。结果:US31核苷酸序列变异较少,大部分是同义突变,氨基酸序列高度保守;其编码蛋白全长为162个氨基酸,相对分子质量20 kD,等电点7.66,为可溶性蛋白,二级结构中以无规则卷曲为主。经综合分析,US31基因的B细胞优势表位可能存在于氨基酸序列N端的5~19、32~47、58~68、110~124区段。结论:多参数预测HCMV pUS31的B细胞优势表位,为进一步研究蛋白特征、制备单克隆抗体及表位疫苗提供依据。
人巨细胞病毒;US31基因;B细胞表位
人巨细胞病毒(human cytomegalovirus,HCMV)属疱疹病毒科β亚科,为双链DNA病毒,基因组全长约230~240 kb,含208个开放读码框,由长独特序列(unique long domains,UL)和短独特序列(unique short domains,US)2个片段组成。US31基因是US1基因家族成员,5’端和3’端分别与US30和US32基因相邻,属于HCMV增殖非关键病毒基因[1-2]。Burgdorf等[3]利用WTC-F HB5和Q548R IE2 C-F HB5病毒感染和空白感染G0期的HFFs细胞,芯片分析结果显示在Q548R IE2 C-F HB5病毒感染HFFS细胞96 h和168 h后,US31基因相对于野生型病毒感染96 h的US31基因表达量,分别上调了3.5倍和5.7倍,定量PCR再次确证芯片分析结果,提示突变的Q548R IE2基因巨细胞病毒能够上调US31基因的表达,随后利用IE2Δ40+60基因缺失的病毒感染HFFs细胞进一步说明并不是IE2 40和IE2 60基因的缺失而是Q548R IE2 86基因的突变上调了US31基因的表达。而IE2 86是HCMV增殖性感染的一个重要蛋白[4],提示US31基因可能在HCMV感染及病毒增殖中发挥了重要的作用。但US31基因变异及序列特征迄今尚未见研究报道。本研究在分析US31基因变异及编码蛋白序列特征基础上,进一步预测US31基因B细胞优势表位,为应用多肽小片段制备单克隆抗体、表位疫苗及研究其蛋白功能提供重要的依据。
1 材料和方法
1.1US31基因序列的获取 选取HCMV标准株Merlin株进行B细胞表位的预测,此株分离自英国加的夫1例HCMV先天感染儿童尿液。US31蛋白氨基酸序列由基因组序列推导得出,检索自Gene Bank(序列号:AY446894),共有486个核苷酸,编码162个氨基酸残基,相对分子质量为20 kD,等电点为7.66。序列如下:MSLLEREESWRRVVDYSHNLWCTCGNWQSHV EIQDEEPNCEQPEPAHWLEYVAVQWQARVRDSHDRWCLCNA WRDHALRGRWGTAYSSGSSASSSGFVAESKFTWWKRLR HSTRRWLFRRRRARYTPSNCGESSTSSGQSSGDESNCSLRTHG VYTRGEQH。另外从NCBI获取20株HCMV US31基因并转译为氨基酸序列进行序列同源性分析,序列号分别为:Toledo(GU937742)、3157(GQ221974)、3301(GQ466044)、AD169(FJ527563)、AF1(GU179291)、BE_10_2010(KC519320)、CZ_2_2012(KP745643)、HAN(JX512204)、JHC(HQ380895)、JP(GQ221975)、Merlin(AY446894)、U11(GU179290)、PAV16(KJ872539)、VR1814(GU179289)、Towne(FJ616285)、U8(GU179288)、UKNEQAS1(KJ361971)、BE_5_2011(KP745667)、6397(JX512197)、TR(KF021605)。
1.2pUS31蛋白的二级结构预测 结合EXPASY服务器提供的GOR4、SOPMA、Scratch Protein Predictor(SPP)程序对US31编码蛋白全长氨基酸序列进行二级结构的分析。
1.3pUS31蛋白跨膜区域预测 应用EXPASY服务器提供的TMHMM、DAS-TMfilter server、HMMTOP程序对US31蛋白跨膜区域进行预测,以非跨膜区域作为抗原表位的待选区。
1.4pUS31蛋白亲水性、极性、抗原性、柔韧性和表面可及性的预测 结合EXPASY服务器提供的Hopp & Woods(亲水性参数)、Zimmerman(极性参数)和DNAstar软件的Protein进行的Emini(表面可及性参数)、Jameson-Wolf(抗原性参数)、Flexible regions(柔韧性参数)、Kolaskar & Tongaonkar Antigenicity Prediction[5](抗原性指数)和Bepipred Linear Epitope Prediction[6](线性B细胞表位预测)方法分析来对US31编码蛋白的B细胞表位进行预测。
1.5综合分析 综合以上预测方法,兼顾各项预测参数推断pUS31蛋白B细胞表位,采用吴玉章等[7]建立的抗原性指数(antigenic index,AI)综合评判HCMV pUS31 B细胞表位的优势区域。
2 结果
2.1US31基因序列分析 经Clustalx中Do Complete Alignment,BioEdit中Sequence Identity Matrix进行多序列比对分析,结果表明US31核苷酸有存在少数碱基的变异,但几乎属于同义突变,无缺失。除U11株(序列号:GU179290)氨基酸序列第155位Y错义突变为C,其他部位与Merlin株序列一致。US31基因核苷酸同源性大小为98.1%~100.0%,氨基酸为99.3%~100.0%,序列高度保守。翻译后修饰位点显示:pUS31蛋白含有1个N-糖基化位点,4个酪蛋白激酶 II磷酸化位点,4个N-十四(烷)酞化位点,3个蛋白激酶C磷酸化位点,2个双组分核定位信号,修饰位点氨基酸序列高度保守(见图1)。通过MEGA5软件的phylogeny,设置bootstrap值为1 000,我们对20株HCMV US31氨基酸序列进行了系统进化树邻接分析(见图2),采用Poisson Correction模型,结果显示HCMV US31氨基酸序列分为2个亚型,我们定义为G1和G2型,其中19株属于G1型,占95.0%,G2型仅1株即U11株,由于核苷酸第464位A突变成G,导致错义突变,仅占5.0%。
2.2pUS31蛋白的二级结构 采用EXPASY服务器的GOR4、SOPMA、SPP 3种方案预测二级结构,提示HCMV US31编码蛋白的二级结构中以无规卷曲为主,少见β-转角、Bend region(见表1)。二级结构柔性区域占总氨基酸数的47.2%,蛋白肽段的柔韧性越大,越易发生扭曲和折叠,易与抗体嵌合,作为抗原表位的概率也就越高。同时在这3种方案的预测结果中,取至少有2种方案预测结果一致的重叠区为准,且与柔韧性参数的预测相结合,可见无规则卷曲主要位于N端的1~3、25~28、35~46、79~82、83~87、88~94、122~136、139~151、159~161(见图3)。
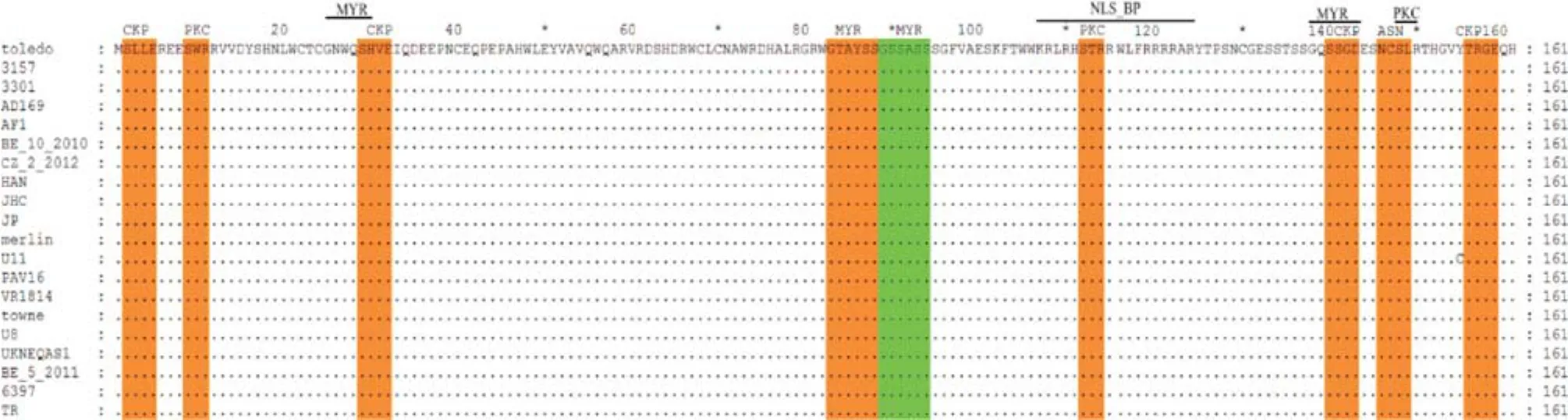
图1 HCMV US31基因编码蛋白氨基酸序列线性化比较

图2 HCMV US31序列种系进化树分析
2.3HCMV pUS31跨膜区域的预测 使用EXPASY的TMHMM、DAS-TMfilter server、HMMTOP方法预测US31跨膜区域,结果显示HCMV pUS31为可溶性蛋白,无跨膜区。因此选择US31全部编码区氨基酸作为预测表位的候选区段。
2.4多参数预测US31蛋白表位 按照Hopp &Woods、Zimmerman、Jameson-Wolf、Flexible regions、Emini方案分别预测US31蛋白的亲水性、极性、抗原性、柔韧性和表面可及性。其中高于阈值的肽段即为预测的抗原表位。综合分析HCMV pUS31的亲水性、极性、柔韧性、表面可及性和抗原性显示(见图4和表2):应用不同参数预测的B细胞抗原表位肽段略有差异,而氨基酸片段5~19、32~45、58~68、86~96、110~124、140~161在多种预测方法中一致(AI≥0,亲水性指数≥0,表面可及性指数≥1,极性指数≥12)。采用Kolaskar & Tongaonkar Antigenicity Prediction(http://tools. immuneepitope.org/bcell/result)预测US31蛋白的B细胞表位,结果显示其N端的11~25、27~33、40~56、65~67、98~101、146~154为可能的B细胞抗原表位(见图5A和表3)。使用Bepipred Linear Epitope Prediction(http://tools.immuneepitope.org/bcell/result/)预测分析US31蛋白的抗原性,发现US31蛋白N端的9~9、32~47、59~62、83~98、123~147、154~161抗原性较强(见图5B和表4)。
2.5pUS31蛋白表位的综合预测 综合以上预测方法及吴玉章等[7]的AI计算方法,计算临床分离株HCMV US31蛋白的B细胞表位平均AI,结果显示该蛋白N端的5~19、32~47、58~68、110~124的平均AI较高,提示其可能为B细胞表位的优势区域(见表5)。
3 讨论

表1 3种方法预测HCMV pUS31的二级结构的构成比[n(%)]

图3 HCMV pUS31二级结构预测
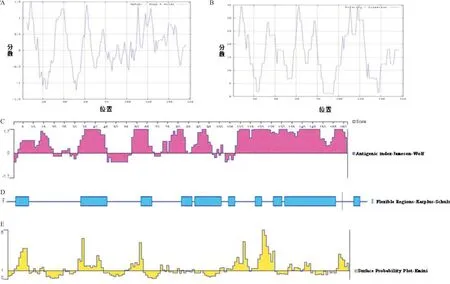
图4 US31蛋白不同参数预测结果

表2 HCMV pUS31亲水性、可及性、抗原性、柔韧性及二级结构参数
US31基因位于HCMV的短独特序列区,与US1基因、US32基因同属于US1家族[8]。研究发现US1基因为即刻早期基因,US32基因为晚期基因且与病毒的潜伏相关[9],但目前与其同属一个家族的US31基因还未见报道。尽管US31基因核苷酸序列存在少数碱基的变异,但几乎属于同义突变,氨基酸序列高度保守,不同病毒株US31蛋白氨基酸序列比对显示,序列同源性高达99.3%~100.0%,提示US31基因将是HCMV感染诊断的候选基因。

表3 HCMV-US31线性B细胞表位预测
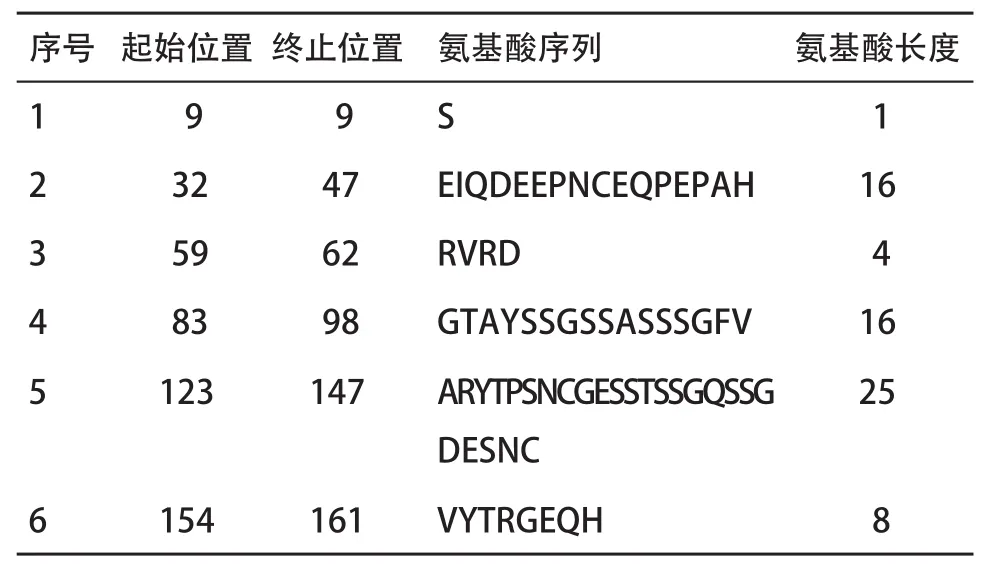
表4 HCMV-US31蛋白抗原性分析
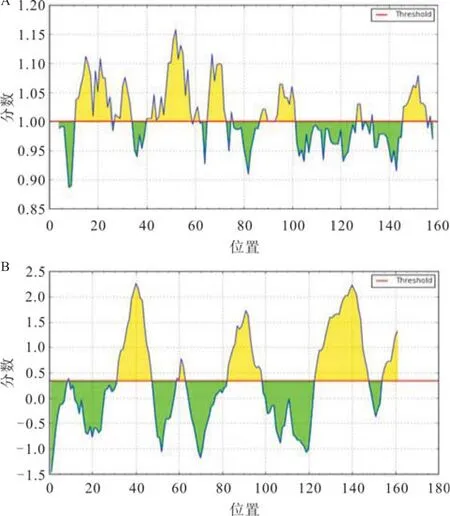
图5 US31蛋白B细胞抗原表位预测和抗原性分析

表5 临床分离株HCMV US31蛋白的B细胞表位平均AI
不同病毒株US31蛋白翻译后修饰位点比较显示,pUS31蛋白含有1个N-糖基化位点,4个酪蛋白激酶 II磷酸化位点,4个N-十四(烷)酞化位点,3个蛋白激酶C磷酸化位点,2个双组分核定位信号,修饰位点氨基酸序列均高度保守,提示US31是一个具有磷酸化修饰调节及核定位的病毒蛋白。此外,20株HCMV US31氨基酸序列系统进化树邻接分析显示,19株属于G1型,占95.0%,提示来自不同病毒株的US31基因亲缘性较近。Sijmons等[10]对来自不同疾病状态的100个HCMV感染患者进行病毒分离与培养、DNA的提取及全基因扩增,序列分析显示US31基因的dN/dS比率为0.030,具有较强的阴性选择,提示US31基因能够很好地适应人宿主,这些现象说明该结构区对于HCMV生物学功能有着重要意义,选择压力保留了这段基因的稳定性。
应用生物信息学技术预测B细胞表位成为目前发现新的B细胞抗原表位的主要方法,并被广泛应用,且均取得了很好的结果[11-12]。而目前常用的B细胞线性表位预测方法主要分为单一参数预测方法和多参数综合预测方法。由于各种预测方法存在差异性和局限性,使得各科研人员预测准确性差异较大,故研究者通过不断地改进方法、开发更好的预测评价体系,使B细胞表位的预测、评价更加标准化[13]。本研究结合单一参数预测方法Hopp & Woods、Zimmerman和DNAstar软件的Protein进行的Emini、Jameson-Wolf、Flexible regions、Kolaskar & Tongaonkar Antigenicity Prediction和多参数综合预测方法Bepipred Linear Epitope Prediction分别预测US31编码蛋白可能的B细胞抗原优势表位,筛选采用了多项参数一致的原则。吴玉章等[7]综合考虑蛋白质的多种性质如片段结构、氨基酸侧链的排列、构象、活动性等,通过计算20种氨基酸在病毒蛋白表位及一般蛋白质中频率的比值,再取对数即可,建立了一种AI的预测方法,用以计算可能的B细胞优势表位平均AI。通过结合以上多种预测方法及吴玉章等的AI计算方法综合分析,结果显示US31蛋白的氨基酸位置5~19区段(EREESWRRVV DYSHN)、32~47区段(EIQDEEPNCEQPEPAH)、58~68区段(ARVRDSHDRWCL)、110~124区段(RHSTRRWLF RRRRAR)平均AI较高,可能是B细胞表位的优势区段。
综上,本研究对HCMV US31基因进行了序列特征分析及B细胞表位预测,一方面有利于认识US31蛋白的结构和理化特性,便于了解US31病毒蛋白的作用,另一方面也有利于为研究者克隆US31基因片段提供依据,为构建高效的US31表位疫苗提供基础。
[1] YU D, SILVA M C, SHENK T. Functional map of human cytomegalovirus AD169 defi ned by global mutational analysis[J]. Proc Natl Acad Sci U S A, 2003, 100(21): 12396-12401.
[2] DUNN W, CHOU C, LI H, et al. Functional profi ling of a human cytomegalovirus genome [J]. Proc Natl Acad Sci U S A, 2003, 100(24): 14223-14228.
[3] BURGDORF S W, CLARK C L, BURGDORF J R, et al. Mutation of glutamine to arginine at position 548 of IE2 86 in human cytomegalovirus leads to decreased expression of IE2 40, IE2 60, UL83, and UL84 and increased transcription of US8-9 and US29-32[J]. J Virol, 2011, 85(21): 11098-11110.
[4] SANDERS R L, CLARK C L, MORELLO C S, et al. Development of cell lines that provide tightly controlled temporal translation of the human cytomegalovirus IE2 proteins for complementation and functional analyses of growth-impaired and nonviable IE2 mutant viruses[J]. J Virol, 2008, 82(14): 7059-7077.
[5] KOLASKAR A, TONGAONKAR P C. A semi-empirical method for prediction of antigenic determinants on protein antigens[J]. FEBS letters, 1990, 276(1-2): 172-174.
[6] LARSEN J E, LUND O, NIELSEN M. Improved method for predicting linear B-cell epitopes[J]. Immunome Res, 2006, 2: 2.
[7] 吴玉章, 朱锡华. 一种病毒蛋白B细胞表位预测方法的建立[J]. 科学通报, 1994, (24): 2275-2279.
[8] VAN DAMME E, VAN LOOCK M. Functional annotation of human cytomegalovirus gene products: an update[J]. Front Microbiol, 2014, 5: 218.
[9] GOODRUM F D, JORDAN C T, HIGH K, et al. Human cytomegalovirus gene expression during infection of primary hematopoietic progenitor cells: a model for latency[J]. Proc Natl Acad Sci U S A, 2002, 99(25): 16255-16260.
[10] SIJMONS S, THYS K, MBONG NGWESE M, et al. Highthroughput analysis of human cytomegalovirus genome diversity highlights the widespread occurrence of gene-disrupting mutations and pervasive recombination[J]. J Virol, 2015, 89: 7673-7695.
[11] 金劲激, 丁玉杰, 王冰冰, 等. MAGE-A家族抗原共同B细胞表位预测分析[J]. 温州医学院学报, 2013, 43(11): 706-710.
[12] SHEN X, JIN J, DING Y, et al. Novel immunodominant epitopes derived from MAGE-A3 and its signifi cance in serological diagnosis of gastric cancer[J]. J Cancer Res Clin Oncol, 2013, 139(9): 1529-1538.
[13] CHEN P, RAYNER S, HU K H. Advances of bioinformatics tools applied in virus epitopes prediction[J]. Virol Sin, 2011, 26(1): 1-7.
(本文编辑:赵翠翠)
Analysis of the characteristics of gene seguence of human cytomegalovirus US31 and pr edication of B cell epitopes
GUO Gangqiang1, XIE Shangdan2, YE Sisi1, SUN Xiangwei3, ZHANG Liang3, ZHANG Lifang1, XUE Xiangyang1. 1.Department of Microbiology and Immunology, Institute of Molecular Virology and Immunology, Institute of Tropical Medicine, Wenzhou Medical University, Wenzhou, 325035; 2.The Second Clinical College of Wenzhou Medical University, Wenzhou, 325035; 3.Department of General Sur gery, the First Aff liated Hospital of Wenzhou Medical University, Wenzhou, 325015
Objective: To analyze the sequence of human cytomegalovirus US31 gene and predict the B cell epitopes of US31 protein. Methods: The hydrophilicity, accessibility, antigenicity and fl exibility index were used to predict the potential B cell epitopes of protein based on the US31 amino acid sequence. Results: Most amino acid sequence of US31 was highly conserved while only a few strains had variation, and most of them were sense mutation. HCMV pUS31 contained 162 amino acid residues, the relative molecular mass was 20 kD and the isoelectric point was 7.66. It’s a soluble protein and its secondary structure occurred more frequently as random coil region. The predicted B-cell epitopes of the pUS31 might exist at N-terminal of amino acid sequence: 5~19, 32~47, 58~68, 110~124. Conclusion: The B-epitopes of pUS31 are predicted successfully, which provides a theoretic basis for the further study of protein characteristics, development of epitopes based vaccine, and preparation of monoclonal antibody against fusion protein.
human cytomegalovirus; US31 gene; B cell epitopes
R373.9
A
10.3969/j.issn.2095-9400.2016.11.001
2016-04-25
国家自然科学基金资助项目(81472308,81672707);浙江省大学生新苗计划(2014R413058,2015R413069)。
郭刚强(1992-),男,云南曲靖人,硕士生。
薛向阳,副教授,硕士生导师,Email:wzxxy001@ 163.com。

