肠道菌群对机体营养物质的代谢研究
郭慧慧,黄帅,王璐璐,韩燕星,蒋建东
·综述·
肠道菌群对机体营养物质的代谢研究
郭慧慧,黄帅,王璐璐,韩燕星,蒋建东
人体肠道内寄居的微生物种类繁多,主要以厌氧菌为主,而且数量巨大,约为人体细胞总和的 10 倍[1]。正常的肠道菌群按一定的比例栖息于肠道内部,彼此之间互相制约,互相依存,共同维持人体肠道系统的平衡和稳定。
人类机体通过摄食从外界获得的各种营养物质经过胃肠道的消化、吸收供给机体各种生命活动,而那些未消化、吸收的营养物质进入结肠后,将主要通过结肠内厌氧菌对其进行发酵,产生各种代谢产物[2]。这些代谢产物对维持肠道内环境起着不可忽视的作用。而且,肠道菌群可以利用机体提供的代谢产物合成多种氨基酸和维生素,补充人体生长所需的营养物质[3]。但是,一旦宿主与肠道微生物之间的稳态被打破,可能会诱发多种疾病,目前已经发现肠道菌群的营养物质代谢与糖尿病、血脂异常、慢性心脑血管疾病、结肠炎甚至癌症的发生、进展和预后都有密切关系。本文主要介绍肠道菌群对糖类、膳食纤维、蛋白质、氨基酸、脂质和维生素等营养物质的代谢以及对机体产生的影响。
1 糖和膳食纤维代谢
食物中的抗性淀粉、低聚糖、非淀粉多糖和膳食纤维等进入人体后,不能在胃肠道酶的作用下消化吸收,它们经消化道进入结肠内,在结肠厌氧菌作用下,生成含 1 ~ 6 个碳原子的短链脂肪酸(short chain fatty acid,SCFA),其中最主要的是乙酸、丙酸和丁酸,三者比例大约为 3∶1∶1[4-6]。不同的肠道菌群产生不同种类的 SCFA,拟杆菌门以生成乙酸和丙酸为主,而厚壁菌门以生成丁酸为主[7]。那些未消化、吸收的糖和纤维素首先被肠道菌水解成单糖,然后经过菌群无氧糖酵解途径生成丙酮酸,再在不同菌群内经过不同的代谢途径生成不同种类的 SCFA[6-7],详细代谢过程和代谢关键酶分别如图 1 和表 1 所示。
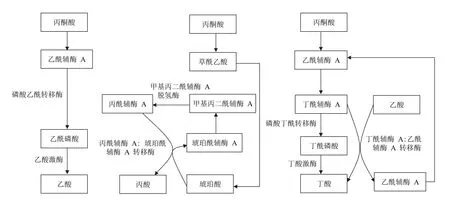
图1 乙酸、丙酸和丁酸在肠道菌群内的合成途径
拟杆菌门细菌首先将丙酮酸氧化成乙酰辅酶 A,然后经磷酸乙酰转移酶、乙酸激酶生成乙酸[8],在生成乙酸时会产生 H2和 CO2,而 CO2经 Wood-Ljungdahl 通路也可生成乙酸,具体为:CO2首先被还原成 CO,然后经甲基转移酶与甲基结合形成乙酸[9]。而且 H2和 CO2在古生菌内可生成甲烷,以免副产物堆积阻碍乙酸的生成。

表1 肠道菌产 SCFA 的基本情况
在拟杆菌门内生成的丙酮酸能与 CO2羧化形成草酰乙酸,经加氢、脱水和还原反应生成琥珀酸,然后转化成甲基丙二酰辅酶 A 后,再经甲基丙二酰辅酶 A 脱氢酶生成丙酰辅酶 A,最后在丙酰辅酶 A∶琥珀酰辅酶 A 转移酶作用下生成丙酸[10];此外,丙酮酸还可通过丙烯酸途径形成丙酸(丙酮酸在乳酸菌内被还原成乳酸,经脱水、加氢还原生成丙酸)[8]。
丁酸是厚壁菌门的主要代谢产物,丙酮酸首先转化成乙酰辅酶 A,经硫解、加氢、脱水、再加氢生成丁酰辅酶 A后,通过两种途径生成丁酸:①丁酰辅酶 A 通过磷酸丁酰转移酶形成丁酰磷酸,然后在丁酸激酶作用下生成丁酸,其中丁酸激酶是该途径的限速酶[8, 11];②丁酰辅酶 A 和乙酸直接经丁酰辅酶 A∶乙酰辅酶 A 转移酶生成丁酸和乙酰辅酶 A[12],该通路将乙酸与丁酸的生成联系在一起。而且,乳酸菌也可利用乳酸转化成乙酰辅酶 A,再通过以上途径生成丁酸[13]。研究还发现,有些产丁酸的肠道菌如Clostridium a cetobutylicum 有两种丁酸激酶 BUK-1 和BUK-2,其中 BUK-1 对乙酰磷酸也有较高亲和性,能催化乙酰磷酸生成乙酸,而 BUK-2 却只能生成丁酸,基于此,敲除该菌的 buk-1 基因能大大提高丁酸产量[14]。
肠道菌群生成的 SCFA,约 95% 被宿主结肠细胞摄取,只有 5% 随粪便排出体外,吸收的 SCFA 可为结肠细胞和机体其他组织提供能量,未吸收部分可调节肠道内环境的氧化还原平衡。近年来的研究发现,短链脂肪酸对肥胖、II 型糖尿病、胰岛素抵抗代谢综合征有显著影响;乙酸和丙酸可抑制 TNF-α 的释放,对结肠炎有良好的治疗效果;而且研究发现丁酸能有效抑制结肠肿瘤细胞增殖、诱导肿瘤细胞分化的凋亡、影响原癌基因的表达,从而预防和治疗结肠癌[15-19]。
2 蛋白质和氨基酸代谢
肠道中未消化的蛋白质和未吸收的氨基酸在肠道菌群作用下可生成多种活性代谢产物,主要包括 NH3、H2S、短链脂肪酸、多胺化合物、吲哚及酚类化合物。未消化的蛋白质先在肠道菌作用下分解成氨基酸,然后再进行氨基酸酵解,而且不同种类的氨基酸经肠道菌内的酶产生不同类型的产物(表 2)。
2.1芳香氨基酸代谢
芳香氨基酸经某些肠道厌氧菌如拟杆菌、乳酸菌、双歧杆菌、梭状芽孢杆菌、消化链球菌等代谢,主要生成酚类和吲哚类物质,但研究发现,仅约 10% 的肠道厌氧菌能代谢芳香氨基酸[20]。酪氨酸和苯丙氨酸经肠道厌氧菌群代谢主要生成苯酚、对甲酚、苯乙酸、苯丙酸等。测定结肠内这些物质的浓度时,发现在结肠近端这些产物的含量微量,而在结肠远端产物的量明显升高,说明能酵解酪氨酸和苯丙氨酸的肠道菌群主要位于结肠远端[21]。

表2 肠道菌对氨基酸的代谢情况
色氨酸在肠道菌群的作用下生成吲哚、吲哚乙酸、吲哚-3-乳酸、3-甲基吲哚(粪臭素)等多种吲哚类化合物(图 2)。色氨酸在色氨酸酶的作用下分解为吲哚、丙酮酸和氨,色氨酸酶是生成吲哚的关键酶,酶的活性易受底物色氨酸的诱导,因此当结肠环境内色氨酸的量发生改变时,机体吲哚的含量会有较大的波动[22];而色氨酸与 α-酮戊二酸在转氨酶作用下生成吲哚-3-丙酮酸,然后再经吲哚丙酮酸脱羧酶生成吲哚乙酸,吲哚乙酸作为粪臭素的前体物质,可在某些肠道菌群如梭菌属和杆菌属作用下脱羧生成具有恶臭味的粪臭素,其中吲哚丙酮酸脱羧酶是生成吲哚乙酸和粪臭素的关键酶[23]。研究发现,能产生粪臭素的肠道菌群在肠道内并不广泛存在,因此正常情况下,人体内粪臭素的量较少[24]。虽然粪臭素是由色氨酸代谢产生,但是大量研究发现,在肠道菌培养基中加入过量的色氨酸并不会显著增加粪臭素的含量,但是当增加吲哚乙酸时,粪臭素的产生量显著增加[25]。正常生理浓度下,这些吲哚类物质并不会对机体造成伤害,但是如果在结肠内过量集聚,会造成人结肠上皮细胞的损伤,甚至引发结肠癌[26],而且如果过量的粪臭素进入血液循环,易引发肺气肿等呼吸道疾病[25]。
肠道菌群除了自身对色氨酸代谢影响机体生理功能外,还可通过其产生的各种代谢产物如 SCFA、脱氧胆酸、γ-氨基丁酸和酪胺等影响肠嗜铬细胞对色氨酸代谢成 5-羟色胺[27];而且某些肠道菌自身也可利用色氨酸转化为 5-羟色胺,但两者的代谢方式不同,在肠道菌内,色氨酸首先经过脱羧反应生成色胺,再羟化成 5-羟色胺,而在肠嗜铬细胞内,首先是在色氨酸羟化酶(限速酶)作用下生成 5-羟色氨酸,然后再脱羧生成 5-羟色胺[28]。肠道菌自身生成和肠道菌介导肠嗜铬细胞生成的 5-羟色胺共同影响机体的肠道运动、血小板聚集和止血等生理功能。
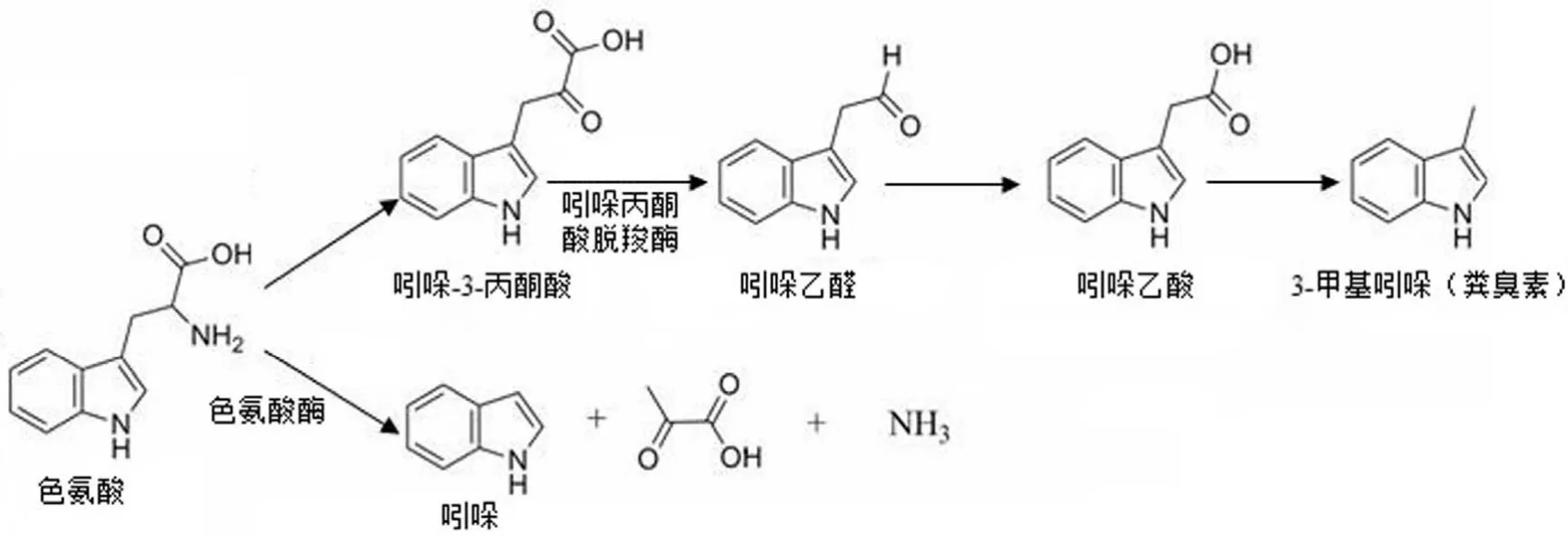
图2 色氨酸在肠道菌的主要代谢途径
2.2含硫氨基酸代谢
半胱氨酸、甲硫氨酸等含硫氨基酸在肠道菌作用下产生比较特殊的产物——硫化物,它们主要是经过肠道菌群脱硫反应产生。半胱氨酸在半胱氨酸脱巯基酶作用下生成H2S[29];甲硫氨酸可在肠道菌作用下转化成 α-酮丁酸、甲硫醇[30]。研究发现大肠内的大肠杆菌、沙门菌、梭状菌和产气肠杆菌等菌群可酵解含硫氨基酸[31]。H2S 在结肠内含量较高时,可穿透结肠上皮细胞的脂质双分子层进入细胞,抑制线粒体内膜上细胞色素 C 氧化酶活性,干扰结肠细胞呼吸,影响能量代谢,甚至可以造成 DNA 链断裂[32];但是生理浓度下的 H2S 对维持结肠细胞的正常能量供应和生命活动有非常重要的作用,因此饮食中含硫氨基酸的过多或过少都会引发机体结肠功能的紊乱,从而引发肠道甚至全身疾病[33]。
2.3脂肪族氨基酸代谢
精氨酸、鸟氨酸和甲硫氨酸在肠道菌群的作用下可产生多胺化合物,包括精胺、亚精胺、腐胺、尸胺,体外培养的类杆菌、乳酸菌、双歧杆菌和梭状菌等多种肠道菌都可产生多胺化合物[34];体内研究也发现对无菌鼠接种不同种类的肠道菌,其肠道内的多胺物质含量都会发生变化[35]。在正常生理浓度范围内,这些多胺类物质可与周围结肠细胞内的多种大分子物质结合,影响细胞的生长和增殖[36-37],如腐胺可促进结肠细胞的有丝分裂,诱发结肠癌[33, 38]。组氨酸、酪氨酸、赖氨酸经肠道菌脱羧反应可分别产生组胺、酪胺、胍丁胺等胺类物质[33, 39]。当菌群发生较大变化时,这些代谢产物作为神经递质或生理活性物质作用于机体,产生不同的效应,如组胺与肠道内的组胺受体结合,使肠道平滑肌收缩,增强胃动力,引发腹痛、腹泻;胍丁胺可抑制结肠癌细胞的增殖,发挥体外抗结肠癌活性[40]。
NH3是几乎所有氨基酸都会产生的一种重要代谢产物。随着饮食中蛋白质含量的增加,大肠内的氨浓度会显著升高[41]。肠道内的 NH3主要是通过肠道菌对氨基酸脱氨或水解尿素所产生。氨在机体有非常重要的作用,如果结肠内产 NH3的肠道菌群过量繁殖,过量的氨可引发中枢性的氨中毒,影响大脑供能[42];过量的氨还会影响肠道内短链脂肪酸的氧化,干扰结肠细胞和某些肠道菌群的能量供应,引发机体的不良反应[17, 43];而且氨还有诱发结肠癌的风险[44]。
各种氨基酸经肠道菌脱氨、转氨作用可产生多种结构不同的 α-酮酸或 α-羟基酸,然后再经过一系列复杂的氧化还原反应产生多种短支链脂肪酸[45],与糖类物质产生的短链脂肪酸一起发挥生物学效应。
3 脂代谢
肠道内寄生的肠道菌群在胆固醇的代谢过程中占有一席之地。肠道菌群对胆固醇的代谢有直接代谢和间接代谢两种形式。直接代谢是指肠道内的某些正常菌群可产生胆固醇氧化酶,可将肠道内的胆固醇氧化成胆固烯酮,进而被降解成粪固醇和胆固烷醇,随粪便排出体外,加速胆固醇的降解[46];间接代谢是指肠道菌群对胆固醇的肝脏代谢产物初级胆汁酸的代谢。正常肠道菌产生的初级胆汁酸水解酶能将结合胆汁酸转变成游离胆汁酸,影响胆汁酸的肠肝循环,使血中的胆固醇更多的被转化,从而降低血胆固醇[47]。而且,某些肠道菌可表达羟化类固醇 7α-脱氢酶,促进初级胆汁酸转化成次级胆汁酸[47],抑制艰难梭状芽胞杆菌的生长和繁殖,有效治疗艰难梭状芽孢杆菌感染[48];但也有研究称,肠道菌产生的次级胆汁酸增多会对机体造成潜在的致癌效应[49]。
肠道菌对食物中的卵磷脂代谢与某些慢性心血管疾病有关。卵磷脂在体内水解形成胆碱后进入肠道,经由肠道菌的作用生成三甲胺,之后三甲胺被吸收进入血液,随血流进入肝脏,在肝脏黄素单加氧酶作用下形成 N-氧化三甲胺。目前研究发现,机体 N-氧化三甲胺的增多与冠状动脉粥样硬化性心血管疾病、慢性收缩性心力衰竭等都有重要的关系[50-51]。
4 维生素的代谢
机体自身缺乏合成大多数维生素的能力,所需维生素主要由食物供给,因此当营养不良或者有不良的饮食习惯时,很容易造成维生素的缺乏。但研究发现,人体肠道内的微生物也是供给机体维生素的一种重要途径,人体肠道内的正常菌群如双歧杆菌、乳酸杆菌等可合成多种 B 组维生素(如维生素 B1、B2、B6、B12、维生素 PP、叶酸、泛酸、生物素)和维生素 K等。
肠道菌合成叶酸主要是利用人肠道提供的 6-羟甲基-7,8-蝶呤焦磷酸(DHPPP)和对氨基苯甲酸(PABA)合成的。双歧杆菌属和乳酸杆菌属内的多种菌都含有合成叶酸的多种酶,而且不同种的菌合成叶酸的能力也大不相同[52-53]。但是 PABA 和 DHPPP 是合成叶酸所必需的底物,当肠道内缺乏这两种物质时,几乎所有菌株都无法合成叶酸[54]。
肠道菌群在机体提供 GTP 和 5-磷酸-D-核酮糖的基础上依靠多种菌群产生的多种酶合成维生素 B2。研究发现,合成维生素 B2的酶并不存在同一种菌株内,而是一种菌只能合成其中的一个或者某几个酶,因此,它的生物合成需要多种菌株共同协作完成[55-56]。目前已知枯草芽孢杆菌、大肠杆菌和沙门菌等可以合成维生素 B2[57]。
生物素可在多种肠道菌如大肠杆菌、球形芽孢杆菌和枯草芽孢杆菌中合成。研究发现,球形芽孢杆菌和枯草芽孢杆菌可利用自身合成的庚二酸,在菌体 bioW 基因编码的庚二酰辅酶 A 合成酶作用下合成庚二酰辅酶 A,之后再在多种氨基酸及各种酶作用下合成生物素[58];而大肠杆菌体内缺乏合成庚二酸的酶,需依赖另外两种基因 bioC 和 bioH表达的酶合成庚二酰辅酶 A 后,再按上述步骤合成生物素[59]。
维生素 K 在维持血液的正常凝固方面有非常重要的生理意义。人体缺乏合成维生素 K 的酶,机体所需主要由食物和肠道菌群供给。而且发现维生素 K1主要由植物合成,而 K2主要由人体肠道细菌合成。肠道中的一些专性厌氧菌如拟杆菌属、真细菌属、丙酸菌属和蛛网菌属是合成维生素 K2的主要菌群,而兼性厌氧菌如大肠杆菌、双歧杆菌属、乳酸菌属、梭菌属等不能合成[60]。
其他 B 族维生素如 B1、B6、B12等也可以在肠道菌群的某种或多种菌株内合成,如在肠道内含有嗜热链球菌ST5 和瑞士乳杆菌 R0052 的动物比不含有这些菌株的动物体内的维生素 B1和 B6的含量要高[61]。
肠道菌群合成的维生素是机体补充维生素、维持维生素水平稳定的一种非常重要的方式,当疾病、抗生素治疗导致肠道菌群失衡时,机体内的维生素水平有可能发生较大的波动,此时,一旦食物无法及时供应足够量的维生素,机体就会出现各种维生素缺乏症状,影响机体的生命活动。
5 展望
Science 就曾预测肠道微生物研究可能成为世界范围内的新热点课题,而且随着肠道微生物基因组计划的实施和肠道宏基因组学、功能基因组学、代谢组学以及蛋白质组学的不断发展,肠道微生物研究越来越受到众多学者的重视[62]。调查研究发现,近期有多个国家的研究组在 Nature、Science等顶尖杂志上发表了有关这一领域的最新研究成果。肠道菌群对机体营养物质代谢产生的各种代谢产物与肥胖、II 型糖尿病、血脂异常、慢性心脑血管疾病、结肠炎和结肠癌等密切相关,因此在肠道微生物的研究中,肠道菌对机体营养物质的代谢研究将会越来越受到关注。
[1] Cani PD, Delzenne NM. Gut microflora as a target for energy and metabolic homeostasis. Curr Opin Clin Nutr Metab Care, 2007,10(6):729-734.
[2] Fischbach MA, Sonnenburg JL. Eating for two: how metabolism establishes interspecies interactions in the gut. Cell Host Microbe,2011, 10(4):336-347.
[3] Xu J, Gordon JI. Honor thy symbionts. Proc Natl Acad Sci U S A,2003, 100(18):10452-10459.
[4] Binder HJ. Role of colonic short-chain fatty acid transport in diarrhea. Annu Rev Physiol, 2010, 72:297-313.
[5] Hijova E, Chmelarova A. Short chain fatty acids and colonic health. Bratisl Lek Listy, 2007, 108(8):354-358.
[6] Flint HJ, Bayer EA, Rincon MT, et al. Polysaccharide utilization by gut bacteria: potential for new insights from genomic analysis. Nat Rev Microbiol, 2008, 6(2):121-131.
[7] Macfarlane S, Macfarlane GT. Regulation of short-chain fatty acid production. Proc Nutr Soc, 2003, 62(1):67-72.
[8] Miller TL, Wolin MJ. Pathways of acetate, propionate, and butyrate formation by the human fecal microbial flora. Appl Environ Microbiol,1996, 62(5):1589-1592.
[9] Ragsdale SW, Pierce E. Acetogenesis and the Wood-Ljungdahl pathway of CO2 fixation. Biochim Biophys Acta, 2008, 1784(12):1873-1898.
[10] Wang Z, Ammar EM, Zhang A, et al. Engineering Propionibacterium freudenreichii subsp. shermanii for enhanced propionic acid fermentation: effects of overexpressing propionyl-CoA: Succinate CoA transferase. Metab Eng, 2015, 27:46-56.
[11] Louis P, Flint HJ. Diversity, metabolism and microbial ecology of butyrate-producing bacteria from the human large intestine. FEMS Microbiol Lett, 2009, 294(1):1-8.
[12] Duncan SH, Barcenilla A, Stewart CS, et al. Acetate utilization and butyryl coenzyme A (CoA): acetate-CoA transferase in butyrate -producing bacteria from the human large intestine. Appl Environ Microbiol, 2002, 68(10):5186-5190.
[13] Duncan SH, Louis P, Flint HJ. Lactate-utilizing bacteria, isolated from human feces, that produce butyrate as a major fermentation product. Appl Environ Microbiol, 2004, 70(10):5810-5817.
[14] Jang YS, Im JA, Choi SY, et al. Metabolic engineering of Clostridium acetobutylicum for butyric acid production with high butyric acid selectivity. Metab Eng, 2014, 23:165-174.
[15] Tedelind S, Westberg F, Kjerrulf M, et al. Anti-inflammatory properties of the short-chain fatty acids acetate and propionate: a study with relevance to inflammatory bowel disease. World J Gastroenterol,2007, 13(20):2826-2832.
[16] den Besten G, van Eunen K, Groen AK, et al. The role of short-chain fatty acids in the interplay between diet, gut microbiota, and host energy metabolism. J Lipid Res, 2013, 54(9):2325-2340.
[17] Darcy-Vrillon B, Cherbuy C, Morel MT, et al. Short chain fatty acid and glucose metabolism in isolated pig colonocytes: modulation by NH4+. Mol Cell Biochem, 1996, 156(2):145-151.
[18] Remely M, Aumueller E, Merold C, et al. Effects of short chain fatty acid producing bacteria on epigenetic regulation of FFAR3 in type 2diabetes and obesity. Gene, 2014, 537(1):85-92.
[19] Layden BT, Angueira AR, Brodsky M, et al. Short chain fatty acids and their receptors: new metabolic targets. Transl Res, 2013,161(3):131-140.
[20] Smith EA, Macfarlane GT. Enumeration of human colonic bacteria producing phenolic and indolic compounds: effects of pH,carbohydrate availability and retention time on dissimilatory aromatic amino acid metabolism. J Appl Bacteriol, 1996, 81(3):288-302.
[21] Macfarlane GT, Macfarlane S. Human colonic microbiota: ecology,physiology and metabolic potential of intestinal bacteria. Scand J Gastroenterol Suppl, 1997, 222:3-9.
[22] Yokoyama MT, carlson JR. Microbial metabolites of tryptophan in the intestinal tract with special reference to skatole. Am J Clin Nutr, 1979,32(1):173-178.
[23] Whitehead TR, Price NP, Drake HL, et al. Catabolic pathway for the production of skatole and indoleacetic acid by the acetogen Clostridium drakei, Clostridium scatologenes, and swine manure. Appl Environ Microbiol, 2008, 74(6):1950-1953.
[24] Cook KL, Rothrock MJ Jr, Loughrin JH, et al. Characterization of skatole-producing microbial populations in enriched swine lagoon slurry. FEMS Microbiol Ecol, 2007, 60(2):329-340.
[25] Sharma N, Doerner KC, Alok PC, et al. Skatole remediation potential of Rhodopseudomonas palustris WKU-KDNS3 isolated from an animal waste lagoon. Lett Appl Microbiol, 2015, 60(3):298-306.
[26] Nowak A, Libudzisz Z. Influence of phenol, p-cresol and indole on growth and survival of intestinal lactic acid bacteria. Anaerobe, 2006,12(2):80-84.
[27] Yano JM, Yu K, Donaldson GP, et al. Indigenous bacteria from the gut microbiota regulate host serotonin biosynthesis. Cell, 2015, 161(2):264-276.
[28] Williams BB, Van Benschoten AH, Cimermancic P, et al. Discovery and characterization of gut microbiota decarboxylases that can produce the neurotransmitter tryptamine. Cell Host Microbe, 2014,16(4):495-503.
[29] Awano N, Wada M, Mori H, et al. Identification and functional analysis of Escherichia coli cysteine desulfhydrases. Appl Environ Microbiol, 2005, 71(7):4149-4152.
[30] Smith EA, Macfarlane GT. Dissimilatory amino Acid metabolism in human colonic bacteria. Anaerobe, 1997, 3(5):327-337.
[31] Kadota H, Ishida Y. Production of volatile sulfur compounds by microorganisms. Annu Rev Microbiol, 1972, 26:127-138.
[32] Vermeiren J, Van de Wiele T, Van Nieuwenhuyse G, et al. Sulfide- and nitrite-dependent nitric oxide production in the intestinal tract. Microb Biotechnol, 2012, 5(3):379-387.
[33] Davila AM, Blachier F, Gotteland M, et al. Intestinal luminal nitrogen metabolism: role of the gut microbiota and consequences for the host. Pharmacol Res, 2013, 68(1):95-107.
[34] Allison C, Macfarlane GT. Influence of pH, nutrient availability, and growth rate on amine production by Bacteroides fragilis and Clostridium perfringens. Appl Environ Microbiol, 1989, 55(11):2894-2898.
[35] Noack J, Dongowski G, Hartmann L, et al. The human gut bacteria Bacteroides thetaiotaomicron and Fusobacterium varium produce putrescine and spermidine in cecum of pectin-fed gnotobiotic rats. J Nutr, 2000, 130(5):1225-1231.
[36] Igarashi K, Kashiwagi K. Modulation of cellular function by polyamines. Int J Biochem Cell Biol, 2010, 42(1):39-51.
[37] Shah P, Swiatlo E. A multifaceted role for polyamines in bacterial pathogens. Mol Microbiol, 2008, 68(1):4-16.
[38] Ray RM, McCormack SA, Johnson LR. Polyamine depletion arrests growth of IEC-6 and Caco-2 cells by different mechanisms. Am J Physiol Gastrointest Liver Physiol, 2001, 281(1):G37-G43.
[39] Gale EF. The production of amines by bacteria: the production of putrescine from l(+)-arginine by Bacterium coli in symbiosis with Streptococcus faecalis. Biochem J, 1940, 34(6):853-857.
[40] Mayeur C, Veuillet G, Michaud M, et al. Effects of agmatine accumulation in human colon carcinoma cells on polyamine metabolism, DNA synthesis and the cell cycle. Biochim Biophys Acta,2005, 1745(1):111-123.
[41] Smith EA, Macfarlane GT. Enumeration of amino acid fermenting bacteria in the human large intestine: effects of pH and starch on peptide metabolism and dissimilation of amino acids. FEMS Microbiol Ecol, 1998, 25(4):355-368.
[42] Andriamihaja M, Davila AM, Eklou-Lawson M, et al. Colon luminal content and epithelial cell morphology are markedly modified in rats fed with a high-protein diet. Am J Physiol Gastrointest Liver Physiol,2010, 299(5):G1030-G1037.
[43] Cremin JD Jr, Fitch MD, Fleming SE. Glucose alleviates ammonia-induced inhibition of short-chain fatty acid metabolism in rat colonic epithelial cells. Am J Physiol Gastrointest Liver Physiol,2003, 285(1):G105-G114.
[44] Ichikawa H, Sakata T. Stimulation of epithelial cell proliferation of isolated distal colon of rats by continuous colonic infusion of ammonia or short-chain fatty acids is nonadditive. J Nutr, 1998,128(5):843-847.
[45] Millet S, Van Oeckel MJ, Aluwé M, et al. Prediction of in vivo short-chain fatty acid production in hindgut fermenting mammals:problems and pitfalls. Crit Rev Food Sci Nutr, 2010, 50(7):605-619.
[46] Li C, Cui LH. Relationship among intestinal flora, high-fat diets, and hyperlipidemia. World Chin J Digestology, 2013, 21(14):1273-1277. (in Chinese)
李超, 崔立红. 高脂血症、高脂饮食与肠道菌群的关系. 世界华人消化杂志, 2013, 21(14):1273-1277.
[47] Ridlon JM, Kang DJ, Hylemon PB. Bile salt biotransformations by human intestinal bacteria. J Lipid Res, 2006, 47(2):241-259.
[48] Buffie CG, Bucci V, Stein RR, et al. Precision microbiome reconstitution restores bile acid mediated resistance to Clostridium difficile. Nature, 2015, 517(7533):205-208.
[49] Gill CI, Rowland IR. Diet and cancer: assessing the risk. Br J Nutr,2002, 88 Suppl 1:S73-S87.
[50] Wang Z, Klipfell E, Bennett BJ, et al. Gut flora metabolism of phosphatidylcholine promotes cardiovascular disease. Nature, 2011,472(7341):57-63.
[51] Tang WH, Wang Z, Shrestha K, et al. Intestinal microbiota-dependent phosphatidylcholine metabolites, diastolic dysfunction, and adverse clinical outcomes in chronic systolic heart failure. J Card Fail, 2015,21(2):91-96.
[52] Pompei A, Cordisco L, Amaretti A, et al. Administration of folate-producing bifidobacteria enhances folate status in Wistar rats. J Nutr, 2007, 137(12):2742-2746.
[53] Laiño JE, Leblanc JG, Savoy de Giori G. Production of natural folates by lactic acid bacteria starter cultures isolated from artisanal Argentinean yogurts. Can J Microbiol, 2012, 58(5):581-588.
[54] Ventura M, Turroni F, Zomer A, et al. The Bifidobacterium dentiumBd1 genome sequence reflects its genetic adaptation to the human oral cavity. PLoS Genet, 2009, 5(12):e1000785.
[55] Ventura M, Canchaya C, Fitzgerald GF, et al. Genomics as a means to understand bacterial phylogeny and ecological adaptation: the case of bifidobacteria. Antonie van Leeuwenhoek, 2007, 91(4):351-372.
[56] Bacher A, Eberhardt S, Fischer M, et al. Biosynthesis of vitamin B2 (riboflavin). Annu Rev Nutr, 2000, 20(1):153-167.
[57] Magnúsdóttir S, Ravcheev D, de Crécy-Lagard V, et al. Systematic genome assessment of B-vitamin biosynthesis suggests co-operation among gut microbes. Front Genet, 2015, 6:148.
[58] Ploux O, Soularue P, Marquet A, et al. Investigation of the first step of biotin biosynthesis in Bacillus sphaericus. Purification and characterization of the pimeloyl-CoA synthase, and uptake of pimelate. Biochem J, 1992, 287(Pt 3):685-690.
[59] Lemoine Y, Wach A, Jeltsch JM. To be free or not: the fate of pimelate in Bacillus sphaericus and in Escherichia coli. Mol Microbiol, 1996,19(3):645-647.
[60] Bentley R, Meganathan R. Biosynthesis of vitamin K (menaquinone)in bacteria. Microbiol Rev, 1982, 46(3):241-280.
[61] LeBlanc JG, Milani C, de Giori GS, et al. Bacteria as vitamin suppliers to their host: a gut microbiota perspective. Curr Opin Biotechnol,2013, 24(2):160-168.
[62] Wang ZK, Yang YS. Intestinal microbiota and human diseases. Med J Chin PLA, 2012, 37(12):1168-1176. (in Chinese)王子凯, 杨云生. 肠道微生物与人类疾病. 解放军医学杂志, 2012,37(12):1168-1176.
10.3969/j.issn.1673-713X.2016.04.010
北京市科委重点实验室项目(Z141102004414062)
100050 北京,中国医学科学院北京协和医学院药物研究所药理室(郭慧慧、黄帅、王璐璐、韩燕星、蒋建东);100050 北京,中国医学科学院北京协和医学院医药生物技术研究所病毒室(黄帅、蒋建东)
韩燕星,Email:hanyanxing@imm.ac.cn;蒋建东,Email:jiang.jdong@163.com
2016-03-04

