猪TRIM11基因克隆及其促进猪伪狂犬病毒在体外增殖的研究
宋 爽,曾 磊,鲁绍芳,郭珍珍,鲁维飞,韩立强,王 江,王月影,褚贝贝,杨国宇
(河南农业大学 农业部动物生化与营养重点开放实验室,郑州 450002)
猪TRIM11基因克隆及其促进猪伪狂犬病毒在体外增殖的研究
宋爽,曾磊,鲁绍芳,郭珍珍,鲁维飞,韩立强,王江,王月影,褚贝贝*,杨国宇*
(河南农业大学 农业部动物生化与营养重点开放实验室,郑州 450002)
为初步研究猪TRIM11的免疫学功能,以猪颌下淋巴结cDNA为模板扩增TRIM11 基因cDNA并对该基因进行了蛋白质结构预测和组织表达谱分析,将该基因与PiggyBac载体相连获得真核表达载体PB-TRIM11,之后转染PK15细胞获得稳定表达细胞系。用猪伪狂犬病毒(PRV)刺激过表达TRIM11 PK15细胞和对照组细胞后,于不同时间收获细胞上清,比较两组细胞上清中病毒滴度变化。结果表明,猪TRIM11的ORF长度为1 407 bp,编码468个氨基酸,该氨基酸序列含有3个TRIM家族保守的结构域。组织表达谱分析显示,猪TRIM11在脂肪组织、肺的表达量较高,而在心、回肠、十二指肠、直肠中的转录量较低。成功克隆了猪TRIM11 cDNA序列并构建了真核转录载体PB-TRIM11,qRT-PCR检测结果表明TRIM11在PK15细胞系中成功表达。检测感染PRV后过表达TRIM11 PK15细胞和对照组细胞上清液病毒的TCID50发现过表达TRIM11组的病毒滴度始终高于对照组。过表达TRIM11有一定的促进PRV增殖的作用,为进一步研究猪TRIM11在天然免疫中的作用奠定了基础。
三基序结合蛋白;猪伪狂犬病毒;病毒增殖;稳定细胞系
三基序结合蛋白(TRIM)家族在人上已发现超过70个家族成员。所有的TRIM蛋白都含有一个由锌指结构、1或2个B-box、1个卷曲螺旋(RBCC)组成的N端三结构域[1]。TRIM蛋白功能多种多样,除了参与细胞增殖、分化、发育、形态发生和细胞凋亡,也在免疫相关信号通路和抗病毒免疫反应中发挥重要作用[2]。TRIM11(含有468个氨基酸残基)是一种典型的RBCC蛋白且形成寡聚物,在细胞质与细胞核中都存在[3]。近年来有研究表明,TRIM11可以与TBK1含有接头蛋白的复合体结合,抑制RIG-Ⅰ介导的IFN-β产生的信号通路,降低Ⅰ型疱疹病毒感染中IFN的产生量,负调控抗病毒免疫反应[4]。
猪伪狂犬病毒(pseudorabies virus,PRV)也称为奥叶基氏病毒或猪Ⅰ型疱疹病毒,是疱疹病毒科中疱疹病毒甲亚科的一员,其基因组是一个双链线性DNA分子[5]。主要感染猪和其他家畜,在偶然情况下也会感染野生动物[6]。猜测猪TRIM11基因在病毒感染细胞过程中也可能起到负调控抗病毒免疫反应作用。作者选择猪的TRIM11基因作为研究对象,对其进行克隆,并构建真核表达质粒PB-TRIM11,之后转染PK15细胞,构建稳定表达猪TRIM11基因的PK15细胞系。通过PRV感染过表达细胞系,对TRIM11基因对PRV增殖的作用进行了初步研究。
1 材料与方法
1.1细胞和病毒
猪伪狂犬病毒(PRV strain Bartha K16),猪肾传代细胞PK15细胞,非洲绿猴肾细胞Vero细胞均由本实验室保存。
1.2主要试剂
猪颌下淋巴结、睾丸、肺等组织提取RNA反转录的cDNA,大肠杆菌(Escherichiacoil)感受态Top10由本实验室保存;氨苄青霉素、嘌呤霉素为美国Sigma公司产品;ExTaq酶、反转录试剂盒PrimeScriptTMRT Reagent Kit with gDNA Eraser(Perfect Real Time)、SYBR®Premix Ex TaqTMⅡ(Tli RNaseH Plus)、DL2000为TaKaRa公司产品;Q5®热启动超保真DNA聚合酶、限制性内切酶NheⅠ、SwaⅠ、T4 DNA连接酶为NEB公司产品;质粒中提试剂盒为QIAGEN公司产品;转染试剂Turbofect为Thermo公司产品。
1.3引物设计
参照GenBank中的猪源TRIM11(序列号:GACC01000116.1)基因序列,设计了包含NheⅠ和SwaⅠ酶切位点的扩增引物,以及用于Q-PCR定量的特异引物。引物均由生物工程(上海)股份有限公司合成(表1)。
表1扩增TRIM11和参照基因引物
Table 1The primers used to amplifyTRIM11 and reference gene
1.4猪TRIM11基因序列和系统进化分析
用NCBI(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)预测猪TRIM11的CDS序列,猪TRIM11分子大小和等电点用DNAStar进行计算,二级结构域模式分析用SMART(http://smart.embl-heidelberg.de)。构建猪TRIM11的系统进化树。首先从NCBI数据库上下载不同物种间TRIM11蛋白序列,然后在MEGA 6 software中构建系统进化树,计算方法为NJ法。NJ法系统进化树的稳健性使用自展内部分枝法(Bootstrapping)评定,Bootstrap Replication为1 000。
1.5猪TRIM11基因的克隆与PB-TRIM11表达载体构建
利用PCR技术,以实验室保存的猪颌下淋巴结组织提取的RNA反转录成的cDNA为模板,扩增得到猪TRIM11基因的cDNA序列。产物通过1%琼脂糖凝胶电泳分离回收大小为1 432 bp左右条带,并进行纯化。用NheⅠ和SwaⅠ分别双酶切载体PiggyBac和纯化得到的目的片段,连接过夜后转化Top10感受态细胞。将转化后的菌液涂布于预先加入Amp的LB琼脂平板,37 ℃过夜。挑取单菌落扩大培养,采用菌液PCR,琼脂糖凝胶电泳筛选阳性克隆,命名为PB-TRIM11,送生物工程(上海)股份有限公司测序。将鉴定正确的质粒按中提试剂盒说明书进行提取,-20 ℃保存,备用。
1.6PB-TRIM11质粒转染PK15细胞
将处于对数生长期的PK15细胞接种于6孔板中,达到80%融合度时,利用Turbofect进行转染。48 h后以PiggyBac系统自带的GFP为依据,荧光显微镜下观察转染效率。更换含有4 μg·mL-1嘌呤霉素的培养基进行筛选,加压筛选4周后,荧光显微镜下可见有绿色荧光且抗嘌呤霉素的阳性细胞克隆,扩增培养即可得到稳定表达猪TRIM11的PK15细胞系。
1.7荧光定量PCR法检测TRIM11 mRNA转录水平
将稳定转染TRIM11基因和稳定转染PiggyBac空载体的PK15细胞,以7.5×105·孔-1分别接种于6孔板中,每组在6孔板中做3个复孔,6 h后收集细胞,Trizol法提取细胞总RNA,按照反转录试剂盒操作说明将RNA反转录为cDNA,以cDNA为模板,荧光定量PCR法检测TRIM11 mRNA表达水平。两组之间比较采用independent-sampleTtest (P<0.05)。
1.8TRIM11对PRV增殖的影响
1.8.1猪伪狂犬病毒(PRV strain Bartha K16)的培养及预处理将实验室保存的一支PRV悬液接种于长满单层PK15细胞的25 cm2细胞瓶内,于37 ℃孵育1 h后,每瓶加8 mL含2%胎牛血清的DMEM维持液,培养至24 h细胞出现明显病变时收获培养物。将培养物反复冻融3次后,1 500 r·min-1离心15 min,收集病毒上清液,置-80 ℃备用。取96孔微量培养板,每孔接种1×104个PK15细胞,待细胞长成单层后,取100 μL制得的病毒悬液,用DMEM培养液进行10倍稀释(10-1~10-10),同时设立阴性对照,接种96孔微量培养板,测定所制病毒悬液的TCID50。TCID50计算按Reed-Muench法进行。
1.8.2过表达猪TRIM11对PRV增殖的影响将稳定转染TRIM11基因和稳定转染PiggyBac空载体的PK15细胞,以7.5×105·孔-1分别接种于6孔板中,每组做3个复孔,待细胞长至单层后接种0.1 MOI PRV,37 ℃孵育1 h后用PBS漂洗细胞去除未吸附的病毒,每孔加入2 mL含2%胎牛血清的DMEM维持液继续培养。于病毒感染12、24、36 h后观察细胞病变并收集细胞上清液测定TCID50。取96孔微量培养板,每孔接种1×104个Vero细胞,待细胞长成单层后,取100 μL待测细胞上清液,用DMEM培养液进行10倍稀释(10-1~10-10),同时设立阴性对照,接种96孔微量培养板,测定TCID50。TCID50计算按Reed-Muench法进行。两组之间比较采用independent sampleTtest(P<0.05)。
2 结 果
2.1猪TRIM11基因的序列分析及蛋白质二级结构域模式
首先从NCBI数据库中搜索预测的猪TRIM11基因的CDS序列(序列号为:GACC01000116.1)。猪TRIM11的CDS区长1 407 bp,编码468个氨基酸(图1),蛋白质的相对分子质量为53 ku,等电点为5.409。TRIM家族的显著特点是具有3个结构域(tripartite motif)[7],蛋白质二级结构域模式分析显示TRIM11蛋白含有3个TRIM家族保守的结构域,这三个结构域从N端到C端的顺序依次是RING结构、2个B-box结构域、一个卷曲结构域(coiled-coil domain),此外该蛋白C端结构域为PRY结构域和SPRY结构域。PRY和SPRY结构域组成了B30.2结构域[8](图2)。
2.2猪TRIM11序列相似性和在不同物种间的系统进化
在TRIM11序列系统进化树上,猪和牛、虎鲸、山羊先聚在一起,然后又与太平洋海象聚成一支,与人和褐家鼠最后也聚成了一支。结果显示,猪和牛、虎鲸、山羊的亲缘关系很近,其相似性高达98%、99%、98%,人和猪的相似性为72%(图3)。
2.3荧光定量PCR分析猪TRIM11基因的组织特异性
为了分析猪TRIM11基因mRNA在不同组织中的转录及分布情况,本研究使用了猪的15个组织提取的RNA反转录的cDNA,采用荧光定量PCR的方法进行了分析,以GAPDH为内参,采用与心组织的比值作为目的基因的相对转录水平,猪TRIM11基因在不同组织中的转录和分布明显不同,在肺、脂肪组织中的转录量较高,而在心、十二指肠、回肠、直肠中的转录量较低(图4)。

划线处为起始密码子,边框处为终止密码子,阴影部分从起始密码子到终止密码子依次为预测的RING、BBOX、Coiled-Coil domain、PRY结构域和SPY结构域。结构域起止用三角符号标注The start codon is underlined and the stop codon is boxed.The shaded parts represent the predicted RING domain,two B-box motifs,coiled-coil region,PRV domain,SPRY domain.Use a small triangle to sign the starting point or the ending point of the predicted domain/motif/region图1 猪TRIM11基因的CDS序列和氨基酸序列Fig.1 The CDS sequence and Amino Acid Sequence of porcine TRIM11

图2 猪TRIM11蛋白二级结构域模式Fig.2 Schematic model of porcine TRIM11

用MEGA 6软件采用邻接法构建系统进化树,不同物种TRIM11蛋白序列从GenBank中获得,序列号为:JAA53691(Sus scrofa),XP_004420550.1(Ceratotherium simum simum),XP_011370226.1 (Pteropus vampyrus),XP_004780674.1 (Mustela putorius furo),XP_848259.1(Canis lupus familiaris),XP_004407123.1(Odobenus rosmarus divergens),XP_004284071.1(Orcinus orca),XP_007986332.1 (Chlorocebus sabaeus),XP_006534642.1 (Mus musculus),XP_003980669.1(Felis catus),NP_001071388.1 (Bos taurus),XP_011542587.1 (Homo sapiens),XP_013820385.1 (Capra hircus),XP_006246567.1 (Rattus norvegicus),XP_006147045.1 (Tupaia chinensis),XP_008250757.1 (Oryctolagus cuniculus),XP_010599060.1 (Loxodonta africana)The MEGA 6 software was used to construct the phylogenetic tree with the neighbor-joining method.The protein sequences of TRIM11 from different species were taken from GenBank,under accession numbers:XP_004420550.1 (Ceratotherium simum simum),XP_011370226.1 (Pteropus vampyrus),XP_004780674.1 (Mustela putorius furo),XP_848259.1(Canis lupus familiaris),XP_004407123.1(Odobenus rosmarus divergens),XP_004284071.1 (Orcinus orca),XP_007986332.1 (Chlorocebus sabaeus),XP_006534642.1 (Mus musculus),XP_003980669.1 (Felis catus),NP_001071388.1 (Bos taurus),XP_011542587.1 (Homo sapiens),XP_013820385.1 (Capra hircus),XP_006246567.1 (Rattus norvegicus),XP_006147045.1 (Tupaia chinensis),XP_008250757.1 (Oryctolagus cuniculus),XP_010599060.1 (Loxodonta africana)图3 不同物种间TRIM11蛋白的系统进化分析Fig.3 Phylogenetic tree of the TRIM11 proteins from different species
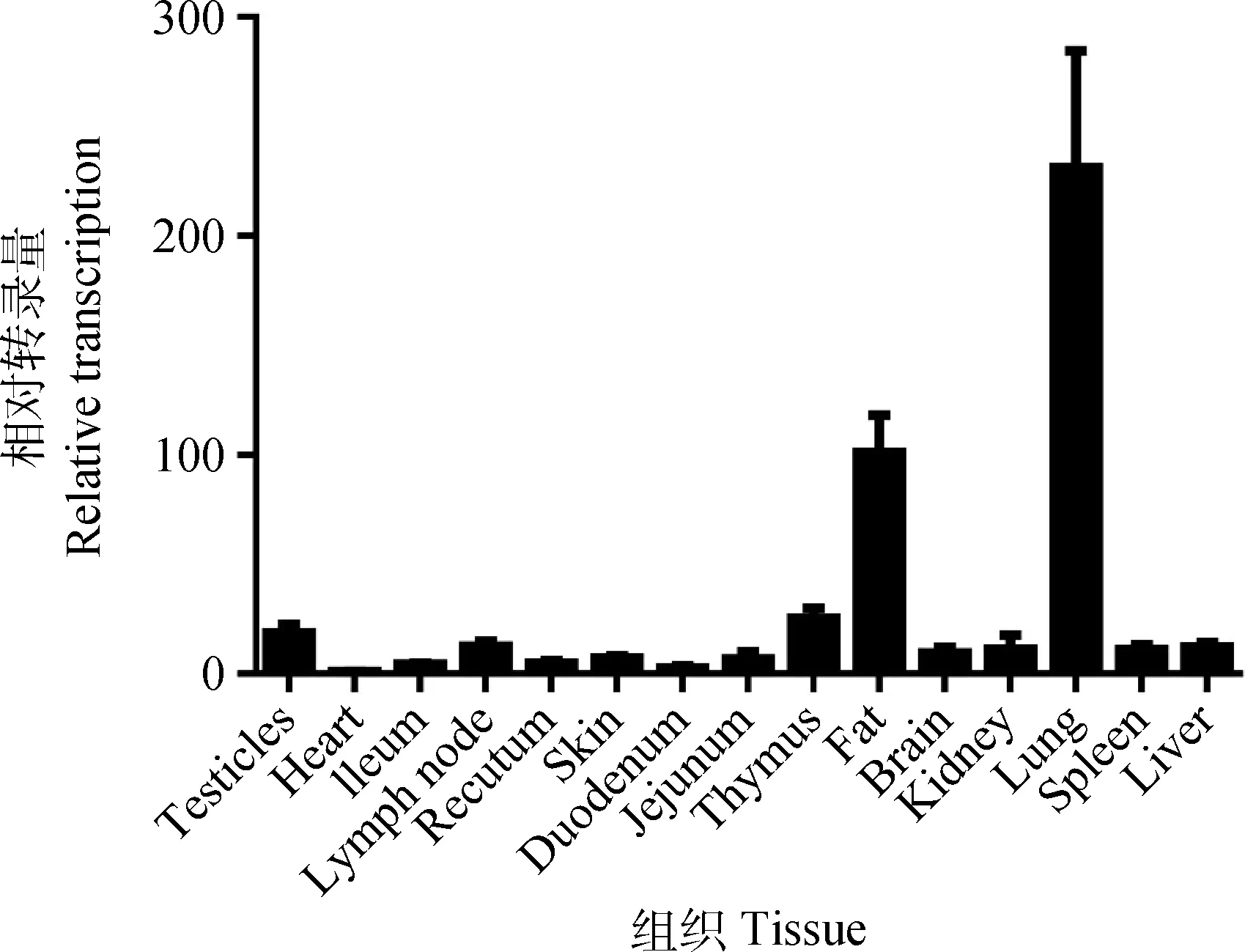
图4 猪TRIM11 mRNA在不同组织中的相对转录量Fig.4 Relative porcine TRIM11 mRNA transcription in different tissues
2.4猪TRIM11基因的克隆及PB-TRIM11重组质粒的构建
采用降落PCR技术,扩增TRIM11基因。PCR扩增产物经1%琼脂糖凝胶电泳检测,获得单一目的条带,目的片段大小与预期的TRIM11基因(1 432 bp)一致(图5)。将经NheⅠ和SwaⅠ双酶切的载体PiggyBac和猪TRIM11基因连接,转化Top10感受态,经氨苄青霉素筛选,进行菌液PCR鉴定。将鉴定为阳性的菌液送至上海生物工程股份有限公司测序,测序结果Blast分析显示,该重组质粒包含了猪TRIM11基因CDS序列与两端的酶切位点序列,提示成功构建了猪PB-TRIM11重组质粒。

M.DNA相对分子质量标准DL2000;1.TRIM11的PCR扩增产物M.DNA marker (DL2000);1.PCR product of TRIM11图5 猪TRIM11基因的PCR扩增产物Fig.5 PCR product of pig TRIM11
2.5PB-TRIM11转染PK15的荧光显微镜观察
将PB-TRIM11重组质粒转染PK15细胞,由于PiggyBac载体上有绿色荧光蛋白GFP,转染48 h后在荧光显微镜下观察转染效果(图6)。经嘌呤霉素筛选4周后,可见单克隆出现,细胞GFP表达稳定。
2.6猪PB-TRIM11重组质粒在细胞中的表达检测
以GAPDH为内参,荧光定量检测稳定转染PB-TRIM11细胞中的TRIM11 mRNA的转录情况。结果显示(图7),和转染PiggyBac组相比,稳定转染PB-TRIM11组的TRIM11转录量显著升高,差异具有统计学意义(P<0.05)。
2.7过表达猪TRIM11对PRV病毒增殖的影响
经过测定,制得的PRV悬液在PK15细胞上的TCID50为10-6.23·0.1 mL-1。过表达TRIM11组与PiggyBac空载组的细胞分别接种0.1 MOI PRV 12、24、36 h后,于显微镜下放大100倍观察细胞病变情况,可以看到两组细胞于12 h开始出现病变,24 h细胞变圆形成合胞体病变明显,36 h细胞脱落明显出现拉丝现象。

A1、B1.PK15细胞;A2、A3.转染PiggyBac空白载体和PB-TRIM11载体48 h的PK15细胞;B2、B3.分别表示转染PiggyBac空白载体和PB-TRIM11载体28 d的PK15细胞A1,B1.PK15 cells;A2、A3.PK15 cells transfected with PiggyBac empty vector or PB-TRIM11 vector after 48 hours;B2,B3.PK15 cells transfected with PiggyBac empty vector or PB-TRIM11 vector after 28 days (200×)图6 荧光显微镜下转染结果(200×)Fig.6 Transfection results under fluorescence microscope (200×)
过表达TRIM11组与PiggyBac空载体组的细胞分别接种0.1 MOI PRV 12、24、36 h后,收取细胞上清液检测病毒滴度发现,过表达TRIM11组的病毒滴度始终高于PiggyBac空载体组,这种差异在12 h时更明显,约1-log10(P<0.05),随着时间的延长而缩小,提示过表达TRIM11在PRV感染早期,一定程度上促进了病毒的增殖(图8)。

Control.转染PiggyBac vector组;*. P<0.05;以GAPDH为内参Control.The PK15 cells stably transfected with PiggyBac vector;* indicated the significant level at 5%;GAPDH was used as internal standard图7 荧光定量PCR检测PK15细胞中TRIM11 mRNA转录水平Fig.7 The transcription level of TRIM11 mRNA in PK15 cells determined by fluorescence quantitative
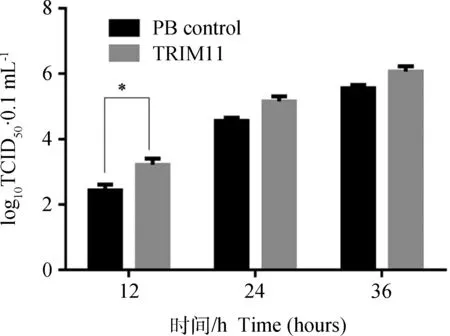
*.P<0.05图8 稳定表达TRIM11的PK15细胞对PRV增殖的影响Fig.8 The PK15 cells stably expressing TRIM11 influnence on PRV multiplication
3 讨 论
最大的含有RING结构域的蛋白家族是三结构域(TRIM)蛋白也称为RING/B-box/Coil(RBCC)蛋白家族。在功能上,许多TRIM蛋白家族成员如TRIM8、TRIM25、TRIM32等的RING结构具有E3泛素连接酶活性[9]。近年来的研究表明TRIM蛋白在病毒感染中调节Ⅰ型干扰素产生的有关信号通路,调控抗病毒免疫反应。例如,最近有研究报道TRIM32参与病毒诱导的MITA泛素化修饰,正调控Ⅰ型干扰素表达信号通路,抑制VSV病毒复制[10]。TRIM25能够泛素化病毒RNA受体视黄酸诱导基因-Ⅰ(RIG-Ⅰ)的N末端,这种改变能够激活RIG-Ⅰ下游配体结合参与线粒体抗病毒信号通路(MAVS)直接引起Ⅰ型干扰素(IFN)的产生从而抑制病毒复制[11]。TRIM8通过介导TAK1 K63连接的聚泛素化,在IL-1β和TNFα诱导的NF-κB激活中充当了一个关键的调节因子,而NF-κB在免疫调节中起着关键作用,TAK1也参与了模式识别受体介导的信号通路[12]。
TRIM11是TRIM/RBCC蛋白家族中E3泛素连接酶中的一员,因其与人体肽相互作用而被知晓。虽然之前也有报道称TRIM11参与了HIV-1复制的抑制[13],但最近在Y.Lee等[4]的研究中发现在HSV-1感染中TRIM11可以和TBK1(RIG-Ⅰ介导的IFN-β产生信号通路的关键因子)的CC2结构域结合抑制IRF3的激活最终导致IFN-β产生的减少,负调控抗病毒免疫反应,这种作用不依赖于其RING结构域。
伪狂犬病毒(PRV)是一种猪的α疱疹病毒,其与人类病原体单纯疱疹病毒Ⅰ型(HSV-1)、HSV-2以及水痘带状疱疹病毒有关[14]。推测在PRV感染中TRIM11也可能抑制抗病毒免疫反应。作者选择猪的TRIM11基因为研究对象,对其进行克隆,构建真核表达载体PB-TRIM11,之后用PB-TRIM11和PiggyBac空载体转染PK15细胞,经嘌呤霉素筛选后,构建稳定细胞系,探究在PK15细胞中过表达 TRIM11 对PK15细胞感染PRV后的影响。结果表明经筛选后的转染PB-TRIM11的PK15细胞比对照组细胞TRIM11基因约过表达1倍。在0.1 MOI的PRV感染细胞0、12、24、36 h时,过表达 TRIM11组上清中的病毒滴度始终高于对照组,且在12 h时这种差异更明显(P<0.05),提示过表达TRIM11在PRV感染早期,一定程度上促进了PRV的增殖。以往有研究[15]RIG-Ⅰ介导了PRV感染早期诱导的IFN-β表达,推测TRIM11促进PRV增殖的作用可能与其负调控RIG-Ⅰ诱导Ⅰ型干扰素产生的信号通路有关,有待进一步研究。
[1]OZATO K,SHIN D M,CHANG T H,et al.TRIM family proteins and their emerging roles in innate immunity[J].NatRevImmunol,2008,8(11):849-860.
[2]仇艳光,王江雁,王沛.TRIM蛋白家族结构与抗病毒功能[J].中国免疫学杂志,2013,29(1):107-110.
QIU Y G,WANG J Y,WANG P.The structure of TRIM family and antiviral function[J].ChineseJournalofImmunology,2013,29(1):107-110.(in Chinese)
[3]ISHIKAWA H,TACHIKAWA H,MIURA Y,et al.TRIM11 binds to and destabilizes a key component of the activator-mediated cofactor complex (ARC105) through the ubiquitin-proteasome system[J].FEBSLett,2006,580(20):4784-4792.
[4]LEE Y,SONG B,PARK C,et al.TRIM11 negatively regulates IFNβ production and antiviral activity by targeting TBK1[J].PLoSOne,2013,8(5):e63255.
[5]YU X L,ZHOU Z,HU D M,et al.Pathogenic pseudorabies virus,China,2012[J].EmergInfectDis,2014,20(1):102-104.
[6]SCOTT A D.Overview of Pseudorabies [J/OL].http://www.merckvetmanual.com/mvm/nervous_system/pseudorabies/overview_of_pseudorabies.html,2014-10.
[7]田利源,陈红星,邓继先.一个新的与泛素化有关的蛋白家族—TRIM家族[J].生物技术通讯,2007,18(2):270-272.
TIAN L Y,CHEN H X,DENG J X.TRIM protein:a family involved in ubiquitination[J].LettersInBiotechnology,2007,18(2):270-272.(in Chinese)
[8]RYU S.Investigation of the FAT10 conjugation pathway[D].Konstanz:University of Konstanz,2012.
[9]PETROSKI M D,DESHAIES R J.Function and regulation of cullin-RING ubiquitin ligases[J].NatRevMolCellBiol,2005,6(1):9-20.
[10]张静.TRIM32调控病毒诱导的Ⅰ型干扰素表达的分子机制[D].武汉:武汉大学,2013.
ZHANG J.The mechanisms of virus-induced expression of type I interferon by TRIM32[D].Wuhan:Wuhan University,2013.(in Chinese)
[11]刘童,安利国,杨桂文.TRIM在病毒免疫防御机制中的作用[J].国际病毒学杂志,2012,19(2):57-60.
LIU T,AN L G,YANG G W.The role of TRIM in the immune defense against virus[J].InternationalJournalofVirology,2012,19(2):57-60.(in Chinese)
[12]LI Q,YAN J,MAO A P,et al.Tripartite motif 8 (TRIM8) modulates TNFα- and IL-1β-triggered NF-κB activation by targeting TAK1 for K63-linked polyubiquitination[J].ProcNatlAcadSciUSA,2011,108(48):19341-19346.
[13]YUAN T,YAO W T,HUANG F,et al.The human antiviral factor TRIM11 is under the regulation of HIV-1 Vpr[J].PLoSOne,2014,9(8):e104269.
[14]BRUKMAN A,ENQUIST L W.Suppression of the interferon-mediated innate immune response by pseudorabies virus[J].JVirol,2006,80(13):6345-6356.
[15]谢立兰.伪狂犬病毒和猪传染性胃肠炎病毒诱导β干扰素产生的分子机制研究[D].武汉:华中农业大学,2011.
XIE L L.Studies on the molecular mechanisms that PRV induced and TGEV induced IFN-β production[D].Wuhan:Huazhong Agricultural University,2011.(in Chinese)
(编辑白永平)
Molecular Cloning of PorcineTRIM11 Gene and Its Promotion Effect on Pseudorabies Virus Replicationinvitro
SONG Shuang,ZENG Lei,LU Shao-fang,GUO Zhen-zhen,LU Wei-fei,HAN Li-qiang,WANG Jiang,WANG Yue-ying,CHU Bei-bei*,YANG Guo-yu*
(KeyLaboratoryofAnimalBiochemistyandNutrition,MinistryofAgriculture,HenanAgriculturalUniversity,Zhengzhou450002,China)
In order to preliminary study the immunologic functions of porcineTRIM11,the cDNA of the target gene was amplified from the cDNA of porcine submandibular lymph node,the protein structure of porcineTRIM11 was predicted and its tissue expression profile was analysed,the cloned porcineTRIM11 cDNA was connected into PiggyBac (PB) transposon vector,then the recombinant vector (PB-TRIM11) was transfected to PK15 cells to establish stably transfected PK15 cell line.The results showed that the coding length of the cloned porcineTRIM11 was 1 407 bp and the sequence code 468 amino acids.Porcine TRIM11 protein was characterized by the presence of the tripartite motif.Tissue transcription profile analysis showed that,the transcription ofTRIM11 was higher in fat,lung,whereas the transcription of porcineTRIM11 was lower in heart,ileum,recutum,duodenum.DNA sequence analysis ofTRIM11 showed that the clonedTRIM11 cDNA was consistent with sequence from GenBank and the recombinant vector (PB-TRIM11) was constructed.The transfected PK15 cell line stably expressing TRIM11 was established successfully and the transcription ofTRIM11 was identified by qRT-PCR.PK15 cell line that stably express porcine TRIM11 and control cells were infected with pseudorabies virus (PRV),respectively,then the cell supernatants were harvested at various time points after infection and the virus titers were detected.The results showed that the viral TCID50of theTRIM11 over-expression group was higher than the control all the time.It indicates that theTRIM11 over-expression cells might be more susceptible to PRV.The study provides the experimental basis for the role of porcine TRIM11 in the innate immunity system in the future.
tripartite motif protein;pseudorabies virus;virus multiplication;stable cell line
10.11843/j.issn.0366-6964.2016.06.020
2015-12-01
国家自然科学基金(3149600030) ;农业部转基因专项(2014ZX0801015B);河南省基础与前沿技术研究计划项目(142300413209)
宋爽(1990-),女,河南许昌人,硕士生,主要从事动物生物化学研究,E-mail:Domhill@163.com
褚贝贝,E-mail: chubeibei_hau@hotmail.com;杨国宇,教授,博士,主要从事动物生物化学研究,E-mail: haubiochem@163.com
S852.4;S852.659.1
A
0366-6964(2016)06-1239-08

