家族性乳腺癌和健康遗传高危人群BRCA1/2基因突变研究
栗辰 ,靳彦文,刘晓静,进淑娟,韩小伟,庞汉民,郝晓鹏,曹诚,黄焰
军事医学科学院 a.附属307医院乳腺外科,北京 100071;b.生物工程研究所,北京 100850
乳腺癌是影响女性健康的主要恶性肿瘤之一。乳腺癌易感基因BRCA1、BRCA2(BRCA1/2)是目前研究最确切的与乳腺癌发病相关的2 个基因,具有明显的家族遗传性。国内至今尚无针对汉族家族性乳腺癌患者的BRCA1/2的大样本筛查,小样本的BRCA1/2基因突变研究表明这2 个基因突变存在地域、种族差异。此外,针对家族性乳腺癌患者健康亲属(即健康遗传性高危人群)的BRCA1/2突变的筛查基本空白。我们采用PCR-DNA 直接测序技术,对54例家族性乳腺癌患者及24例健康遗传性高危人群进行BRCA1/2全编码外显子基因序列检测,旨在进一步研究中国汉族女性家族性乳腺癌患者及健康高危人群中BRCA1/2基因的突变情况。
1 资料与方法
1.1 入组标准及研究对象
1.1.1 家族性乳腺癌患者 ①本人经病理学证实为乳腺癌患者;②具有血缘关系的近亲(一级、二级、三级亲属)中至少1人患乳腺癌和(或)卵巢上皮癌;③个体出现2 个原发性乳腺癌;④具有血缘关系的男性近亲患有乳腺癌。
1.1.2 健康遗传性高危人群 ①本人无乳腺癌和(或)卵巢上皮癌;②有符合以上家族性乳腺癌患者条件的一级或二级亲属;③三级亲属中至少有2名患乳腺癌和(或)卵巢上皮癌。
1.1.3 研究对象 分为2 组:①2013 年12 月~2014年10月就诊于解放军307医院乳腺外科的汉族女性乳腺癌患者共54例;②24例健康高危人群主要来自同时期解放军307 医院乳腺外科家族性乳腺癌患者的健康女性亲属。所有研究对象知情同意后,每人抽取外周静脉血5 mL 于EDTA 抗凝管,-20℃冰箱保存备用。
1.2 全血基因组提取
用QIAquick Gel Extraction kit(QIAGEN)提取样本全血基因组DNA,-20℃冰箱保存备用。
1.3 PCR扩增
根据GenBank 中BRCA1、BRCA2基因序列,由北京奥科鼎盛公司合成引物序列,覆盖BRCA1、BRCA2基因的所有编码外显子。用Veriti Thermal Cycler(Applied Biosystems)PCR 扩增仪进行所有扩增反应。PCR 反应总体积为50μL,包括模板DNA 2μL,上、下游引物各2μL,ddH2O 19μL,2×TaqPCR MasterMix 25μL。PCR反应条件:95℃1 min预变性,然后以95℃变性30 s、55℃退火30 s、72℃延伸30 s 行6 循环,以95℃变性30 s、60℃退火15 s、72℃延伸55 s行40循环,72℃延伸7 min。
1.4 琼脂糖凝胶电泳分析
1.0%琼脂糖凝胶电泳后在紫外灯下观察判断正确条带,对琼脂糖凝胶电泳分析未出现亮带的PCR样品重新行PCR 扩增,由北京奥科鼎盛生物科技有限公司进行DNA直接测序。
1.5 数据分析
应用Genious6.0 软件进行核苷酸序列比较,核酸序列位置编码参考美国国家生物技术信息中心网站(NCBI,http://www.ncbi.nih.gov)上GenBank 中野生型cDNA 序列BRCA1(U14680)和BRCA2(U43746)。所检测到的位点与乳腺癌信息中心网站数据库(Breast Cancer Information Core,BIC)对照,判断是否为新发现的突变。不同分组间采用卡方检验进行统计学分析,P<0.05时具有统计学意义。
2 结果
2.1 家族性乳腺癌患者资料
共收集54例家族性乳腺癌患者,年龄28~74岁,≤40 岁的13人(24.1%),>40 岁的41人(75.9%);淋巴结转移的19人(35.2%),淋巴结未转移的35人(64.8%);三阴性乳腺癌7人(13%),非三阴性乳腺癌47人(87%)。
2.1.1BRCA1/2基因突变情况 54例家族性乳腺癌患者中共发现8例基因突变,其中BRCA1突变患者共6例,分别位于外显子3、11、15,其中4例(4/6,66.7%)位于外显子11;BRCA2突变患者共2例,分别位于外显子11、14(表1)。8例均未在BIC 及其他文献中报道,可能为新发现的突变位点。突变类型均为单个碱基置换突变,其中BRCA1突变中3 外显子146G>A 造成天冬氨酸被丝氨酸代替;11 外显子1019A>T 造成赖氨酸被异亮氨酸代替,1405C>T 造成脯氨酸被丝氨酸代替,3551A>G 造成赖氨酸被精氨酸代替,3295G>C造成甘氨酸被精氨酸代替;15外显子4908C>G 造成丝氨酸被精氨酸代替。BRCA2突变中11 外显子2317A>G 造成苏氨酸被丙氨酸代替,14外显子7058C>G造成丙氨酸被甘氨酸代替。
2.1.2BRCA1/2基因突变在不同临床特征中的表达情况 54例家族性乳腺癌患者中BRCA1和BRCA2的总突变率为15.8%(8/54)。其中6例位于BRCA1(11.1%,6/54),2例位于BRCA2(3.7%,2/54)。所有家族性乳腺癌患者中≤40 岁的共13人,BRCA1、BRCA2突变率分别为15.4%和7.7%;>40 岁的共41人,BRCA1、BRCA2突变率分别为21.9%和9.8%。家族中有2人患乳腺癌的患者BRCA1、BRCA2总突变率为10.6%,而有3名或3名以上患乳腺癌的患者总突变率为42.9%。淋巴结有转移的患者共有4例基因突变,突变率为21.1%。
2.1.3BRCA1/2基因突变与三阴性乳腺癌 三阴性乳腺癌中BRCA1/2基因突变较为常见。本研究免疫组化结果符合三阴性乳腺癌的患者总突变率为28.6%(2/7),非三阴性乳腺癌患者的总突变率为12.8%(6/47),二者差异具有统计学意义(P<0.05)。BRCA1突变的患者中2例为三阴性乳腺癌,4例为非三阴性乳腺癌;BRCA2基因突变的患者均为非三阴性乳腺癌(表2)。
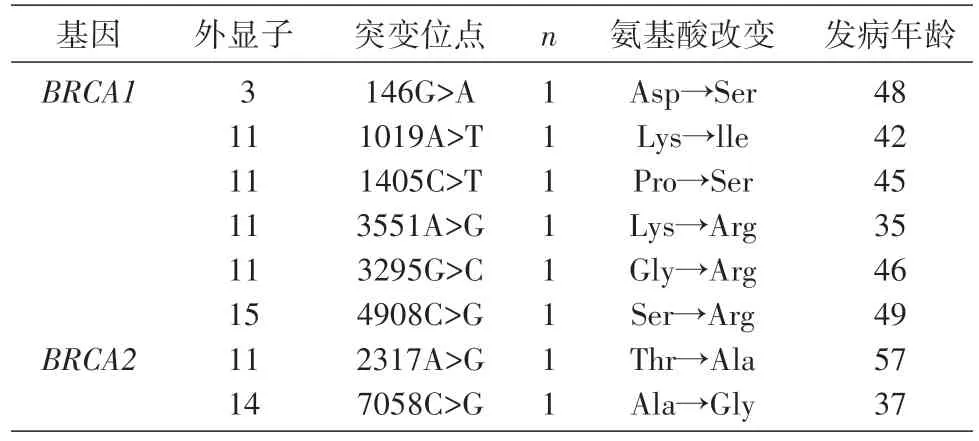
表1 54例家族性乳腺癌患者BRCA1/2突变
2.2 健康遗传性高危人群中BRCA1/2突变
24例健康遗传性高危人群中共发现2例致病性突变,均位于BRCA1的11 外显子,突变率为8.3%(2/24)。突变类型为碱基置换突变,2417T>C 造成异亮氨酸被苏氨酸代替,2618C>T 造成脯氨酸被亮氨酸替换(表3)。BRCA2未发现基因突变。
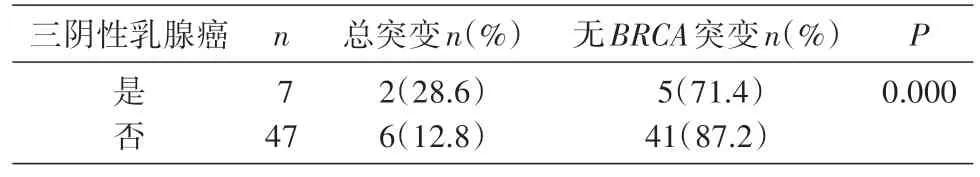
表2 BRCA1/2基因突变与三阴性乳腺癌

表3 遗传性高危人群BRCA1/2突变
3 讨论
乳腺癌的发生、发展是由多因素影响共同产生的结果,遗传因素是其中很重要的一个方面。家族性乳腺癌是指具有家族聚集性的乳腺癌,占所有乳腺癌的5%~10%。家族性乳腺癌中50%~60%具有明确的遗传因素(如基因突变)。
乳腺癌易感基因BRCA1[1]、BRCA2[2]自20 世纪初发现以来,对其研究逐渐深入。BRCA1/2作为肿瘤抑制基因,在维护染色体结构和数目稳定方面具有重要作用。当基因突变发生后,通过改变其蛋白质结构促使染色体不稳定从而失活,进而促进肿瘤的发生和发展[3-5]。目前BRCA1/2突变检查已纳入乳腺癌NCCN指南、ESMO 指南和NCCN 乳腺癌降低风险指南,用于常规指导家族性乳腺癌的筛查、预防与治疗。
国外研究显示,殴裔人群中家族性乳腺癌占全部乳腺癌的5%~10%,BRCA1基因突变率可高达45%[6],在乳腺癌与卵巢癌均高发的家族中BRCA1突变率可高达90%[7]。在欧裔健康人群中,BRCA1/2基因突变率可以高达30%左右,是遗传性乳腺癌发病的极高危因素[8]。BRCA1/2基因突变携带者患乳癌的风险可高达40%~85%[9-10]。Brohet等[11]针对582例BRCA1和176例BRCA2突变的家族进行的患病风险研究发现,截至70 岁,BRCA1突变人群患乳腺癌的风险是45%,BRCA2突变为27%,突变位点和家族遗传强弱与患癌的风险相关。
国内现有家族性乳腺癌相关研究样本量普遍偏小[12-13],BRCA1/2基因突变率约为10%。已报道的家系乳腺癌最大样本量的研究为北京肿瘤医院张娟[14]等对北方地区409例家族性乳腺癌患者进行的BRCA1/2基因突变检测,发现BRCA1/BRCA2突变率为10.5%。乳腺癌患者BRCA1/2基因突变存在明显的种族、地区差异[15]。BRCA1/2可发生多形式、多位点的基因突变,突变可分布于整个编码区域,突变形式大多为移码、缺失或插入。这种形式的基因突变引起乳腺癌发病的危险度可达80%~90%,另存在小部分的碱基置换突变,各种突变形式共同导致同义突变、错义突变和无义突变的结果。BRCA1/2基因突变位点众多,种类复杂,每个种族和地区有其自己的突变特征和规律。综上对国内现有的BRCA1/2基因突变的研究发现:约50%的突变位点为中国人所特有,与白种人的突变位点不一致;与白种人相比,突变检出率普遍偏低。
BRCA1/2是与家族性或遗传性乳腺癌发生发展密切相关的基因,在乳腺癌健康遗传性高危人群中,BRCA1/2基因突变被视为须进行预防干预的极高危因素,但BRCA1/2基因在中国汉族健康遗传性高危人群中的研究基本空白,截至目前,国内针对健康遗传性高危人群仅见河北对家系健康成员的小样本研究[16]。来自香港的Kwong 等[17]首次对69例乳腺癌遗传性高危人群进行1 年随访,发现基因突变检测的人群定期复查率比未检测前明显增高,但仅有个别人选择预防性手术切除,大多数还是选择中药进行预防。除香港外,国内未见任何遗传性高危人群发病风险及预防干预的报道。目前国内对于健康遗传性高危人群BRCA1/2基因突变检测并未出现大样本量的研究,更没有关于健康遗传性高危人群的发病风险的研究,因而对于健康遗传性高危人群BRCA1/2突变的基本特征不明了,对其未来发病风险的情况也无法评估。
因此,本研究共收集54名家族性乳腺癌患者外周血并提取基因组DNA,应用PCR-DNA 直接测序技术对BRCA1/2编码外显子进行直接测序,共发现8例致病性突变,总突变率为14.8%,其中BRCA1和BRCA2突变频率分别为11.1%和3.7%,BRCA1突变是BRCA2突变频率的3 倍(11.1% vs 3.7%)。以上结果略高于国内报道水平。这可能由于国内的大多数研究是基于欧美人群的突变高发区域展开的热点测序,在中国人BRCA1/2突变规律尚未明确的前提下可能会对热点突变区域有所遗漏。另外,国内既往报道中采用的突变检测方法多为PCR-SSCP(single-strand conformation polymorphism,单链构象多态性)或PCR-DHPLC(denaturing high performance liquid chromatography,变性高效液相色谱分析)技术,技术自身的限制性及BRCA1/2基因突变的复杂性导致一部分突变不能被发现,从而造成漏检。本研究使用一代DNA 直接测序技术(金标准),不仅可以准确测定基因组片段中存在的所有突变,并能够覆盖全部编码外显子,所以检测的基因突变率可能会有所提高。本研究中发现的突变类型均为碱基置换突变,但国内外研究报道BRCA1/2基因突变多为移码突变,其他突变类型虽存在但与移码突变相比较少。这可能与检测方法及BRCA1/2突变本身数目庞大、类型多样、具有地域差异等因素相关。三阴性乳腺癌在乳腺癌中所占的比例为15%~20%[18],是一类雌、孕激素受体及人表皮生长因子受体-2 均表达阴性的乳腺癌。研究显示[19],BRCA1/2基因突变在三阴性乳腺癌中较为常见。本研究入组患者中共7例三阴性乳腺癌,BRCA1/2基因总突变率为28.6%,非三阴性乳腺癌患者的总突变率为12.8%。三阴性乳腺癌患者的突变率是非三阴性乳腺癌患者的2.2倍,二者之间差异具有统计学意义,与国内外研究基本一致。
另外,BRCA1/2突变检测对于健康遗传性高危人群的早期预防和预测发病风险具有重要意义,然而国内相关研究却十分罕见。基于此研究的重要性,我们收集24例健康遗传性高危人群,对其BRCA1/2全编码外显子进行DNA 直接测序,结果发现2例致病性突变,突变位点均位于BRCA1,突变率为8.3%,低于国外研究结果[8],可能与样本量较少、种族差异等原因有关。在健康遗传性高危人群中发现BRCA1/2基因突变,意味着今后对该人群值得进行更大样本量的BRCA1/2突变检测,以期能够评估健康高危人群BRCA1/2携带者的发病风险。
综上,我们不仅对中国汉族家族性乳腺癌患者进行了BRCA1/2基因突变检测,进一步了解家族性乳腺癌中BRCA1/2基因突变情况,更重要的是弥补了对健康遗传性高危人群BRCA1/2基因突变检测的空白,对中国健康高危人群乳腺癌的预防及风险评估具有重要意义。
[1]Hall J M,Lee M K,Newman B,et al.Linkage of early-onset familial breast cancer to chromosome 17q21[J].Science,1990,250(4988):1684-1689.
[2]Wooster R,Neuhausen S L,Mangion J,et al.Localization of a breast cancer susceptibility gene,BRCA2,to chromosome 13q12-13[J].Science,1994,265(5181):2088-2090.
[3]Morris J R,Boutell C,Keppler M,et al.The SUMO modification pathway is involved in the BRCA1 response to genotoxic stress[J].Nature,2009,462(7275):886-890.
[4]De Siervi A,De Luca P,Byun J S,et al.Transcriptional autoregulation by BRCA1[J].Cancer Res,2010,70(2):532-542.
[5]Venkitaraman A R.Cancer suppression by the chromosome custodians,BRCA1 and BRCA2[J].Science,2014,343(6178):1470-1475.
[6]Hall M J,Reid J E,Burbidge L A,et al.BRCA1 and BRCA2 mutations in women of different ethnicities undergoing testing for hereditary breast-ovarian cancer[J].Cancer,2009,115(10):2222-2233.
[7]Hall M J,Reid J E,Burbidge L A,et al.BRCA1 and BRCA2 mutations in women of different ethnicities undergoing testing for hereditary breast-ovarian cancer[J].Cancer,2009,115(10):2222-2233.
[8]Konstantopoulou I,Tsitlaidou M,Fostira F,et al.High prevalence of BRCA1 founder mutations in Greek breast/ovarian families[J].Clin Genet,2014,85(1):36-42.
[9]Wang F,Fang Q,Ge Z,et al.Common BRCA1 and BRCA2 mutations in breast cancer families:a meta-analysis from systematic review[J].Mol Biol Rep,2012,39(3):2109-2118.
[10]Roy R,Chun J,Powell S N.BRCA1 and BRCA2:different roles in a common pathway of genome protection[J].Nat Rev Cancer,2012,12(1):68-78.
[11]Brohet R M,Velthuizen M E,Hogervorst F B,et al.Breast and ovarian cancer risks in a large series of clinically ascertained families with a high proportion of BRCA1 and BRCA2 Dutch founder mutations[J].J Med Genet,2014,51(2):98-107.
[12]Rao Nan-Ya,Zhou Jie,Zhao Lin,et al.BRCA1 and BRCA2 deleterious mutation in 219 Han Chinese hereditary breast cancer patients[J].China Oncol,2008,18(5):370-375.
[13]宋传贵,胡震,袁文涛,等.中国上海家族性乳腺癌BRCA1和BRCA2基因的突变[J].中华医学遗传学志,2006,23(1):27-31.
[14]Zhang J,Pei R,Pang Z.Prevalence and characterization of BRCA1 and BRCA2 germline mutations in Chinese women with familial breast cancer[J].Breast Cancer Res Treat,2012,132(2):421-428.
[15]Cao W,Wang X,Li J C.Hereditary breast cancer in the Han Chinese population[J].J Epidemiol,2013,23(2):75-84.
[16]李军改,回天立,李峥,等.家族性乳腺癌家系成员乳腺癌易感基因突变的研究[J].中华乳腺病杂志(电子版),2012,6(6):631-639.
[17]Kwong A,Chu A T,Wu C T,et al.Attitudes and compliance of clinical management after genetic testing for hereditary breast and ovarian cancer among high-risk Southern Chinese females with breast cancer history[J].Fam Cancer,2014,13(3):423-430.
[18]Yao-Lung K,Dar-Ren C,Tsai-Wang C.Clinicopathological features of triple-negative breast cancer in Taiwanese women[J].Int J Clin Oncol,2011,16(5):500-505.
[19]Anders C K,Zagar T M,Carey L A.The management of early-stage and metastatic triple-negative breast cancer:a review[J].Hematol Oncol Clin North Am,2013,27(4):737-749.puffer fish(Takifugu rubripes)[J].Food Chem,2002,78(2):173-177.

