新疆酸驼乳NSLAB的筛选及鉴定
樊哲新,李宝坤,李开雄,朱 敏,蒋琰洁,李王强,宁孔卵
(石河子大学食品学院,新疆石河子832000)
新疆酸驼乳NSLAB的筛选及鉴定
樊哲新,李宝坤*,李开雄*,朱敏,蒋琰洁,李王强,宁孔卵
(石河子大学食品学院,新疆石河子832000)
以新疆牧民家庭自然发酵酸驼乳为原料,采用改良的ROGOSA培养基筛选出20株非发酵剂乳酸菌(Non Starter Lactic Acid Bacteria,NSLAB),以产酸、自溶度、氨肽酶活力、蛋白水解能力为指标复筛性能优良的NSLAB,为新疆特色干酪的现代化改造提供新的菌株资源。结果表明:从酸驼乳中复筛得到六株NSLAB(编号X-4、X-5、X-7、X-12、X-13、X-15)。据多元统计分析六株菌综合得分X-5>X-4>X-15>X-13>X-12>X-7。其中X-5ΔpH(24h)、自溶度、氨肽酶活力、蛋白水解能力分别为1.08%、28.26%、18.88、3.21U/mL,有较好的应用前景。基于菌株生理生化特性,结合16S rDNA基因序列分析,构建菌株系统发育树明确其分类地位,X-4、X-5、X-13、X-15鉴定为植物乳杆菌(Lactobacillus plantarum),X-7鉴定为发酵乳杆菌(Lactobacillus fermentum),X-12确定为乳酸片球菌(Pediococcus acidilactici)。
发酵乳制品,非发酵剂乳酸菌,酸驼乳,菌种筛选
非发酵剂乳酸菌(NSLAB)是干酪微生物中一种独特的菌群,属次生菌群。通过添加NSLAB加速干酪成熟已成为研究热点[1]。干酪发酵过程NSLAB的产酸、凝乳影响很小;在成熟过程中,通过直接代谢或释放各种酶类到干酪中,从而影响干酪风味及质构[2-3]。有研究发现NSLAB含有柠檬酸盐代谢系统及脂蛋白酯酶,酯酶将脂肪分解为脂肪酸,脂肪酸转化成其他的芳香组分,如甲基酮和内酯,另外NSLAB中含有的肽酶、氨基肽酶可将酪蛋白分解为多肽和游离氨基酸,增强干酪的风味[4-7]。但也有研究表明某些菌株会对干酪风味产生消极影响。传统发酵乳制品的最终质量取决于发酵过程中主发酵剂和非发酵剂乳酸菌及其他菌的相互作用[8]。因此,筛选适宜的NSLAB就显得非常重要。
近二十几年来,国内外学者对NSLAB的组成,影响其生长的因素等方面进行了研究,发现原料乳和发酵乳品是筛选NSLAB的主要来源。NSLAB群体主要由嗜温乳杆菌组成,部分片球菌属、肠球菌属及明串珠球菌属也包括在内[9]。董晓婉等[10]从新疆地区酸乳中分离出5株乳酸菌,但未对NSLAB进行确认。目前,尚未有从新疆传统发酵乳制品中筛选NSLAB的研究报道。新疆地区传统发酵乳品如酸奶、酥油、奶酪中微生物资源丰富,从中筛选NSLAB具有可行性。本研究通过改良的ROGOSA固体培养基从伊犁、博州地区采集的12份酸驼乳中筛选NSLAB。由于菌株的生物学特性具有相关性,故以菌株产酸能力、自溶度、氨肽酶活力和蛋白质水解能力为指标,通过多元统计方法复筛出性能优良的NSLAB,为研究NSLAB对干酪成熟过程中风味、质构的影响提供基础。
1 材料与方法
1.1材料与仪器
12份酸驼乳样品采集新疆牧民家庭传统工艺制造,其中精河小海子1份、阔岱管护站1份、伊犁国营牧场3份、那仁和布克牧场2份、伊克乌图布拉格牧场2份、巴音傲瓦乡3份,采集50mL酸驼乳样品装于无菌离心管内,封严后放入车载冰箱于4℃运回实验室,立即进行实验;ROGOSA培养基、MRS培养基、糖发酵培养基等参照相关资料配制[11-12],其中葡萄糖天津盛奥化学试剂有限公司;乳糖天津兴复精细化工研究所;蛋白胨、酵母浸膏北京奥博星生物技术有限责任公司;麦芽糖、甘露糖上海蓝季科技发展有限公司;甘油、柠檬酸铵天津致远化学试剂有限公司;硝酸钠天津天达净化材料精细化工厂;无水碳酸钠天津市北辰方正试剂厂;吐温80天津市福晨化学试剂厂;硫酸镁、硫酸锰天津市福晨化学试剂厂;牛肉膏、琼脂粉北京奥博星生物技术公司;以上试剂均为分析纯;细菌基因组总DNA提取试剂盒、PCR产物柱式纯化试剂盒北京全式金生物技术有限公司;溶菌酶、琼脂糖Sigma公司;2×Taq PCR Master Mix广州东盛生物科技有限公司;Gold View、T载体、DH5α宝生物工程有限公司;L-亮氨酸-对硝基苯胺、酪氨酸、其他常规试剂均购自北京奥博星生物技术有限责任公司。
电热恒温培养箱上海精宏实验设备公司;高速冷冻离心机Eppendorf公司;PCR扩增仪Techne公司;电热蒸汽灭菌锅上海申安医疗器械厂;恒温恒湿培养箱德国BINDER公司;超净工作台苏州苏洁净化设备有限公司;凝胶成像仪美国Bio-Rad公司;细胞破碎仪昆山市超声仪器有限公司;紫外可见光光度计美国GE Amersham Biosciences公司;pH计北京哈纳科仪科技有限公司。
1.2NSLAB的初筛
将酸驼乳样品用无菌的2%柠檬酸三钠(w/v)乳化,并做10倍递增稀释,取10-2、10-3、10-4三个稀释度,分别吸取200μL于调整的ROGOSA固体培养基(4g/100mL NaCl,pH5.0),30℃培养5d。根据菌落的颜色、大小、光泽、光滑度等,挑取单菌落划线接种至MRS固体培养基上,30℃培养1~2d,多次划线分离纯化,得到纯菌株。进行革兰氏染色,镜检观察菌体大小、形状、排列方式,同时进行接触酶实验,参照《乳酸细菌分类鉴定及实验方法》将革兰氏染色阳性、接触酶阴性的菌株初步定为NSLAB。纯菌株斜面保藏,同时用40%甘油保存,置于-20℃保存备用。
1.3NSLAB的复筛
以菌株产酸能力、自溶度、氨肽酶活力和蛋白水解四个特性为指标,采用多元统计法复筛。
1.3.1产酸能力测定将连续活化两代的菌株按照3%(v/v)的接种量接种到150mL高温瞬时灭菌的驼乳中,30℃培养24h。分别测定培养0、24h驼乳的pH,以ΔpH(24h)表示菌株的产酸能力。
1.3.2自溶度的测定[13]将连续活化两代的菌株,低温离心(10000r/min,5min,4℃),将收集到的菌体悬浮于磷酸钠缓冲液(0.1mol/L,pH6.8)中,将菌体悬浮液吸光值(OD650)调整到0.4~0.6,30℃培养24h后测定OD650值。

式中,A1—菌体初始悬浮液吸光度值,A2—菌体培养24h吸光度值。
1.3.3氨肽酶活力的测定[14]用细胞破碎仪制备无细胞提取物,测定无细胞提取物氨肽酶活力。氨肽酶活性的测定以L-亮氨酸-对硝基苯胺为底物,参照Deborah和Storey的方法采用LNT法在405nm处测定酶活力,空白对照不放入菌液。在37℃每分钟生产1μmol对硝基苯胺为一个酶活力单位,用U表示。
1.3.4蛋白水解能力的测定连续活化两代的菌株以3%接种于12g/mL的脱脂乳中,30℃培养12h,以未接菌的脱脂乳作为空白。参照Frank,Church方法作适当修改[15],在340nm处测定培养物的吸光度,测定菌株蛋白水解能力。绘制0.1~3mmol/L酪氨酸标准曲线,对应标准曲线得出蛋白水解活性相当于酪氨酸的量。在37℃每分钟分解牛奶蛋白产生1μmol酪氨酸所需的酶量为一个酶活力单位,用U表示。

式中,K—酶液稀释倍数;W—为生成酪氨酸量,μmol;V—反应酶液体积,mL;t—反应时间,min。
1.3.5数据分析由于菌株产酸能力、自溶度、氨肽酶活力和蛋白质水解能力有一定的相关性,数据在一定程度上具有重叠性。采用SPSS 19.0统计软件对此四个指标进行主成分(PCA)分析。
1.4NSLAB种属的鉴定
1.4.1菌株生理生化特性对纯菌株的生理生化特性的测定参照文献进行[11-12]。
1.4.2分子生物学鉴定
1.4.2.1纯菌株DNA的提取按照细菌基因组总DNA提取试剂盒(全式金)说明书的要求操作(略作改动,第二步添加溶菌酶,水浴2h,用来破坏NSLAB的细胞壁)提取NSLAB菌株的DNA。
1.4.2.216S rDNA片段的PCR扩增对NSLAB16S rDNA进行PCR扩增。PCR采用50μL扩增体系:DNA稀释液2μL,上游、下游引物各2μL,2×PCR Mixture 25μL,ddH2O 19μL;上游引物为U968(5-AACGCGA AGAACCTTAC-3)和L1401(5-CGGTGTGTACAAGA CCC-3)。PCR反应条件:94℃预变性5min,30个循环(94℃预变性30s,56℃退火30s,72℃延伸30s),72℃延伸10min。PCR扩增产物经1%琼脂糖凝胶电泳检测,采用全式金纯化试剂盒割胶纯化回收后,与pMD18-T载体连接,转化入E.coli DH5α中。经蓝白斑初筛,再以T载体通用引物(M13-47和RV-M)进行菌落PCR筛选,将阳性克隆子送到上海捷瑞生物技术有限公司进行测序。利用CHECK-CHIMERA筛除嵌合体,通过NCBI上的Blast程序进行序列同源性分析。
2 结果与分析
2.1NSLAB初筛结果
经改良的ROGOSA选择培养基分离纯化,筛得20株菌株,菌落多呈现圆形、扁平或针尖状,不透明或者半透明,表面光滑,白色或乳黄色。分离得到的杆菌细胞为短杆状或长杆状;球菌细胞为圆形,以单个或成对出现。

图1 菌株革兰氏染色荧光显微镜图片(100×)Fig.1 The images of fluorescence microscope from isolates of gram staining(100×)
2.2NSLAB复筛
2.2.1菌株产酸能力分析NSLAB是一种独特的菌群,在干酪生产过程中,几乎不产生酸,故筛选产酸能力弱或不产酸的菌株[2]。由图2可知,X-8产酸能力最弱,ΔpH(24h)为0.66%,X-9、X-17、X-3的产酸能力较弱,ΔpH(24h)分别为0.71%、0.98%、0.98%;65%的菌株ΔpH变化在1.01%~1.08%之间,说明了所分离的NSLAB在发酵过程对产酸贡献不大。X-15产酸能力最强,ΔpH为1.17%,X-4、X-16的ΔpH(24h)依次为1.16%、1.11%,虽然这三株菌株产酸能力相对较大,但依然符合NSLAB的生物特性。
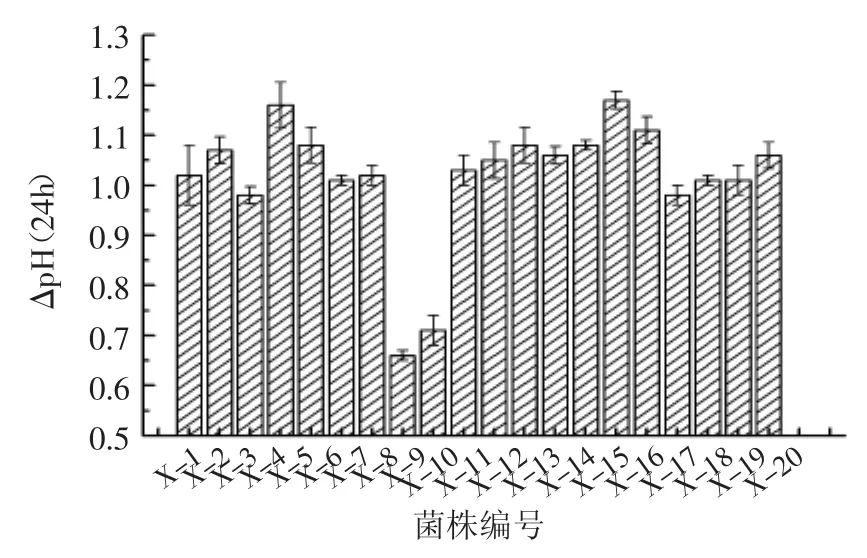
图2 20株NSLAB产酸力Fig.2 Change of pH in culture solution for 20 strains NSLAB
2.2.2菌株自溶能力分析干酪成熟过程中,NSLAB细胞死亡自溶,释放出大量的胞内酶并保持活性,这些酶可将乳中的蛋白质降解为菌体生长所必须的小肽和氨基酸,形成特定风味物质前体或者直接形成干酪的风味[16]。但在发酵过程中菌株自溶又降低了NSLAB的活菌数,这可能会影响乳制品的发酵成熟。NSLAB的自溶特性是由菌株的性质决定的,不同菌株的自溶性强弱有很大的差异。对干酪成熟有促进作用的NSLAB要结合产酸能力来选择。据图3知,菌株自溶度在14.58%~32.02%范围内变化,有较大的差异。X-3自溶度高达32.02%,X-4、X-5、X-13、X-15的自溶度分别是24.31%、28.26%、26.21%、27.57%,自溶度最低的是X-1,仅为14.58%。

图3 20株NSLAB自溶度Fig.3 The extent of autolysis of 20 strains NSLAB
2.2.3菌株氨肽酶活力分析NSLAB产生的肽酶具有降解疏水性氨基酸和降低苦味的潜力[17],添加低产酸能力、高肽酶活力的菌株对干酪成熟具有重要作用。氨肽酶活力适当的NSLAB添加到干酪生产过程中,通过对酪蛋白及其降解产物的作用,提高干酪风味和品质。如图4所示,X-8氨肽酶活力最高,是19.02U/mL,X-5与之活力相当,为18.88U/mL;X-4和X-15氨肽酶活力相差不大,分别为13.63、13.21U/mL,明显要低于X-8;X-2氨肽酶活力最低,仅为7.90U/mL。

图4 20株NSLAB氨肽酶活力Fig.4 The aminopeptidase activity of 20 strains NSLAB
2.2.4菌株蛋白水解能力分析蛋白质水解速率在很大程度上决定着干酪成熟期的长短,加快蛋白质水解速率可以缩短成熟时间。游离氨基酸的增加会增强干酪风味的强度,对不同特色干酪形成独特风味起到积极作用。但如果菌株的蛋白水解能力太强,会使蛋白质水解产生的某些小分子多肽具有苦味,影响了干酪的口味[18-20]。据图5可看出,X-5蛋白水解活力最高,是3.21U/mL;X-2最低,是1.35U/mL;X-12、X-13蛋白水解能力相近,仅比X-5低0.2U/mL;X-15蛋白水解能力为2.64U/mL。筛选蛋白水解能力适中的菌株(蛋白水解能力在2.3~3.1U/mL之间),使干酪在成熟期产生较少的苦味多肽,使口味柔和,更利于我国消费者接受。

表1 主成分的初始特征值及累积贡献率Table 1 Initial characteristic values and cumulative contribution of principal components

图5 20株NSLAB氨肽酶活力Fig.5 The proteolysis ability of 20 strains NSLAB
2.2.5NSLAB生物学特性多元统计筛选通过SPSS 19.0的PCA对四个指标进行降维分析,结果见表1。由表1可知,前两个主成分的特征值均大于1,其中第一主成分的方差贡献率为47.467%,第二主成分的方差贡献率为33.738%,累积方差贡献率为81.207%。说明这两个主成分可以反应原始变量的主要信息。
根据主成分载荷矩阵与特征向量之间的关系,计算出前两个特征向量,得到两个主成分表达式:F1=0.679X1+0.249X2+0.917X3+0.729X4;F2=0.408X1-0.889X2+0.044X3+0.629X4,其中X1~X4分别表示各原始变量经标准化处理后的数值。以各主成分的特征值占所提取主份特征值之和的比例作为权重系数,建立主成分综合得分模型:F=0.584F1+0.416F2。根据主成分综合得分模型,计算综合得分值,并对其进行排序(见表2)。筛选出六株优良性状的NSLAB,即X-4、X-5、X-7、X-12、X-13、X-15。

表2 20株NSLAB的主成分得分及排序Table 2 Principal component scores and ranking of 20 strains NSLAB

表3 NSLAB生理生化项目与糖发酵实验Table 3 Biochemical characteristics and fermentation of sugar of NSLAB
2.3NSLAB种属的鉴定
2.3.1NSLAB菌株生理生化特性实验分别以干酪乳杆菌,乳酸乳球菌作为对照菌株,筛选得到的6株NSLAB进行生理生化特性测定与糖发酵实验,结果见表3,表明X-4、X-5、X-13、X-15不可利用鼠李糖,但可发酵乳糖、麦芽糖、甘露糖和葡萄糖等,与Lb.plantarum(植物乳杆菌)性质相近,故鉴定为Lb.plantarum(植物乳杆菌)。X-12不可利用乳糖、麦芽糖、蔗糖产酸,但可以利用鼠李糖,与P.acidilactici(乳酸片球菌)性质相似。X-7可发酵利用阿拉伯糖、甘露糖,但不发酵甘露醇、山梨醇,与Lb.fermentum(发酵乳杆菌)性质一致。
2.3.2菌株的16S rDNA基因序列通过对NSLAB 16S rDNA序列的PCR扩增,得到清晰的特异性条带,片段大小为450bp左右,电泳图谱结果如图6所示。
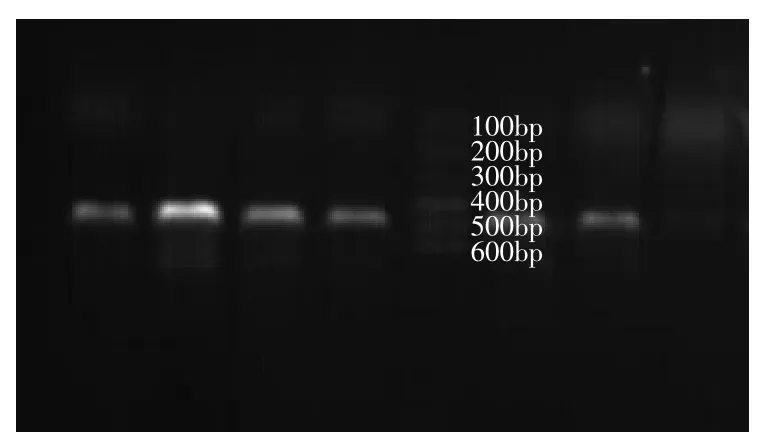
图6 菌株16S rDNA的PCR扩增电泳图Fig.6 PCR amplification pattern of isolates 16S rDNA
复筛得到的6株NSLAB的16S rDNA基因序列与NCBI数据库中已知序列进行比对,选择与其相似性最高且有效发表的典型菌株的16S rDNA基因序列,结合常规生理生化鉴定,确定与实验菌株亲缘关系最近的菌属,使用Mega5.0的邻接法(Neighbor-joinig)构建系统进化树(图7),自展值(bootstrap)为1000。由系统发育树可以看出X-7与Lactobacillus fermentum亲缘性最近,序列同源性为100%,该菌可鉴定为Lactobacillus fermentum;X-4、X-5、X-13和X-15与Lactobacillus plantarum亲缘性最近,序列相似性为100%,确定为Lactobacillusplantarum;X-12与Pediococcus acidilactici亲缘关系为99%,可将该菌定为Pediococcus acidilactici。
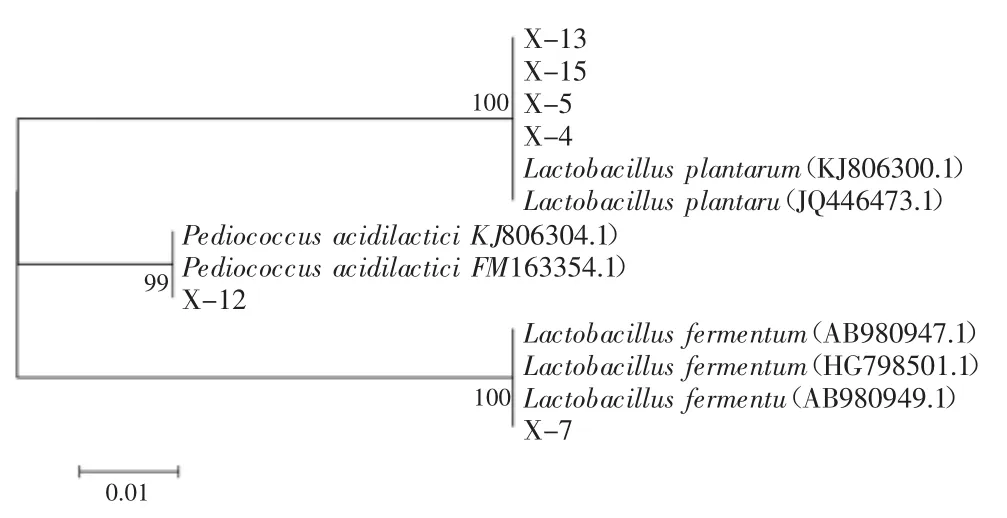
图7 基于16S rDNA序列的6株菌的系统发育树Fig.7 Phylogenetic tree of the 6 strains based on Partial16S rDNA sequences
3 结论
从新疆不同地区酸驼乳中筛选出6株具有优良性状的NSLAB(X-4、X-5、X-7、X-12、X-13、X-15),其中X-5的ΔpH(24h)、自溶度、氨肽酶活力、蛋白水解能力分别为1.08%、28.26%、18.88、3.21U/mL,是性能最优良的NSLAB。16S rDNA基因序列比对结果与生理生化特性鉴定结果相吻合,X-4、X-5、X-13、X-15菌株鉴定为植物乳杆菌(Lactobacillus plantarum),X-7菌株确定为发酵乳杆菌(Lactobacillus fermentum),X-12菌株认定为乳酸片球菌(Pediococcus acidilactici)。由于NSLAB的不可预测性及本身的动力学变化特性,今后应综合考虑干酪成熟期杂菌生长、营养物质匮乏和菌株自身特性等因素对NSLAB促进干酪成熟的影响,并尝试从基因角度分析NSLAB影响干酪风味的机理。
[1]El-Soda M A,Hantira A A,Ezzat N I,et al.Accelerated ripening of ras cheese using freeze-shocked mutant strains of Lactobacillus casei[J].Food Chemistry,1992,44:179-184.
[2]Luca Settanni,Giancarlo Moschetti.Non-starter lactic acid bacteria used to improve cheese quality and provide health benefits[J].Food Microbiology,2010,27:691-697.
[3]Françoise Irlinger,Jérôme Mounier.Microbial interactions in cheese:implications for cheese quality and safety[J].Current Opinion in Biotechnology,2009,20:142-148.
[4]Palles,Beresford,Condon,et al.Citrate metabolism in Lactobacillus casei and Lactobacillus plantarum[J].Journal of Applied Microbiology,2002,85(1):147-154.
[5]Collins Y F,McSweeney P L H,Wilkinson M G.Lipolysis and free fatty acid catabolism in cheese:a review of current knowledge[J].International Dairy Journal,2003,13:841-866.
[6]Upadhyay V K,McSweeney P L H,Magboul A A A,et al. Proteolysis in cheese during ripening[J].In Cheese:Chemistry,Physics and Microbiology,2004,1(3):391-434.
[7]Vassiliadis A,Psoni L,Snikolaou S,et al.Changes in microbial populations,kindsoflacticacidbacteriaandbiochemical characteristics of Greek traditional feta cheese during ripening[J]. International Journal of Dairy Technology,2009,62(1):39-47.
[8]Bassirou Ndoye,Eric Andriamahery Rasolofo,Gisele LaPointe,et al.A review of the molecular approaches to investigate the diversity and activity of cheese microbiota[J].Dairy Science& Technol,2011,91:495-524.
[9]Michael G Casey,Jean Pierre Häni,Josef Gruskovnjak,et al. Characterisation of the non-starter lactic acid bacteria(NSLAB)of Gruyère PDO cheese[J].Lait,2006,86(6):407-414.
[10]董晓婉,李宝坤,卢士玲,等.传统分离培养结合PCR-DGGE技术分析传统乳品中的乳酸菌[J].食品与发酵工业,2014(3):97-101.
[11]东秀珠,蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社,2001:364-385.
[12]凌代文.乳酸菌分类鉴定及实验方法[M].北京:中国轻工业出版社,1999:1-25.
[13]Girbe Buist,Harma Karsens,Arjen Nauta,et al.Autolysis of Lactococcus lactis caused by induced overproduction of its major autolysin,acma.[J].Appl Environ Microbiol,1997,63(7):2722-2728.
[14]Deborah S,Storey R.Proteolytic and trypsin inhibitor activity in germinating jojoba seeds(simmondsia chinensis)[J].Plant Physiology,1981,68(6):1339-1344.
[15]Church F C,Swaisgood H E,Porter D H.Spectrophotometric assay using o-phthaldialdehyde for determination of proteolysis in milk and isolated milk proteins[J].Journal of Dairy Science,1983,66(6):1219-1227.
[16]王秋宇.添加修饰的植物乳杆菌对契达干酪成熟的影响[D].哈尔滨:东北农业大学,2013:13-16.
[17]赵建.半硬质干酪附属发酵剂的筛选及其对干酪风味影响的研究[D].江苏:江南大学,2007:10-13.
[18]Paolo Pirainoa,Teresa Zottaa,Annamaria Ricciardia,et al. Acid production,proteolysis,autolytic and inhibitory properties of Lactic acid bacteria isolated from pasta filata cheeses:amultivariate screening study[J].International Dairy Journal,2008,18:81-92.
[19]Law J,Fitzgerald G F,Daly C,et al.Proteolysis and flavor development in cheddar cheese made with the single starter strains Lactococcus lactis ssp.lactisUC317 or Lactococcus lactis ssp cremoris HP[J].Journal of Dairy Science,1992(75):1173-1185.
[20]倪春梅.乳酸菌分离筛选及其对新型半硬质干酪品质特性影响[D].内蒙古:内蒙古农业大学,2013:9-16.
Screening and identification of NSLAB in Xinjiang acid camel milk
FAN Zhe-xin,LI Bao-kun*,LI Kai-xiong*,ZHU Min,JIANG Yan-jie,LI Wang-qiang,NING Kong-luan
(College of Food Science and Technology,Shihezi University,Shihezi 832000,China)
A total of 20 strains Non Starter Lactic Acid Bacteria(NSLAB)were isolated from home-made fermented camel milk samples of Xinjiang through improved ROGOSA medium.Comprehensive 4 physiological indices including the ability of acidity and the proteolysis,extent of autolysis,the aminopeptidase activity,of NSLAB were used to evaluate the composite score and select evaluation indices in tested high properties NSLAB.By multivariate statistical analysis and composite scores(X-5>X-4>X-15>X-13>X-12>X-7).The results showed that ΔpH(24h),the extent of autolysis,the aminopeptidase activity,the proteolysis ability of X-5 were 1.08%,28.26%,18.88,3.21U/mL,respectively.The isolates were identified by physiology-biochemistry characteristics and the molecular method of 16S rDNA and a phylogenetic tree was contructed.X-4,X-5,X-13,X-15 were identified as Lactobacillus plantarum,X-7,X-12 were identified as Lactobacillus fermentum,Pediococcus acidilactici,respectively.
fermented milks;Non Starter Lactic Acid Bacteria(NSLAB);fermented camel milk;screening
TS261.1
A
1002-0306(2015)12-0170-06
10.13386/j.issn1002-0306.2015.12.027
2014-09-15
樊哲新(1987-),女,在读硕士研究生,研究方向:畜产品加工与安全。
李宝坤(1979-),男,博士,副教授,研究方向:畜产品加工与质量安全。李开雄(1956-),男,教授,研究方向:畜产品加工与质量安全。
国家自然基金青年项目(31201395);新疆生产建设兵团博士资金专项(2014BB005);石河子大学高层次人才启动项目(RCZX201223);国家自然基金地区项目(31460007);国家自然基金地区项目(30960001)。

