几丁质结合蛋白基因克隆、表达与纯化
李美玉,曹洪玉,张庆芳,王晓辉*(大连大学生命科学与技术学院,辽宁大连116622)
几丁质结合蛋白基因克隆、表达与纯化
李美玉,曹洪玉,张庆芳,王晓辉*
(大连大学生命科学与技术学院,辽宁大连116622)
该研究通过聚合酶链反应(PCR)方法从假交替单胞菌属(Pseudoalteromonassp.)DL-6菌株中成功克隆了几丁质结合蛋白基因。PCR测序结果表明,该基因全长1 596 bp,编码531个氨基酸,其理论分子质量为58.517 ku,等电点(pI)4.35,命名为CBP58(Gen-Bank登录号KF234016)。结构域分析结果表明,该蛋白包括1个33家族碳水化合物结合模块(CBM),2个类型3几丁质结合域(ChtBDs);用Insight II 2005软件以同源建模的方法构建CBP58蛋白CBM33结构域的三维结构模型,Ramachandram图谱检测和三维结构评估显示模型结构合理,整体相容性较为可信;将CBP58基因构建到pET23b载体,并转化至大肠杆菌(Escherichia coli)BL21(DE3)中诱导表达;利用镍柱亲和层析纯化获得重组蛋白。十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)检测显示目的蛋白可溶表达,为后期几丁质结合蛋白(CBP)的生化性质表征奠定理论基础。
几丁质结合蛋白;假交替单胞菌DL-6;克隆;表达;纯化
几丁质(chitin)是海洋环境中含量最丰富,自然界中仅次于纤维素的第二大可再生的生物质来源[1]。几丁质的降解产物为高附加值的几丁寡糖(chitin oligosaccharides),其具有良好的生物相容性、生物降解性,而且还表现出调节免疫力、抗菌、抗肿瘤、诱导植物抗病性和促进植物生长等生物活性,可广泛应用于医药工业、食品工业、农业和养殖业等众多领域[2]。开发利用海洋环境含量丰富的可再生资源几丁质,对于解决目前面临的资源、环境和能源危机具有重大意义[3]。微生物降解几丁质主要是通过糖苷水解酶系[4]来完成,即几丁质结合蛋白(chitin-binding proteins,CBP)分散几丁质糖链,内切几丁质酶随机切割多糖链,外切几丁质酶沿糖链末端降解几丁质(见图1)[2],生成的几丁寡糖在β-N-乙酰己糖胺酶或壳二糖酶作用下生成单糖。
几丁质结合蛋白是能与几丁质特异结合的一类蛋白质[3]。CBP在几丁质酶系中单独存在、仅具有几丁质结合域、不具有酶催化活性。CBP不但可以结合几丁质,使其结构变松散,还可以增强酶活性,大大提高降解效率,既节省资源,又绿色环保,具有很高的应用价值[4]。此外,由于几丁质结合蛋白具有结合单糖和多糖底物的能力,从而赋予相应蛋白杀菌,抗真菌活性[5-6]。CAZy数据库根据氨基酸序列相似性,将几丁质结合域归为碳水化合物结合模块(carbohydrate-binding modules,CBM)第1、2、5、6、12、13、14、16、18、19、50、54和55家族。不具有酶催化活性、仅具有几丁质结合域、独立存在的CBP属于第14、18和33家族。2010年,VAAJE-KOLSTAD G等[7]首次报道CBP21具有依赖铜离子溶解性多糖单加氧酶(copper-dependent lytic polysaccharide monooxygenase,LMPO)活性,通过氧化作用断裂几丁质的糖苷键,生成氧化的糖链末端和新的非还原糖链端,使得结晶底物的结构趋于松散,为之后糖苷水解酶的进一步作用提供基础。本研究拟钓取几丁质结合蛋白基因,构建其pET系列的原核表达载体,成功表达并纯化获得大量可溶的表达蛋白,为今后研究其底物结合、协同降解,以及单氧酶功能等奠定理论基础。

图1 粘质沙雷氏菌几丁质酶降解系统图[2]Fig.1 Degradation system diagram ofSerratia marcescenschitinase
1 材料与方法
1.1材料与试剂
1.1.1菌株、质粒与引物
假交替单胞菌(Pseudoalteromonassp.DL-6):中国辽宁大连渤海海域近海底泥6~100 m;E.coliTop 10菌株:实验室保存;质粒pMDTM19-T Vector Cloning Kit:宝生物工程(大连)有限公司[4];表达载体pET23b购自Novagen公司;引物合成和基因测序由生工生物工程(上海)股份有限公司完成。本实验所用的引物见表1。
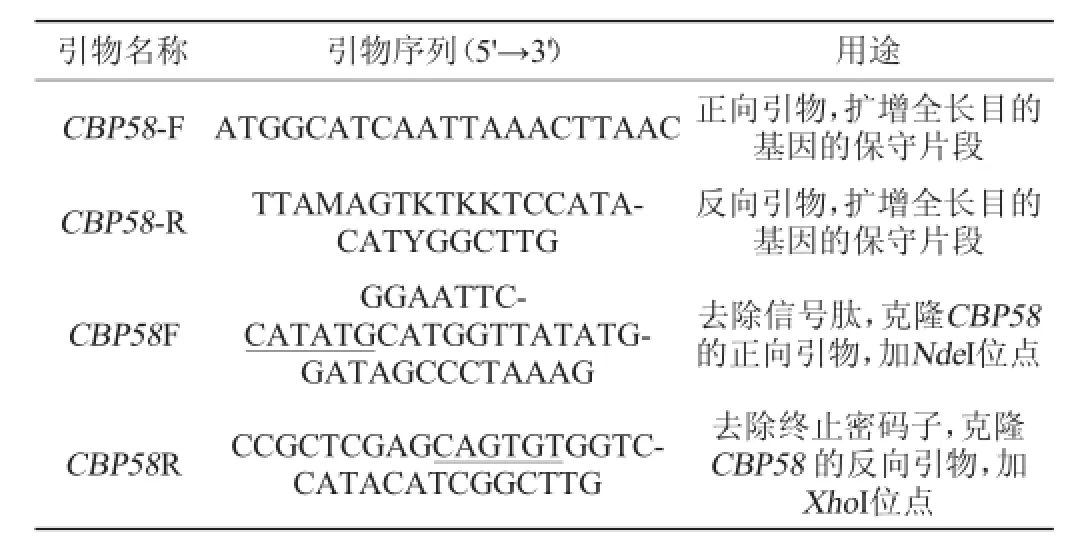
表1 实验所用引物Table 1 Primers used in this experiment
1.1.2培养基
LB液体培养基:胰蛋白胨10 g/L,酵母提取物5 g/L, NaCl 10 g/L,蒸馏水1 000 mL,pH 7.0。
LB固体培养基:向LB液体培养基内加入2%琼脂。
LA培养基:LB液体或固体培养基中加入终质量浓度为100 μg/mL氨苄青霉素。
1.1.3化学试剂
DNA限制性内切酶:美国Amresco公司;细菌基因组DNA提取试剂盒、小量质粒抽提试剂盒、DNA纯化试剂盒、T4连接酶:宝生物工程(大连)有限公司;BM5000 DNA Marker:北京博迈德基因技术有限公司;异丙基硫代半乳糖苷(isopropyl thiogalactoside,IPTG)、氨苄青霉素、二硫苏糖醇、苯甲基磺酰氟:生工生物工程(上海)有限公司;预染蛋白Marler PageRuler Prestained Protein Ladder:美国Thermo公司;镍柱(Ni2+-NTA):美国Novagen公司。其他试剂均为国产分析纯。
1.2仪器与设备
TC-25/HPCR仪:美国ThermoHybaid公司;BOT-860SR紫外凝胶-显微成像仪和扫描仪:上海复日生物技术研究所研制;PowerPac 300蛋白电泳仪:美国Bio-Rad公司;Centrifuge 20PR-52D台式高速冷冻离心机:日本HITACHI公司;MilliQ plus超纯水系统:美国MILLIPORE公司;50 mL超滤离心管(3 ku):生工生物工程(上海)有限公司。
1.3方法
1.3.1CBP58基因的克隆
从平板上挑取菌株Pseudoalteromonassp.DL-6单菌落,接种于50mL种子培养基,20℃、150r/min振荡培养20h,12 000 r/min离心10 min收集菌体,按细菌基因组DNA提取试剂盒操作步骤提取基因组DNA。以基因组DNA为模板,根据美国国家生物技术信息中心(National Center of Biotechnology Information,NCBI)同源序列设计简并引物如下:上游引物(5′-ATGGCATCAATTAAACTTAAC-3′);下游引物(5′-TTAMAGTKTKKTCCATACATYGGCTTG-3′)。聚合酶链反应(polymerase chain reaction,PCR)反应条件为:94℃、2 min,1个循环;94℃、30 s,55℃、30 s,68℃、7 min,30个循环;68℃、15 min,1个循环。PCR扩增产物经1%琼脂糖电泳检测,将PCR产物大小正确的克隆送生工(上海)生物工程股份有限公司进行测序。
1.3.2CBP58基因生物信息学分析
采用Vector NTI Suite软件进行开放阅读框(open reading frames,ORF)分析。序列的同源分析通过NCBI上的本地序列基本搜索工具(basic local alignment search tool,BLAST)完成。采用在线的简单模块构架搜索工具(simple modular architecture research tool,SMART)进行序列结构域分析。采用Align X软件进行氨基酸序列的多重比对。
1.3.3同源模建
将CBP58蛋白的CBM33结构域的氨基酸序列导入Insight II 2005软件中BLAST Search(DS Server)模块,搜索PDB结构数据库选择同源模建的蛋白。根据搜索结果得到与CBP58同源性最高的晶体结构4GBO(来源菌株褐色嗜热裂孢菌(Thermobifida fusca)的E7)构建目标蛋白的空间结构。利用Insight II 2005平台同源建模,Ramachandran plot和Verify-3D评价模型。
1.3.4CBP58表达与纯化
CBP58基因(去除信号肽)和原核表达载体pET23b(+)的PCR产物经NdeI/XhoI双酶切后用T4 DNA连接酶连接,构建重组原核表达载体pET23b-CBP58,并转化至大肠杆菌感受态细胞E.coliTop 10中。双酶切鉴定正确的阳性克隆接入含氨苄的LB培养基中,37℃培养14 h。按1%的接种量接种到含氨苄的LB培养基中。当OD600nm值达到0.6~0.8左右,加入0.2 mmol/L的IPTG,16℃、100 r/min低温诱导6h。诱导表达结束后,4℃、5 000×g离心5 min,收集菌体,用10 mL,20 mmol/L Tris-HCl,50 mmol/L NaCl(pH 8.0)洗涤菌体3次。最后用10mL含1mmol/L二硫苏糖醇,1mmol/L苯甲基磺酰氟,20 mmol/L Tris-HCl和50 mmol/L NaCl(pH 8.0)的缓冲液重悬菌体。离心管置于冰上超声(400 W)破碎3次,每次30 s,每次间隔1 min。4℃、8 000×g离心20min,收集上清液,即为粗蛋白。粗蛋白用镍柱(Ni2+-NTA)进行亲和层析纯化,洗脱蛋白液转至超滤管(3 ku)浓缩后于50 mmol/L Tris-HCl(pH 8.0)缓冲液中4℃保存备用。蛋白样品经十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecylsulfate-polyacrylamidegelelectrophoresis,SDS-PAGE)[15]检测,其中浓缩胶含量5%,分离胶含量12%,考马斯亮蓝R-250染色。预染蛋白Marker用于SDS-PAGE。
1.3.5蛋白质含量测定
蛋白含量通过Bradford蛋白浓度测定试剂盒进行测定。
2 结果与分析
2.1CBP58全长基因的克隆

图2 CBP58基因保守区片段的电泳图谱Fig.2 Electrophoresis pattern of the conserved region fragment ofCBP58
以Pseudoalteromonassp.DL-6菌株基因组DNA为模板,利用引物CBP58-F和CBP58-R,扩增出一条大小约为1 509 bp的DNA条带(见图2),与预计片段大小相近。PCR产物测序表明该基因序列全长1 596 bp,编码531个氨基酸,等电点为4.35,理论分子质量为58.517 ku,根据相对分子质量大小命名为CBP58。该基因提交至NCBI数据库,登录号为KF234016。SMART数据库结构域分析显示,CBP58的N端1-29位氨基酸构成信号肽,30-228位氨基酸构成几丁质结合3区域(chitin_bind_3,CBM33),441-483位和489-531位氨基酸构成类型3几丁质结合域(ChtBD3),即CBP58为多结构域的33家族几丁质结合蛋白。
2.2CBP58基因生物信息学分析
为分析CBP58基因的保守氨基酸残基信息,利用Align X生物信息学软件将编码氨基酸序列与已知晶体结构或研究较透彻的几丁质结合蛋白序列进行比对。所选序列包括粘质沙雷氏菌(Serratia marcescens)CBP21(PDB:2BEM_A),解淀粉芽孢杆菌(Bacillus amyloliquefaciens)CBP33(PDB:2YOW_A),粪肠球菌(Enterococcus faecalis)CBP33(PDB:4A02_A),乳酸乳球菌(Lactococcus lactis)LICBP33A,橄榄绿链霉菌(Streptomyces olivaceoviridis)CHB1,天蓝色链霉菌(Streptomyces coelicolor)CBP33(PDB:4OY7_A)和褐色嗜热裂孢菌(Thermobifida fusca)E7(PDB:4GBO_A)。GUSTAN V K等[8-9]解析了Serratia marcescens菌株CBP21晶体结构,并通过定点突变验证了发挥几丁质结合活性的关键氨基酸(酪氨酸Tyr54、谷氨酸Glu55、谷氨酸Glu60、组氨酸His114、天冬氨酸Asp182和天冬酰胺Asn185),在几丁质结合过程中发挥至关重要的作用。

图3 细菌几丁质结合蛋白CBM33结构域的氨基酸多重序列比对Fig.3 Multiple sequence alignment of the bacteria chitin bindingprotein CBM33 domain
由图3可知,CBP58中的CBM33结构域中存在2个高度保守氨基酸(组氨酸His144和天冬氨酸Asp215),其他4个被谷氨酰胺Gln77、丙氨酸Ala78、酪氨酸Tyr85和谷氨酸Glu219取代,推测这些氨基酸在CBP58与底物结合中发挥关键作用。序列比对发现本研究CBP58具有CBP21发挥氧化酶作用的2个高度保守的活性组氨酸(His30和His144)[10-12]。此外,CBP58还包含CBP21中3个保守的芳香族氨基酸簇酪氨酸Tyr138、色氨酸Trp149、色氨酸Trp211,早期报道该保守区直接位于铜离子结合位点的下方,推测其在电子传递过程[12-13]中发挥作用。本研究的CBP58蛋白不仅具有CBM33结构域,还包含两个ChtBD结构域,这在已报道的CBP结构域中较少见,由此表明CBP58为CBM33家族一名新成员。
应用Insight II 2005软件同源建模几丁质结合蛋白CBP58的CBM33结构域。同源比对分析,系统以褐色嗜热裂孢菌E7的晶体结构(PDB:4GBO_A)为模板生成CBM33结构域的三维结构模型。模型的拉曼图谱(Ramachandran plot evaluation)分析和评估评分(Profile-3D)都表明建模可信合理。由图4可知,CBP58与第一个晶体解析来源于粘质沙雷氏菌33家族几丁质结合蛋白CBP21结构类似,该结构为一个“芽”状纤连蛋白III型褶皱,即3个短的α螺旋组成一个65个氨基酸的“芽”位于β-折叠“三明治”构象的β1和β2之间。比对发现CBP58蛋白结合底物的保守氨基酸(谷氨酰胺Gln77、丙氨酸Ala78、酪氨酸Tyr85、组氨酸His144、天冬氨酸Asp215和谷氨酸Glu219)位于蛋白表面,推测其可特异结合几丁质底物,发挥酶协同降解等生理功能。

图4 CBP58的拉曼图(A)和三维结构(B)分析Fig.4 Ramachandran plot(A)and three-dimensional structure(B)analysis of CBP58
2.3CBP58基因的表达与纯化
2.3.1重组原核表达质粒pET23b-CBP58的酶切鉴定
通过质粒提取试剂盒提取1、2、3、4、6、7、9和10号白斑的重组质粒,用限制性内切酶NdeI/XhoI进行双酶切,酶解产物全量上样1%琼脂糖凝胶电泳检测,结果如图5所示。由图5可知,重组质粒酶切产物片段大小分别约为1.6 kbp和3.7 kbp左右,即表达的CBP58基因和原核表达载体pET23b(+),说明成功地构建了CBP58基因原核表达载体。

图5 重组质粒的双酶切鉴定结果Fig.5 Identification results of recombinant plasmid by double digestion
选取1、4、6和9号构建的重组质粒以通用引物为模板进行PCR测序,测序结果表明1号和4号有一定的点突变,而6号和9号测序结果和PCR产物测序结果完全一致。
2.3.2 CBP58在大肠杆菌内的重组表达
将酶切鉴定与测序鉴定正确的6号重组质粒转化至E.coliBL21(DE3)中异源表达,通过SDS-PAGE分析目标蛋白表达情况。如图6所示。

图6 转化株E.coliBL21(DE3)/pET23b-CBP58表达CBP58Fig.6 Detection of CBP58 expressed by transformantsE.coliBL21(DE3)/pET23b-CBP58
由图6可知,加入0.2 mmol/L IPTG后成功诱导外源蛋白的表达。以没有装载CBP58的E.coliBL21(DE3)/pET23b菌体作为对照,在E.coliBL21(DE3)/pET23b-CBP58菌体裂解液全菌、上清液和沉淀的约58 ku位置清晰可见出现一条蛋白带,该条带分子质量大小与重组CBP58的理论分子质量(58.517 ku)相一致,由此推测该蛋白带为诱导表达的重组蛋白CBP58。
2.3.3 CPB58的Ni-NTA亲和纯化
表达菌株超声破碎后的上清液使用Ni2+-NTA亲和层析柱纯化重组蛋白CPB58,SDS-PAGE检测洗脱收集液,如图7所示。由图7可知,目标蛋白被150 mmol/L、300 mmol/L咪唑溶液洗脱,将150~300 mmol/L咪唑洗脱液合并后用3 ku超滤管超滤浓缩(用50 mmol/L Tris-HCl,pH 8.0缓冲液替换咪唑溶液)。SDS-PAGE检测显示该重组蛋白纯化后得到单一条带,大小约58 ku,与预期大小一致。纯化蛋白质量浓度为1.7 mg/mL,说明目标蛋白CBP58纯度较高且浓度较大。
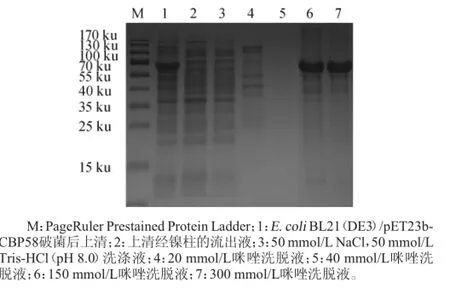
图7 重组CBP58不同洗脱条件下的SDS-PAGE电泳图谱Fig.7 The SDS-PAGE electrophoresis pattern of recombinant CBP58in different elution conditions
3 结论
目前已报道大部分33家族几丁质结合蛋白仅由单独的CBM33模块构成,本研究CBP58除CBM33外还包含两个类型3几丁质结合域(ChtBD3),这些结构域的功能已有文献报道,但具体机制尚未完全阐述清楚[2]。BORASTON A B I等[14]验证CBM的底物结合特性,但是CBM家族到底如何结合底物、提高底物转化效率的机制等并不完全清楚。UNI F等[15]定点突变芽孢杆菌(Bacillussp.J813)几丁质酶中ChtBD的Trp541和Trp542,结果降低了该酶结合与水解粉状晶体几丁质聚合物的效率。KEZUKA Y等[16]突变灰色链霉菌(Streptomyces griseus)HUT6037几丁质酶C上ChtBD的Trp59和Trp60得到同样结论。WATANABE T等[17-20]研究表明ChtBD3在几丁质酶降解粉状晶体几丁质中发挥关键作用,ChtBD3更利于几丁质酶催化域与底物结合、可持续作用几丁质并具有脱乙酰功能[21-24]。
本研究成功克隆了来源海洋菌株的几丁质结合蛋白基因,对基因序列进行了生物信息学分析,同源建模其三维结构,推测该蛋白的功能。并将其在大肠杆菌中实现了过量表达,通过镍亲和柱纯化获得高纯度的目的蛋白,目前深入的研究该蛋白的底物结合、协同降解等生物学功能,为实现其与几丁质酶协同降解几丁质规模化制备几丁寡糖等奠定理论基础。
[1]SOUZA C P,ALMEIDA B C,COLWELL R R,et al.The importance of chitin in the marine environment[J].Mar Biotechnol,2011,13(5):823-830.
[2]VAAJE-KOLSTAD G,HORN S J,SØRLIE M,et al.The chitinolytic machinery ofSerratia marcescens-a model system for enzymatic degradation of recalcitrant polysaccharides[J].FEBS J,2013,280(13):3028-3049.
[3]FREDERIKSEN R F,PASPALIARI D K,LARSEN T,et al.Bacterial chitinases and chitin-binding proteins as virulence factors[J].Res Microbiol,2013,159(Pt5):833-847.
[4]MERINO S T,CHERRY J.Progress and challenges in enzyme development for biomass utilization[J].Adv Biochem Engin/Biotechnol,2007,108:95-120.
[5]AAMER MEHMOOD M H,KHADIM L,FAROOQ R T,et al.Synergistic action of the antifungal-chitin binding protein CBP50 fromBacillus thuringiensiswith bacterial chitinases[J].Curr Proteomics,2014,11(4):23-26.
[6]JAN VAN PARIJS,WILLEM F B,IRWIN J G,et al.Hevein:an antifungal protein from rubber-tree(Hevea brasiliensis)latex[J].Planta,1991,183:258-264.
[7]VAAJE-KOLSTAD G,WESTERENG B,HORN S J,et al.An oxidative enzyme boosting the enzymatic conversion of recalcitrant polysaccharides[J].Sci,2010,330(6001):219-222.
[8]GUSTAV V K,HOUSTON D R,ANN H K R,et al.Crystal structure and binding properties of theSerratia marcescenschitin-binding protein CBP21[J].J Biol Chem,2005,280(12):11313-11319.
[9]GUSTAV V K,HORN S J,DAAN M F V A,et al.The non-catalytic chitin-binding protein CBP21 fromSerratia marcescensis essential for chitin degradation[J].J Biol Chem,2005,280(31):28492-28497.
[10]QUINLAN R J,SWEENEY M D,LO LEGGIO L,et al.Insights into the oxidative degradation of cellulose by a copper metalloenzyme that exploits biomass components[J].P Natl Acad Sci USA,2011,108(37):15079-15084.
[11]FORSBERG Z,MACKENZIE A K,SØRLIE M,et al.Structural and functional characterization of a conserved pair of bacterial cellulose-oxidizing lytic polysaccharide monooxygenases[J].PNAS,2014,111(23):8446-8451.
[12]HEMSWORTH G R,DAVIES G J,WALTON P H.Recent insights into copper-containinglyticpolysaccharide mono-oxygenases[J].Curr Opin Struct Biol,2013,23(5):660-668.
[13]HEMSWORTH G R,TAYLOR E J,KIM R Q,et al.The copper active site of CBM33 polysaccharide oxygenases[J].J Am Chem Soc,2013,135(16):6069-6077.
[14]BORASTON A B I,BOLAM D N,GILBERT H J,et al.Carbohydrate-binding modules:fine-tuning polysaccharide recognition[J].Biochem J,2004,382(Pt3):769-781.
[15]UNI F,LEE S,YATSUNAMI R,et al.Mutational analysis of a cbm family 5 chitin-binding domain of an alkaline chitinase fromBacillus sp.J813[J].Biosci Biotechnol Biochem,2012,76(3):530-535.
[16]KEZUKA Y,OHISHI M,ITOH Y,et al.Structural studies of a two-domain chitinase fromStreptomyces griseusHUT6037[J].J Mol Biol,2006,358(2):472-484.
[17]WATANABE T,ITO Y,YAMADA T,et al.The roles of the c-terminal domain and type-Ⅲdomains of chitinase a1 fromBacillus circulans Wl-12 in chitin degradation[J].J Bacteriol,1994,176(15):4465-4472.
[18]NEERAJA C,MOERSCHBACHER B,PODILE A R.Fusion of cellulose binding domain to the catalytic domain improves the activity and conformational stability of chitinase inBacillus licheniformisDSM13[J].Bioresource Technol,2010,101(19):3635-3641.
[19]MORIMOTO K,KARITA S T,KIMURA S,et al.Cloning,sequencing,and expression of the gene encodingClostridium paraputrificumchitinase chib and analysis of the functions of novel cadherin-like domains and a chitin-binding domain[J].J Bacteriol,1997,179(23):7306-7314.
[20]WANG F P,LI Q,ZHOU Y,et al.The c-terminal module of chi1 from Aeromonas caviaeCB101 has a function in substrate binding and hydrolysis[J].Proteins,2003,53(4):908-916.
[21]KIKKAWA Y,TOKUHISA H,SHINGAI H,et al.Interaction force of chitin-bindingdomainsontochitinsurface[J].Biomacromolecules,2008,9(8):2126-2131.
[22]BORASTON A B,BOLAM D N,GILBERT H J,et al.Carbohydratebinding modules:fine-tuning polysaccharide recognition[J].Biochem J,2004,382(Pt3):769-781.
[23]NIMLOS M R,BECKHAM G T,MATTHEWS J F,et al.Binding preferences,surface attachment,diffusivity,and orientation of a family 1 carbohydrate-binding module on cellulose[J].J Biol Chem,2012,287(24):20603-20612.
[24]EIJSINK V G,VAAJE-KOLSTAD G,VÅRUM K M,et al.Towards new enzymes for biofuels:lessons from chitinase research[J].Trends Biotechnol,2008,26(5):228-235.
Cloning,expression and purification of chitin-binding protein gene
LI Meiyu,CAO Hongyu,ZHANG Qingfang,WANG Xiaohui*
(School of Life Science and Biotechnology,Dalian University,Dalian 116622,China)
Chitin-binding protein gene was cloned successfully fromPseudoalteromonassp.DL-6 by polymerase chain reaction(PCR).The PCR sequencing results indicated that the gene was 1 596 bp in length,and encoded 531 amino acid,and its theoretical molecular mass and isoelectric point(pI)was 58.517 ku and 4.35,respectively.It was named asCBP58(Genebank no.KF234016).The structural domain analysis results showed that the chitin binding protein contained one 33 family carbohydrate-binding modules(CBM),two copies of chitin-binding domain type 3 classified(ChtBDs). The three-dimensional structure model of the homology structure of CBM33 structural domain of the CBP58 protein was built by homologous modeling on Insight II 2005.The diagram of Ramachandram plot and Verify-3D showed that the structure of model was reasonable and the overall compatibility of this model was credible.TheCBP58gene was constructed into the pET23b vector and then transformed intoEscherichia coliBL21(DE3)to be induced for expression.The recombinant proteins were purified by Ni-NTA resin.CBP58was expressed successfully by the analysis of sodium dodecyl sulphate-polyacrylamide gel electrophoresis(SDS-PAGE).The study laid a theoretical foundation for the biochemical characterization of the CBP.
chitin binding protein;Pseudoalteromonassp.DL-6;cloning;express;purification
Q93
A
0254-5071(2015)11-0041-06
10.11882/j.issn.0254-5071.2015.11.010
2015-10-19
国家自然科学基金项目(31500039);大连大学大学生创新创业训练计划项目(2014259)
李美玉(1993-),女,本科生,研究方向为微生物学与酶工程。
王晓辉(1981-),女,讲师,博士,研究方向为微生物与酶工程。

