猪IRS4基因多态性、表达差异及其与脂肪沉积性状的关联分析
马焕班,刘先先,杨 杰,黄卫兵,段艳宇,郭源梅,麻骏武
(江西农业大学猪遗传改良与养殖技术国家重点实验室,南昌 330045)
猪IRS4基因多态性、表达差异及其与脂肪沉积性状的关联分析
马焕班,刘先先,杨 杰,黄卫兵,段艳宇,郭源梅*,麻骏武*
(江西农业大学猪遗传改良与养殖技术国家重点实验室,南昌 330045)
本研究旨在检测猪IRS4基因多态性及其在17种组织中的表达水平,寻找基因内与脂肪沉积性状QTL效应相关的分子标记。本研究采用比较测序方法搜寻到IRS4基因的3个SNPs,分别是在启动子上的g.96C>G,外显子上的同义突变g.1829T>C和错义突变g.1794T>C(p.Phe432Leu)。RT-PCR检测后发现,IRS4基因在垂体、下丘脑中高度表达,在其他组织中弱表达或不表达。在白色杜洛克×二花脸F2资源家系、苏太猪群体、二花脸群体、杜长大三元杂商品猪及中国地方野猪中,用PCR-RFLP方法对IRS4的3个SNPs位点进行判型,然后分析它们的基因频率及其与4点(肩、胸、腰、臀部)背膘厚、腹脂重、肌内脂肪含量和眼肌面积的相关性。结果发现,g.96C>G位点在F2家系(含有SSCX QTL)中分离程度很低,被首先排除作为潜在因果突变(QTN)的可能性。g.1794T>C和g.1829T>C位点在所有检测的西方猪种(白色杜洛克和杜长大)是均为TT基因型,而它们的CC基因型频率在二花脸猪中分别为65%和100%。在不同群体中标记关联分析结果显示,g.1794T>C与F2个体所有脂肪沉积表型均极显著相关(P<0.01),但它在苏太猪中仅与IMF显著相关(P<0.05),且与二花脸猪表型关联不显著(P>0.05);g.1829T>C则不仅与F2个体所有脂肪沉积表型均极显著相关(P<0.01),而且与苏太猪的4点膘厚和眼肌面积呈显著或极显著相关。此外,生物信息学分析发现,g.1794T>C错义突变可能对IRS4蛋白的功能影响不大。结果提示,该错义突变也不太可能是影响脂肪沉积的QTN,而g.1829T>C与F2和苏太猪的脂肪沉积性状都显著关联,故其可作为这些性状选育的分子标记,并值得深入研究。
猪;IRS4基因;QTL;多态性;表达差异;脂肪沉积性状
脂肪沉积性状是猪生产及育种中最为重要的经济性状指标之一。猪皮下脂肪的含量,如背膘厚度和腹脂重,主要决定胴体品质;而肌内脂肪含量(IMF)主要影响猪肉的嫩度、多汁性及加工后的风味等肉质特征。
国内外多个研究小组利用不同西方猪种和中国地方品种梅山猪为祖代杂交构建的资源家系,先后在猪X号染色体近着丝粒区域上定位到了与猪脂肪沉积、生长和胴体组成等性状相关的数量性状位点(QTL)。D.Milan等[1]利用所构建的大白和梅山猪资源家系,在X染色体上标记SW1994附近检测到了显著影响眼肌重和背膘重的QTL区域,该QTL效应分别可以解释36%(眼肌重)和41%(背膘重)的表型变异。H.Ai等[2]利用白色杜洛克(白杜)×二花脸资源家系F2群体,也在SW1994周边定位到了与脂肪沉积性状相关的QTL。J.Ma等[3]曾发现该QTL区域内存在一个较大的重组冷点区(约34 Mb),这使得利用传统的精细定位方法很难进一步缩小QTL的置信区间。M.Perez-Enciso等[4]对来自6个品种(野猪、梅山、大白、长白、伊比利亚、皮特兰)和5个杂交系约3 000头个体的合并分析,发现有利于脂肪沉积的基因起源于亚洲猪种,区间界定在SW1994~SW1943区域。S.Cepica等[5-7]对利用以野猪和梅山为基础的资源家系通过增加标记密度同样将该QTL精细定位于SW1994~SW1943,且QTL最高峰位于IRS4和ACSL4基因周边。本研究组与法国Milan组合作通过合并分析白杜×二花脸资源家系F2群体和法国INRA大白×梅山资源家系群体背膘厚QTL的置信区间缩小至6~7 cM,区域内包含ACSL4、SERPINA7(TBG)、IRS4等几个重要的位置候选基因[8]。
IRS4基因编码胰岛素受体底物-4,并在下丘脑组织中高度表达[9]。有研究表明,胰岛素受体底物-4会耦合到瘦素受体上,而瘦素和胰岛素都能够通过作用于下丘脑神经元调节生物体内的能量消耗和葡萄糖稳态,从而参与控制食物摄入,长期控制肥胖,促性腺激素的分泌以及繁殖性能的调控[10-11]。因此,IRS4基因的多态性可能引起脂肪沉积表型变异,是SSCX QTL区域内值得研究的候选基因。
S.Cepica等[5-7]利用比较测序方法,在IRS4基因启动子区和外显子区分别搜寻到g.96C>G和g.1829T>C(GenBank:FN424076.1)两个多态位点(SNPs);并在梅山×大白资源家系F2群体中发现这两个SNPs与背膘均存在极显著关联性,且1829T>C与眼肌深度存在显著关联性[12]。J.Ma等[8]在对脂肪沉积候选基因的研究中,检测了这两个位点在法国农业科学研究院(INRA)所构建的大白×梅山家系F0和F1代个体中的基因型,结果发现,g.96C>G位点等位基因G仅来源于梅山猪,但其在祖代梅山猪的频率很低(≤0.08),而g.1829T>C位点在所检测的梅山猪和大白猪分别只携带等位基因C和T。
猪IRS4基因与人IRS4基因(NM_003604)具有高度同源性,其编码序列与人IRS4基因编码序列相似度为87%,基因所编码的含1 269个氨基酸的蛋白序列(CAZ66650.1)与人IRS4基因编码的含1 257个氨基酸蛋白序列(AAC51738.1)具有92%的高度同源性。因此,分析候选基因IRS4与目标QTL之间的关系,有助于揭示猪脂肪沉积性状的分子遗传机理,并为人类肥胖病遗传研究提供一定的借鉴。
本研究以猪IRS4基因作为SSCX QTL位置候选基因,先通过比较测序,对猪IRS4外显子区域进行SNPs筛查,再在白杜×二花脸F2群体、苏太猪(杜洛克和二花脸杂交18代以上的培育品种)及二花脸猪群体中分析该基因SNPs与猪脂肪沉积性状的相关性,以期鉴别与猪脂肪沉积性状相关联的分子标记,为今后猪育种的标记辅助选择提供确切的遗传学证据。
1 材料与方法
1.1 试验动物
试验动物包括1个本实验室前期构建的白杜×二花脸资源家系F2群体、1个苏太猪群体(522头)和1个纯种二花脸猪群体(336头)。其中,F2资源家系群体是以2头白杜公猪和17头二花脸母猪杂交,产生9头F1公猪和59头F1母猪,再由这些F1代个体随机交配(避免全同胞交配)产生1 912个F2个体而成的。苏太猪群体和二花脸猪群体分别分批购自于苏州市苏太猪育种中心和江苏省常州市焦溪二花脸猪专业合作社,购买时仔猪均在2~3月龄左右,之后被运至实验室租赁的猪场(位于江西省南昌市郊区),并配以相同的饲养模式进行隔离饲养。试验动物在达到所要求的日龄后,均按生猪标准化屠宰程序进行屠宰,同时进行相关表型的测定。
此外,本研究中还检测了50头杜长大三元杂商品猪和15头中国野猪个体的基因型,所检测个体的DNA样本均来源于本实验室基因库。
1.2 方法
1.2.1 表型测定与DNA样本制备 表型测定则由专人负责以减少系统误差,详细测定过程见参考文献[13]。本研究所测定的肉质性状包括:4点背膘厚(Backfat depth)、肌内脂肪(Intramuscular fat,IMF)含量、眼肌面积(Loin eye area,LEA)、腹脂重(Abdomental weight)和后腿重(Ham weight)。用游标卡尺测定240日龄屠宰个体右边胴体4点背膘厚(肩部为颈椎与胸椎结合处,胸部为第6~7胸椎处,腰部为最后肋骨处,臀部为腰荐结合部)。肌内脂肪含量测定使用索氏脂肪抽提法进行测定。眼肌面积的测量采用硫酸方格纸描绘、Leica软件进行计算的方法。腹脂重、后腿重采用电子天平进行称量。
屠宰个体样本采集同样按采样规范操作,用耳号钳夹取30~40 mg的耳组织样本,装在含1 mL 75%酒精的Eppendorf 离心管内,-20 ℃保存。
采用苯酚/氯仿抽提、乙醇沉淀的方法从耳组织中提取全基因组DNA,Nanodrop-1000用于检测DNA样本质量。本试验中按20 ng·μL-1的浓度标准在96孔板中制成DNA模板备用。
1.2.2 SNPs的搜寻 参考NCBI所提供的猪IRS4基因序列(GenBank:FN424076.1)设计4对测序引物进行测序,扩增片段覆盖IRS4基因约3 300 bp的序列,引物的设计采用Premier5.0软件,引物均由上海生工合成,引物序列、退火温度等详细信息见表1。
选取2头杜洛克和4头二花脸个体的DNA为模板,进行PCR扩增。采用改良型的降落PCR(Touch down PCR)扩增程序进行扩增,PCR反应体系为25 μL:40 ng基因组DNA、0.2 mmol·L-1dNTPs、0.2 μmol·L-1的引物、1.5 mmol·L-1Mg2+,1×PCR缓冲液和2UTaqDNA聚合酶(TaKaRa)。降落PCR程序:94 ℃预变性5 min;然后进入PCR扩增程序,94 ℃变性30 s,68~54 ℃ 30 s,72 ℃ 50 s,每降1 ℃循环2次,共28个循环;94 ℃ 30 s,53 ℃ 30 s,72 ℃ 30 s,19次循环;72 ℃延伸1 min,最后降至4 ℃。所得到的产物经2%琼脂糖凝胶电泳检测后,委托上海生工生物工程有限公司测序,测序结果采用DNAStar软件包的Seqman程序进行分析,通过序列比对鉴定该片段上存在的多态位点。
1.2.3 SNPs的基因型检测 使用PCR-RFLP方法检测多态位点基因型,使用Primer5.0软件设计引物,在IRS4基因3个多态位点附近区域进行扩增,引物信息如表2所示。PCR反应体系25 μL:40 ng基因组DNA、0.2 mmol·L-1dNTPs、0.2 μmol·L-1的引物、1.5 mmol·L-1Mg2+,1×PCR缓冲液和2 UTaqDNA聚合酶。PCR程序设计同样为降落PCR:94 ℃预变性5 min,94 ℃变性30 s,68~54 ℃ 30 s,72 ℃ 50 s,每降1 ℃循环2次;94 ℃ 30 s,53 ℃ 30 s,72 ℃ 30 s,19次循环;72 ℃延伸1 min,最后降至4 ℃。
扩增产物用于酶切基因判型。15 μL的酶切反应体系中包含PCR产物5 μL,限制性内切酶2 U,10×缓冲液1.5 μL和去离子水8.3 μL,37或65℃温育6 h,反应产物经4.0%的琼脂糖凝胶电泳检测,从而判断这3个位点的基因型。PCR-RFLP中所使用的限制性内切酶、温育温度和基因型片段大小如表2所示。

表1 SNP搜寻引物序列及相关信息
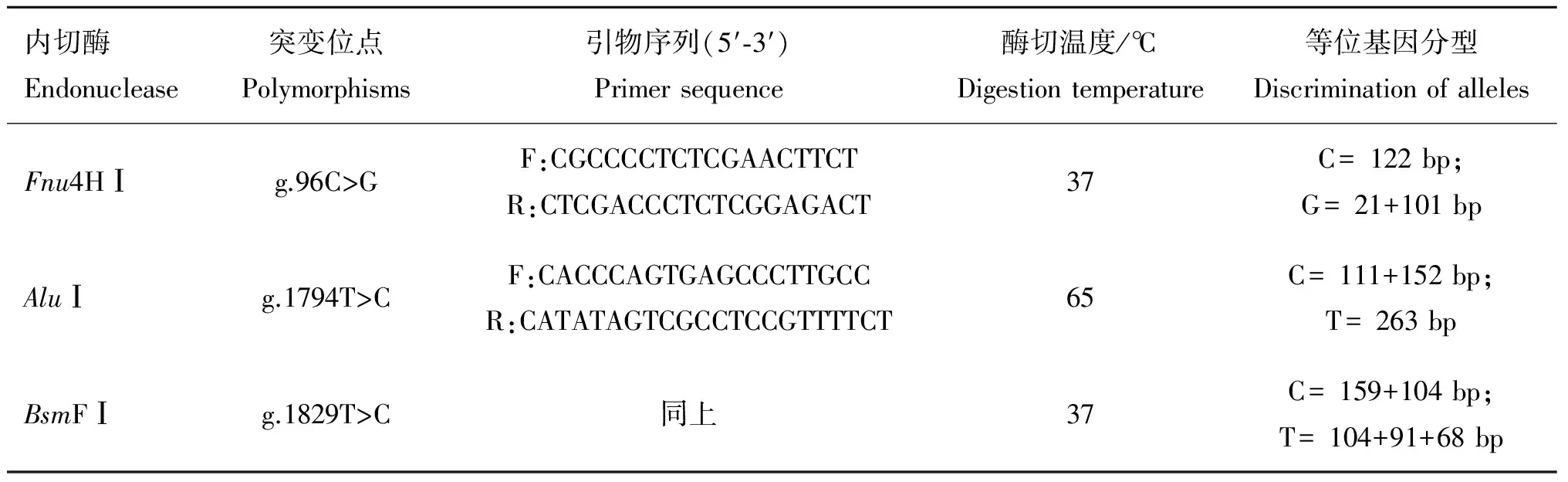
表2 PCR-RFLP方法判定IRS4基因SNPs基因型
1.2.4 组织表达分析 利用半定量RT-PCR(Semi-Quantitative RT-PCR)分析不同组织中IRS4基因表达情况,基因组织表达的17种组织:肾上腺、肾、肺、垂体、卵巢、板油、前列腺、睾丸、心、胸腺、附睾、小肠、胃、甘油、下丘脑、甲状腺、膀胱。所设计的引物序列如表3所示,采用GAPDH看家基因作为反应内参。实时荧光定量PCR(qRT-PCR)检测IRS4基因在不同基因型母猪下丘脑组织表达差异情况,所设计的引物扩增区域包含外显子的两个突变位点,引物信息如表3所示。
1.2.5 统计分析 利用SAS软件的MIXED过程确定分析模型中分别需要考虑的固定效应和协变量,结果将性别和批次作为固定效应,而将胴体重作为协变量,用基因型亲缘系数剔除多基因效应。对不同群体SNP多态性基因型及单倍型与表型进行关联分析,分析模型为加性效应模型,模型:
y=μ+sex+batch+Cw+gkin+snp/hapo+e
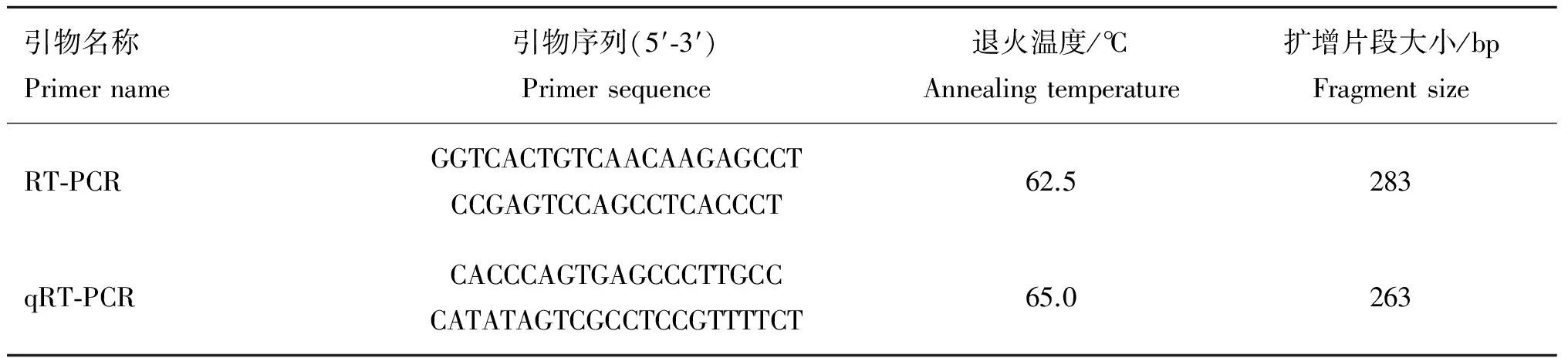
表3 RT-PCR引物信息
其中,y表示表型值,μ为群体平均值,sex和batch分别表示性别和批次,为固定效应,C胴体重回归系数,w为胴体重协变量,gkin表示基于基因型的个体间亲缘关系,snp/hapo为候选基因SNP效应或SNP所构建的单倍型效应,e表示残差。基因位点与表型之间的关联性是基于模型进行判别,如果关联性P值达到或超过显著水平的临界值,说明该位点为显著关联位点。
2 结 果
2.1 SNPs搜寻
通过序列比对,搜寻到了3个SNPs位点,其中两个位点(启动子上的g.96C>G位点和一个同义突变位点g.1829T>C)与此前报道相一致;第3个位点为新发现的位点,它位于同义突变位点g.1829T>C的5′端上游35 bp处,是一个错义突变g.1794T>C(p.Phe432Leu),该位点突变导致相应的氨基酸由苯丙氨酸转变为亮氨酸。
2.2 多态位点的判型与基因型分布频率
使用PCR-RFLP方法判定IRS4基因3个SNPs的基因型。使用限制性核酸内切酶Fnu4HI、AluI和BsmFI分别对g.96C>G、g.1794T>C和g.1829T>C这3个位点PCR扩增产物进行酶切,各位点基因型判定结果见图1。
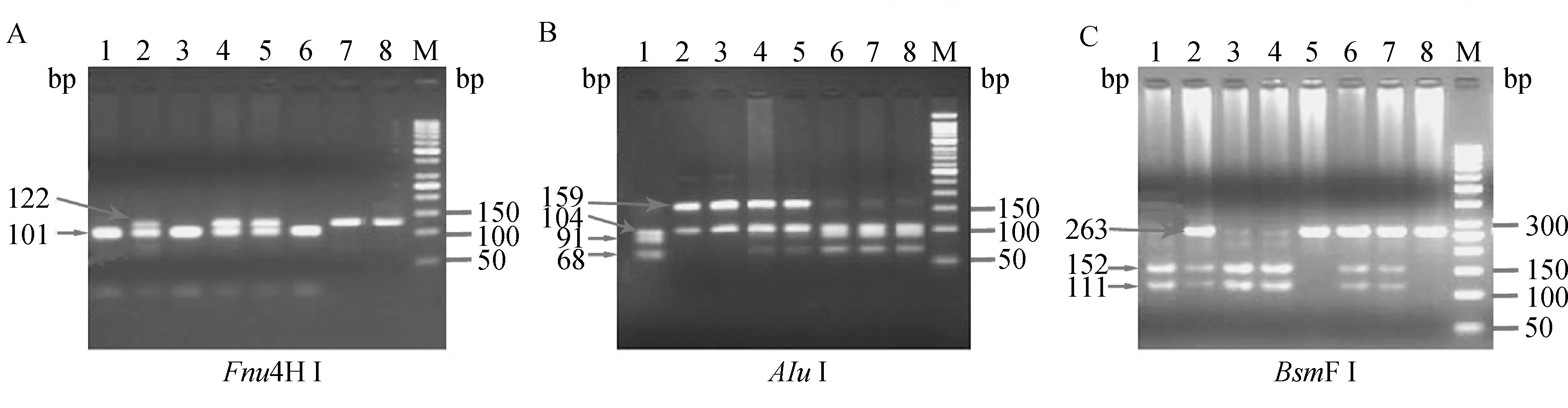
A.限制性内切酶Fnu4HI对g.96C>G分型结果:7、8.CC;1、3、6.GG;2、4、5.CG。B.AluI对g.1794T>C分型结果:2、3.CC;1、6、7、8.TT;4、5.CT。C.BsmFI对g.1829T>C分型结果:1、3、4.CC;5、8.TT;2、6、7.CT。M.DNA相对分子质量标准A.Genotyping results for g.96C>G were obtained by Fnu4HIrestriction enzyme digestion:CC.7,8;GG.1,3,6;GG.2,4,5.B.Genotyping results for g.1794T>C were obtained by AluIrestriction enzyme digestion:CC.2,3;TT.1,6,7,8;CT.2,6,7.C.Genotyping results for g.1829T>C were obtained by BsmFIrestriction enzyme digestion:CC.1,3,4;TT.5,8;CT.2,6,7.M.50 bp ladder DNA marker图1 IRS4基因3个多态位点酶切分型结果Fig.1 The RCR-RFLP-based genotyping results for the 3 IRS4 SNPs
基于上述标准,分别在各研究群体中对这3个位点进行判型,各群体中多态位点的等位基因型频率统计信息见表4。其中,g.96C>G位点在资源家系F0代个体中分离程度较低,仅有2头二花脸母猪为CG型,另外2头白杜公猪和15头二花脸母猪均为CC型,所检测的15头野猪个体也全为CC型。g.1794T>C位点等位基因C在资源家系F0代中仅来源于二花脸母猪,2头白杜公猪和另外检测的50头杜长大三元杂商品肉猪全为TT型。g.1829T>C位点在所有检测的纯种二花脸猪中都为CC型;而在F0代白杜和杜长大三元杂商品猪中都为TT型,野猪群体中没有检测出TT基因型。
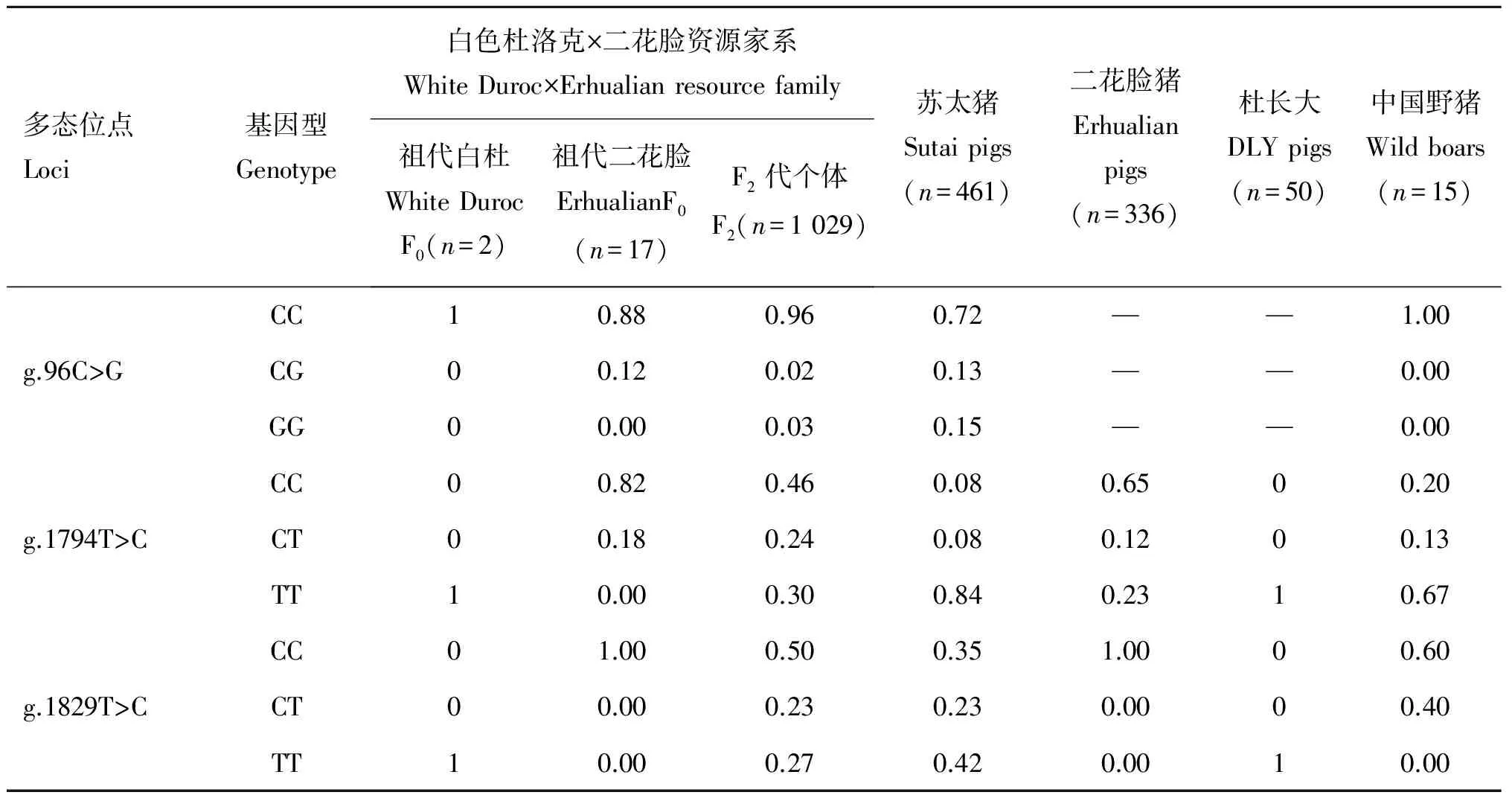
表4 3个SNP位点在不同群体中的基因型分布频率
2.3IRS4在17种不同组织中表达差异性分析
试验结果表明(图2),在猪的17种不同组织cDNA样品中,IRS4基因在垂体、下丘脑高度表达,在卵巢、板油、睾丸、甲状腺中弱表达,在其它组织中微弱或不表达。
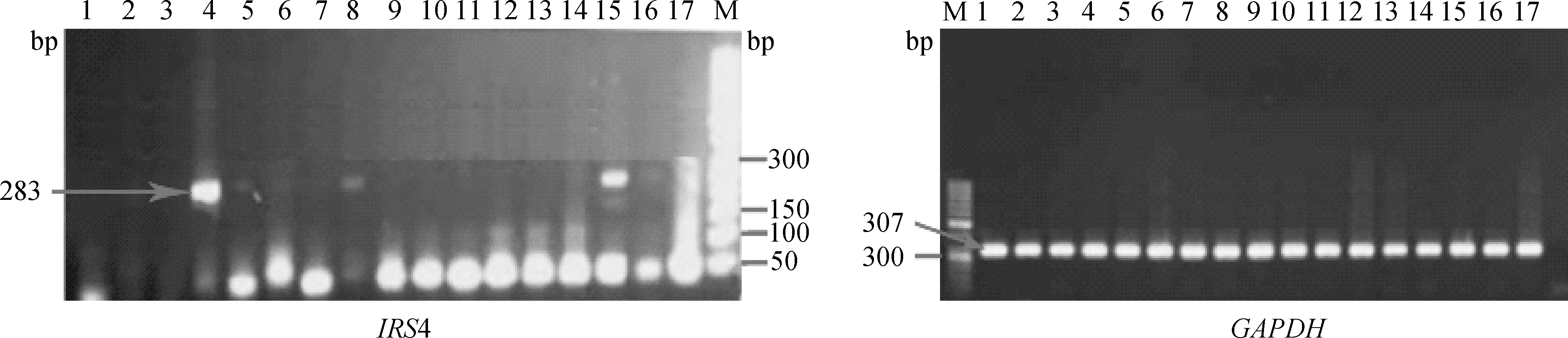
1.肾上腺;2.肾;3.肺;4.垂体;5.卵巢;6.板油;7.前列腺;8.睾丸;9.心;10.胸腺;11.附睾;12.小肠;13.胃;14.肝;15.下丘脑;16.甲状腺;17.膀胱;M.DNA相对分子质量标准1.Adrenal;2.Kidney;3.Lung;4.Pituitary;5.Ovary;6.White adipose tissue;7.Prostate;8.Testis;9.Heart;10.Thymus;11.Epididymis;12.Small intestine;13.Stomach;14.Liver;15.Hypothalamus;16.Thyroid;17.Bladder;M.50 bp ladder DNA marker图2 IRS4基因在猪17种组织中的表达差异Fig.2 The expression pattern of IRS4 gene in 17 porcine tissues
根据错义突变位点g.1794T>C的分型结果,选取CC和CT基因型F2母猪各6头(无TT型母猪样品),分析IRS4基因在下丘脑组织中的表达差异情况。结果显示(图3),CC基因型个体IRS4平均表达量高于CT型,分别为(2.07±4.45)和(0.95±0.43),但两者差异不显著(P=0.15)。这可能是由于检测个体数偏少,且同种基因型个体基因表达差异也较大的缘故。
2.4 关联分析结果
由于g.96C>G位点在资源家系F0代个体中分离程度较低,白杜和二花脸的主等位基因都为G,这与SSCX上脂肪沉积性状QTL等位基因在两个祖代品种各自趋近纯合估计不符,因此排除了该位点作为影响猪脂肪沉积因果突变位点(QTN)的可能性。在后续的分析中,集中探讨了g.1794T>C和g.1829T>C与脂肪沉积性状的相关性。
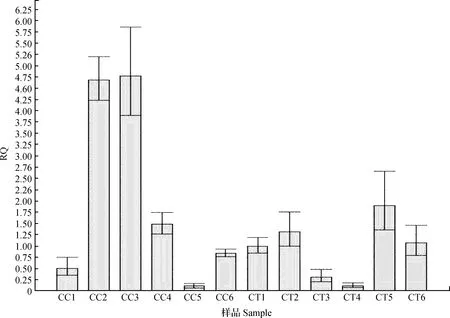
图3 比较g.1794T>C位点CC和CT基因型个体的下丘脑组织中IRS4基因的表达差异Fig.3 The difference in expression level of IRS4 gene in porcine pituitary tissue between pigs with CC and CT genotypes for g.1794T>C
2.4.1 多态位点g.1794T>C和g.1829T>C与脂肪沉积性状的关联分析 在资源家系F2群体、苏太猪群体和纯种二花脸猪群体中,分析了IRS4基因多态位点g.1794T>C和g.1829T>C与脂肪沉积性状的相关性(表5),结果表明,错义突变g.1794T>C与F2群体所有脂肪沉积性状均极显著相关(P<0.01),仅与苏太猪群体IMF显著相关(P<0.05),但与二花脸猪群体所有脂肪沉积性状相关性均不显著;g.1829T>C与F2群体所有脂肪沉积性状均极显著相关(P<0.01),且与苏太猪群体4点膘厚、眼肌面积和后腿重显著或极显著相关。因g.1829T>C在纯种二花脸中不分离,所以无法在二花脸猪中检测该位点的效应。
2.4.2 F2与苏太群体中IRS4单倍型关联分析 根据g.1794T>C和g.1829T>C在资源家系F0代中的基因型判定结果,推出两个位点的相互强连锁(r2= 0.91),并构建了C-C、T-T、T-C共3种单倍型。其中T-C、C-C单倍型来源于二花脸母猪,频率分别为0.18和0.82,T-T单倍型则全部来源于白杜公猪。在苏太群体F0代中,共构建出了G-T-C、C-T-T、C-T-C、C-C-C 4种单倍型,其中G-T-C、C-C-C、C-T-C及C-T-T单倍型来源于二花脸母猪,频率分别为0.29、0.15、0.13和0.43,C-T-T及C-T-C来源于白杜。
在F2群体中,对上述3种单倍型与四点膘厚、腹脂重和肌内脂肪含量做相关性分析,结果发现(表6),二花脸起源的单倍型T-C与C-C效应相近(P> 0.05),且均与杜洛克起源的T-T单倍型效应差异显著(P<0.05),提示在F2群体中g.1829T>C位点比g.1794T>C位点更符合QTL等位基因分离特征或与QTN的连锁不平衡程度更高。在苏太群体中,根据4种单倍型与四点背膘、腹脂重和肌内脂肪含量的相关性分析发现(表7),单倍型主要对臀部膘厚与肌内脂肪含量是有显著影响的。
3 讨 论
有关猪脂肪沉积性状的基因定位和候选基因研究,国内外已有大量的报道[13-16]。已知的影响猪脂肪沉积性状的QTL主要集中在2、4、6、7、X染色体上,但迄今为止仅少数主效基因(如IGF2)被证实[17]。X染色体上与猪脂肪沉积、生长和胴体组成等性状相关的QTL大多定位在近着丝粒区域,但是由于在该区域存在重组冷点区,使得利用传统的精细定位方法很难进一步缩小QTL区间。而且,X染色体存在失活、异质性和性染色体剂量补给等现象,这使得对哺乳动物性染色体的QTL和功能基因研究比常染色体更加困难。目前,X染色体上影响猪脂肪沉积性状QTL的几个候选基因(包括AR、SERPINA7、ACSL4等[18-21])的研究相对较多,但有关猪IRS4基因的研究相对较少。而在人类研究中,IRS4基因多与Ⅱ型糖尿病、代谢紊乱、精神分裂症等疾患相联系。据报道,缺乏IRS4的小鼠会表现出生长、繁殖和糖代谢轻度缺陷[22-24]。
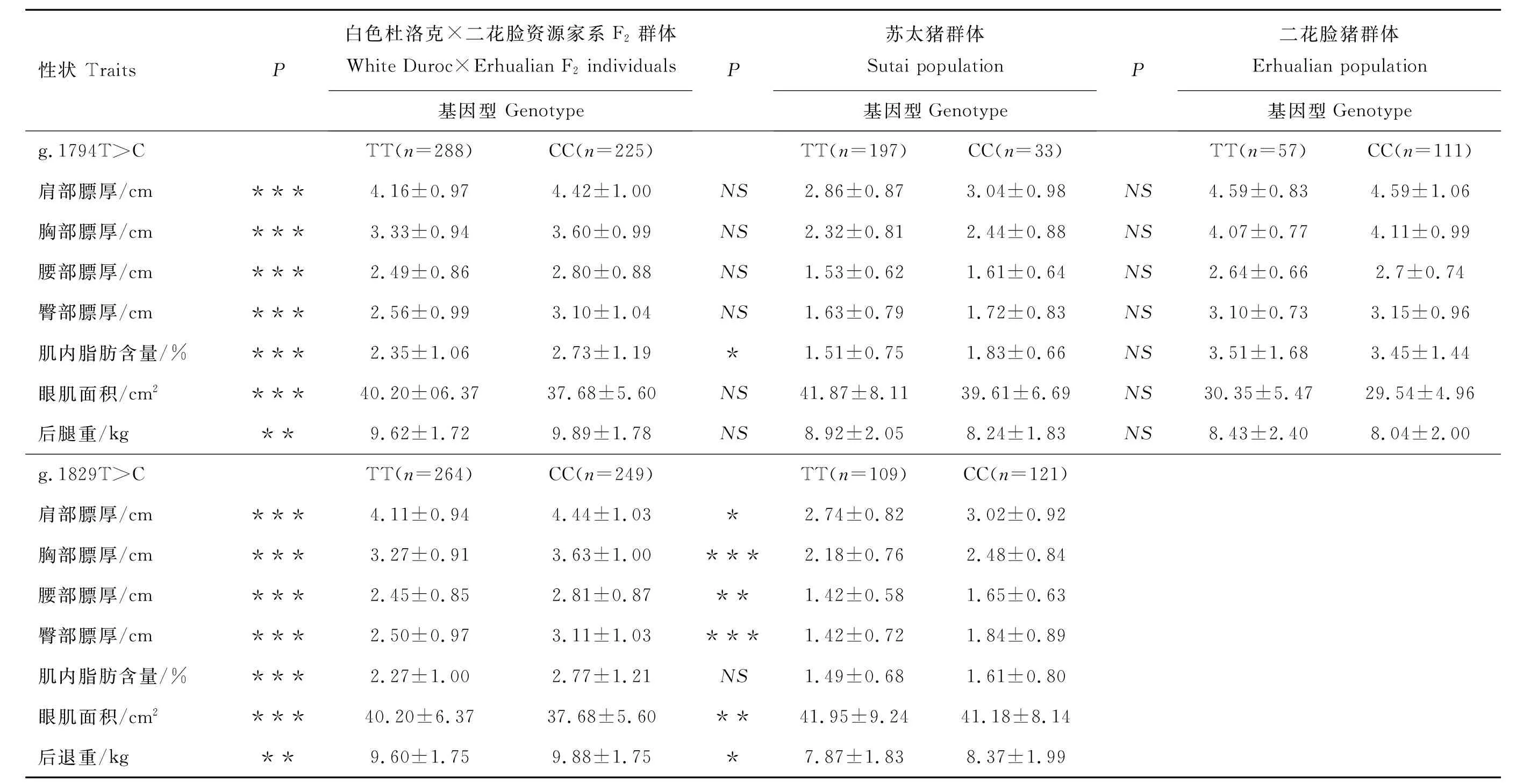
表5 猪IRS4基因两个SNPs位点与3个试验群体公猪的脂肪沉积性状的关联分析
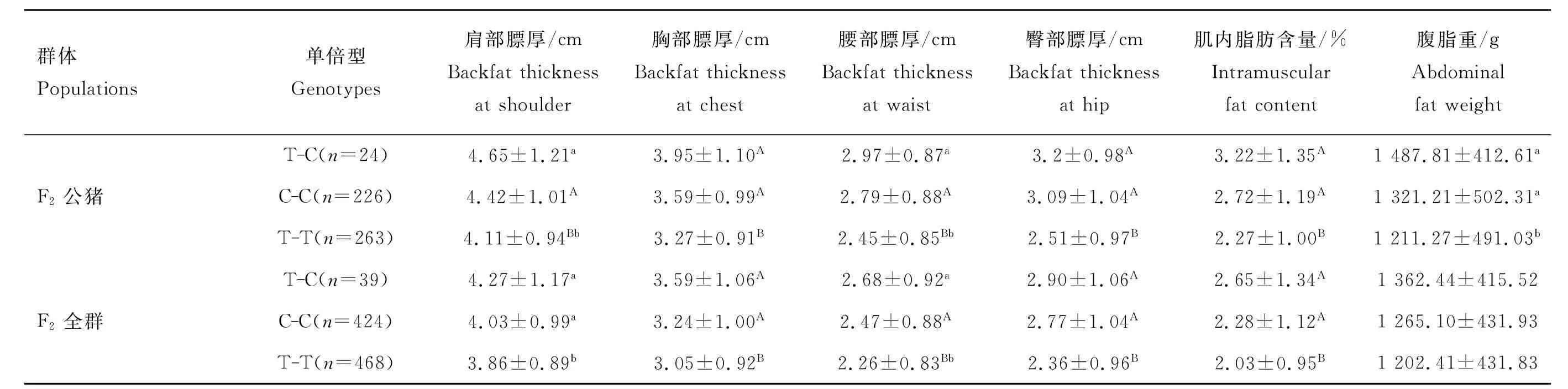
表6 在F2群体中IRS4单倍型与脂肪沉积性状的关联分析
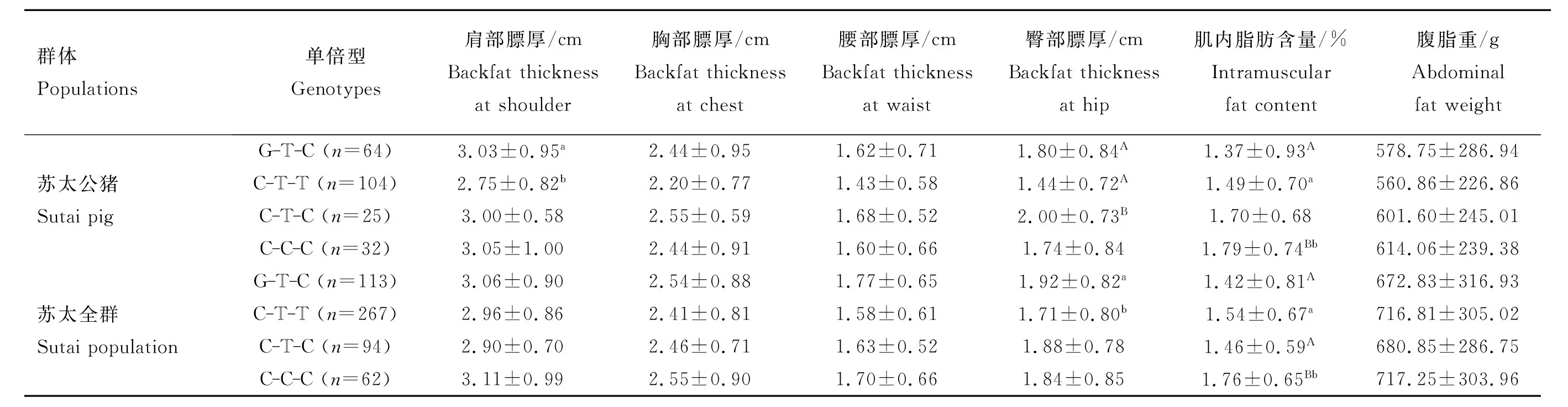
表7 在苏太群体中IRS4单倍型与脂肪沉积性状的关联分析
本试验采用DNA比较测序方法,对IRS4基因外显子区域约3 300 bp片段进行了多态位点搜查。结果除搜寻到了1个S.Cepica等[5-7]所报道同义突变位点g.1829T>C,还搜寻到了1个错义突变位点g.1794T>C,该位点的突变导致该蛋白质第432位氨基酸由苯丙氨酸转变为亮氨酸(Phe432Leu)。然而,这两个位点均位于该基因外显子非保守结构域内。
组织表达差异分析结果显示IRS4基因在垂体、下丘脑高丰度表达、在卵巢、板油、睾丸以及甲状腺弱表达,在其它组织中微弱或不表达,提示IRS4可能与垂体和下丘脑激素分泌有关,从而影响神经和体液的调节系统。由于错义突变可能通过影响蛋白质的翻译机制来影响基因的表达,本研究根据错义突变位点g.1794T>C的分型结果,选择了资源家系F2母猪CC、CT两种基因型个体的下丘脑组织分析其表达差异情况,结果发现个体间的基因表达差异均较大。可能的原因是基因的表达本身存在一定的时空性,或者,由于屠宰时个体的应激程度不同,体内激素水平的差异也可能会引起基因的表达差异。另外,除结构突变,基因调节元件突变、甲基化和miRNA等其它变异也可能导致基因表达差异,甚至影响表型。
本研究还在不同群体中分析IRS4基因3个SNPs的多态性。结果发现,启动子上的g.96C>G突变在中西方猪种中其主等位基因相同(表4),这不符合先前对SSCX QTL两个等位基因在F0代白杜和二花脸品种各自趋近纯合的判断,因此它不被视作潜在的QTN,可以被首先排除。错义突变位点g.1794T>C在所检测的资源家系F0白杜及杜长大商品猪种中均为TT基因型,在苏太猪和二花脸群体中则表现出偏态分布;其中,苏太猪TT基因型频率达到0.84,接近其祖代杜洛克的TT基因型频率,提示该位点可能与表型存在一定的关联性,并在品种培育过程中受到了一定强度选择。在同义突变位点g.1829T>C上,西方猪种均为TT基因型,而二花脸猪均为CC基因型,这与QTL等位基因在中西猪种中分离特征相近。基于此,作者进一步对IRS4基因这两个位点与脂肪沉积性状开展关联分析。关联分析结果显示,在资源家系F2群体中,这两个位点均与脂肪沉积表型极显著相关,表明在该群体中它们与QTL因果突变(QTN)强连锁。然而,在苏太和二花脸群体中,错义突变g.1794T>C对它们的脂肪沉积性状影响基本不显著。反而是,同义突变g.1829T>C与苏太公猪多数脂肪沉积性状呈显著相关,所以今后需要进一步研究分析它是否具有调节功能。同时,我们根据这两个位点在资源家系中的分型结果,构建了来源于F0代杜洛克公猪的T-T单倍型和来源于二花脸母猪的C-C、T-C两种单倍型。在资源家系F2群体中,通过单倍型关联分析,发现T-C单倍型与C-C单倍型效应相近,都能增加脂肪沉积,类似于QTL等位基因“Q”,而T-T单倍型则作用相反,可减少脂肪沉积,相当于QTL等位基因“q”。因此,这一结果提示,同义突变位点g.1829T>C与QTL等位基因分离更一致。此外,T-C单倍型表型值略高于C-C单倍型(P>0.05,表6),这说明最好是利用两个突变位点构成的单倍型作为脂肪沉积性状分子育种标记。
4 结 论
本研究运用DNA比较测序方法,对猪IRS4基因外显子区域约3 300 bp片段进行了单核苷酸多态性检测,结果在IRS4基因外显子区域鉴别到一个错义突变g.1794T>C和一个同义突变g.1829T>C。IRS4基因在垂体、下丘脑组织中高丰度表达,而且不同个体下丘脑组织中IRS4基因表达量差异也较大。PCR-RFLP检测结果表明g.96C>G在中西方猪种中其主等位基因相同,g.1794T>C和g.1829T>C位点在中西方猪种中存在一定的偏态分布,在中外杂交猪群中这两个位点与脂沉积表型存在显著的相关性,因此,可以将两者构成的单倍型应用于脂肪沉积性状的分子育种中。
[1] MILAN D,BIDANEL J P,IANNUCCELLI N,et al.Detection of quantitative trait loci for carcass composition traits in pigs[J].GenetSelEvol,2002,34(6):705-728.
[2] AI H,REN J,ZHANG Z,et al.Detection of quantitative trait loci for growth- and fatness-related traits in a large-scale White Duroc×Erhualian intercross pig population[J].AnimGenet,2012,43(4):383-391.
[3] MA J,IANNUCCELLI N,DUAN Y,et al.Recombinational landscape of porcine X chromosome and individual variation in female meiotic recombination associated with haplotypes of Chinese pigs[J].BMCGenomics,2010,11:159.
[4] PEREZ-ENCISO M,MERCADE A,BIDANEL J,et al.Large-scale,multibreed,multitrait analyses of quantitative trait loci experiments:the case of porcine × chromosome[J].JAnimSci,2005,83(10):2289-2296.[5] CEPICA S,BARTENSCHLAGER H,GELDERMANN H.Mapping of QTL on chromosome X for fat deposition,muscling and growth traits in a wild boar × Meishan F2family using a high-density gene map[J].AnimGenet,2007,38(6):634-638.
[6] CEPICA S,MASOPUST M,KNOLL A,et al.Linkage and RH mapping of 10 genes to a QTL region for fatness and muscling traits on pig chromosome X[J].AnimGenet,2006,37(6):603-604.
[7] CEPICA S,ROHRER G A.Linkage and radiation hybrid mapping of the porcine PIK3R1 gene to chromosome 16[J].AnimGenet,2003,34(4):313-315.
[8] MA J,GILBERT H,IANNUCCELLI N,et al.Fine mapping of fatness QTL on porcine chromosome X and analyses of three positional candidate genes[J].BMCGenet,2013,14:46.
[9] FANTIN V R,WANG Q,LIENHARD G E,et al.Mice lacking insulin receptor substrate 4 exhibit mild defects in growth,reproduction,and glucose homeostasis[J].AmJPhysiol-EndocM,2000,278(1):E127-133.
[10] VALASSI E,SCACCHI M,CAVAGNINI F.Neuroendocrine control of food intake[J].NutrMetabCardiovas,2008,18(2):158-168.
[11] WAUMAN J,DE SMET A S,CATTEEUW D,et al.Insulin receptor substrate 4 couples the leptin receptor to multiple signaling pathways[J].MolEndocrinol,2008,22(4):965-977.
[12] MASOPUST M,VYKOUKALOVA Z,KNOLL A,et al.Porcine insulin receptor substrate 4 (IRS4) gene:cloning,polymorphism and association study[J].MolBiolRep,2011,38(4):2611-2617.
[13] REN J,GUO Y,MA J,et al.Growth and meat quality QTL in pigs with special reference to a very large white Duroc×Erhualian resource population[C].In:Proceedings of the 8th World Congress on Genetics Applied to Livestock Production,Belo Horizonte,Minas Gerais,Brazil,2006:13-11.
[14] 苏玉虹,熊远著,张 勤,等.猪胴体脂肪沉积性状的QTL定位[J].遗传学报,2002,29(8):681-684. SU Y H,XIONG Y Z,ZHANG Q,et al.Mapping quantitative trait loci for fat deposition in carcass in pigs[J].ActaGeneticaSinina,2002,29(8):681-684.(in Chinese)
[15] BIDANEL J P,MILAN D,IANNUCCELLI N,et al.Detection of quantitative trait loci for growth and fatness in pigs[J].GenetSelEvol,2001,33:289-309
[16] CEPICA S,ROHRER G A,KNOLL A,et al.Linkage mapping of four genes (OTC,SERPINA7,SLC25A5 and FMR1) on porcine chromosome X[J].AnimGenet,2001,32:106-109.
[17] ROTHSCHILD M F,HU Z L,JIANG Z.Advances in QTL mapping in pigs[J].IntJBiolSci,2007,3(3):192-197.
[18] MERCADE A,ESTELLE J,PEREZ-ENCISO M,et al.Characterization of the porcine acyl-CoA synthetase long-chain 4 gene and its association with growth and meat quality traits[J].AnimGenet,2006,37(3):219-224.
[19] NONNEMAN D,ROHRER G A,WISE T H,et al.A variant of porcine thyroxine-binding globulin has reduced affinity for thyroxine and is associated with testis size[J].BiolReprod,2005,72(1):214-220.
[20] PONSUKSILI S,MURANI E,SCHELLANDER K,et al.Identification of functional candidate genes for body composition by expression analyses and evidencing impact by association analysis and mapping[J].BiochimicaetBiophysicaActa,2005,1730(1):31-40.[21] TRAKOOLJUL N,PONSUKSILI S,SCHELLANDER K,et al.Polymorphisms of the porcine androgen receptor gene affecting its amino acid sequence and expression level[J].BiochimicaetBiophysicaActa,2004,1678(2-3):94-101.
[22] MELKERSSON K,PERSSON B.Association between body mass index and insulin receptor substrate-4 (IRS-4) gene polymorphisms in patients with schizophrenia[J].NeuroEndocrinolLett,2011,32(5):634-640.
[23] SESTI G.Insulin receptor substrate polymorphisms and type 2 diabetes mellitus[J].Pharmacogenomics,2000,1(3):343-357.
[24] SESTI G,FEDERICI M,HRIBAL M L,et al.Defects of the insulin receptor substrate (IRS) system in human metabolic disorders[J].FASEBJ,2001,15(12):2099-2111.
(编辑 郭云雁)
PorcineIRS4 Gene:Polymorphism,Differential Expression and Its Associations with Fat Deposition Traits
MA Huan-ban,LIU Xian-xian,YANG Jie,HUANG Wei-bing,DUAN Yan-yu,GUO Yuan-mei*,MA Jun-wu*
(NationalKeyLaboratoryforSwineGenetics,BreedingandProductionTechnology,JiangxiAgriculturalUniversity,Nanchang330045,China)
This study aimed to detect polymorphisms of porcineIRS4 gene and its expression pattern in 17 tissues,and to identify gene markers associated with the QTL for fat deposition traits.Comparative sequence analysis of theIRS4 revealed 3 SNPs,including g.96C>G in the promoter region,a synonymous substitution g.1829T>C and a missense substitution g.1794T>C (p.Phe432Leu).Through RT-PCR assay,we found thatIRS4 gene was abundantly expressed in hypothalamus and pituitary,but weakly expressed in other tissues.Furthermore,we genotyped the 3 SNPs in White Duroc×Erhualian F2intercross,Sutai pigs,Erhualian pigs and DLY[Duroc×(Landrace×Yorkshire)]three-way crossbred pigs and Chinese local wild boar by PCR-RFLP,then calculated allele frequencies and estimated the associations between those SNPs and backfat thicknesses (BFTs) at shoulder,chest,waist and hip,abdominal fat weight (AFW),intramuscular fat content (IMF) and loin eye area (LEA) in these populations.The SNP g.96C>G was almost fixed in the F2intercross,so it was first excluded as a causative mutation (QTN) underlying this QTL.For both SNPs g.1794T>C and g.1829T>C,all western breeds (White Duroc and DLY) only had TT genotype,while,in the Erhualian population,the frequencies of CC genotypes at g.1794T>C and g.1829T>C were 65% and 100%,respectively.The results of association analysis showed that g.1794T>C was highly associated with all investigated traits in the F2intercross (P<0.01),but associated only with IMF in Sutai pigs(P<0.05)and had no significant effect on Erhualian’s phenotypes;g.1829T>C was significantly associated with several fatness traits (e.g.BFT and LEA) in the F2intercross and Sutai pigs.Moreover,bioinformatics analysis demonstrated that the potential influence of the missense variant g.1794T>C on IRS4 protein function was limited.The results suggest that the SNP g.1794T>C is unlikely to be QTN,and the SNP g.1829T>C associated with fat deposition traits across populations could be used as marker for selection of these traits.
pig;IRS4 gene;QTL;polymorphism;differential expression;fat deposition traits
10.11843/j.issn.0366-6964.2015.07.002
2014-10-16
国家“973”计划项目(2012CB124702);江西省重大科技项目(20114ACB01100)
马焕班(1990-),男,江西上饶人,硕士,主要从事猪肉质性状的遗传解析工作,E-mail:jiangximhb@sina.com
*通信作者:麻骏武,副教授,Tel/Fax:0791-83813080,E-mail:ma_junwu@hotmail.com;郭源梅,副研究员,Tel/Fax:0791-83813080,E-mail:gyuanmei@hotmail.com
S828.2
A
0366-6964(2015)07-1084-11

