猪博卡病毒(重庆株)NP1部分基因的克隆测序及生物信息学分析
叶星宇,聂 奎,聂福平
(1.西南大学动物科技学院,重庆 北碚400715;2.四川省广元市动物疫病预防控制中心,四川 广元628000;3.重庆市出入境检验检疫局,重庆 江北400020)
2009年,瑞典的Blomstroem等人从患PMWS病猪的淋巴结中分离得到一株新的细小病毒,将其暂命名为猪博卡样病毒(Porcine Boca-like Virus)[1]。随后,通过对其接近全长的基因组进行扩增和测序,证明了该病毒属于博卡病毒属,与犬博卡病毒的亲缘关系较近,因此正式命名为猪博卡病毒(Porcine Bocavirus,PBoV)[2]。目前在欧洲、北美、中国、中国香港已经有PBoV感染猪群的报道,并且PBoV在患病猪中的感染率高于健康猪中的感染率,因而推测PBoV可能与某些病原体起到协同致病的作用。但幸运的是,至今尚未见有PBoV感染人的报道。
研究证实,PBoV为单股线状DNA,其基因组大小为5.2 kb左右,包含3个开放阅读框,ORF1编码非结构蛋白(NS1),ORF2编码2个衣壳蛋白(VP1和VP2),中间开放阅读框(ORF3)编码一个高度磷酸化的非结构性蛋白质(Nuclear Protein 1,NP1)[3-4]。稳定的NP1基因为博卡病毒所特有,对于NP1遗传进化的研究有助于了解PBoV在国内猪群的流行的传染源以及其遗传变异规律,为PBoV的科学防控提供参考。本试验通过设计一对特异性引物,对重庆地区猪群中猪博卡病毒部分NP1基因进行克隆及遗传变异分析,报告于下。
1 材料与方法
1.1 血清来源 血液样本采自重庆市某规模养殖场,离心后分离血清,分装并置于-20℃冻存备检。
1.2主要试剂 DNA提取试剂盒、r Taq DNA聚合酶、DNA片段回收试剂盒、感受态细胞JM109、pMD19-T载体试剂盒,均购自宝生物工程(大连)有限公司;UNIQ-10柱式质粒小量抽提试剂盒,购自Omega公司;氨苄青霉素(Amp),购自Invitrogen公司。
1.3引物的设计 根据GenBank已公布的PBoV-NP1基因序列,利用DNAStar和Primer 5.0软件在其保守序列设计一对特异性引物。上游引物PBoV-F:5'-AAGGACATCTCCGAAAC-3',下游引物PBoV-R:5'-ATGAATGCCAGTGAAA-3',预计扩增片段大小为257 bp。引物由宝生物工程(大连)有限公司合成。
1.4 病原体DNA抽提和PCR扩增 按TaKaRa公司(大连)的病毒基因组提取试剂盒说明书,从猪血清提取病毒DNA。以提取的DNA样品为模板,进行PCR扩增,反应条件为:95℃5min;94℃30 s、56℃30 s、72℃60 s,共40个循环;72℃延伸10min。PCR产物用10 g/L琼脂糖凝胶电泳检测。
1.5 PCR产物的克隆与测序 用DNA回收试剂盒回收PCR产物,与pMD19一T载体进行连接;将连接产物转化到大肠埃希菌JM109感受态细胞,37℃培养12 h,挑取上述转化平皿中的白色菌落,接种到含有Amp(100μg/mL)的LB液体培养基中,37℃振荡培养过夜至混浊,提取质粒送宝生物工程(大连)有限公司测序。
1.6 生物信息学分析 从GenBank获取已公布的PBoV序列及HBoV、PPV序列(见表1),并对本试验的PboV-NP1(CQRC1-7)基因序列与公布的PBoV基因序列及HBoV、PPV基因序列进行同源性比较和系统进化分析。
2 试验结果
2.1 NP1部分基因的克隆 用PCR方法扩增出PBoV-NP1部分基因片段,大小约为257 bp,与预期片段大小一致(图1)。
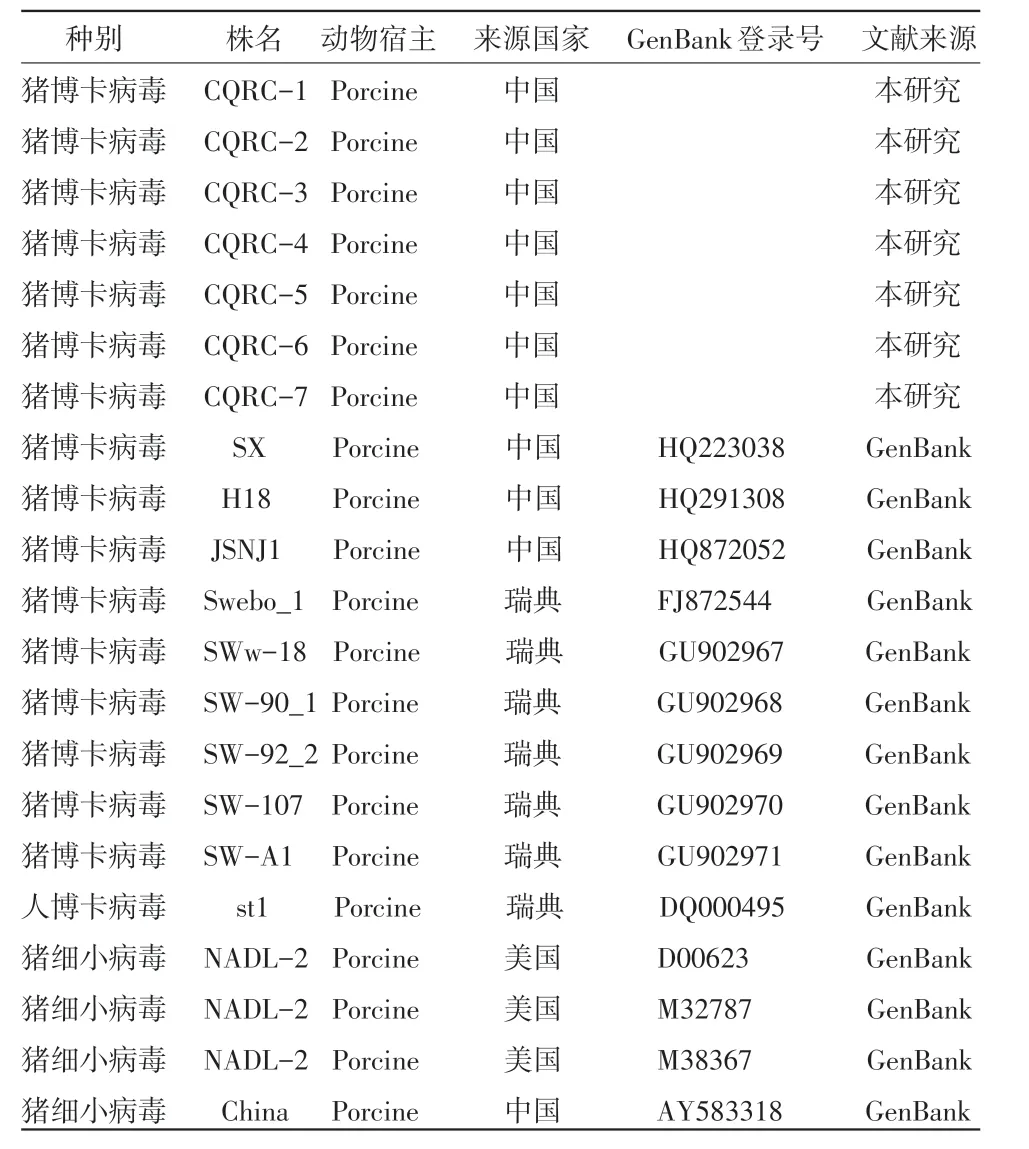
表1 本试验中用于PBoV系统进化分析的序列信息
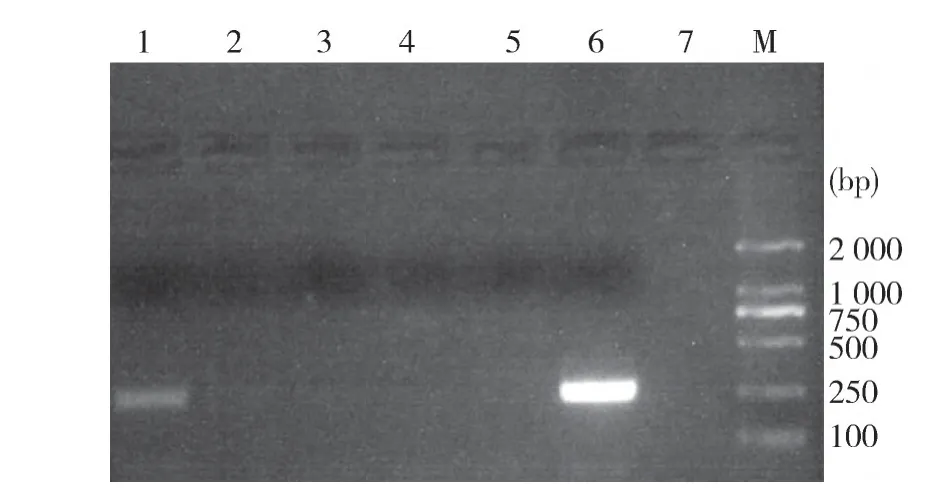
图1 重庆地区部分猪血清样本PBoV的PCR检测结果
2.2 PBoV-NP1基因核苷酸序列分析
2.2.1 PBoV-NP1基因序列同源性比较 测得7个猪博卡病毒重庆株NP1部分基因片段序列,拟命名PboV-NP1(CQRC1-7)。测序结果显示,PboVNP1(CQRC1-5)及PboV-NP1(CQRC7)与PBoV瑞典标准株GU902968序列完全一致,PBoV-NP1(CQRC6)在NP1基因的第236位碱基及第269位碱基与GU902968仅有2个碱基不同(图2)。
CQRC1-7与PBoV瑞典株GU902968同源性为99.2%-100%,CQRC-6与CQRC1-5及CQRC-7仅有2个碱基不同,同源性为99.2%。CQRC1-7与其他PBoV瑞典株及中国株的同源性均在97.3%以上,CQRC1-7与HBoV瑞典株同源性仅为32.3%~34.6%,与PPV的同源性较低,表明本试验扩增区域内的基因较为保守(图3)。
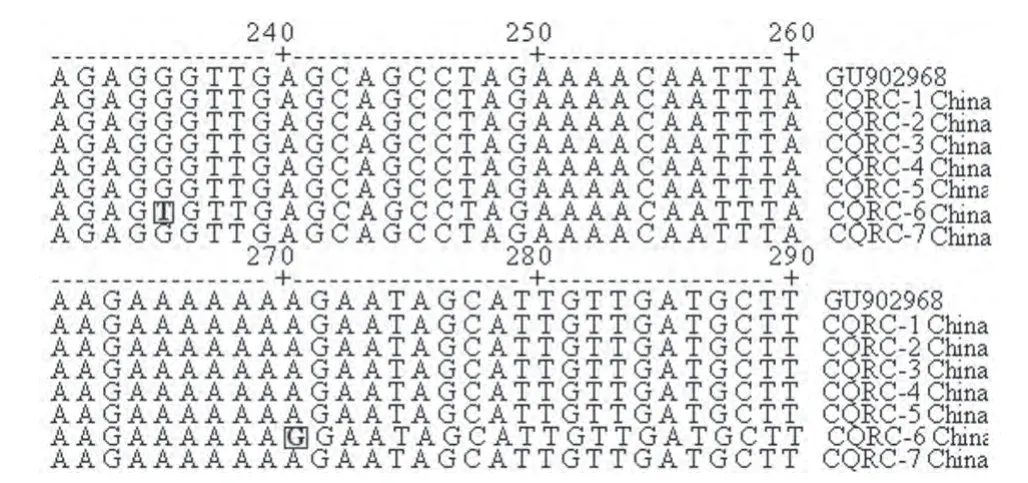
图2 PBoV-NP1(CQRC1-7)与PBoV瑞典标准株NP1部分基因序列比对

图3 猪博卡病毒NP1部分基因同源性比较
2.2.2 PBoV-NP1基因遗传变异分析 PBoV-NP1(CQRC1-7)基因遗传变异分析结果显示,PBoVNP1(CQRC1-7)与HBoV st1株形成一簇,3株PPV NADL-2株与PPV中国株亲缘关系很近,但其与博卡病毒属成员的亲缘关系较远,所以这4株PPV聚在一起形成另一簇;PBoV-NP1(CQRC1-7)与GenBank公布的9个PBoV瑞典株及中国株在一个分支上,HBoV st1株单独形成一个分支;其中,PBoV-NP1(CQRC1-5)及PBoV-NP1(CQRC7)与PBoV瑞典株(GU902968)亲缘关系非常近,聚在一个分支上,PBoV-NP1CQRC6与PBoV-NP1(CQRC1-5)及PBoV-NP1(CQRC7)属于不同的分支。PBoV-NP1(CQRC1-7)与PBoV瑞典株及中国株在同一分支上,表明PBoV的NP1基因遗传变异较小(图4)。
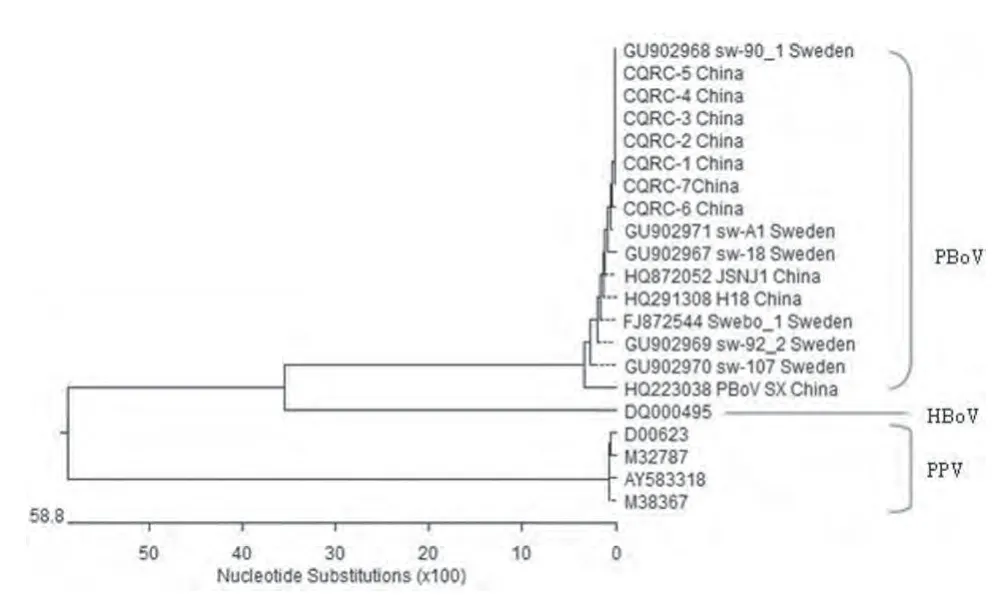
图4 猪博卡病毒NP1基因发育进化树
2.3 PBoV-NP1基因氨基酸序列分析 PBoV瑞典株sw90_1株NP1基因推导,NP1蛋白全长218个氨基酸,本次试验获得的基因片段编码NP1基因第30位点至111位点的82个氨基酸,根据16株PBoV的NP1基因的核苷酸序列推导了其所编码的氨基酸序列。结果显示,各毒株氨基酸序列之间比较,共有10个位点的氨基酸发生了变异,同源性在87.8%~100%之间。
3 讨论
2010年,Blomstroem等发现了PBoV的一段编码218个氨基酸的开放阅读框,并证实为NP1基因,且与人类博卡病毒NP1基因序列相近,基因组两端具有2个分别编码NS蛋白和NP蛋白的开放阅读框NP1基因具有较好的稳定性,是成为疫苗和药物作用的良好靶点[5]。通过对本次试验分离的7个病毒株NP1部分基因的测序与分析,发现7个PBoV-NP1部分基因片段其与NCBI公布的PBoV序列同源性达到97.3%~100%,其中与瑞典标准株(GU902968)同源性最高,为99.2%~100%,表明重庆地区流行的猪博卡病毒可能与PBoV标准株的亲缘关系很近,推测重庆地区猪群中感染的PBoV可能是通过国外品种引进或者进口国外动物性产品而传播感染猪群,因此,应当加强国外引种及进口动物性产品的检验检疫,加强境岸检疫,防止特定病原传入国内。该试验也表明PBoV的NP1基因核酸序列可能比较保守,遗传进化速度较慢,与同科的其他病毒做遗传变异分析,形成相对独立的一个分支,为猪博卡病毒NP1基因功能的进一步研究奠定了一定的基础。
[1]Blomstrom A,Belak S,Fossum C,et al.Detection of a novel porcine bocalike virus in the background of porcine circovirus type 2 induced postewaningmultisystemic wasting syndrome[J].Virus Research,2009,146:125-129.
[2]Blomstrom A,Belak S,Fossum C,etal.Studies of porcine circovirus type 2,porcine boca-like virus and torque teno virus indicate the presence ofmultiple viral infections in postweaningmultisystemic wasting syndrome pigs[J].Virus Research,2010,152:59-64.
[3]林峰,曾爱平,杨恩,等.WLL-1株博卡病毒(Boeavirus)全基因组序列分析[J].病毒学报,2007,23(1):1-3.
[4]罗迪贤,刘巧突,林应标,等.人类博卡病毒基因组序列与进化分析[J].实用预防医学,2006,13(6):1430-1432.
[5]曾松林.猪新型细小病毒PHoV和PBoV诊断方法的建立、分子流行病学调查及基因组序列分析[D].武汉:华中农业大学,2010:4-5.

