MSAP荧光检测初始数据的自动分析研究
陈秋博,邹智元,李金龙,张竞涛,徐青
北京交通大学 a.生命科学与生物工程研究院;b.软件学院;北京 100044
DNA 甲基化是真核细胞基因组重要的表观遗传学修饰方式之一,能在DNA 序列不发生变化的前提下调控基因的表达[1]。DNA 甲基化修饰主要发生在基因组CCGG 位点[2-3],与细胞分化、染色体失活及基因印记等生物学过程密切相关[4]。目前,检测DNA 甲基化的方法主要有酶切法、亚硫酸盐测序法、甲基化特异性PCR、基因组限制性酶切扫描法等。其中,以酶切和PCR 为基础的甲基化敏感扩增片段多态性(methylation sensitive amplified polymorphism,MSAP)不但敏感性强,而且只需要常规的仪器,操作比较简单,适用于没有任何信息的全基因组水平的甲基化分析。MSAP 方法由扩增片段长度多态性(AFLP)方法改进而来[5],随后被广泛应用于动植物基因组甲基化相关研究中。过去,一般采用聚丙烯酰胺凝胶电泳(PAGE)银染方法检测MSAP的选择性扩增产物,但因PAGE 法存在操作复杂、检测通量低和分析时间长等缺点,逐渐被以荧光标记为基础的测序胶和毛细管电泳技术所替代。
随着自动化程度和灵敏度的增强,毛细管电泳技术可以更灵敏、更有效、更高通量地获得MSAP 的检测数据。例如,本课题组使用以毛细管电泳技术为基础的ABI 3730xl 遗传分析仪,平均90 min 内可以获得96 泳道的荧光初始检测数据,与传统方法相比,检测时间缩短至约1/12,但产生的数据量庞大,约为PAGE-银染检测的8~10 倍。如果每条泳道平均产生200 条数据,那么96 泳道可产生近20 000 条数据,而往往一个普通的实验设计需要检测约10 个96 泳道。如此巨大的数据量如果依赖传统的人工手动分析,几乎不可能完成。因此,为了建立能够实现MSAP 荧光检测初始数据自动分析的方法,我们采用毛细管电泳荧光MSAP 检测技术,分析了北京油鸡基因组的甲基化程度,对产生的MSAP 初始数据采用标准的人工转换方法及3 种不同的计算机自动转换方法进行处理,比较和分析了人工转换方法和计算机自动转换方法获得的结果,确定了能够替代人工转换方法的自动转换方法,从而实现了MSAP 荧光检测初始数据的自动分析,并进行编程,创建了适用于MSAP 荧光检测初始数据自动分析的在线软件。
1 材料和方法
1.1 材料
选择10只17周龄北京油鸡作为实验材料,提取基因组DNA。接头与引物序列设计参照徐青等[6]方法,由上海生工生物工程技术服务有限公司合成。
1.2 方法
MSAP 方法包括酶切反应、连接反应、扩增反应和荧光检测共4 个主要步骤。本实验中,酶切反应、连接反应和扩增反应条件与徐青等[6]所用条件相同。
毛细管电泳荧光检测与数据分析方法如下:在96 孔板的各孔中分别加入3μL 选择性扩增产物、6.67μL甲酰胺、0.33μL ROX800分子量内标,95℃变性5 min,冰上放置10 min,离心1 min,在ABI 3730xl DNA 分析仪上进行毛细管电泳(10 kV 预电泳1 min,3 kV 进样15 s,10 kV 电泳70 min),收集初始数据,系统将各峰值的位置与其泳道中的ROX800 分子量内标做比较,通过GeneMapper 4.0软件对收集的初始数据进行分析。
2 结果
2.1 MSAP 扩增片段荧光信号的转换和初始数据的获得
应用GeneMapper 4.0 软件将MSAP 扩增片段荧光检测信号转换为MSAP 扩增片段的实体数据。首先对所有泳道的分子量内标的荧光图谱进行校正,依据每个泳道校正后的内标获得相应泳道的所有MSAP 扩增DNA 片段的大小,将结果导至Excel 表格中。Excel 表格的初始数据包括峰位置(Size)、峰高值(Height)、峰面积(Area)和数据点(Data Point)等4项有效数据。其中,Height是MSAP扩增片段的荧光信号强度,代表扩增片段的拷贝数,在本研究中,Height 的阈值设为50,荧光强度小于50 的片段不予考虑。Size表示检测样品MSAP扩增片段的长度,基本上为非整数值。在实验样品的MSAP 荧光图谱上,扩增片段集中在50~800 bp,所以随后的数据分析只统计长度为50~800 bp 的片段。在本研究中,定义Size≥50 的值为扩增产物在该位点出现,而Size=0 或不出现为扩增产物在该位点缺失。这样,通过定义Height和Size 值的区间,对Excel 表格的初始数据进行过滤,获得用于进行实验样品DNA 甲基化分析的初始数据,如图1A所示。
2.2 MSAP荧光标记检测初始数据的转换
2.2.1 MSAP 荧光标记检测初始数据的人工转换在MSAP 图谱上,每个样本基因组对应2 条泳道,其中H 泳道是用HpaⅡ和EcoRⅠ处理的组织DNA 样品,M 泳道是用MspⅠ和EcoRⅠ处理的组织DNA 样品。根据扩增产物在2 条泳道出现的情况,个体基因组甲基化带型可分为3 种:TypeⅠ为非甲基化模式(条带在H和M 泳道同时出现),TypeⅡ为全甲基化模式(条带在M 泳道出现而在H 泳道缺失),TypeⅢ为半甲基化模式(条带在H 泳道出现而在M 泳道缺失)。个体基因组的甲基化水平为TypeⅡ+TypeⅢ与TypeⅠ+TypeⅡ+TypeⅢ的比值。
在MSAP 图谱中,H和M 泳道的扩增片段对应于MSAP 荧光标记检测初始数据表中的H和M 数据列的Size 值。如图1A 所示,初始数据中每个扩增片段的Size 值几乎全为非整数值,而实际扩增片段应为整数值,所以初始数据的非整数Size 值要转换为对应的整数Size 值。初始数据Size 值的转换处理对于样品基因组的甲基化水平估算具有非常大的影响。在本研究中,我们定义人工转换为初始数据的标准处理。如图1C 所示,初始数据的标准转换是通过直接比对样本毛细管电泳的荧光吸收峰图谱和MSAP 荧光标记检测初始数据Excel 表格中的Size值,来确认每个扩增位点H和M 数据列的Size 值的有或无。MSAP 检测中,每个样品每对引物每条泳道平均获得约200 条目的峰,完成1 个样品10 对引物获得的MSAP 荧光标记检测初始数据的直接转换需要大约3 h。
2.2.2 MSAP 荧光标记检测初始数据的自动转换由于人工转换是对样本毛细管电泳的荧光吸收峰图谱进行一一比对来确定初始数据Excel 表格中的Size 值的有无,所以是最准确的转换方法。当样品数量较少时,可以采用这种方法。但是,在实际研究中,为了获得较为准确的结果,样本量需要达到一定规模。大的样本量获得的初始数据量非常庞大,人工转换需要大量时间,很难完成。以人工转换方法获得的甲基化水平数据为标准,我们设计了3 种可通过计算机编程实现初始数据自动转换的方法,用于替代人工转换方法。这3 种方法可通过计算机编程实现初始数据自动转换,确定实验样品基因组对应位点的甲基化带型,计算实验样品基因组的甲基化水平。
2.2.2.1 直接取整法 将所有Size 值四舍五入为整数值,判断每一位点H和M 泳道Size 值的有无,确定该位点的甲基化状态。直接取整法的数据转换流程为:程序读取初始数据→实现所有位点Size 值的取整→设置每条泳道中50~800长度为1的区间依次递增标识→将取整后Size 值填入对应标记的位置中→计算甲基化水平。如图2DI所示,M 列中172这一值应当填入区间171,但是由于取整丢失了数据精确度,所以存在一定缺陷。
2.2.2.2 整区间放置法 将直接取整法的泳道整数位置标识调整为区间位置标识,数据转换流程为:程序读取初始数据→设置每条泳道中50.00~800.99 长度为1 的区间依次递增标识→初始Size 值直接填入所属区间标识的位置中→程序智能校正填入的初始Size 值,处理重复的Size 值→计算甲基化水平。整区间放置法没有对初始Size 值直接进行四舍五入,所以保留了数据精度,从而可以使用程序对Size 值进行智能校正。如图2WR 所示,M 列中167.91 应当填入区间167.00~167.99,但167.91 与168.07 的差值小于0.5,经过智能校正,该值被填入区间168.00~168.99。168.9和169.89同样是这种情况。
2.2.2.3 半区间放置法 与整区间放置法的数据转换流程相同,但对泳道区间位置标识进行了调整。每条泳道中的位置从49.50 到800.50 用长度为1.00的区间依次标识。如图2HR 所示,M 列中171.88 应当填入区间171.50~172.50,但171.88 与171.48 的差值小于0.5,经过智能校正,该值被填入170.50~171.50区间。
在后文中,用4 种处理方法的英文单词首字母缩写代表这4 种方法。AA 为人工分析法,DI为直接取整法,WR为整区间放置法,HR为半区间放置法。
2.3 4种数据转换方法计算结果比较
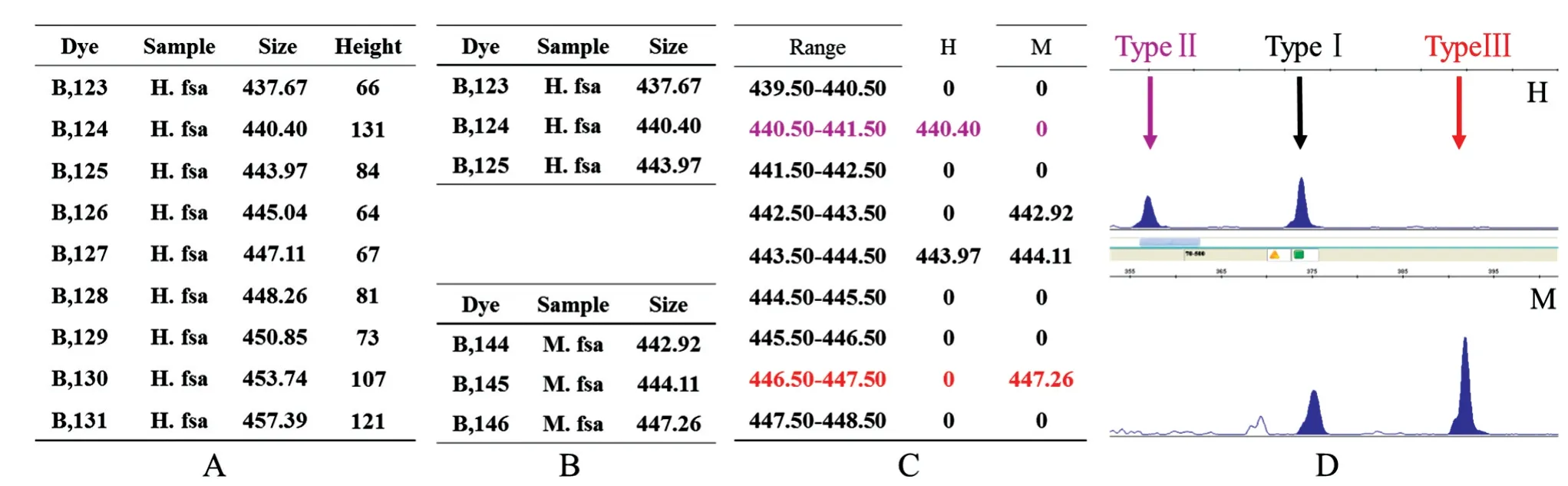
图1 初始数据人工转换流程图
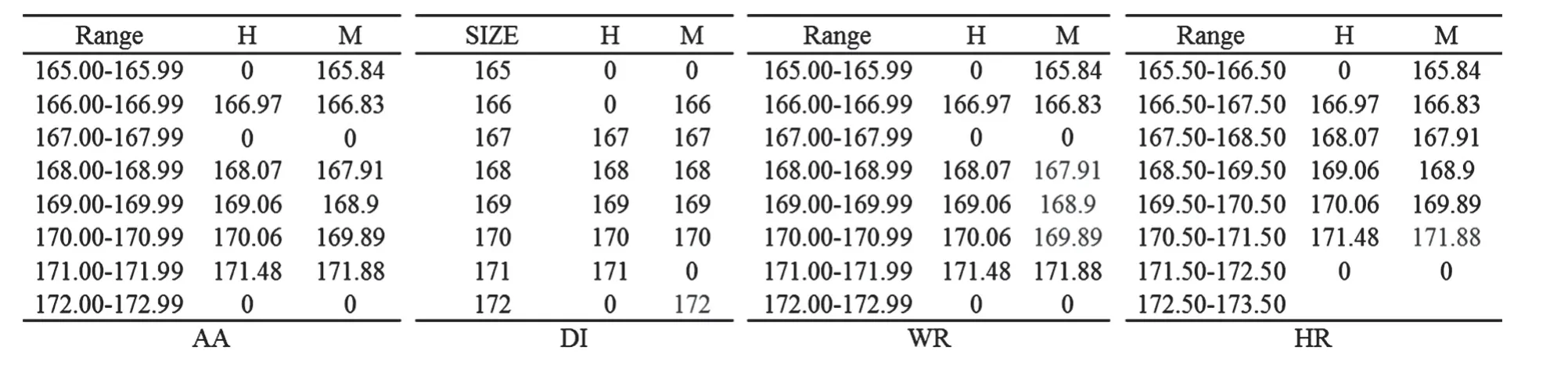
图2 4种方法泳道位置标识及峰位置值填入
北京油鸡基因组DNA甲基化程度MSAP荧光检测初始数据通过人工转换和3 种自动转换方法进行分析,4 种数据转换方法处理结果见表1。方差分析显示,对于非甲基化位点和半甲基化位点的分析,4种转换方法获得的结果存在显著差异(P<0.05);对于全甲基化位点,4 种转换方法获得的结果没有显著差异。Duncan 多重比较分析显示,对于非甲基化位点和全甲基化位点的数据转换,DI 与其他3 种方法之间差异显著(P<0.05),而AA、WR和HR 三种方法之间没有显著差异,HR 与AA 方法之间差异最小。综合以上结果,对于MSAP 荧光检测初始数据的转换,与AA 标准转换相比,HR 转换结果与AA 最接近,WR 次之,DI 差异最大达显著水平(P<0.05),所以在处理较大量的数据时,可以采用HR和WR 来替代AA方法,不建议使用DI方法。
2.4 用于MSAP荧光检测初始数据的自动分析的程序编写
MSAP 方法已被广泛应用于动植物基因组的甲基化研究。随着毛细管电泳技术在MSAP 上的应用,越来越庞大的数据量使研究人员的分析遇到了难题,初始数据的计算机自动化处理已成为高通量MSAP 分析的迫切需要。目前一些公司提供的荧光图谱收集仪器和配套软件已经初步使初始数据的获得自动化,但是在获得这些数据之后,研究人员应结合实际意义进行二次处理,目前还没有此类成型软件。为了解决这个问题,结合本研究分析结果,我们在半区间放置法的基础上编写了一个数据分析软件——MSAP Analyst。
该软件采用Microsoft.Net 4.0架构,使用C#语言开发。软件分为界面模块、数据处理模块和算法模块,处理的数据文件格式为“.csv”或“.xls”。软件的使用流程如图3 所示,主要分为三步:①打开MSAP初始数据表格,启动软件,点击左上角菜单“文件-打开”,在弹出的窗口中选择一个或多个初始数据表格并打开;②点击左上角菜单“分析-设置导出选项”,如图所示,在弹出的新窗口中可以勾选需要统计的泳道(与96 孔板上样孔一一对应),点击“确定”完成;③选择导出方式并导出,点击“分析-生成统计表”可对一个原数据文件进行统计,点击“分析-生成所有”则统计当前打开的所有原数据文件。在生成统计表时,还可以根据实验样本实际放置的位置,即对应关系,选择“横向”或“纵向”生成统计表。我们应用MSAP Analyst 模拟进行了多种类似数据的处理,都获得了理想的分析结果,因此以半区间放置法为基础的MSAP Analyst 软件可以应用于DNA 甲基化的MSAP 荧光检测初始数据的自动分析。另外,为了方便大家使用,我们设计了MSAP Analyst 软件的在线分析网站(http://www.fly2leo.cn),支持软件、测试数据和使用说明书的下载。

表1 4种数据转换方法处理的结果

图3 MSAP Analyst数据处理软件的操作界面
3 讨论
人工分析法是直接将2 种酶切反应的荧光图谱进行重合比较,根据荧光图谱的差异确定甲基化类型,计算甲基化程度。数据处理过程中,可以及时地进行人工调整,所以能够最大程度地减少误差,得到的分析结果最准确,因而把这个结果定义为标准结果,把人工分析法定义为标准方法。而对于3 种自动转换方法来说,由于碱基的最小值是1 bp,所以设置的区间跨度只能为1,因此3种分析方法都会出现2 个峰位置值在同一泳道位置(距离较短,近似重合)重复出现的情况,即重复位置值,重复位置值的出现会导致甲基化水平的计算误差。直接取整法中,对峰位置值直接进行简单的四舍五入,忽略了初始数据的小数部分,产生了没有进行任何纠正的重复位置值,导致了较大的计算误差。为了减小重复位置值的出现,引入区间放置的概念。整区间放置法通过程序智能校正峰位置值,减少了部分重复位置值,所以甲基化程度的计算误差比直接取整法小。根据人工分析法获得的经验,对整区间放置法进行了改进,得到半区间放置法。半区间放置法本身与样品峰值图吻合度最高,而且经过程序智能校正,重复位置值进一步减少,虽然不能完全消除重复位置值的产生,但可以保证甲基化程度的计算误差最小。为了最大程度地减少重复位置值的出现,我们设计的软件中可以将每个泳道的重复值进行标识,使用人员可根据原始的峰位置图进行手动纠正这些重复位置值,以进一步减小自动分析与标准分析之间的误差。
[1]Jones P A.Functions of DNA methylation:islands,start sites,gene bodies and beyond[J].Nat Rev Genet,2012,13(7):484-492.
[2]Smith Z D,Meissner A.DNA methylation:roles in mammalian development[J].Nat Rev Genet,2013,14(3):204-220.
[3]Moore L D,Le T,Fan G.DNA methylation and its basic function[J].Neuropsychopharmacology,2013,38(7):23-38.
[4]Gupta R,Nagarajan A,Wajapeyee N.Advances in genomewide DNA methylation analysis[J].Biotechniques,2010,49(4):iii-xi.
[5]Xu M,Li X,Korban S S.AFLP-based detection of DNA methylation[J].Plant Mol Biol Rep,2000,18(4):361-368.
[6]徐青,张沅,孙东晓,等.应用MSAP 方法检测鸡不同组织基因组的甲基化状态[J].遗传,2011,33(6):620-626.

