飞行时间二次离子质谱在生物材料和生命科学中的应用(下)
孙立民
(上海交通大学分析测试中心, 上海 200240)
1 化学成像
二次离子质谱的第2个主要操作模式是成像分析。高度聚焦的一次离子束,一般为液态金属源(如Cs、Ga、Au、Bi、In),按照一定次序“扫描”式的在样品表面所选择的区域里轰击。与此同时,质谱检测器检测并记录被轰击点上所产生的二次离子及其强度。单个二次离子在扫描范围内每个轰击点上的强度用颜色深浅表达出来,就得到该离子强度分布的二维图像,也就是二次离子质谱(SIMS)图像。SIMS图像可以直接或间接地描述元素或分子的二维化学分布(定性或半定量)。质谱检测器根据工作原理的不同,可分为四极杆、磁式和飞行时间等,它们的主要区别之一是二次离子被检测的顺序不同。 使用四极杆或磁式检测器, 二次离子需要单个被检测;而使用飞行时间检测器, 所有二次离子可同时被检测。因此,在成像分析中,飞行时间检测器的优势是从一次常规检测过程中就可获得所有可以检测到的离子的成像。 SIMS成像技术是质谱技术和表面成像技术的结合。理论上讲,质谱图中所检测到的每个谱峰的二维强度分布都能够通过成像技术而获得,但是,如果要求图像有一定的空间分辨率,则只有相当强的谱峰才可以产生有较高信噪比的图像。这主要是由于为了保证空间分辨率,需要减小一次离子束斑大小,从而造成离子束流密度大大降低。在这种条件下,谱图中强度较小的峰就不能在成像模式下被检测出来。基于减小一次离子束斑的原理,ION TOF 公司的TOF-SIMS 仪器设计了Burst Alignment模式来做高空间分辨率的成像检测。在这种模式下,最佳空间分辨率可以达到小于400 nm,然而却以损失谱图质量分辨率的1 u水平为代价[1],由此可见,单纯依靠减小一次离子束斑大小不是最好的解决空间分辨率的手段。近年来,为了解决小束斑离子源带来的信噪比差的问题,新型金属团簇离子源,如Aun+, Bin+不断地被开发,并应用于TOF-SIMS仪器设计上。实验证明,金属团簇离子源的使用大大提高了二次离子产额,尤其是对较高质量数的有机碎片离子和分子离子的效果更为显著。
随着TOF-SIMS仪器设计的不断改进,SIMS的成像功能被广泛应用于科学与技术研究中[2],在生物生命科学领域的研究与开发更是仪器厂家与科学家们关注的热点[3]。目前,TOF-SIMS成像技术在生物芯片[4],药物输运载体[5],细胞[6],器官组织[7],植入体内的生物材料[8],组合化学法多肽固相合成[9]等多种生命生物科学的研究中成功应用。本工作就TOF-SIMS成像技术在生物芯片、细胞、生物体组织3方面的应用研究做简略的介绍。
1.1 生物芯片
生物分子微阵列芯片的制作通常是经过多步且空间被分辨的表面杂化反应过程完成的。 因此,需要在制备的每个步骤中对反应产物进行空间上可分辨的化学结构鉴定和表征[10]。当空间阵列的尺度在微米~亚微米范围时,TOF-SIMS化学成像技术的空间分辨率可满足表征单个及多个阵列点上分子及元素的化学信息要求。TOF-SIMS 成像技术已被应用于制备微米甚至纳米尺度的DNA[4,10-12],蛋白质[13-15],及多糖微阵列[16-17]的工作中。
Attavar等[10]在制备DNA 铝基金属芯片时,使用PO2-和PO3-的叠加图像来表征被烷基磷酸所钝化的区域,同时使用SiCO2H3-离子图像表征硅烷化处理后硅氧烷分子分布的区域。这些图像显示烷基磷酸分布于Al表面区域,而硅氧烷分子仅仅分布于正方形窗口处玻璃衬底区域(200 μm×200 μm)。由此可以看出,TOF-SIMS图像可以较为便利地完成空间上可分辨的化学结构鉴定。Tuccitto等[11]在使用甲苯三联吡啶为标签制备图案密度可控的寡核苷酸阵列时,使用TOF-SIMS的正性和负性离子图像来监测ssDNA-ttpy探针的锚定反应,其陈列表面的SIMS图像示于图1。由乙二醇基自组装膜产生的多个特征峰图像叠加在一起显示出自组装膜仅在图案区以外分布; ssDNA-ttpy探针产生的特征峰的叠加图像显示,ssDNA-ttpy分子仅分布在长方形的图案区域内。采用图像叠加的目的是提高信噪比。Lee 等[12]研究了固定在聚丙烯酰胺型膜的单链DNA(ssDNA)微阵列的制备。利用XPS技术的成像、可定量性及小面积分析等功能,表征了阵列(样点直径在100~150 μm)的化学组成及目标DNA分子的杂化效率;同时,使用TOF-SIMS离子图像表征
了DNA和衬底信号的空间分布(空间分辨率为2 μm)。对比未杂化和杂化后阵列样品的几个DNA分子产生的二次离子图像(成像面积为200 μm×200 μm),包括 PO-,PO2-, PO3-, H2PO4-, 以及4个DNA 碱基的离子峰 (adenine (Ade-H),m/z134; thymine (Thy-H),m/z125; guanine (Gua-H ),m/z150 和 cytosine (Cyt-H),m/z110),可以看出 DNA均分布在非接触式打印样点区域内,并且单个样点内DNA成分的分布均匀。
在一个有化学反应活性的聚合物表面进行微观盖印(microstamping),被称为MAPS技术。使用这种技术可以将生物配体和蛋白质图案化到聚合物表面,所制备的阵列的空间分辨率可小于5 μm,且制备方法具有良好的重现性。 MAPS过程需要多步的化学反应处理。在Yang等[13]的工作中,TOF-SIMS的离子图像除了用于表征生物素(biotin)和链霉亲和素(streptavidin)被固定在盖印区域外,还可以有效地检验出每一步反应处理后是否有反应试剂残留于阵列表面。比如,从二次离子图像可以发现,分子质量小的聚二甲基硅氧烷(PDMS)及 Tween 20表面活性剂残留在阵列表面。能够检测阵列上微量残留试剂是TOF-SIMS成像技术的优势,这要归功于TOF-SIMS所具有的高检测灵敏度及优秀的化学结构分辨能力, 这也是其他技术(如荧光成像和XPS成像)所远远不及的。
当TOF-SIMS用于表征吸附到衬底上的蛋白质分布时,会遇到蛋白质分子离子和蛋白质特征碎片离子产额低的问题,因此,常使用氨基酸离子峰或CN离子峰来表征蛋白质的空间分布。但这些离子峰不具有特异性,即不能直接表明产生它们的蛋白质结构。 另外,CN离子峰的强度通常远大于其他的碎片峰,因它的图像有优秀的信噪比而常被用来表征蛋白质的空间分布。但是,由于任何含N物质都能产生CN离子峰,因此,CN图像的使用也会受到干扰。解决这一问题可采用15N同位素标记技术。Belu等[14]在研究链霉亲和素与图案化的生物素(点直径为40 μm,或线宽大约100 μm)的键合反应中,使用了15N同位素标记的链霉亲和素。C15N离子图像清楚地显示出经过“培养”反应后,靶链霉亲和素主要分布在生物素覆盖的阵列样点区域。
碳氢化合物微阵列可用于研究多聚糖与其他生物分子的相互作用。结合TOF-SIMS、XPS、SEM和AFM等多种测试手段,可探讨制备工艺对碳氢化合物微阵列质量的影响[16-17]。Scurr等[17]研究了喷涂工艺对微阵列上单个斑点内糖分子,链接剂和盐类分子的化学成分分布均匀性的影响。K+离子图像代表磷酸盐缓冲液(PBS)中盐类分子的分布;S-和HS-加和图像显示含硫链接剂的分布;C4H7+图像显示基底马来酰亚胺基团的分布;而C2H3O+及C5H7O3+代表糖类分子的分布。在基底效应可忽略的情况下,图像的颜色强度可以代表二次离子的相对强度,图像亮度的均匀性可以说明化学成分的均匀性。结果表明,糖分子浓度对喷涂点内化学成分分布的均匀性有较大的影响。
生物芯片阵列新工艺的研究也常借助TOF-SIMS作为有效的检测手段[18-20]。Sanni等[18]在开发微透镜阵列图案法(microlens array) 制备聚丙烯胺(PAAm)微阵列过程中,使用TOF-SIMS研究在样品点处(硅氧化物)的PAAm选择性吸附率以及吸附上的PAAm与4种常见的生物偶联分子,包括glutaric anhydride(GA), phenylenediisothiocyanate(PDITC), biotin NHS ester(BNE)和oligonucleotide (DNA)的化学反应。Sanni着重研究了在PAAm溶液中添加离子型和非离子型表面活性剂对PAAm在亲水区域和憎水区域(烷基单分子层) 吸附的影响。研究中, CN-离子图像代表PAAm的化学分布; S-和 CSN-离子图像代表PDITC被固定后的分布; S-和SH-离子图像代表BNE被固定后的分布; CNO-离子图像代表GA被固定后的分布;PO3-离子图像代表DNA被固定后的分布。Bearinger等[19]使用基于光催化作用的平版印刷术(PCL)方法,在聚(丙烯硫醚)嵌段共聚物薄膜(涂在金衬底上)表面制备了线宽为4 μm的“L”形图案阵列。对比代表共聚合物膜的信号C2H5O+和SO3-与代表金衬底的信号AuO-的TOF-SIMS图像可以看出,在L形状图案区域处所发生的光催化反应导致聚合物薄膜的移除和金表面的暴露。Dubey等[21]将活性表面耦合剂N-羟基丁二酰亚胺|(NHS)与“反应惰性”2-甲氧基乙胺(MEO)共同图案到高分子涂层表面制备出双组分微阵列。TOF-SIMS成像技术用于检测反应性及残留物。m/z98 (C4H4NO2-)和m/z114 (C4H4NO3-)为NHS的5-环结构的碎片特征峰,m/z58 (C2H2O2-)和m/z43 (C2H3O-)为NHS水解后形成的羧基基团的碎片特征峰,m/z107为光阻材料的特征碎片峰。新鲜制备、水解处理和再生样品的m/z98 (C4H4NO2-)和m/z114 (C4H4NO3-)离子图像示于图2。对比图像发现,水解后NHS特征信号消失,而再生样品重新出现NHS基团。另外,还对反应过程中化学试剂的残留情况进行了研究。从m/z107离子图像可以看出,虽然使用溶剂清洗光阻材料,但在阵列表面依然部分残留。
1.2 脂质细胞膜仿生体系
在应用TOF-SIMS成像技术研究生物分子的实例中,研究对象包括蛋白质、DNA、糖分子和脂质,其中对脂质的研究最多也最成功。从生物学的角度来讲,脂质双分子构成的细胞膜在生命体系中有至关重要的生物功能,然而对于脂质和其他构成细胞膜的生物分子在细胞上/内的相对空间分布还缺乏充分的描述。TOF-SIMS成像技术所具有的亚微米空间分辨率及分子识别能力对这类研究有较好的应用前景。常用的模仿细胞膜内外侧结构的单分子层和双分子层模型体系可以使用LB技术在平整衬底上制备,借助这些模型体系可研究脂质与蛋白质以及脂质与脂质分子间的相互作用。 Zheng 等[22]使用LB技术制备了支撑型三组分膜的模型体系,包含(神经)鞘磷脂 (sphingomyelin,SM)、卵磷脂(phosphatidylcholine,PC)和胆固醇(cholesterol,CH),并借助TOF-SIMS成像技术研究了胆固醇结构域的形成和卵磷脂和胆固醇之间的相互作用。胆固醇,鞘磷脂以及卵磷脂的分布分别由它们的特征碎片峰m/z369、264、224 的图像来描绘。由图像可观察到胆固醇结构域以及每个组分的相对分布,还能发现烷基链饱和度对膜结构有重要的影响。Baker 等[23]研究了蛋白质与细胞膜单分子层仿生结构的相互作用。该研究采用三组分混合体系,包含二棕榈酰磷脂酰胆碱 (dipalmitoyl-phosphatidylcholine,DPPC)或二棕榈酰-磷脂乙醇胺(dipalmitoyl-phosphatidyl ethanolamine, DPPE)、 胆固醇及糖蛋白(glycophorin)。DPPC构成细胞膜外侧单分子层仿生结构,而DPPE构成细胞膜内侧单分子层仿生结构。3种组分的相对空间分布分别由它们的特征碎片峰DPPC(m/z184),DPPE(m/z551), CH(m/z369)和glycophorin(m/z59)的图像来描绘。结果发现,在DPPC/CH/glycophorin 体系中3组分形成单一相,只有DPPE与胆固醇形成结构域,而糖蛋白构成连续相。
Gunnarsson等[24]研究了TOF-SIMS一次离子源对表征脂质结构的影响。发现采用Bi3+一次离子源(80 KeV)对于支撑型脂质双分子层中尖锐边缘的TOF-SIMS图像极限空间分辨率可达60 nm;图像可以分辨间距小于1 μm、直径为300 nm的单个囊泡,并可以做选区(region of
interest, ROI)的谱图分析。空间分辨率一般受表征离子的检测效率和一次离子源束斑大小的限制。Bi3+相比于C60+有更高的二次离子产额,能使用更小的离子束斑,因此,Bi3+所产生的高空间分辨率的二次离子图像更适合于脂质结构的表征。
1.3 单个细胞
TOF-SIMS成像技术在对细胞膜表面和细胞内的生物分子成分分布方面也有应用报道,其中研究较多的是脂质分子的分布。在对单个细胞进行表征时,为了维持其在超高真空腔体内细胞结构的稳定性,常常采用低温条件下的检测方法。这要求样品台在检测过程中维持低温,同时需要使用冷冻断裂法制备样品。Heien等[25]详细描述了细胞膜上或细胞内脂质分子的样品冷冻制备方法、数据收集及结果分析,并给出识别不同种类脂质分子的特征碎片离子峰表,列于表1。
在常用的冷冻制样方法基础上,Parry等[26]发明了trehalose基质辅助冷冻断裂的制样手段。脱水的trehalose是一种具有高粘度的玻璃态物质,可以阻止细胞组分的移动和扩散,从而使细胞有效地“冻结”在某一状态。 将细胞在含有trehalose的环境中孵化后进行冷冻,在超高真空测试条件下干燥并断裂,然后在室温下进行 TOF-SIMS成像检测。结果表明,横向分辨率小于1 μm时,可以观察到细胞亚结构。由于trehalose基质取代了冰,在常规冷冻制样过程中,不存在水重新在样品表面结冰的现象,温度不再需要严格控制,因此,样品的检测可以在室温下进行,从而大大简化了TOF-SIMS的实验操作,增大了TOF-SIMS成为一种常规生化分析工具的可能。
另外, 在细胞膜研究中,除采用冷冻样品制备方法外,Sjovall等[27]发明了将细胞膜较为完整地转移到Ag表面的“印记”(imprint)方法。将Ag箔放置在被细胞覆盖的玻璃衬底上,使用弹性高分子半球从Ag箔背面施加几秒钟的压力,然后,使用TOF-SIMS在常温下对“印”在Ag箔上的细胞膜成分分布进行成像观察。Sjovall观察了磷脂酰胆碱(卵磷脂)与胆固醇的相对空间分布, 发现高浓度胆固醇分布在质膜上,而磷脂酰胆碱倾向于分布在核膜处。

表1 常见脂质分子的碎片离子峰[25]
在细胞内生物成分及其空间分布的研究中,断开/切开细胞而使其内部成功地暴露于表面的样品制备是TOF-SIMS实验的关键。常用的断开/切开细胞实验手段包括冷冻断裂[28-29]和聚焦离子束(FIB)切割(milling)单个细胞。Szakal等[30]报道了TOF-SIMS表征完整哺乳动物细胞表面及在亚微米尺度上细胞内的元素、分子及自身细胞膜标记分子的分布。在这项研究中,使用Ga+聚焦离子束切开单个细胞,观察到了细胞内部的高蛋白质背景。
应用半定量TOF-SIMS成像技术对单个细胞的研究也有报道。 Ostrowski等[6]首先使用原位荧光显微镜定位2种J774细胞,然后使用TOF-SIMS表征单个细胞上胆固醇的相对含量。其中一种细胞经过胆固醇与β-环糊精的预制配合物(Chol-BCD)处理而含有更高的胆固醇含量。 在m/z366~370范围内的胆固醇碎片离子峰被用于表征胆固醇分布及定量分析。该研究以C5H9(m/z69)峰为内标,采用内标法定量分析。胆固醇碎片峰面积与该峰面积的比值作为归一化(normalization)结果。C5H9+是由脂质脂肪酸侧链所产生的一般性碎片,如果忽略基底效应,其强度对于处理和未处理的J774s细胞没有变化。结果表明,经过Chol-BCD处理后,J774 细胞膜的胆固醇含量增加(135±34)%。这种相对定量分析方法可以在单细胞水平上评估不同的药物处理和孵化条件对plasma 膜的化学作用程度,因此, 该方法有望在探索细胞正常生物功能、疾病引发原因、 抗药性和疾病治愈方法的研究中提供有价值的信息。
采用等离子体涂层技术使样品表面金属化的制样手段,会增强SIMS检测过程中生物分子的解吸附和离子化,这种方法也被称为金属辅助(MetA)SIMS。 Alterlaar等[31]使用溅射的方法将金粒子涂到成神经细胞瘤细胞培养物样品后,获得了高空间分辨率细胞的TOF-SIMS图像。图3为几个选择的离子图像,显示了几种被测细胞上成分分布的模式,其中已经明确的生物成分为胆固醇和甘油二酯。
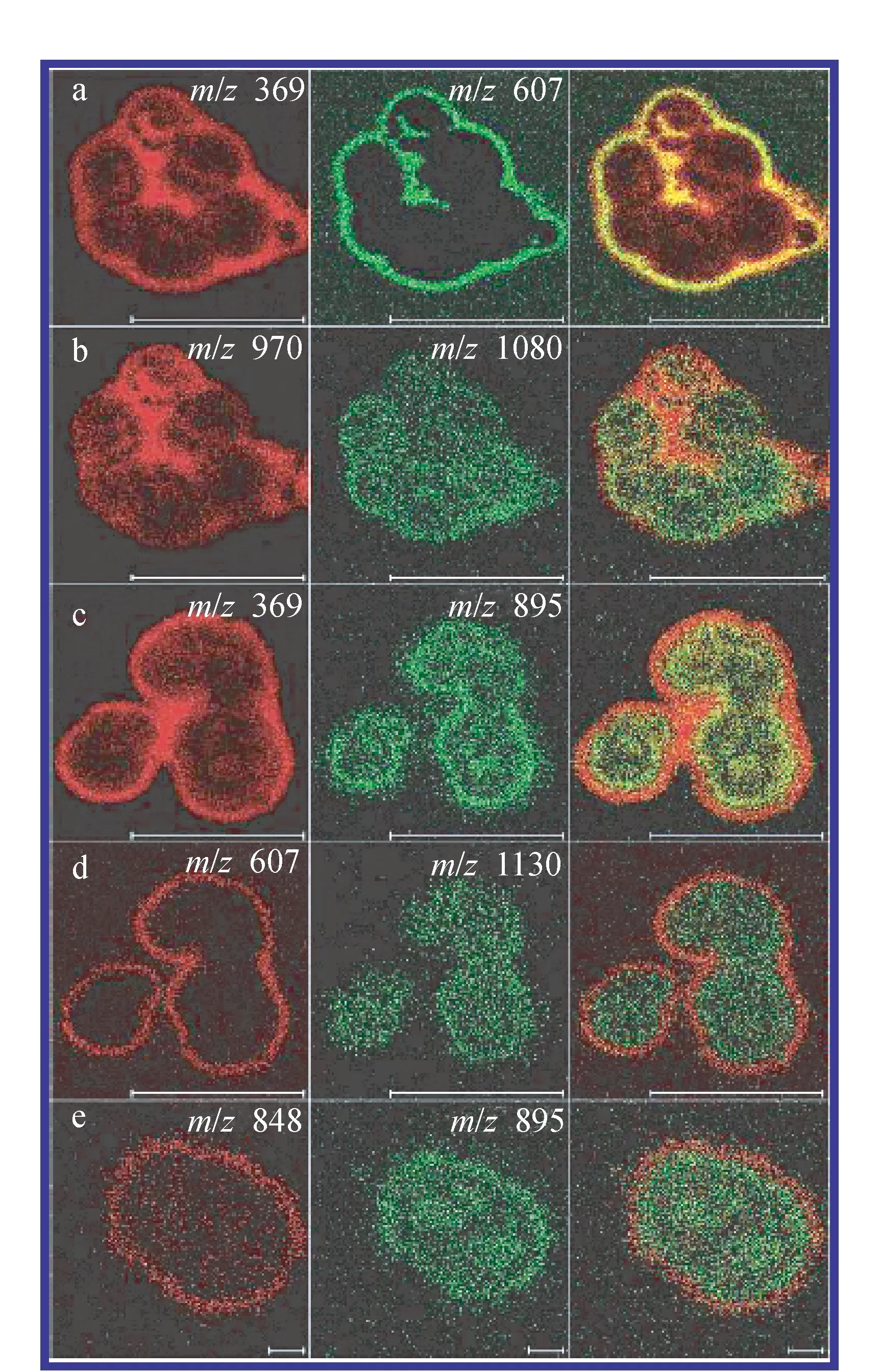
注:a~d图的比例尺为1∶100 μm,e图的比例尺为1∶10 μm图3 MetA-SIMS 图像显示成神经细胞瘤细胞的内部成分分布Fig.3 Cellular localization of MetA-SIMS selected ion count signals from neuroblastoma cells
1.4 组织切片
应用TOF-SIMS成像技术对组织样品检测时,在超高真空条件下,样品自身的化学性质及组织结构应尽可能保持不变。因此,组织样品也需要做“固定”处理,以防止离子(如 K, Ca, Na和蛋白质)的扩散。 另外,在做固定处理时,需要避免异物对样品的污染[32]。较为标准的组织样品制备方法为快速冷冻加低温脱水。
TOF-SIMS 成像技术在脂质、 胆固醇、卵磷脂及氨基酸分子的分布方面的研究较多。Toubul等[33]对老鼠大脑切片上脂质、胆固醇、维生素E等生物分子的分布做了TOF-SIMS 成像,示于图4。 Sjovall等[34]使用Au3+作为一次离子源对经过真空干燥的老鼠大脑冷冻切片进行TOF-SIMS成像表征,图像显示出胆固醇与卵磷脂有高度互补性的分布。为了提高组织切片的成像信号强度,还采用添加MALDI制样剂[35]以及添加金粒子等实验手段。Altelaar等[31]使用电喷雾方法将MALDI基质沉积到神经组织切片的表面上。这样,在TOF-SIMS检测中不仅分子离子峰的强度被大大提高,同时由于基质液滴很小且均匀分散在冷冻切片上,避免了待分析分子被湿润后有较大的横向扩散而造成空间分辨率的降低。
对比正常与患病人或动物的组织切片中生物组成的差异或它们空间分布的差异,是探究疾病由来的有效方法。应用TOF-SIMS成像技术研究患病组织的目的在于探索TOF-SIMS成为一种新型疾病诊断手段的可能。目前已有应用TOF-SIMS成像技术研究皮肤的光老化和自然老化作用[36],直肠癌组织[36-37],慢性肾疾病人的脂肪性组织[38]和脂肪肝患者的肝组织[39]等报道。Lee等[36]使用TOF-SIMS成像技术对经过不同时间的UV光照后又被活体解剖下来的人皮肤的冷冻切片进行了表征。从图像可以看出,

注:h~j为正离子图像;k~n 为负离子图像;n.三色叠加图像:C16:0 carboxylate ion (红色), vitamin E (绿色) 和 [ST d18:1/18:0-H]_ (蓝色);o.老鼠大脑切片的光学成像图4 老鼠大脑切片的TOF-SIMS离子图像Fig.4 TOF-SIMS ion images of a sagittal rat brain section
多肽和脂质分子的碎片离子峰随光照时间不同均有很大变化。例如,在真皮区域里,与未照射的皮肤相对照,经过24 h UV照射后的皮肤的胶原特异性的羟基脯氨酸 (OH-Pro,m/z86.06, C4H8NO+)信号强度明显降低,但经过48 h和72 h照射后,又恢复正常水平。在表皮区域里,经过24 h UV照射后,脂质分子的碎片离子峰(如C5H14NO+,m/z104.12, choline) 强度有很大提高,但经过48 h和72 h照射后维持不变。该研究表明,应用TOF-SIMS 成像技术能够在空间和时间上观察皮肤组分的改变。
2 深度剖析
深度剖析是SIMS的另一种操作模式,其检测需要溅射离子源和一次离子源2个离子束源。溅射离子源产生高束流密度离子束,用于在样品选定区域内做扫描式溅射轰击,使在一定厚度内的物质被逐渐剥离样品表面;一次离子源产生低束流密度的高聚焦离子束,用于常规的SIMS谱图或成像分析。循环交替式进行轰击-谱图采集或轰击-成像数据的采集操作过程,最终得到一系列深度下的谱峰或图像数据。根据这些数据,可以分析样品在不同表面深度的化学成分变化(depth profiling),或化学成分分布的变化,即三维成像。 深度剖析技术最常用于半导体材料表面掺杂元素的成分分析,但这种检测中刻蚀离子束的束流密度高,检出的信号为元素离子。而本工作所讨论的深度剖析检测是应用于有机物质(比如高分子材料和生命生物材料)的表征,因此,在采谱或成像检测之前,刻蚀离子束的轰击是否已经造成样品结构的破坏关系到测试的成功与否。为解决样品易被破坏的问题,近年来仪器制造商不断开发团簇离子束轰击技术,比如使用C60+和SF5+作为刻蚀离子束。一些实验已经证明了团簇离子束对有机体系具有低损伤效果[40],理论模拟计算也对在同样动能下,团簇离子束对有机表面损害程度低于单原子离子束的现象做了合理的解释。团簇离子束在撞击样品表面时,发生了自身分裂而引起传输能量降低,并导致一次离子穿透表面的深度变浅,最终对样品有较小的破坏作用。 目前,与其他液体金属团簇离子束相比,C60+离子束更适于应用在生物样品深度剖析或3D生物成像的研究中,如生物分子、组织、细胞和载药医用高分子涂层等。
2.1 蛋白质及多肽膜
为了探究蛋白质与基底的界面化学特性,Mouhib等[41]对吸附在不同亲水性的硅衬底表面的牛血清蛋白(BSA)做了深度剖析检测和PCA的数据分析。深度剖析图可以看到特征性的分子峰随着刻蚀深度的变化基本保持稳定,直至衬底信号出现后开始衰减。为了研究衬底对蛋白质构象的影响,使用PCA方法对衬底信号出现前的谱图数据进行分析,结果表明,在憎水性硅衬底上主要检测到憎水性氨基酸, 而在亲水性硅衬底上检测到带正电荷的亲水性氨基酸。
Cheng等[42]将多肽分子与trehalose混合,并使用旋涂手段制备成薄膜,并以此为模型体系研究在三维空间里表征生物体系的最佳仪器构造和测量参数。结果表明,C60团簇离子束与trehalose基质的联合使用,使得多肽分子的分子离子峰强度在深度剖析过程中基本保持不变。他们认为刻蚀速度大于化学损伤速度是导致分子离子峰强度保持稳定的原因。
2.2 细胞和组织切片的生物成分深度分布及三维成像
Breitenstein等[43]使用Bi3+作为取谱或成像的一次离子束,C60+为轰击离子束对NRK细胞(正常老鼠肾脏克隆52E)做三维微区检测。样品的制备采用戊二醛和甲醇/醋酸的化学固定方法。从被轰击到一定深度的XY断面图像可以清楚的看到卵磷脂分子和氨基酸分子的分布。但在三维图像中,细胞看起来在Z方向上有所变形,即使经过数学矫正处理,Z方向上的变形依然没有明显改善。另外,Breitenstein对溴乙啡锭二聚体(核酸荧光探针)标记的细胞做了三维检测,对比共聚焦激光扫描显微镜图像发现,荧光探针分子不能被TOF-SIMS三维所表征。
Malmberg等[44]分别使用SF5+和C60+为轰击离子束,Bi3+为一次离子束对单个培养细胞和完整的肠道上皮细胞组织做三维成像表征。 C60+轰击组织产生的图像显示,胆固醇分子在最表层,并随深度增加而降低。对比轰击与未轰击的样品,发现胆固醇分子的分布没有明显改变,但是胆碱磷酸分子却受到轰击的影响而峰强减弱。使用SF5+和C60+轰击单细胞后,可观察到胆碱磷酸分子的碎片峰,而C60+轰击效果受细胞环境中盐份的影响小于SF5+。
Jones等[45]使用40 keV C60+离子束做轰击,Au+离子束做质谱分析,对老鼠大脑切片完成了深度剖析实验。此项工作探究了在超出静态极限条件下,即高C60+束流密度下,离子束被正确用于研究生物样品的可能性。希望在不损伤样品的前提下,通过高束流密度轰击来提高二次离子产额,从而改善检出限并获得较小且有价值的横向空间分辨率。在实验中发现,样品中的盐分子对C60+轰击有较大影响,比如,脂质分子信号降低,而Na 和 K的加和离子(adducted ions)强度增加。因此,采用甲酸铵清洗方法对样品进行除盐份的处理,从而除去干扰。组织切片经过了约4 μm深度刻蚀的深度剖析结果示于图5。可以看到,在刻蚀深度内,氨基酸信号相对稳定(图5a),但是胆固醇信号却似乎可分成2个区域(图5b中m/z184 曲线)。另外,3D图像显示,胆固醇信号(m/z184离子)在灰物质区域开始较强,但随后降低至零,而在白色物质区域其强度却一直增高。

注:a.氨基酸信号,alanine亚胺离子m/z 44,proline m/z 70, m/z 56 为来自钢衬底的Fe+ ;b.胆固醇信号m/z 369和 m/z 184;c.轰击坑AFM 的图片,坑的深度为4 μm;d~f为3D图像,其中d为胆固醇m/z 369信号, e为胆固醇m/z 184信号, f为氨基酸的亚胺离子图5 组织切片的深度剖析结果Fig.5 Depth profiling analysis results of a tissue section
2.3 载药医用高分子涂层
Braun等[46]对涂在心脏支架表面的含有紫杉醇聚(苯乙烯-β-异丁烯-β-苯乙烯)三嵌段共聚物膜做了深度剖析研究。用15 keV Ga+和 20 keV C60+两种离子源,既作为一次离子源又作为轰击离子源。由于实际支架在测试腔体内的对准和对其检测区域的选择较为困难,所以使用涂在PET衬底上的高分子涂膜替代支架样品。这些涂膜经过多重检测手段的分析,证明与支架上的涂层一样。实验结果发现,使用Ga+不仅不能检测到紫杉醇的分子峰,而且由于荷电效应严重,也不能获得深度剖析曲线。而使用C60+离子源可获得较强的紫杉醇分子离子峰。从紫杉醇和高分子基底的深度分布曲线可以清楚看出,紫杉醇分子在表面有轻度的富集现象,而高分子的分布随深度的变化基本保持稳定。另外,C60+离子源的轰击可有效去除常污染表面的 PDMS分子层,却不影响下面材料的化学性质。轰击坑深度可达2 μm,并且从AFM和轮廓仪图像上可以看出,坑底上没有发生相转移和大的形貌改变,证明轰击过程中样品台不需转动。
3 TOF-SIMS在生物生命材料领域应用的展望
如果以对科学问题回答的结果及回答过程的简易程度来作为评估方法实用性的标准,那么TOF-SIMS 的表面质谱和化学成像功能有望成为常规的生物生命科学分析的表征手段。它们可以用于对分布在材料表面的分子进行结构鉴定和二维分布的表征,作为生物芯片制备过程的检查手段,用于对细胞及组织切片中脂质分子和其他生物分子的二维表征。但是,深度剖析技术对生物样品表征的实用性还有待更多的探讨。
在表面质谱对有机结构表征的应用方面,TOF-SIMS仪器有优异的质量分辨率(约8 000 u), 这使得TOF-SIMS谱图数据能提供精确的分子质量及同位素分布信息,进而可用于确认样品中未知成分的化学式。为了开发TOF-SIMS对样品中未知的有机和生物分子的结构鉴定潜能,需要建立有机分子和生物分子的谱图数据库。另外,虽然定量分析方法已被应用于医用高分子的生物降解机理等方面的研究,但其应用范围还需进一步的拓展。在化学成像的应用方面,提高空间分辨率和分子离子峰的产额仍然是仪器制造人员努力的目标。如果分子离子峰图像的信噪比可以大程度的增强,则可以直接使用分子离子峰对生物芯片的化学反应进行观察,而不再局限于使用无特异性的小碎片离子峰(如CN-、POx-、CnHn-及SOx-等)间接地表征带有氨基、磷酸基、硫及饱和碳氢的化合物。在生物质谱成像方面,与TOF-SIMS相似的是MALDI成像。相比于MALDI成像,TOF-SIMS的优点是有较高的空间分辨率 (亚微米),而MALDI的空间分辨率在30~100 μm范围内。然而MALDI成像的优点是它的离子产额远远高于TOF-SIMS,特别是有较高荷质比的离子峰也会有较为合适的离子产额,能够产生信噪比强的图像。为了克服离子产额低的弱点,除了不断提高仪器性能外,还可以通过特殊制样的方法来实现,比如,ME-SIMS, MetA-SIMS技术。另外,如何使生物样品制备更易操作,使低温测试所需的配套实施更易实现,也是推广化学成像应用于细胞和生物组织表征的关键问题。
参考文献:
[1] CHAURAND P. Imaging mass spectrometry of thin tissue sections: A decade of collective efforts[J]. Journal of Proteomics, 2012, 75(16): 4 883-4 892.
[2] BRISON J, ROBINSON M A, BENOIT D S W, et al.
TOF-SIMS 3D imaging of native and non-native species within hela cells[J]. Analytical Chemistry, 2013, 85 (22): 10 869-10 877.
[3] BOXER S, MOON D W, BREITENSTEIN D, et al.
General discussion [J]. Applied Surface Science, 2008, 255: 1 271-1 272.
[4] GRAF N, GROSS T, WIRTH T, et al. Application of XPS and TOF-SIMS for surface chemical analysis of DNA microarrays and their substrates [J]. Analytical and Bioanalytical Chemistry, 2009, 393(8): 1 907-1 912.
[5] BELU A M, DAVIES M C, NEWTON J M, et al.
TOF-SIMS characterization and imaging of controlled-release drug delivery systems[J]. Analytical Chemistry, 2000, 72(22): 5 625-5 638.
[6] OSTROWSKI S G, KURCZY M E, RODDY T P,et al. Secondary ion MS imaging to relatively quantify cholesterol in the membranes of individual cells from differentially treated populations[J]. Analytical Chemistry, 2007, 79(10): 3 554-3 560.
[7] BRUNELLE A, TOUBOUL D, LAPRÉVOTE O.
Biological tissue imaging with time-of-flight secondary ion mass spectrometry and cluster ion sources[J]. Journal of Mass Spectrometry, 2005, 40(8): 985-99.
[8] KLERK L A, DANKERS P Y W, POPA E R, et al.
TOF-secondary ion mass spectrometry imaging of polymeric scaffolds with surrounding tissue after in vivo implantation[J]. Analytical Chemistry, 2010, 82(11): 4 337-4 343.
[9] XU J Y, BRAUN R M, WINOGRAD N. Applicability of imaging time-of-flight secondary ion MS to the characterization of solid-phase synthesized combinatorial libraries[J]. Analytical Chemistry, 2003, 75(22): 6 155-6 162.
[10] ATTAVAR S, DIWEKAR M, LINFORD M R, et al. Passivation of aluminum with alkyl phosphonic acids for biochip applications[J]. Applied Surface Science, 2010, 256(23): 7 146-7 150.
[11] TUCCITTO N, GIAMBLANCO N, GHOSH S, et al. Controlled density patterning of tolylterpyridine-tagged oligonucleotides[J]. Langmuir, 2011, 27 (14): 8 595-8 599.
[12] LEE C Y, HARBERS G M, GRAINGER D W, et al. Fluorescence, XPS, and TOF-SIMS surface chemical state image analysis of DNA microarrays[J]. Journal of the American Chemical Society, 2007, 129 (30): 9 429-9 438.
[13] YANG Z P, BELU A M, LIEBMANN-VINSON A, et al. Molecular imaging of a micropatterned biological ligand on an activated polymer surface [J]. Langmuir, 2000, 16 (19): 7 482-7 492.
[14] BELU A M, YANG Z P, ASLAMI R, et al. Enhanced TOF-SIMS imaging of a micropatterned protein by stable isotope protein labeling[J]. Analytical Chemistry, 2001, 73(2): 143-150.
[15] HYUN J, ZHU Y,LIEBMANN-VINSON A, et al.
Microstamping on an activated polymer surface: Patterning biotin and streptavidin onto common polymeric biomaterials[J]. Langmuir, 2001, 17(20): 6 358-6 367.
[16] DIETRICH P M, HORLACHER T, GROSS T, et al. Surface analytical characterization of carbohydrate microarrays[J]. Surface and Interface Analysis, 2010,42(6/7): 1 188-1 192.
[17] SCURR D J, HORLACHER T, OBERLI T A, et al. Surface characterization of carbohydrate microarrays[J]. Langmuir, 2010, 26(22): 17 143-17 155.
[18] SAINI G, GATES R, ASPLUND M C, et al. Directing polyallylamine adsorption on microlens array patterned silicon for microarray fabrication [J]. Lab on a Chip, 2009, 9(12): 1 789-1 796.
[19] BEARINGER J P, STONE G, HIDDESSEN A L, et al. Phototocatalytic lithography of poly(propylene sulfide) block copolymers: Toward high-throughput nanolithography for biomolecular arraying applications[J]. Langmuir, 2009, 25(2): 1 238-1 244.
[20] WENDELN C, HEILE A, ARLINGHAUS H F,et al. Carbohydrate microarrays by microcontact printing[J]. Langmuir, 2010, 26(7): 4 933-4 940.
[21] DUBEY M, EMOTO K, CHENG F, et al. Surface analysis of photolithographic patterns using TOF-SIMS and PCA[J]. Surface and Interface Analysis, 2009, 41 (8): 645-652.
[22] ZHENG L, MCQUAW C M, BAKER M J, et al.
Investigating lipid-lipid and lipid-protein interactions in model membranes by TOF-SIMS[J]. Applied Surface Science, 2008, 255(4): 1 190-1 192.
[23] BAKER M J, ZHENG L, WINOGRAD N, et al.
Mass spectral imaging of glycophospholipids, cholesterol, and glycophorin a in model cell membranes[J]. Langmuir, 2008,24(20): 11 803-11 810.
[24] JOHANSSON B. TOF-SIMS imaging of lipids in cell membranes [J]. Surface and Interface Analysis, 2006, 38(11): 1 401-1 412.
[25] RUBAKHIN S S, SWEEDLER J V. Mass spectrometry imaging-principals and protocols, methods in molecular biology 656[M]. New York: Humana Press, 2010 : 85-97.
[26] PARRY S, WINOGRAD N. High-resolution TOF-SIMS imaging of eukaryotic cells preserved in a trehalose matrix[J]. Analytical Chemistry, 2005, 77(24): 7 950-7 957.
[27] SJOVALL P, LAUSMAA J, NYGREN H, et al.
Imaging of membrane lipids in single cells by imprint-imaging time-of-flight secondary ion mass spectrometry[J]. Analytical Chemistry, 2003, 75(14): 3 429-3 434.
[28] RUBAKHIN S S, SWEEDLER J V. Mass spectrometry imaging-principals and protocols, methods in molecular biology 656[M]. New York: Humana Press, 2010: 113-130.
[29] CHANDRA S. Challenges of biological sample preparation for sims imaging of elements and molecules at subcellular resolution[J]. Applied Surface Science, 2008, 255(4): 1 273-1 284.
[30] SZAKAL C, NARAYAN K, FU J, et al. Compositional mapping of the surface and interior of mammalian cells at submicrometer resolution[J]. Analytical Chemistry, 2011, 83(4): 1 207-1 213.
[31] ALTELAAR A F M, KLINKERT I, JALINK K,et al. Gold-enhanced biomolecular surface imaging of cells and tissue by SIMS and MALDI mass spectrometry[J]. Analytical Chemistry, 2006, 78(3): 734-742.
[32] VICKERMAN J C, BRIGGS D. TOF-SIMS: Sur-face analysis by mass spectrometry[M]. Chichester: IM Publications, SurfaceSpectra, 2001: 616-620.
[33] TOUBOUL D, BRUNELLE A, LAPRÉVOTE O.
Mass spectrometry imaging: Towards a lipid microscope? [J]. Biochimie, 2011, 93(1): 113-119.
[34] SJOVALL P, LAUSMAA J, JOHANSSON B. Mass spectrometric imaging of lipids in brain tissue [J]. Analytical Chemistry, 2004, 76(15): 4 271-4 278.
[35] ALTELAAR A F M, MINNEN J V, JIMENEZ C R, et al. Direct molecular imaging of lymnaea stagnalis nervous tissue at subcellular spatial resolution by mass spectrometry[J]. Analytical Chemistry, 2005,77(3): 735-741.
[36] LEE T G, PARK J W, SHON H K, et al. Biochemical imaging of tissues by sims for biomedical applications[J]. Applied Surface Science, 2008, 255(4): 1 241-1 248.
[37] PARK J W, SHON H Y, YOO B C, et al. Differentiation between human normal colon mucosa and colon cancer tissue using TOF-SIMS imaging technique and principal component analysis[J]. Applied Surface Science, 2008, 255(4): 1 119-1 122.
[38] SJOVALL P, JOHANSSON B, BELAZI D, et al.
TOF-SIMS analysis of adipose tissue from patients with chronic kidney disease[J]. Applied Surface Science, 2008, 255(4): 1 177-1 180.
[39] DEBOIS D, BRALET M P, Le NAOUR F, et al.
In situ lipidomic analysis of nonalcoholic fatty liver by cluster TOF-SIMS imaging[J]. Analytical Chemistry, 2009, 81(8): 2 823-2 831.
[40] MAHONEY C M. Cluster secondary ion mass sp-ectrometry of polymers and related materials[J]. Mass Spectrometry Reviews, 2010, 29(2): 247-293.
[41] MOUHIB T, DELCORTE A, POLEUNIS C, et al.
C60SIMS depth profiling of bovine serum albumin protein-coating films: A conformational study[J]. Surface and Interface Analysis, 2010, 42(6/7): 641-644.
[42] CHENG J, WINOGRAD N. Depth profiling of peptide films with TOF-SIMS and a C60probe[J]. Analytical Chemistry, 2005,77(11): 3 651-3 659.
[43] BREITENSTEIN D, ROMMEL C E, STOLWIJK J,et al. The chemical composition of animal cells reconstructed from 2D And 3D TOF-SIMS analysis[J]. Applied Surface Science, 2008, 255(4): 1 249-1 256.
[44] MALMBERG P, KRIEGESKOTTE C, ARLIN-GHAUS H F, et al. Depth profiling of cells and tissues by using C60+and SF5+as sputter ions[J]. Applied Surface Science, 2008, 255(4): 926-928.
[45] JONES E A, LOCKYER N P, VICKERMAN J C. Depth profiling brain tissue sections with a 40 Kev C60+primary ion beam[J]. Analytical Chemistry, 2008, 80(6): 2 125-2 132.
[46] BRAUN R M, CHENG J, PARSONAGE E E, et al. Surface and depth profiling investigation of a drug-loaded copolymer utilized to coat taxus express(2) stents[J]. Analytical Chemistry, 2006, 78(24): 8 347-8 353.

