基于基质辅助激光解吸-电离质谱的磷脂分析和成像的基质体系与制样方法
周丽华,陈安珣,赵肃清,于会娟
(广东工业大学轻工化工学院,广东 广州 510006)
磷脂分析除了应用薄层色谱、气相色谱、液相色谱等传统方法外,已出现不少新技术,如色谱-质谱联用、软电离质谱、 P31核磁共振和荧光共振能量转移技术等,其中,基于质谱的分析方法发展尤为突出。随着鸟枪法、纳米次级离子质谱法、组织切片直接分析法等质谱技术的出现,脂质组学在疾病脂生物标志物的识别、疾病诊断、药物靶点及先导化合物的发现、药物作用机制研究等方面已展现出广泛的应用前景。基质辅助激光解吸-电离成像质谱法(MALDI-IMS) 是最具代表性的磷脂组织切片直接分析技术,由Caprioli等[1]在基质辅助激光解吸-电离质谱(MALDI-MS)基础上建立起来的。该技术将之前的质谱只能分析提取或合成目标物,扩展到不经任何分离纯化手段就可直接分析细胞、生物组织切片,甚至是整个生物组织。MALDI-IMS利用离子扫描技术与专业图像处理软件,产生任意指定质荷比(m/z)化合物的二维离子密度图,可对组织中化合物的组成、相对丰度及分布情况进行高通量、全面、快速的分析,已成为质谱在临床蛋白质组学、糖组学、脂质组学及药物体内代谢研究中的重要发展方向。MALDI-IMS的基本工作流程示于图1,主要包括4部分:组织切片的制备(A);基质体系的确立与覆盖(B);质谱分析方法(C);数据统计及处理(D)。该技术能够精确、快速识别不同表达水平的特定磷脂-蛋白细胞,能够确定正常和患病器官中不同部位(包括肿瘤部位)的特定磷脂或蛋白质的位置及相对浓度,能够跟踪包括癌症在内的多种疾病的治疗药物及其代谢产物的空间分布,因此,在了解组织病理特征、疾病诊断、药物疗效及发现生物标志物等方面可能成为一种强有力的全新研究工具。此外,通过比较正常与疾病组织的分子分布,质谱成像技术在肿瘤早期诊断、预后评估等临床应用中将扮演重要角色。目前,已有利用MALDI-IMS开展细胞或器官中磷脂分析和成像工作的研究。
基质体系及其覆盖方式是MALDI-IMS的关键部分,是获得高质量成像的瓶颈。由于测定对象复杂性的提高,如组织切片中杂质含量高、目标物结构变化复杂且含量不均一、目标物分子质量变化范围大等因素,对基质体系的性能,如基质浓度、基质溶剂、基质覆盖方式、基质高真空稳定性等要求与MALDI-MS不一样。为了从复杂的生物体系中直接获取高质量的分子分布图,基质体系的建立更加严格,除了要满足作为MALDI基质的几个基本特征,如低的化学噪音和基质相关离子峰的影响、在特定波长有较好的吸收率、在特定溶剂中有较好的溶解度、合适的酸碱性、较高的分析物分子电离效率外,同时还应具备以下重要性质:1)基质必须有足够的真空稳定性,以保证较长的数据采集时间,且具有较好的耐激光辐射能力,尤其是耐高频激光;2)基质体系能产生均匀的结晶并能较好的覆盖于组织表面,晶体的尺寸必须小于质谱成像的分辨率,从而获得高空间分辨率;3)基质体系在相应的基质覆盖方式下不引起明显的分析物置换或位移[2]。

图1 MALDI-IMS基本工作流程图Fig.1 Workflow of MALDI-IMS
与其他生物分子相比较,磷脂自身有比较明显的特点:1)有较低的分子质量(<1 500 u) 和相近的质荷比,可能与MALDI-MS/IMS中的基质体系相关峰发生重叠,不利于磷脂的识别。2)水溶性低,要求所用溶剂对所测磷脂有一定的溶解性。3)多样性的分子结构,如根据极性头的不同可分为磷脂酰胆(PC)、磷脂酰乙醇胺(PE)、磷脂酰丝氨酸(PS)、磷脂酰肌醇(PI)、磷脂酰甘油(PG)、甘油磷脂酸(PA)等;根据Sn-1和Sn-2位上(不饱和)脂肪酸链与甘油骨架之间的连接键,酯键、烷酰基键和烯烃酰基键等的不同,可分为多个亚类。以上不同类型的磷脂往往同时存在,但极性头的不同决定了它们不可能被同一检测模式一次性检测,如PC和PS易正离子化,而PE、PI、PA和PG则易负离子化。4)不同结构磷脂的生物组织含量有明显差异性。磷脂的上述特点要求建立适合自身的MALDI基质体系。尽管目前已有不少化合物被证明可作为磷脂MALDI-MS分析的基质,其详细结构示于图2,但适用于MALDI-IMS的只有少数基质体系。针对磷脂特点,为了获得高质量的MALDI-IMS谱图,主要采用新基质体系结合不同的基质覆盖方式提高对生物组织质谱成像的质量。近10年来,为了建立具有高真空稳定性(稳定存在2 h以上)、耐高频率激光辐射、无基质峰干扰、分布均匀且不引起分析物位移、能全面解吸-电离不同结构与浓度的磷脂并准确表征其空间分布情况的高效磷脂基质体系,不少研究者进行了大量的工作。本工作从基质组成类型出发,简述MALDI-MS/IMS用于生物组织磷脂分析和成像的基质体系及制样方法的相关进展,并展望其发展前景。
1 单组分有机基质体系及相关制样方法
在MALDI-MS应用发展过程中,单一组分有机固体化合物是最常见的用于分析各类目标物的基质类型,磷脂的MALDI-MS/IMS分析也不例外。
1.1 酸性有机基质
肉桂酸衍生物(如芥子酸(SA)、α-氰基-4-羟基肉桂酸(HCCA))和苯甲酸类衍生物(如2,5-二羟基苯甲酸(DHB))是最早用于磷脂分析的基质类型。SA和HCCA 由于在较低m/z范围内(m/z<1 500)易产生强的基质干扰峰,并且在有机溶剂(如甲醇)中溶解度较低,在磷脂的MALDI-MS/IMS中较少使用,但HCCA的使用偶见报道。刘祥等[3]以含0.2%三氟乙酸(TFA)的HCCA 50%甲醇水溶液为基质,结合Image Prep基质覆盖仪,以正离子模式检测了小鼠肝组织切片上700~900 u之间的5类共13种磷脂类分子,并指出了这些磷脂分子在组织内分布的不均匀性。α-氰基-4-氯肉桂酸(ClCCA)是HCCA邻位羟基被氯取代后的产物,由于氯原子的引入,使ClCCA相对HCCA具有较低的质子亲和力[4-5]。因此,ClCCA比HCCA表现出更好的基质效果,可测定范围更广、种类更多的物质。如ClCCA可高效检测到常用磷脂基质不能离子化的氯化PE,为直接分析炎症机体脂质过氧化过程中产生的中间体(一氯或二氯PE)提供可能性,打破了之前氯胺只能采用ESI-MS分析的局面,ClCCA检测氯化PE的能力可能是因为它能引导气相PE磷脂分子内部的重排[6]。苯甲酸类衍生物DHB因其信噪比高、分析物碎片离子少、相关基质峰不影响分析物鉴定的特点,被视为目前最适用于磷脂分析的基质。此外,DHB具有较高的真空稳定性和良好的有机溶剂溶解性能,这些特点也是其成为磷脂MALDI-IMS最常用基质的关键因素。DHB可用乙醇-水(50∶50或90∶10,V/V,或添加0.1%三氟乙酸TFA)、乙腈-水(50∶50,V/V,或添加0.1%TFA)、甲醇-水(70∶30 或90∶10,V/V,或添加0.1%甲酸)、氯仿-甲醇(2∶1,V/V)等溶剂体系配制基质溶液,以达到最大化解吸-电离磷脂的效果。少量TFA的存在可有效抑制磷脂的分解[7],并在一定程度上增强脂质的溶解性,有效提高磷脂(特别是PC和PI)的信噪比[7-8]。另外,进行磷脂正离子模式MALDI-MS/IMS检测时,在DHB基质溶液中适当添加阳离子试剂,如碘化钠[9]、醋酸钠[10]、氯化铯[11]等,可通过减少每个PL产生的阳离子络合物类别而达到简化谱图的目的,此时,磷脂[M+Na]+峰或[M+Cs]+峰将取代[M+H]+、[M+Na]+和[M+K]+成为主要谱峰。
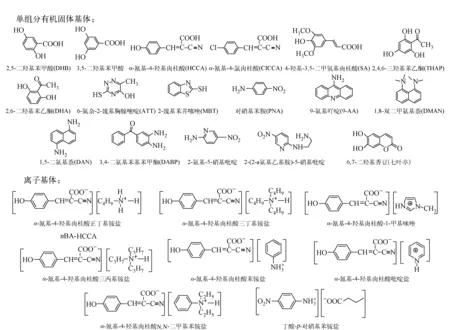
图2 磷脂MALDI-MS/IMS 有机基质的名称和化学组成Fig.2 Structures and names of organic matrices for profiling and imaging of phospholipids by MALDI-IMS
基质在组织分布中的均匀度是控制IMS实际空间分辨率的最主要因素。DHB最大的缺点是容易形成大针状结晶,导致较低的点与点重复性和空间分辨率。为了弥补这一缺点,已有研究报道了几种不同的基质覆盖方式,并取得了不错的效果,其装置示于图3。其中,基于溶剂的基质覆盖方法有:1)仪器自动微点阵法,一般借助化学喷墨式打印机(如CHIP-1000)进行,装置示于图3a。此方法可提高检测灵敏度,但空间分辨率会有一定程度上的降低[12],且相对费时(一般需要1 h以上)。2)喷雾法可在较短时间内(一般0.5~1 h)实现在切片上覆盖相对小且分布均匀的基质晶体[13],其方法主要有自动喷雾法(如电喷雾覆盖法)、碰撞雾化法、自动雾化喷雾器(如Image Prep基质覆盖仪法,示于图3b)、手动喷雾法(如薄层色谱溶剂喷雾法,示于图3c)[14-15]和人工空气喷枪法(示于图3d)。上述几种典型的基质覆盖方法的操作过程、注意事项及优缺点在文献[16]中有较详细的描述,本工作则重点综述各方法在磷脂分析中的应用。 Amstalden van Hove 等[14]采用Image Prep对含0.1%TFA 的DHB乙腈水溶液(50∶50,V/V)进行覆盖,对转移性乳腺癌病变模型进行了PC类磷脂的分析和成像研究[14]。相对于手动喷雾法,自动喷雾法对仪器的要求高,费用相对昂贵。薄层色谱溶剂喷雾法因其价格低廉、操作简便成为较常用的喷雾方法,而人工喷枪法则更加经济实用。使用人工喷枪法,利用100 g/L的DHB乙醇水溶液(90∶10,V/V)对大鼠脑磷脂分析和成像时,可获得更均匀的基质分布,且能获得比薄层色谱溶剂喷雾法更清晰明确的正离子PCs及负离子PIs的分布图[17]; 为了解实施肝脏切割术(PH)后生物机体磷脂的变化,Miyamura等[18]利用人工喷枪法,将50 g/L含0.1%TFA的DHB甲醇水溶液(70∶30,V/V)均匀喷雾到实施肝脏切割术24 h后的小鼠肝脏和脂肪组织切片上,同时喷雾到组织磷脂类物质分离用的TLC 板上,通过IMS技术对每片组织及TLC板上所分布的磷脂进行成像分析,发现小鼠耳垂上PC类磷脂的变化与肝脏位置的变化相似,从而推测受伤后的肝脏可能会发送不可查明的信息给生物脂肪组织来管理脂质代谢;Garrett等[19]使用人工喷枪法,将40 g/L含10 mmol/L 醋酸钠的DHB甲醇水溶液(70∶30,V/V)均匀喷雾到采用平铺技术处理后的眼睛组织上,分析其中磷脂的种类和分布,同时排查眼睛组织保存时受到的污染等情况,从而评价捐赠眼睛的质量。基质溶液中的醋酸钠作为离子化试剂,使磷脂分子钠离子化,有利于磷脂分子在离子阱分析器中更稳定的存在,便于在谱图中提供更多的目标物信息。人工喷枪法对操作人员技术要求不高,但要达到最佳基质覆盖效果需注意以下几点[15]:1)喷雾应在稳定室温,且有适宜湿度的环境中进行。2)选用带0.2 mm喷嘴的金属空气喷枪。3)喷嘴与切片的距离适当。若距离太近,会导致过多基质溶液喷雾到组织上,形成不均匀基质晶体覆盖,且导致分析物原位位移;若距离太远,则无足够基质溶液喷雾到组织上,达不到润湿组织,将分析物原位提取的目的。

a.化学打印机[16];b.自动雾化喷雾器[16];c.TLC 喷雾器[16];d.人工喷枪[16];e.毛细管振荡雾化法(OCN)[20];f.基质升华法[22]图3 用于磷脂MALDI-IMS 基质覆盖装置Fig.3 Matrix application instruments for MALDI-IMS of phospholipids
MALDI-IMS的质量分辨率、灵敏度、空间分辨率和再现性取决于基质溶液覆盖的效果。基质溶液覆盖方式对于脂类物质,特别是能溶于甲醇、乙醇和乙腈等的磷脂物质尤为重要。但基质溶液中的有机溶剂在一定程度上会导致脂类在组织切片上产生迁移,严重影响IMS的空间分辨率。因此,一种经济且溶剂用量少的毛细管振荡雾化法(OCN)被开发报道。此法利用毛细管尖端产生较小粒径的小水滴或雾滴雾化基质溶液,其主要特征是能够产生较干燥的微小基质颗粒,并最大可能地减少溶剂与组织的接触,从而减少分析物的迁移,避免分子空间信息的损失,其装置示于图3e。通过OCN法将30 g/L含0.1%TFA 的DHB乙腈水溶液(50∶50,V/V),覆盖于患有hexb 基因特性的Tay-Sachs和Sandhoff 疾病模型的大鼠脑部切片上,能够清晰地显示出多个PCs、PS、PI分子在鼠脑中的分布轮廓图[20]。
为了更有效地避免溶剂的使用,并消除溶剂给IMS法带来的分析物扩散问题,引入了两种新型的无溶剂基质覆盖法。其中之一是基质升华法,利用基质升华达到蒸汽均匀分布在切片上的目的[21],装置示于图3f。此法的显著特点是能产生基质微晶形态,增加基质的纯度和分布均匀性。Hankin等[21]利用此法获得了比普通基质覆盖法质量更高的鼠脑切片磷脂成像图。2010年,该课题组再用此法详细讨论了23~70岁男性眼睛水晶体膜中磷脂,特别是随着年龄增加SM类的变化情况[22]。基质升华法虽然需要特殊的仪器装置,但可有效地提高分析物的信号,形成均匀细小的基质覆盖层,减少由于基质晶体尺寸对成像分辨率的限制。此法对操作者的技术要求不高,且分析速度快,可在几分钟内完成,更重要的是不需要加入任何溶剂。另一种是无溶剂干燥涂层基质覆盖法[23],将DHB粉末通过20 μm不锈钢筛过滤,得到细小固体后直接涂于组织上。此法可获得直径为1~40 μm DHB晶体,由于分析物迁移量得到最小化,基质覆盖均匀且组织覆盖率超过80%,从而可获得更高质量的磷脂MALDI-IMS成像图。
DHB作为常用的磷脂基质,因其自身结构特性存在着不可避免的弱点:由于其酸性较高,负离子模式采集磷脂特别是PAs、PSs、PIs、PGs和PEs时,常因检测灵敏度较低只能获得信噪比低或无信号的谱图;此外,在负离子模式下采集PLs混合物时,去质子化的DHB会使目标分子以比自身分子质量大153 u的离子形式出现,增加谱图解析的难度。
1.2 中性、低酸性和碱性有机基质
为了克服酸性基质的弱点,几种中性、低酸性和碱性化合物被报道,用以进一步提高磷脂分子负离子检测的离子化效率和的灵敏度。图1所示的基质中,2,4,6-三羟基苯乙酮(THAP)较适合分析各类脂类物质,但因其易形成较大结晶而较少用于IMS分析中。2,6-二羟基苯乙酮(DHA)是最常用的中性基质,添加0.1%TFA的50%~70%乙醇或甲醇水溶液是其常用的溶剂,添加碘化铯[24]或氯化锂[25]作为离子化试剂有利于磷脂的MS/MS分析。如对鼠脑切片心磷脂(CLs)[24]和PCs[25]的分析,基于各个磷脂分子的Cs+或Li+离子峰可获得更多的结构信息和更准确的MS/MS数据。另外,Cs+的存在可在一定程度上提高组织上CLs的检测丰度[24]。DHA相对DHB,在正、负离子下都有更好的检测灵敏度、更高的分辨率和信噪比。DHA与中性基质6-氮杂-2-硫代胸腺嘧啶(ATT)最突出的优点是可检测磷脂的非共价复合物,如可检测到鼠脑切片中PC32∶0和PC34∶1与存在于基质溶液中的氯异吲哚铵形成的非共价复合物,但高酸性基质(如SA和HCCA)则无法检测到相关信息[26]。然而,DHA在高真空30 min内通常会升华,真空不稳定,一般不适用于较长时间的 IMS数据采集(通常不超过1 h)。DHA升华所导致的基质损失有时可通过增加基质浓度解决[27-28]。如30 g/L含0.1%TFA 的DHA甲醇水溶液(70∶30,V/V)结合人工喷枪基质喷雾法,对日本患者的300 μm×300 μm组织切片进行IMS分析,成功揭示了人类结肠癌肝转移病变中磷脂的非正常分布情况[27]。 中性基质ATT与基质添加剂,如柠檬酸盐(抑制磷脂金属加合物)和离液试剂,氯化胍(GUA)(增加磷脂水溶性和OxPL的稳定性)等组合,可高效地解吸-电离氧化磷脂(如OxPC、OxPE、OxPS等),对人血浆痕量OxPL的检测灵敏度可达fmol级[29]。 中性化合物2-巯基苯并噻唑(MBT)可分析各类脂类,相对DHB、DHA、PNA,在分析各类型PLs及脑和肝样品原位磷脂成像时,MBT具有更好的基质效果[30],其较低的蒸汽压能够承受数小时的高频率激光照射,可获得轮廓分明的组织切片磷脂分布成像图,其中性性质可在负离子模式下鉴定出更多的脑和肝切片磷脂。通过喷雾覆盖法,MBT甲醇饱和溶液可在切片上形成细而均匀的覆盖层,获得比DHB更好的空间分辨率和点与点的重复性。MBT易在m/z<500范围内产生基质相关峰,通过添加CsCl使分析物因加Cs+而电离,从而使各磷脂相关峰在高出m/z132时出现,避免基质相关峰的干扰。另外,CsCl的加入可使MBT检测到更多磷脂分子,如肝脏中的GPEtn类。但利用MBT获得的磷脂谱峰在m/z780~880进行归属时存在不确定因素。此外,在利用MBT进行IMS成像时发现,MBT易在负离子模式下产生大量高质荷比(激光稍强时m/z可达3 000)的基质簇峰,严重影响磷脂分析。
由于对硝基苯胺(PNA)对紫外光有较高的吸收,是一种可以代替DHB的碱性基质。PNA可获得比DHB更高的灵敏度,特别是在负离子模式下,PNA不仅可获得更高信噪比的PGs和PSs信号,还可鉴定PEs和PEps,即使这些物质同时存在于含有SMs和PCs的混合物中,也可获得理想的分析效果[31]。但PNA较高的蒸汽压制约了其在IMS中的应用,尽管在PNA中添加CsCl能够在一定程度上抑制其蒸发[32],但仍不能满足对较大组织切片进行较长时间(约2 h)IMS分析的要求。
9-氨基吖啶(9-AA)作为基质最先由 Vermillion-Salsbury 和 Hercules于2002年测定肽解液时引入[33],因其产生的基质干扰峰较少,主要用于测定分子质量小的物质,如代谢产物或磷脂。9-AA对磷脂的测定,在正、负离子模式下均比DHB的检测灵敏度高,磷脂以单一质子化峰电离,无加钠离子峰干扰。测定脂类混合物时,9-AA不电离胆固醇、三酰基甘油酯类物质,可有效地降低对脂类混合物中磷脂分析的复杂性[34]。9-AA(pK=9.99)相对其他常用基质具有更强的碱性,这有利于PE类磷脂以负离子模式测定,可避免正离子模式测定中PC对PE信号的抑制问题。9-AA基质溶剂的组成对获得的磷脂谱图有较大影响,通过优化溶剂组成可改善基质与分析物的作用力。甲醇、乙腈、异丙醇相比,异丙醇-乙腈混合溶剂(60∶40,V/V)可使9-AA对PC测定的信噪比提高30倍[34-35]。另外,在无溶剂的情况下,9-AA 干粉可与冻干组织膜,如嗜盐古生菌(halobacterium salinarum)一起研磨,通过压片法形成细薄片样后直接分析,此法免去了破膜、磷脂分离提纯等步骤,可快速获得膜上的磷脂信息,开创了生物膜直接测定的新方法[36]。但在负离子模式下,9-AA易将PCs类磷脂去甲基后离子化,常被误为PEs,导致归属错误[37],因此,在测定含有PC和PE的混合样品时应注意此点。尽管如此,9-AA卓越的负离子化PLs能力引起了广泛的关注。Cerruti等[38]采用喷雾法,利用10 g/L的9-AA乙醇水溶液(70∶30,V/V)对鼠脑切片磷脂进行了负离子模式的IMS分析,与HCCA、DHB、THAP相比,更多的磷脂分子被9-AA成像分析,包括PA、PE、PS、PG、PI、PIP、ST在内的28个脂质分子得到清晰的分布图,并通过MS/MS分析得到归属确认。另外,也有9-AA用于老鼠肾脏器官中SM3与SB1的硫脂复合物的IMS分析报道[39]。
Calvano等[40]采用另一强碱性物质1,8-双二甲基氨基萘(DMAN),又称 “质子海绵”(proton sponge),在无任何分离提纯步骤的情况下,直接分析生物膜中磷脂,对格兰氏阳性菌(乳酸菌)Lactobacillussanfranciscensis和胚芽乳汁杆菌L.plantarummicroorganisms的分析结果表明,此基质在负离子模式下可在低于皮摩尔的检测限下离子化磷脂,并且几乎无基质干扰峰。苗楠等[41]采用碱性化合物3,4-二氨基苯基苯甲酮(DABP)含3%HCl的甲醇水溶液(80∶20,V/V)为基质体系,分析不同种类的磷脂,发现此基质体系能有效抑制碱金属离子加成,简化谱图,从而利于磷脂的鉴定。此体系与DHB 和HCCA相比,能以更低的激光能量解吸-电离磷脂,获得更高的信噪比和更低的检测限。
2012年,Thomas等[42]详细讨论和对比了DHB、HCCA、DHA、THAP、3-羟基吡啶甲酸、DMAN、1,8,9-蒽三酚(DIT)、1,5-二氨基萘 (DAN)、PNA、9-AA、2-MBT等12种有机物质作为升华法的基质效果,发现另一个高效碱性基质DAN能在正、负离子采集模式下获得更清晰的鼠脑及鱼切片磷脂成像谱图,且具有更高的真空稳定性及小于20 μm的空间分辨率。
2 多元有机基质体系及相关制样方法
2.1 离子型基质体系
具有低熔点的盐类物质,或称“室温离子液体”RTILs(room temperature ionic liquids),具有极高的真空稳定性(可忽略的蒸汽压)和热稳定性,且溶液分布均匀,是极具应用前景的一类基质体系。基于HCCA发展的离子基质,如HCCA/正丁胺[43-44]、HCCA/1-甲基咪唑、HCCA/嘧啶、HCCA/三丙胺、HCCA/三丁胺和HCCA/苯胺,在离子化效率、信噪比、灵敏度、分辨率、重现性、检测物数量、耐污染程度等方面均表现出比单一基质更优越的性能[44],磷脂在离子基质的辅助下可更加稳定,不易碎裂[43]。 将40 g/L含0.1%TFA的HCCA/正丁胺的甲醇水溶液(75∶25,V/V)与DHB/正丁胺(DHBB)离子基质一同被用于小鼠肝脏与小脑切片磷脂分析和成像[45],利用人工喷枪喷雾覆盖,两种离子基质均比HCCA、DHB、DHBB、PNA、DHAP、9-AA、CHCAB 获得了更高的分析信噪比,检测到更多的磷脂分子。Calvano等[46]利用另一离子基质HCCA/三丁胺的甲醇溶液,通过修改Bligh-Dyer脂质提取程序,对含有榛子油(HO)的特级初榨橄榄油(EVOO)进行磷脂提取和富集,建立基于MALDI-MS的EVOO掺假分析。在此研究中,HCCA/三丁胺既作为磷脂提取溶剂,又作为MALDI 基质,仅选择性地从脂类混合物中高效解吸/电离磷脂。此法创立了基于PLs含量不同,鉴别具有高度相似性的HO和EVOO的方法。
由于ClCCA表现出比HCCA更优秀的基质效果,基于ClCCA发展起来的离子液体基质(ILM)或液体支持基质(liquid support matrix:LSM)[47]若被用于磷脂成像分析,则有望取得更佳的效果。
基于PNA化合物发展起来的离子基质PNA/丁酸能以[M+H]+的方式离子化溶血PC、PC和PAF,以[M+Na]+离子化PG、PA、PS,更大程度上简化谱图[48]。在正离子模式下,PNA/丁酸可同时检测含PCs、SMs、PAFs、PEs的磷脂混合物,能消除这些不同类磷脂之间的离子化抑制效应。
2.2 多元混合有机基质体系
多元混合有机基质由两种或两种以上化合物混合而成,有效的多元混合有机基质体系不仅能够充分发挥单一基质的优点,而且通过优化制样可克服单一基质的缺点。
2011年,课题组与Shanta等[49]共同研究报道了一个新混合基质,每毫升含7 mg HCCA和7 mg DHB的甲酸水溶液(70∶30,V/V),利用Image Prep进行基质覆盖,对鼠脑磷脂进行了IMS分析。此基质体系与单一基质相比,虽在基质峰干扰上未有明显改进,但其具有更高的真空稳定性,一次进样可获得正、负两种模式的成像数据。哌啶的存在可使基质溶液更有效地将磷脂从切片上原位萃取出来,可检测到更多磷脂分子,获得更高的分辨率。同年,Kim等[50]采用手动滴液干燥法,将此基质体系溶液滴加到患有肝内肝胆管型肝癌和其他可能患肝癌的腺癌病人的组织切片上,进行磷脂表达差异性分析。2012年,Shanta等[51]再次利用此基质体系,并结合Image Prep法,通过IMS对正常鼠脑及脑缺血切片进行磷脂表达差异性分析,获得了与脑缺血疾病相关的GPC、PC、PE、SM磷脂类变化的生物标记物的相关信息。
10 g/L含0.1%TFA的HCCA乙腈水溶液(50∶50,V/V)与同浓度的9-AA乙腈水溶液(50∶50,V/V)等体积混合,形成了另一个可用于磷脂分析的混合基质体系,其特点是在正离子模式下能以较低激光能量解吸-电离PC,无基质相关峰干扰[52]。
3 “无基质”体系及相关制样方法
为进一步避免低质荷比区来自基质相关峰的干扰,提高分析灵敏度、重复性、空间分辨率,避免分析物原位位移,不同形式的“无基质”的LDI-MS/IMS被引入。
3.1 基于各种材料的“无基质”体系
近年来,基于硅、碳、金属等元素发展起来的“无基质”材料体系,因具有独特的热、电、光物理和化学性能,相比有机化合物更有利于各类物质的LDI-MS分析。各种“无基质”材料体系的发展,大大促进了LDI-MS/IMS在基因组学、蛋白质组学、糖组学、代谢组学及其他小分子(特别是分子质量小于1 500 u)物质分析领域中的应用[53-57]。描述这些“无基质”的LDI-MS新名词有:表面(或材料)辅助(或加强)激光解吸-电离质谱(SALDI-MS、SELDI-MS和MELDI-MS),凝胶溶胶辅助激光解吸-电离质谱(SGALDI-MS),纳米颗粒辅助激光解吸-电离质谱(nano-PALDI-MS)等。
基于硅元素的材料有多孔硅(DIOS)、10~40 nm硅纳米线、硅酸盐介孔材料、SiO2芯片、纳米级SiO2颗粒及基于硅氧烷结构的聚合物凝胶溶胶体系等。He等[58]将DIOS与待测组织或细胞接触,将分析物转移到DIOS表面后分析,成功获得了HEK293细胞的生物磷脂标志物PC的特征碎片离子峰m/z184.1,并对HEK293细胞进行了鉴定。DIOS常用恒电流蚀刻法获得主要用于分析质量小于3 000 u的分析物,分析物通过纳米孔隙捕捉,同时吸收激光能量,完全避免了基质峰背景干扰,表面光滑且具较大孔径的硅材料则无此基质效果[59]。DIOS的主要缺点是使用寿命短(一般不到1年),再利用率低,对污染物敏感[60]。
基于碳元素的材料有石墨、碳纳米管、富勒烯、富力烯、类纳米金刚石型碳等。Peng等[61]将悬浮于异丙醇的胶态石墨悬浊液通过TLC喷雾器覆盖在分离磷脂的TLC板上,进行PCs和SMs分析。Cha等[62]采用人工喷枪将上述胶态石墨悬浊液用异丙醇稀释4倍后,覆盖于鼠脑切片上进行磷脂成像分析,通过固定喷枪和样品靶位置即可在切片上覆盖均匀的石墨异丙醇胶态溶液。此材料体系若过多覆盖或受到较大激光能量照射,会增加来源于胶态石墨中碳簇峰的背景噪音及m/z413、449未知峰的干扰。他们还发现,胶态石墨能高效解吸-电离脑苷脂,这是一类DHB等常用基质在正离子模式下很难检测到的脂类物质。富勒烯(C60)功能化后可提高其作为基质材料的灵敏度,并克服“热点”现象。Woods等[63]采用C60(C11H23)n(n=6)成功地分析了各类磷脂,特别是硫酸脑苷脂,通过离子淌度分析器获得的二维谱,能明确区分脂类和其他类物质。C60(C11H23)n不易产生背景干扰,且在氯仿中有极高的溶解性能,适合脂类等脂溶性物质的分析。2008年,类纳米金刚石碳材料覆盖的DVD整体装置首次被Naiam-ul-hag等[64]引入到脂类LDI-MS分析中。该装置由纳米金刚石类型碳材料涂层(DLC),金属钼溅射层和数字多功能光盘层(DVD)组成。DLC特殊的热、电、光及化学性能和涂层中存在的大量空格可有效辅助LDI分析低分子质量的肽类和代谢物质,如PC等。DLC层表面粗糙,含Csp3和Csp2,对305~330 nm波长有较强吸收,可避免使用长脉冲激光,从而减少分析物的裂解。DLC表面极高的疏水性可对分析物起到预浓缩的效果,提高分析灵敏度;中间Mo层可避免DVD受激光照射,减少背景干扰,同时可对DLC层进行电荷耗散。这一体系可获得的分析灵敏度高于DIOS,与硅酸盐介孔材料相当,具有高重复性、强耐盐性、易处理、可重复使用且不易污染等特点,还可避免在使用碳纳米管和富勒烯材料时,常出现的质谱数据产生较大质量漂移的现象。
基于金属及其氧化物纳米颗粒的材料有:Au、Ag、Mn、Zn、Fe2O3、TiO2、ZnO 等。如Jackson等[65]利用人工喷枪将金胶体(5.5 nm)的乙醇溶液覆盖在鼠脑切片上进行脑苷脂分析[65]。Taira等[66]利用功能化的硅酸盐材料侧边修饰2个赤铁矿(Fe2O3)单元电池(刚玉结构),获得功能化的纳米颗粒(fNPs),采用人工喷枪喷雾对鼠脑切片进行多肽和脂类物质分析,特别是PC类的IMS分析。fNPs直径为3.7~0.1 nm,表面修饰有羟基和氨基,可大大增加分析物的离子化效率。使用fNPs可获得低于15 μm的空间分辨率,并可避免分析物原位迁移。另外,在m/z0~10 000区域内不会产生任何来源于fNPs的背景信号。2009年,该研究小组再次使用fNPs在负离子模式下对大鼠海马组织中硫脂及PSs类磷脂进行了IMS分析[67]。
3.2 基于其他光源解吸-电离的“无基质”体系
红外激光直接解吸-电离磷脂是另一“无基质”体系的方法[68-69],分析前将醋酸钾溶液喷雾覆盖于切片上,可对神经组织膜中磷脂和糖脂类物质进行正交IR-LDI-IMS分析。该方法极少有脂类碎片产生,可获得高空间分辨率的成像谱图,不会导致分析物在切片上扩散或迁移。
4 展望
在MALDI-MS/IMS的应用及发展进程中,与基质相关的研究一直是分析工作的重点和难点。为进一步促进MALDI-MS/IMS对磷脂类物质的分析和成像技术进展,重点仍在于减少背景峰干扰、增加制样均匀度、提高分析灵敏度和重复性、提高成像的空间分辨率、减少分析物组织上的位移,这些都有待于新基质,尤其是高效新基质或特殊“基质”的发现及相应基质覆盖方法的引入。在以有机化合物为主要成分的基质体系发展中,单组分有机基质将侧重于对已有高效基质的结构改造,或基于此的多元及离子基质的制备,以获得更高基质性能的新体系,或改善其真空稳定性(如DHA、PNA、THAP),满足IMS较长时间采样的需要;多元组成及离子基质,特别是RTILs基质,在磷脂中有较高的利用空间和开发应用前景;“无基质”的材料体系在磷脂分析中的报道还相对较少。另外,在磷脂成像过程中的基质覆盖方法不多,可直接引用其他类型物质成像过程中使用的基质覆盖方法,如喷雾-滴液二层法[70]及干-湿二层法[71]等。
参考文献:
[1] CAPRIOLI R M, FARMER T B, GILE J. Molecular imaging of biological samples:Localization of peptides and proteins using MALDI-TOF MS[J]. Analytical Chemistry, 1997, 69(23): 4 751-4 760.
[2] FRANCK J, ARAFAH K, BARNES A, et al. Im-
proving tissue preparation for matrix-assisted laser desorption ionization mass spectrometry imaging. Part 1: Using microspotting[J]. Analytical Chemistry, 2009, 81(19): 8 193-8 202.
[3] 刘 辉,陈国强,王艳英,等. 应用超高分辨质谱成像技术研究脂类分子在小鼠肝组织中的分布[J].分析化学, 2011, 39(1): 87-90.
LIU Hui, CHEN Guoqiang, WANG Yanying, et al. Imaging and identification of phospholipids in mouse liver tissue by matrix-assisted laser desorption ionization-fourier transform ion cyclotron resonance mass spectrometry[J]. Chinese Journal of Analytical Chemistry, 2011, 39(1): 87-90(in Chinese).
[4] JASKOLLA T W, PAPASOTIRIOU D G, KAR-
AS M. Comparison between the matricesα-cyano-4-hydroxycinnamic acid and 4-chloro-α-cyanocinnamic acid for trypsin, chymotrypsin, and pepsin digestions by MALDI-TOF mass spectrometry[J]. Journal of Proteome Research, 2009, 8(7): 3 588-3 597.
[5] PORTA T, GRIVET C, KNOCHENMUSS R, et al.
Alternative CHCA-based matrices for the analysis of low molecular weight compounds by UV-MALDI-tandem mass spectrometry[J]. Journal of Mass Spectrometry, 2011, 46(2): 144-152.
[6] JASKOLLA T, FUCHS B, KARAS M, et al.
The new matrix 4-chloro-α-cyanocinnamic acid allows the detection of phosphatidylethanolamine chloramines by MALDI-TOF mass spectrometry[J]. Journal of the American Society for Mass Spectrometry, 2009, 20(5): 867-874.
[7] PETKOVIC′ M, SCHILLER J, MÜLLER M, et al.
Detection of individual phospholipids in lipid mixtures by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry: Phosphatidylcholine prevents the detection of further species[J]. Analytical Biochemistry, 2001, 289(2): 202-216.
[8] SCHILLER J, AMHOLD J, BENARD S, et al. Lipid analysis by matrix-assisted laser desorption and ionization mass spectrometry: A methodological approach[J]. Analytical Biochemistry, 1999, 267(1): 45-56.
[9] ISHIDA Y, NAKANISHI O, HIRAO S, et al. Di-
rect analysis of lipids in single zooplankter individuals by matrix-assisted laser desorption/ionization mass spectrometry[J]. Analytical Chemistry, 2003, 75(17): 4 514-4 518.
[10] GARRETT T J, PRIETO-CONAWAY M C, KO-
VTOUN V, et al. Imaging of small molecules in tissue sections with a new intermediate-pressure MALDI linear ion trap mass spectrometer[J]. International Journal of Mass Spectrometry, 2007, 260(2/3): 166-176.
[11] SCHILLER J, SUBB R, PETKOVIC' M, et al. CsCl as an auxiliary reagent for the analysis of phosphatidylcholine mixtures by matrix-assisted laser desorption and ionization time-of-flight mass spectrometry (MALDI-TOF MS)[J]. Chemistry and Physics of Lipids, 2001, 113(1/2): 123-131.
[12] AERNI H R, CORNETT D S, CAPRIOLI M R. Automated acoustic matrix deposition for MALDI sample preparation[J]. Analytical Chemistry, 2006, 78(3): 827-834.
[13] AGAR N Y, YANG H W, CARROLL R S, et al.
Matrix solution fixation: Histology-compatible tissue preparation for MALDI mass spectrometry imaging[J]. Analytical Chemistry, 2007, 79(19): 7 416-7 423.
[14] AMSTALDEN van HOVE E R, BLACKWELL T R, KLINKERT I, et al. Multimodal mass spectrometric imaging of small molecules reveals distinct spatio-molecular signatures in differentially metastatic breast tumor models[J]. Cancer Research, 2010, 70(22): 9 012-9 021.
[15] GOTO-INOUE N, HAYASAKA T, ZAIMA N, et al. Imaging mass spectrometry for lipidomics[J]. Biochimica et Biophysica Acta (BBA) - Molecular and Cell Biology of Lipids, 2011, 1 811(11): 961-969.
[16] SETOU M. Imaging mass spectrometry: Protocols for mass microscopy[M]. Tokyo: Springer, 2010: 72.
[17] WANG H J, POST S N J J, WOODS A S. A minimalist approach to MALDI imaging of glycerophospholipids and sphingolipids in rat brain sections[J]. International Journal of Mass Spectrometry, 2008, 278(2/3): 143-149.
[18] MIYAMURA N, NAKAMURA T, GOTO-INOUE N, et al. Imaging mass spectrometry reveals characteristic changes in triglyceride and phospholipid species in regenerating mouse liver[J]. Biochemical and Biophysical Research Communications, 2011, 408(1): 120-125.
[19] GARRETT T J, MENGER R F, DAWSON W W, et al. Lipid analysis of flat-mounted eye tissue by imaging mass spectrometry with identification of contaminants in preservation[J]. Analytical and Bioanalytical Chemistry, 2011, 401(1): 103-113.
[20] CHEN Y, ALLEGOOD J, LIU Y, et al. Imaging MALDI mass spectrometry using an oscillating capillary nebulizer matrix coating system and its application to analysis of lipids in brain from a mouse model of tay-sachs/sandhoff disease[J]. Analytical Chemistry, 2008, 80(8): 2 780-2 788.
[21] HANKIN J A, BARKLEY R M, MURPHY R C. Sublimation as a method of matrix application for mass spectrometric imaging[J]. Journal of the American Society for Mass Spectrometry, 2007, 18(9): 1 646-1 652.
[22] DEELEY J M, HANKIN J A, FRIEDRICH M G, et al. Sphingolipid distribution changes with age in the human lens[J]. Journal of Lipid Research, 2010, 51(9): 2 753-2 760.
[23] PUOLITAIVAL S M, BUMUM K E, CORNE-
TT D S, et al. Solvent-free matrix dry-coating for MALDI imaging of phospholipids[J]. Journal of Proteome Research, 2008, 19(4): 882-886.
[24] WANG H J, JACKSON S N, WOODS A S. Direct MALDI-MS analysis of cardiolipin from rat organs sections[J]. Journal of the American Society for Mass Spectrometry, 2007, 18(3): 567-577.
[25] JACKSON S N, WANG H J, WOODS A S. In situ structural characterization of phosphatidylcholines in brain tissue using MALDI-MS/MS[J]. Journal of the American Society for Mass Spectrometry, 2005, 16(12): 2 052-2 056.
[26] JACKSON S N, WANG H, WOODS A S, et al. Direct tissue analysis of phospholipids in rat brain using MALDI-TOFMS and MALDI-ion mobility-TOFMS[J]. Journal of the American Society for Mass Spectrometry, 2005, 16(2): 133-138.
[27] SHIMMAA S, SUGIURA Y, HAYASAKA T, et al. MALDI-based imaging mass spectrometry revealed abnormal distribution of phospholipids in colon cancer liver metastasis[J]. Chromatogr B,2007, 855(1): 98-103.
[28] HAYASAKA T, GOTO-INOUE N, SUGIURA Y, et al. Matrix-assisted laser desorption/ionization quadrupole ion trap time-of-flight (MALDI-QIT-TOF)-based imaging mass spectrometry reveals a layered distribution of phospholipid molecular species in the mouse retina[J]. Rapid Communications in Mass Spectrometry, 2008, 22(21): 3 415-3 426.
[29] STUBIGER G, BELGACEM O, REHULKA P, et al. Analysis of oxidized phospholipids by MALDI mass spectrometry using 6-aza-2-thiothymine together with matrix additives and disposable target surfaces[J]. Analytical Chemistry, 2010, 82(13): 5 502-5 510.
[30] ASTIGARRAGA E, BARREDA-GMEZ G,
LOMBARDERO L, et al. Profiling and imaging of lipids on brain and liver tissue by matrix-assisted laser desorption/ionization mass spectrometry using 2-mercaptobenzothiazole as a matrix[J]. Analytical Chemistry, 2008, 80(23): 9 105-9 114.
[31] ESTRADA R, YAPPERT M C. Alternative approaches for the detection of various phospholipid classes by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry[J]. Journal of Mass Spectrometry, 2004, 39(4): 412-422.
[32] RUJOI M, ESTRADA R, YAPPERT M C. In situ MALDI-TOF MS regional analysis of neutral phospholipids in lens tissue[J]. Analytical Chemistry, 2004, 76(6): 1 657-1 663.
[33] VERMILLION-SALSBURY R L, HERCULES D M. 9-Aminoacridine as a matrix for negative mode matrix-assisted laser desorption/ionization[J]. Rapid Communications in Mass Spectrometry, 2002, 16(16): 1 575-1 581.
[34] SUN G, YANG K, ZHAO Z, et al. Matrix-assisted laser desorption/ionization time-of-flight mass spectrometric analysis of cellular glycerophospholipids enabled by multiplexed solvent dependent analyte-matrix interactions[J]. Analytical Chemistry, 2008, 80(19): 7 576-7 585.
[35] TEUBER K, SCHILLER J, JAKOP U, et al.
MALDI-TOF mass spectrometry as a simple tool to determine the phospholipid/glycolipid composition of sperm: Pheasant spermatozoa as one selected example[J]. Animal Reproduction Science, 2011, 123(3/4): 270-278.
[36] ANGELINI R, BABUDRI F, LOBASSO S, et al.
MALDI-TOF/MS analysis of archaebacterial lipids in lyophilized membranes dry-mixed with 9-aminoacridine[J]. Journal of Lipid Research, 2010, 51(9): 2 818-2 825.
[37] FUCHS B, BISCHOFF A, SÜβ R, et al. Phosphatidylcholines and ethanolamines can be easily mistaken in phospholipid mixtures: A negative ion MALDI-TOF MS study with 9-aminoacridine as matrix and egg yolk as selected example[J]. Analytical and Bioanalytical Chemistry, 2009, 395(8): 2 479-2 487.
[38] CERRUTI C D, BENABDELLAH F, LAPRÉV-
OTE O, et al. MALDI imaging and structural analysis of rat brain lipid negative ions with 9-aminoacridine matrix[J]. Analytical Chemistry, 2012, 84(5): 2 164-2 171.
[39] MARSCHING C, ECKHARDT M, GRÖNE H, et al. Imaging of complex sulfatides SM3 and SB1a in mouse kidney using MALDI-TOF/TOF mass spectrometry[J]. Analytical and Bioanalytical Chemistry, 2011, 401(1): 53-64.
[40] CALVANO C D, ZAMBONIN C G, PALMISANO F. Lipid fingerprinting of Gram-positive lactobacilli by intact cells-matrix-assisted laser desorption/ionization mass spectrometry using a proton sponge based matrix[J]. Rapid Communications in Mass Spectrometry, 2011, 25(12): 1 757-1 764.
[41] 苗 楠, 张 宇, 张建业, 等. 3,4-二氨基苯基苯甲酮在基质辅助激光解析离子化质谱分析磷脂中的应用 [J].分析化学, 2011, 39(5), 605-610.
MIAO Nan, ZHANG Yu, ZHANG Jianye, et al. Matrix of 3, 4-diaminobenzophenone for analysis of phospholipids by matrix-assisted laser desorption/ionization-time of flight-mass spectrometry[J]. Chinese Journal of Analytical Chemistry, 2011, 39(5): 605-610( in Chinese).
[42] THOMAS A, CHARBONNEAU J L, FOURN-
AISE E, et al. Sublimation of new matrix candidates for high spatial resolutionimaging mass spectrometry of lipids: enhanced information in both positive and negative polarities after 1,5-diaminonapthalene deposition[J]. Analytical Chemistry, 2012, 84( 4): 2 048-2 054.
[43] JONES J J, BATOY S M A B, WILKINS C L, et al. Ionic liquid matrix-induced metastable decay of peptides and oligonucleotides and stabilization of phospholipids in MALDI FTMS analyses[J]. Journal of the American Society for Mass Spectrometry, 2005, 16(12): 2 000-2 008.
[44] LI Y L, GROSS M L, HSU F. Ionic-liquid matrices for improved analysis of phospholipids by MALDI-TOF mass spectrometry[J]. Journal of the American Society for Mass Spectrometry, 2005, 16(5): 679-682.
[45] SHRIVAS K, HAYASAKA T, GOTO-INOUE N, et al. Ionic matrix for enhanced MALDI imaging mass spectrometry for identification of phospholipids in mouse liver and cerebellum tissue sections[J]. Analytical Chemistry, 2010, 82(21): 8 800-8 806.
[46] CALVANO C D, CEGLIE C D, ACCOLTI L D, et al. MALDI-TOF mass spectrometry detection of extra-virgin olive oil adulteration with hazelnut oil by analysis of phospholipids using an ionic liquid as matrix and extraction solvent[J]. Food Chemistry, 2012, 134(2): 1 192-1 198.
[47] TOWERS M W, MCKENDRICK J E, CRAMER R. Introduction of 4-chloro-α-cyanocinnamic acid liquid matrices for high sensitivity UV-MALDI-MS[J]. Journal of Proteome Research, 2010, 9(4): 1 931-1 940.
[48] HAM B M, JACOB J T, COLE R B. MALDI-TOF MS of phosphorylated lipids in biological fluids using immobilized metal affinity chromatography and a solid ionic crystal matrix [J]. Analytical Chemistry, 2005, 77(14): 4 439-4 447.
[49] SHANTA S R, ZHOU L, PARK Y S, et al. Binary matrix for MALDI imaging mass spectrometry of phospholipids in both ion modes[J]. Analytical Chemistry, 2011, 83(4): 1 252-1 259.
[50] PARK Y S, YOO C W, LEE S C, et al. Lipid profiles for intrahepatic cholangiocarcinoma identified using matrix-assisted laser desorption/ionization mass spectrometry[J]. Clinica Chimica Acta, 2011, 412(21/22): 1 978-1 982.
[51] SHANTA S R, CHOI C S, LEE J H, et al. Global changes in phospholipids identified by MALDI-MS in rats with focal cerebral ischemia[J]. Journal of Lipid Research, 2012, 53(9): 1 823-1 831.
[52] GUO Z, HE L. A binary matrix for background suppression in MALDI-MS of small molecules[J]. Analytical and Bioanalytical Chemistry, 2007, 387(5): 1 939-1 944.
[53] RAINER M, QURESHI M N, BONN G K. Matrix-free and material-enhanced laser desorption/ionization mass spectrometry for the analysis of low molecular weight compounds[J]. Analytical and Bioanalytical Chemistry, 2011, 400(8): 2 281-2 288.
[54] PILOLLI R, PALMISANO F, CIOFFI N. Gold nanomaterials as a new tool for bioanalytical applications of laser desorption ionization mass spectrometry[J]. Analytical and Bioanalytical Chemistry, 2012, 402(2): 601-623.
[55] XU S Y, LI Y F, ZOU H F, et al. Carbon nanotubes as assisted matrix for laser desorption/ionization time-of-flight mass spectrometry[J]. Analytical Chemistry, 2003, 75(22):6 191-6 195.
[56] PAN C S, XU S Y, ZOU H F, et al. Carbon na-
notubes as adsorbent of solid-phase extraction and matrix of laser desorption/ionization[J]. Journal of the American Society for Mass Spectrometry, 2005, 16(2):263-270.
[57] PAN C S, XU S Y, HU L G, et al. Using oxidized carbon nanotubes as matrix for analysis of small molecules by MALDI-TOF MS[J]. Journal of the American Society for Mass Spectrometry, 2005, 16(6): 883-892.
[58] LIU Q, GUO Z, HE L. Mass spectrometry imaging of small molecules using desorption/ionization on silicon[J]. Analytical Chemistry, 2007, 79(10): 3 535-3 541.
[59] SHEN Z, THOMAS J J, AVERBUJ C, et al. Porous silicon as a versatile platform for laser desorption/ ionization mass spectrometry[J]. Analytical Chemistry, 2000, 73(3): 612-619.
[60] CREDO G, HEWITSON H, BENEVIDES C, et al.
Development of a porous silicon product for small molecule mass spectrometry[M]. Symposium Proceedings: Materials Research Society, 2004, 808: 471-476.
[61] PENG S, EDLER M, AHLMANN N, et al. A new interface to couple thin-layer chromatography with laser desorption/atmospheric pressure chemical ionization mass spectrometry for plate scanning[J]. Rapid Communications in Mass Spectrometry, 2005, 19(19): 2 789-2 793.
[62] CHA S, YEUNG E S. Colloidal graphite-assisted laser desorption/ionization mass spectrometry and MSnof small molecules 1.Imaging of cerebrosides directly from rat brain tissue[J]. Analytical Chemistry, 2007, 79(6): 2 373-2 385.
[63] UGAROV M V, EGAN T, KHABASHESKU D V, et al. MALDI matrices for biomolecular analysis based on functionalized carbon nanomaterials[J]. Analytical Chemistry, 2004, 76(22): 6 734-6 742.
[64] NAJAM-UL-HAQ M, RAINER M, HUCK C W,
et al. Nanostructured diamond-like carbon on digital versatile disc as a matrix-free target for laser desorption/ionization mass spectrometry[J]. Analytical Chemistry, 2008, 80(19): 7 467-7 472.
[65] JACKSON S N, UGAROV M, EGAN T, et al. MALDI-ion mobility-TOFMS imaging of lipids in rat brain tissue[J]. Journal of Mass Spectrometry, 2007, 42(8): 1 093-1 098.
[66] TAIRA S, SUGIURA Y, MORITAKE S, et al. Nanoparticle-assisted laser desorption/ionization based mass imaging with cellular resolution[J]. Analytical Chemistry, 2008, 80(12): 4 761-4 766.
[67] AGETA H, ASAI S, SUGIURA Y, et al. Layer-specific sulfatide localization in rat hippocampus middle molecular layer is revealed by nanoparticle-assisted laser desorption/ionization imaging mass spectrometry[J]. Medical Molecular Morphology, 2009, 42(1): 16-23.
[68] WOODS A S, UGAROV M, JACKSON S N, et al.
IR-MALDI-LDI combined with ion mobility orthogonal time-of-flight mass spectrometry[J]. Journal of Proteome Research, 2006, 5(6): 1 484-1 487.
[69] DREISEWERD K, LEMAIRE R, POHLENTZ G, et al. Molecular profiling of native and matrix-coated tissue slices from rat brain by infrared and ultraviolet laser desorption/ionization orthogonal time-of-flight mass spectrometry[J]. Analytical Chemistry, 2007, 79(6): 2 463-2 471.
[70] SUGIURA Y, SHIMMA S, SETOU M. Two-step matrix application technique to improve ionization efficiency for matrix-assisted laser desorption/ionization in imaging mass spectrometry[J]. Analytical Chemistry, 2006, 78(24): 8 227-8 235.
[71] FERGUSON L, BRADSHAW R, WOLSTENHOLME R, et al. Two-step matrix application for the enhancement and imaging of latent fingermarks[J]. Analytical Chemistry, 2011, 83(14): 5 585-5 591.

