短乳杆菌谷氨酸脱羧酶的生物信息学分析
齐育平,孙 蕾,刘琴英,蒋冬花
(浙江师范大学化学与生命科学学院,浙江金华 321004)
乳酸菌(Lactic acid bacteria,LAB)是指一类能够通过同型发酵或异型发酵而产生乳酸的细菌[1]。大多数乳酸菌是有着重要经济价值的食品级微生物[2]。谷氨酸脱羧酶[3](Glutamate decarboxylase,EC 4.1.1.15,简称GAD)在动物、植物和微生物中广泛存在,催化谷氨酸的α-脱羧反应,生成γ-氨基丁酸(GABA),是GABA唯一的合成酶[4],对维持整个GABA能系统稳定地运转起着至关重要的作用。乳酸菌是一种优良、高产、安全的菌种,利用乳酸菌生产GABA已经成为一种较为理想的生物合成方法。目前已有多种乳酸菌的谷氨酸脱羧酶基因被克隆[5-8]。生物信息学(bioinformatics)是一门利用应用数学、信息学、统计学和计算机科学的方法研究生物学问题的学科。生物信息学的研究工具是计算机,研究方法包括对生物学数据的搜索(收集和筛选)、处理(编辑、整理、管理和显示)及利用(计算、模拟)。目前主要的研究方向有序列比对、基因识别、基因重组、蛋白质结构预测、基因表达、蛋白质反应的预测以及建立进化模型。本文以1株高产GABA的短乳杆菌(Lactobacillus brevis)Lb-2菌株[9]为研究对象,克隆了其GAD基因,并利用生物信息学方法预测分析了其氨基酸组成、理化性质、信号肽及其高级结构等,以期指导分析该菌株的高产GABA特性、后续目的基因的表达纯化以及研究该酶的生物学特性等。
1 材料与方法
1.1 材料
1.1.1 菌种 短乳杆菌(Lactobacillus brevis)Lb-2菌株为本实验室筛选保存的1株高产γ-氨基丁酸(GABA)的菌株。
1.1.2 培养基 改良的MRS培养基(g):蛋白胨10,牛肉提取物10,酵母提取物5,葡萄糖5,乙酸钠2,柠檬酸二胺2,MgSO4·7H2O 0.2,MnSO4·H2O 0.05,吐温-80 1 mL,蒸馏水1 L,pH 6.50。
1.1.3 主要数据库及软件 NCBI blast:http://blast.ncbi.nlm.nih.gov/、ExPASy:http://www.expasy.org/、SignalP:http://www.cbs.dtu.dk/services/SignalP/、GOR Ⅰ:http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_gor.html、PSORT WWW Server:http://psort.hgc.jp/、SWISS-MODEL:http://swissmodel.expasy.org/;LaserGene、MEGA4、DNAMAN。
1.2 方法
1.2.1 GAD基因克隆 以本实验室分离得到的短乳杆菌Lb-2菌株cDNA为模板,参照已发表的短乳杆菌GAD核苷酸序列设计引物,上游引物Forward:5'-ATG TTA CAC AGG CAC GGT TCT AAG CAG AAG-3';下游引物Reverse:5'-TCA ACA TGT TCC TCT ATA GTT TCT C-3')进行PCR扩增,引物由上海生工公司合成。
1.2.2 GAD基因氨基酸序列及保守结构域 利用综合性序列工具软件LaserGene中的ORF Finder确定其完整的编码序列(complete coding sequence,CDS),然后保存为Translation Sequence格式,输出其氨基酸序列。利用NCBI的Conserved Domains检测该序列的保守结构域。
1.2.3 ProtParam预测GAD的理化性质 用Ex-PASy[9]的ProtParam[10]工具对GAD氨基酸序列进行分析,如分子量、等电点、氨基酸组成、摩尔消光系数、半衰期、稳定性等。
1.2.4 GAD 的基本信息分析 Motifscan[11]预测一级结构中糖基化、脂酰化、磷酸化、硫酸化、GPI锚着位点等修饰位点和模序;用SignalP 4.1[12]在线进行信号肽预测;用PSORT WWW Server中的PSORT Prediction工具进行细胞定位;用ExPASy的ProtScale进行在线蛋白质疏水性分析。
1.2.5 GAD 二级结构预测 多肽链的螺旋和折叠使氨基酸形成稳定的空间结构,从而使蛋白具有特定理化性质和生物学活性,利用GORⅠ完成蛋白质的二级结构预测。
1.2.6 GAD 三级结构同源建模 SWISS-MODEL[13-14]是一种使用同源建模方法来预测蛋白质三维结构的工具,利用Automated Mode将GAD与蛋白结构数据库中的蛋白质三维结构进行匹配,预测GAD的三级结构模型。
1.2.7 系统进化树的构建 通过构建系统进化树可以比较不同物种间的进化距离,进而判断它们的亲缘关系。检索GenBank中已公布的GAD氨基酸序列,使用MEGA4[15]软件进行对比分析并构建系统树[15]。
1.2.8 多序列比对 利用DNAMAN软件将获得的GAD氨基酸序列与NCBI里已公布的4种短乳杆菌GAD氨基酸进行多重序列比对,分析所克隆到的GAD与其他短乳杆菌菌株GAD的异同。另4种分别为短乳杆菌877G菌株(Lb-877G,登录号:AFU61547.1)、短乳杆菌 BH2菌株(Lb-BH2,登录号:ABU55419.1),短乳杆菌CGMCC 1306菌株(Lb-CGMCC 1306,登录号:ADG02973.1)和短乳杆菌ATCC 367菌株(Lb-ATCC 367,登录号:YP_795941.1)。
2 结果与分析
2.1 GAD 基因的克隆
本实验以短乳杆菌(图1)cDNA为模板经PCR扩增和琼脂糖凝胶电泳分析获得一长度为1 407 bp的PCR产物(图2)。

图1 短乳杆菌Lb-2显微形态Fig.1 The morphology of Lb-2

图2 PCR产物电泳图Fig.2 The electrophoresis of PCR products
2.2 GAD 基因的氨基酸序列
将上述片段回收、测序,最终得到一条全长为1 407 bp的序列(图3),经软件分析,该序列为一个完整的阅读框,起始密码子为ATG,终止密码子为TAA,编码468个氨基酸。将该序列进行Blast检索,发现其与短乳杆菌877G菌株的GAD基因(登录号:JX545343.1)相似性为99%,与短乳杆菌BH2菌株的GAD基因(登录号:EU084998.1)相似性为98%,推测该序列为短乳杆菌GAD基因序列。将此氨基酸序列提交到Conserved Domains检测保守结构域,结果显示(图4),该序列的保守结构域类似多巴脱羧酶家族,该家族主要包括酪氨酸脱羧酶、组氨酸脱羧酶和谷氨酸脱羧酶。
2.3 短乳杆菌GAD氨基酸序列的理化性质
短乳杆菌GAD共有468个氨基酸(表1),65个带负电氨基酸残基,46个带正电荷氨基酸残基,亮氨酸(Leu)所占比重最高,为9.2%;理论分子量和等电点(PI)分别为53 517.8 u和5.42,分子式为C2412H3675N637O703S21;含有6个半胱氨酸,假设形成3对二硫键,则该蛋白在水溶液中280 nm处的摩尔消光系数为87 125 M-1cm-1;蛋白浓度为l g/L时,半胱氨酸未形成二硫键吸光系数(Abs)为1.621。当成熟肽氨基端的第一个氨基酸为甲硫氨酸(Met)时,GAD在哺乳动物网状红细胞体外表达的半衰期为30 h,在酵母和大肠埃希菌中体内表达的半衰期分别大于20 h和10 h。在溶液中的不稳定指数为27.11,低于阈值40,表明在溶液中性质稳定。脂溶指数为85.04,疏水指数为-0.297,初步判断该蛋白是亲水性蛋白。
2.4 短乳杆菌GAD的信息分析结果
GAD翻译后的修饰位点分析结果显示:GAD含有3个潜在的N-糖基化位点;3个酪蛋白激酶Ⅱ磷酸化位点;5个潜在的N-肉豆蔻酰位点;4个潜在的蛋白激酶C(PKC)磷酸化位点;2个酪氨酸激酶磷酸化位点;1个组氨酸脱羧酶活化位点,位于38~428位氨基酸;1个L-酪氨酸脱羧酶活化位点,位于45~445位氨基酸。
PSORT Prediction预测该蛋白位于细胞质。TMHMM Server 2.0预测其没有跨膜区域。SignalP 4.1 Server预测结果显示该蛋白不含信号肽,可以推断GAD在细胞质中合成后,不进行蛋白转运;TargetP结果未发现线粒体、过氧化物酶体、溶酶体和细胞核等亚细胞定位序列。

表1 短乳杆菌GAD的氨基酸组成Table1 The amino acid composition of GAD of Lb-2

图3 短乳杆菌Lb-2的GAD基因及氨基酸序列Fig.3 The gene and amino acid sequence of GAD of Lb-2

图4 短乳杆菌Lb-2的GAD的保守结构域Fig.4 The conserved domains of GAD of Lb-2
ExPASy的ProtScale在线预测,疏水性氨基酸为正值,亲水性氨基酸则为负值,氨基酸的绝对值越高则表明亲疏水性越强。图5显示该GAD疏水性最小值约为-2.589,最大值约为2.544,整个多肽链中分值较低的氨基酸占多数,即亲水性氨基酸比例高于疏水性氨基酸,因此推断该蛋白为亲水性蛋白,与ProtParam预测结果一致。
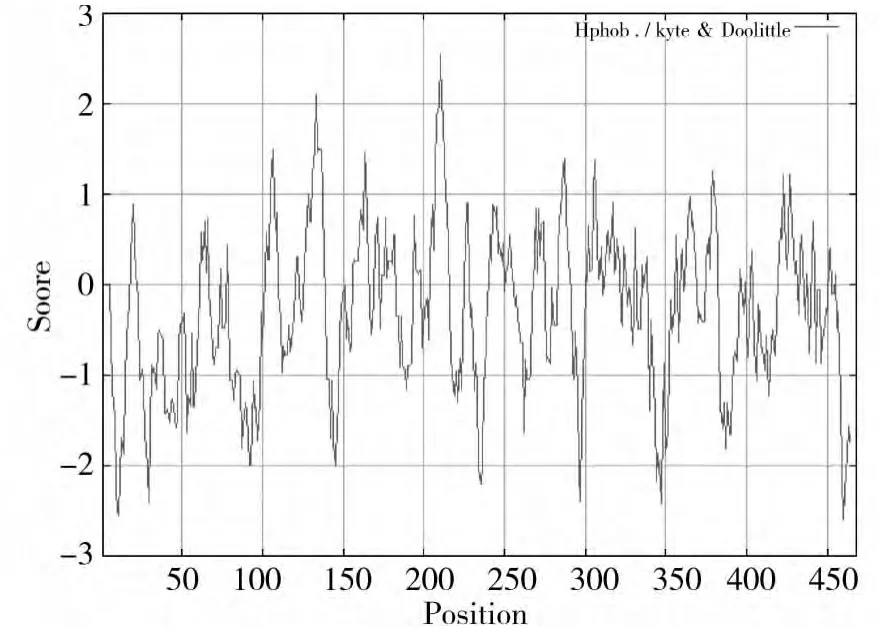
图5 短乳杆菌Lb-2的GAD氨基酸序列亲水性分析Fig.5 The hydrophilicity analysis of the amino acid sequence of GAD from Lb-2
2.5 二级结构分析
蛋白质二级结构预测方法有SOPM、SOPMA、HNN、PHD和GOR等,其中GOR方法将蛋白质序列当作一连串的信息值来处理,不仅考虑了被预测位置本身氨基酸种类的影响,而且考虑了相邻氨基种类对该位置构象的影响,预测效果较好。本文利用GORⅠ预测蛋白二级结构,结果显示(图6),该蛋白二级结构中α-螺旋比例为51.71%,延伸链比例为31.20%,β-转角占8.12%,无规卷曲占8.97%。
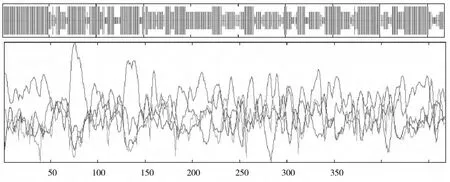
图6 GORⅠ预测的短乳杆菌GAD二级结构Fig.6 The predicted secondary structure of GAD by GORⅠ
2.6 三级结构建模
三级结构是由α-螺旋、β-折叠等二级结构再折叠成一个球形的、包裹紧密的立体空间结构,可使一级结构中2个离得较远的氨基酸残基通过折叠使它们的侧链相互靠近,并且通过疏水作用等构成活性位点。采用SwissModel Automatic Modelling Mode预测短乳杆菌Lb-2的GAD三级结构模型,系统自动选取了拟南芥(Arabidopsis thaliana)GAD1基因作为模板,两者序列相似度为40.23%,Evalue值为 0.00e-1,QMEAN Z-Score为-2.54,四级结构信息显示该模型为单链模型,没有配体。模型(图7)显示该GAD蛋白含有大量的α-螺旋和β-折叠,与二级结构的预测结果相符。
2.7 系统进化树的构建
MEGA是一款主要集中于进化分析进而获得综合序列信息的软件,可以编辑序列数据、序列比对、构建系统发育树、推测物种间的进化距离等。在构建进化树时可提供4种方法:最大简约法、邻接法、最小进化法、算术平均的非加权对群法。本文采用MEGA对乳杆菌目中12种菌的GAD氨基酸序列基于邻位相连法构建系统树,结果如图8所示,短乳杆菌GAD与植物乳杆菌(Lactobacillus plantarum)和德氏乳酸杆菌(Lactobacillus delbrueckii)亲缘关系最近。

图7 短乳杆菌Lb-2的GAD的三级结构预测Fig.7 The predicted tertiary structure of GAD from Lb-2
2.8 多重序列比对
5种短乳杆菌GAD氨基酸序列多重序列比对结果见图9。从图9中可以看出,5种短乳杆菌GAD氨基酸序列的长度一致,都是467个;短乳杆菌种间的GAD氨基酸序列差异很小,只有个别氨基酸的不同;本文得到的短乳杆菌Lb-2的GAD氨基酸序列在416位上为丙氨酸(A),与其他序列有差异,但与短乳杆菌BH2菌株相同。
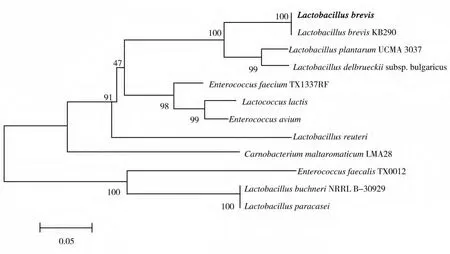
图8 12种乳杆菌GAD的系统进化树Fig.8 The phylogenetic trees of 12 GADs
3 讨论
生物信息学研究的材料是生物学的数据,通过情报学的方法,综合运用数学、计算机科学、情报信息等工具,对生物信息加工后得到相关信息。生物信息学是新兴的学科,是当今国内外研究的热点,在生物技术、生物医学、农业、食品、环境、能源等研究领域发挥了重要作用[16]。
本研究克隆了短乳杆菌GAD基因序列,其全长为1 407 bp,编码468个氨基酸,有酪氨酸脱羧酶结构域,理论分子量和等电点(PI)分别为53 517.8 u和5.42,位于细胞质,为亲水性蛋白,不含信号肽。二级结构预测显示α-螺旋比例为51.71%,延伸链比例为31.20%,β-转角占8.12%,无规卷曲占8.97%,构建系统发育树发现该短乳杆菌GAD基因与植物乳杆菌(Lactobacillus plantarum)和德氏乳酸杆菌(Lactobacillus delbrueckii)的GAD基因亲缘关系最近。
应用软件预测蛋白的结构和功能有一定的局限性,虽然不可能做到完全正确,但总体上近似。此外,由于在蛋白质性质分析过程中采用的方法不同,也会导致分析结果有差异。因此,本研究基本采用目前生物领域较为认可的信息学分析方法,通过这些生物信息学的分析方法所得到信息有一定的参考价值,有助于更加深入地认识乳杆菌谷氨酸脱羧酶,为研究谷氨酸脱羧酶的表达、分离、纯化提供借鉴。
[1]金世琳.乳酸菌的科学与技术[J].中国乳品工业,1998,26(2):14-20.
[2]闫肃,吕嘉枥,郜洪涛.乳酸菌在食品工业中的应用[J].中国酿造,2010,12:1-3
[3]林谦,杨胜远,陆兆新,等.嗜热链球菌谷氨酸脱羧酶基因及其侧翼区序列分析[J].食品科学,2011,32(3):121-125.
[4]杜昭,李世峰,李逸平.谷氨酸脱羧酶在神经系统及雄性生殖系统中的功能[J].生命的化学,2013,33(2):96-100.
[5]Masaru N,Ikuyo N,Yasuhita F,et al.Lactococcus lactiscontains only one glutamate decarboxylase gene[J].Microbiology,1999,145(6):1375-1380.
[6]Park K B,Oh S H.Cloning,sequencing and expression of a novel glutamate decarboxylase gene from a newly isolated lactic acid bacterium,Lactobacillus brevisOPK-3[J].Bioresource Technology,2007,98(2):312-319.
[7]Park K B,Heung O S.Cloning and expression of a full-length glutamate decarboxylase gene fromLactobacillus plantarum[J].Journal of Food Science and Nutrition,2004,9(4):324-329.
[8]Kim S H,Shin B H,Kim Y H,et al.Cloning and expression of a full-length glutamate decarboxylase gene fromLactobacillus brevisBH2[J].Biotechnology and Bioprocess Engineering,2007,12(6):707-712.
[9]缪存影,蒋冬花,徐晓波,等.酸菜中高产 γ-氨基丁酸乳酸菌的筛选和鉴定[J].微生物学杂志,2010,30(2):28-32.
[10]Elisabeth G,Alexandre G,Christine H,et al.ExPASy:the proteomics server for in-depth protein knowledge and analysis[J].Nucleic Acids Research,2003,31(13):3784-3788.
[11]Ayan S,Md J A,Muhammad A K U,et al.Computational analysis of bovine alpha-1 collagen sequences[J].Bioinformation,2013,9(1):42-48.
[12]Shi M,Wang Y N,Zhu N,et al.Four heat shock protein genes of the endoparasitoid wasp,cotesia vestalis,and their transcriptional profiles in relation to developmental stages and temperature[J].Plos One,2013,8(3):1-10
[13]Petersen T N,Brunak S,Von Heijne G,et al.SignalP 4.0:discriminating signal peptides from transmembrane regions[J].Nature Methods,2011,8(10):785-786.
[14]Arnold K,Bordoli L,Kopp J,et al.The SWISS-MODEL Workspace:A web-based environment for protein structure homology modelling[J].Bioinformatics,2006,22:195-201.
[15]Kiefer F,Arnold K,Künzli M,et al.The SWISS-MODEL Repository and associated resources[J].Nucleic Acids Research,2009,37:D387-D392.
[16]Koichiro T,Joel D,Masatoshi N,et al.MEGA4:Molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Molecular biology and evolution,2007,24(8):1596-1599.
[17]孟双,徐冲,陈丽媛,等.生物信息学在生物学研究领域的应用[J].微生物学杂志,2011,31(1):78-81.