自然反义转录物Lmo4as对神经系统重要编码基因Lmo4的调节作用
张 靖,吴 朝,朱丽媛,阴 彬,侯 琳,彭小忠,2*
(中国医学科学院1.基础医学研究所北京协和医学院基础学院医学分子生物学国家重点实验室;2.神经科学中心,北京100005)
长非编码RNA 是指长度大于200 nt、不具有蛋白编码功能的转录产物。近年转录组研究发现不同的物种中都有大量的长非编码RNA,其在表观遗传、疾病发生、个体发育、代谢调节等多个生命过程具有重要的调节作用[1]。自然反义转录物(nature antisense transcript,NAT)是长非编码RNA 中的一类,因由蛋白编码基因的互补链转录而得名[2]。这种双向转录的现象最早于1976年在病毒中发现,随后在不同物种中均有发现[3]。NAT 在转录组中的比例随着物种的进化而升高,提示它们在进化中可能具有重要功能[3]。已有研究结果表明,一些自然反义转录物和正义转录物形成互补双链,通过遮蔽、降解或增加其稳定性等多种方式影响正义转录物的生成和稳定性[4-5]。
神经系统发育是个体发育中最为复杂的过程,转录因子在这一过程中发挥着关键作用[6]。前期研究表明,在大脑皮层发育过程中部分重要的转录因子存在NAT[7]。Lmo4 是一个拥有两个LIM 结构域(Zinc- binding domain present in Lin-11,Isl-1,Mec-3,LIM domain)的连接蛋白,在小鼠的神经系统发育过程中具有重要作用[8]。其基因敲除小鼠有致死表型,并出现脑部发育不全、神经管不能闭合等缺陷[9]。目前尚无Lmo4 的自然反义转录物(Lmo4as)的相关报道。
1 材料与方法
1.1 实验动物
SPF 级ICR 品系孕鼠与E12.5、E14.5、E16.5和E18.5 的胎鼠,以及P1 和P7 的幼鼠[孕鼠购自北京大学医学部动物中心,合格证号:SCXK(京)2006-0008;于中国医学科学院基础医学研究所动物中心培养]。
1.2 材料
Taq DNA 聚合酶、DNA 凝胶回收纯化试剂盒和质粒DNA 提取试剂盒[北京天根(TIANGEN)公司];反转录试剂盒、T4 DNA 连接酶、DNA 分子质量标准、RNase 抑制剂和RNase-Free DNaseI(TaKa-Ra 公司)。
1.3 细胞与组织RNA 的提取及RT-qPCR
对于贴壁生长的细胞,先用PBS 洗细胞1 ~2次,然后按照每(5 ~10)× 106个细胞:1 mL Trizol的比例直接将其加入细胞培养皿中;取新鲜鼠脑组织在液氮中研磨,制成1 mg 组织:1 mL Trizol 的比例,氯仿-异丙醇抽提,75%乙醇漂洗后,空气干燥,加入适量DEPC 处理过的水溶解。使用DNA 酶Ⅰ对RNA 进行处理。每消化10-3mg RNA 用1 U 的DNA 酶Ⅰ,37 ℃处理0.5 h,然后用异丙醇沉淀过夜,纯化RNA 样品。用等量的cDNA 作为模板,用GAPDH 为内参。
1.4 cDNA 末端快速扩增(3'RACE 和5'RACE)
使用Invitrogen 公司的3'RACE 及5'RACE 试剂盒(#18373-019、#18374-058)进行,具体实验步骤详见试剂盒说明书。RACE 后巢式PCR 产物经纯化连入T 载体测序,获得转录本3'RACE 及5'RACE 序列。
3'RACE 引物:GSP1:5'-CTTTGTCATGGGCGGA GC-3';GSP2:5'-GCGGAGCAACATCAGGACAAG-3';GSP3:5'-ATTAGCTTCATTAGTCATTTCCCTCC-3';GSP4:5'-GAGGGTAGAAAGCAGATTGGTGATG-3'; GSP5:5'-CCATCTCCCATTAGCCCAGGTTC-3'。5'RACE 引物:GSP1:5'-GAACCTGGGCTAATGG-3';GSP2:5'-CATCACCAATCTGCTTTCTACCCTC-3';GSP3:5'-AT GAGTTAAAAGGAAAGAGATGTGGC-3' 。
1.5 组织原位杂交
探针制备:将包含有目的转录本探针序列的pGEM-3zf 质粒酶切线性化后,进行体外转录(T7 转录酶)。制备小鼠胚胎脑各时期的组织切片(厚度18 ~22 μm),经50 ℃烘片30 min、焦碳酸二乙酯(DEPC)处理的4%多聚甲醛溶液中固定20 min、1 ×焦碳酸二乙酯处理的PBS 洗片后覆盖杂交液,杂交盒中预杂交1 h。85 ℃5 min 变性探针,替换预杂交液为含探针的杂交液(0.1 ~0.2 ng/μL),65 ~70 ℃杂交过夜(12 ~16 h)。预热0.2 ×SSC,65 ℃洗片;转移至缓冲液B1,室温洗2 次;缓冲液B2 室温封闭1 h;入缓冲液B2 +地高辛抗体(anti-DIG-Ab;1 ∶5 000),4 ℃过夜。缓冲液B1 中洗片(3 × 20 min),缓冲液B3 平衡;200 μL 缓冲液B4显色。ddH2O 停止反应,入100%甲醇中处理15 min~30 min 后封片,观察。
1.6 荧光素酶报告基因实验
双荧光素酶的测定采用Promega 公司的双荧光素酶报告系统,具体操作参见试剂盒说明书。载体质粒为经过改造的pcDNA3.1,使用XhoⅠ和XbaⅠ两个酶切位点,插入Lmo4-3'UTR。构建Lmo4as 过表达质粒,同时准备以下材料:1)TK 质粒(海参荧光质粒),作为共转染的内参。2)miRNA 的过表达质粒,质粒的浓度约500 ng/μL。构建Lmo4-3'UTR-luciferase 使用引物:Lmo4-3UTR-Up 5'-CCCTCGAGTTGGGAATAGCGGTGCTTGC-3';Lmo4-3UTR-Down 5'-GCTCTAGACAACATCAGGACAAGAA CAC-3';产物长度820 bp。
1.7 统计学分析
2 结果
2.1 Lmo4as 的全长序列扩增及验证
经3'RACE 及5'RACE 实验,初步确定Lmo4as的全长1 226 bp,含29 个A 的poly-A 尾,并克隆获得其全长转录本(图1A)。使用UCSC 数据库(mm9)比对结果如图1B 所示,结合3'RACE 结果证明了Lmo4as 具有多聚腺苷酸尾(图1B)。其全长序列信息如图1C 所示。
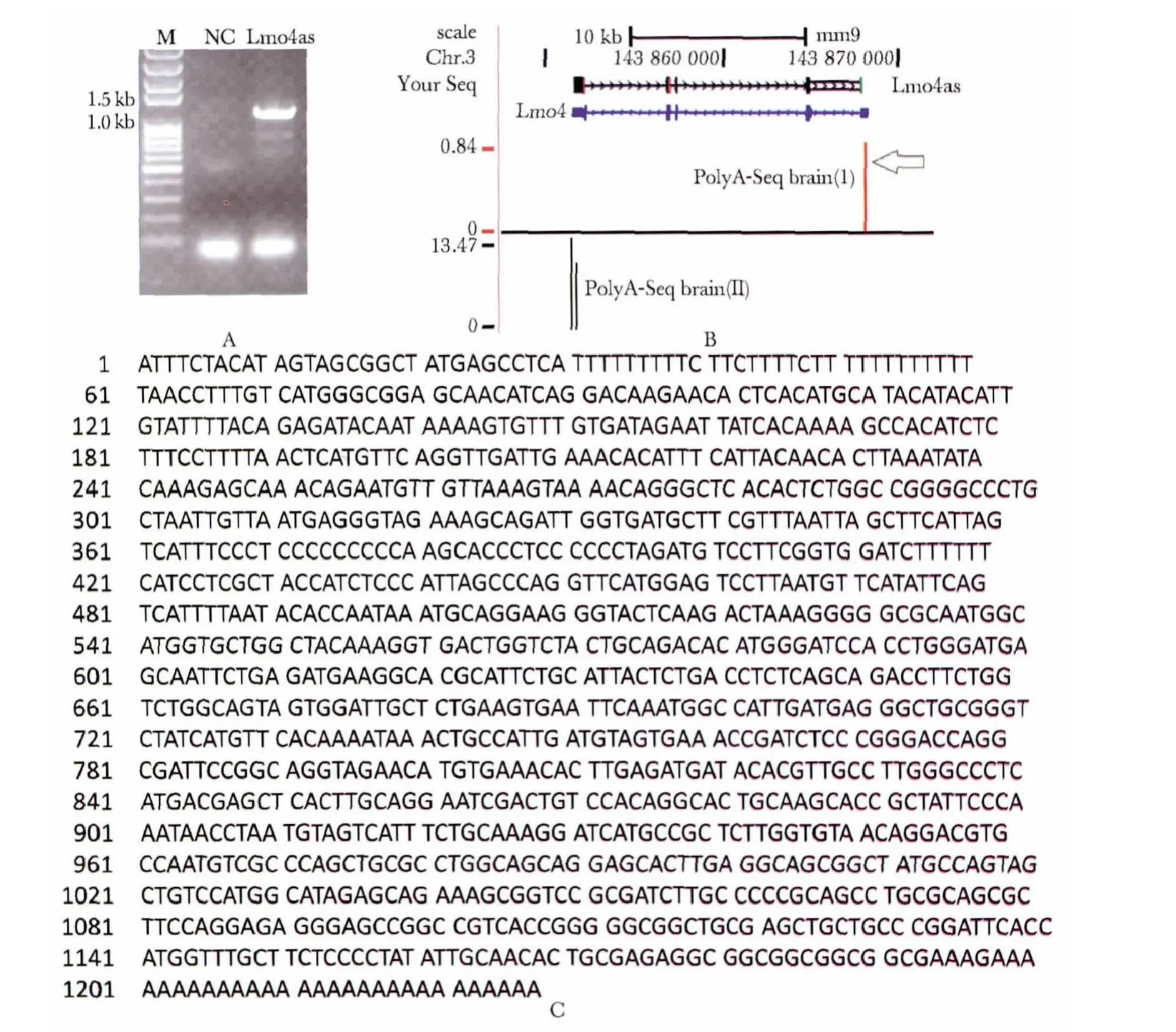
图1 Lmo4as 的全长序列扩增Fig 1 The full length of Lmo4as
2.2 Lmo4as 与Lmo4 mRNA 在小鼠大脑发育阶段的表达情况
E14.5 的原位杂交结果显示,Lmo4as 与Lmo4 的mRNA 在空间分布上有所不同,NAT 的信号相对比较弥散,呈广泛分布(图2A 箭头所示)。Lmo4 的mRNA主要分布于端脑腹侧和局部外侧皮层,而Lmo4as 以室周区较高。多个发育时间点的胎鼠全脑实时定量PCR 检测结果显示二者表达水平随时间变化的特征(图2B)。
2.3 Lmo4as 与miR-124 负性调节Lmo4 mRNA
本研究首先利用Target scan 软件(http://www.targetscan.org/)预测到Lmo4 转录本的3'UTR 区域可能是mmu-miR-124 的靶序列,且在多种物种中极其保守(图3A)。进而克隆Lmo4 转录本的3'UTR,并检测miR-124 mimics、miR-124-inhibitor 及Lmo4as 对含有Lmo4 的3'UTR 萤光素酶报告基因活性的影响。结果显示,miR-124 mimics 可导致萤光素酶活性下降3 倍左右(P<0.01);而miR-124 的inhibitor 可使该萤光素酶的活性升高至1.5 倍(P<0.05);过表达Lmo4as 后,萤光素酶活性下降约3 倍(P<0.05)(图3B)。
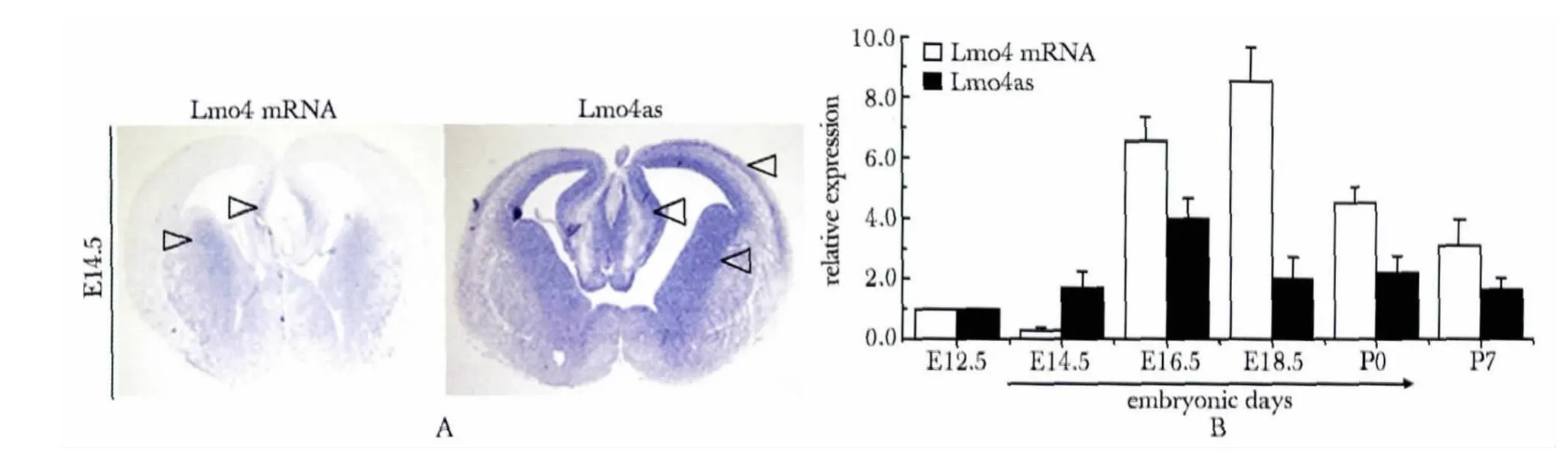
图2 Lmo4 mRNA 及NAT 的时空表达特征分析Fig 2 The spatio-temporal analysis of Lmo4as in the developing embryonic brain of mice
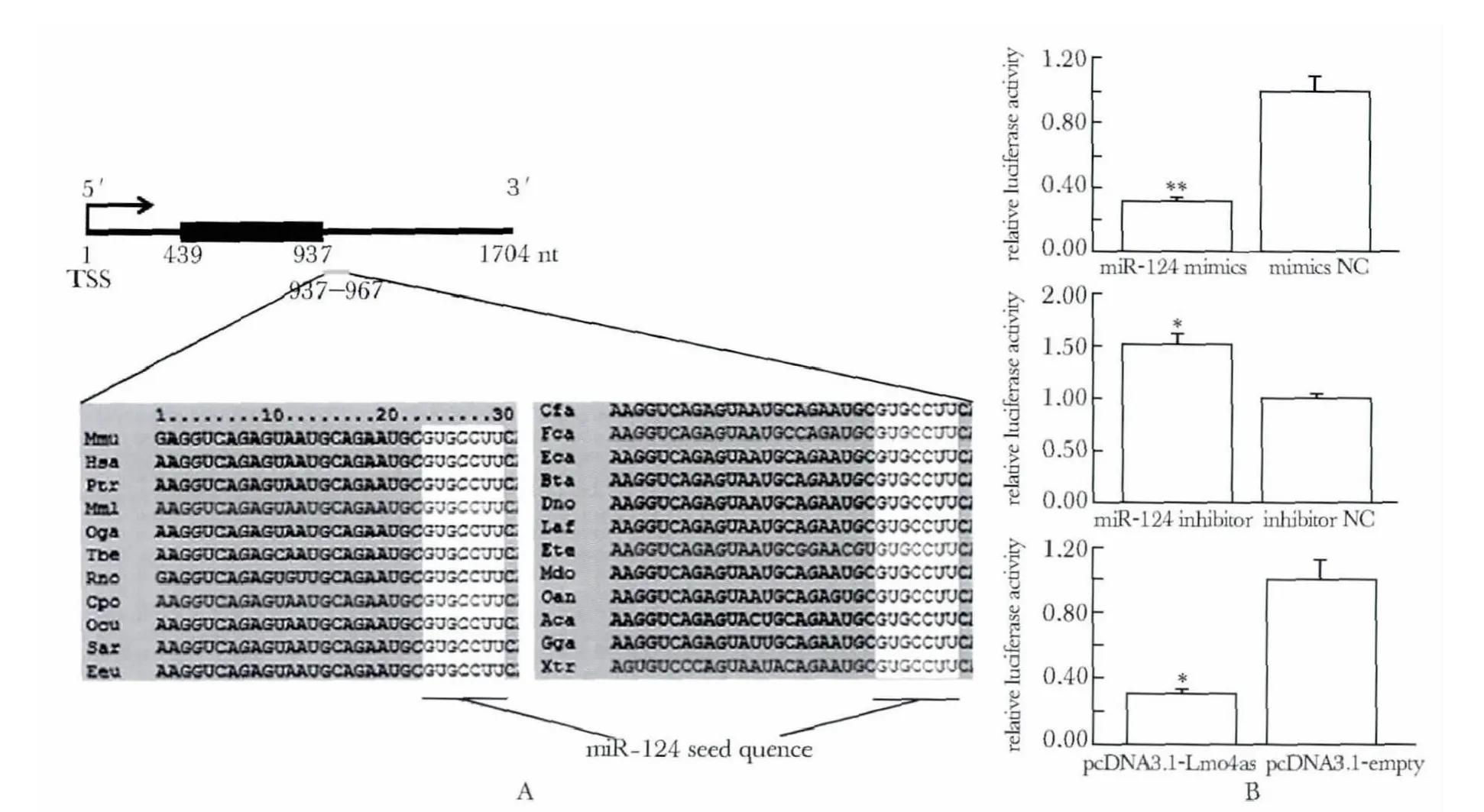
图3 miR-124 和Lmo4as 对Lmo4 的mRNA 进行负性调节Fig 3 Lmo4as negatively regulates Lmo4 at the post-transcriptional level,similar function with miR-124
3 讨论
神经系统发育依赖于在特定时空条件下的复杂基因表达调控网络,而基因表达的动态调节对于细胞命运的转换和谱系的形成都具有重要的作用。越来越多的证据表明长非编码RNA 可参编码基因的表观遗传、转录、转录后调节,且具有时空特异性。近期使用艾伦脑图(Allen Brain Atlas)数据库的数据进行的研究表明,在总数为1 328 的长非编码RNA 中,有849 个表达在成年小鼠大脑中,大部分(623 个)选择性地表达于特定脑区、细胞类型或者亚型中[10]。本研究原位杂交结果也提示Lmo4as可能在发育的一定时空发挥基因表达调控功能。
根据原位杂交和实时定量PCR,可观察到Lmo4as 与Lmo4 mRNA 在时空表达上有一定的关联,提示二者之间可能存在调控关系。进而通过荧光素酶报告基因离体实验的结果,发现Lmo4as 对Lmo4 mRNA 具有顺式的负性调节作用。近期一项研究发现AS1DHRS4 可以通过顺/反式的作用沉默编码基因DHRS4[11],从而提示NAT 不仅可以直接结合正义转录物来发挥作用,还可以通过其他间接方式来实现对正义转录物的调控,因此具有更复杂的分子机制,所以Lmo4as 是否还有反式调节的功能还需要进一步的研究探讨。
除了转录因子与长非编码RNA 的关系以外,RNA 其他成员的存在让这个调控网络变得更加复杂。长非编码RNA 和miRNAs 之间可以产生拮抗作用,例如在阿尔兹海默病中,主要的致病因素是淀粉样肽Aβ,淀粉样前体蛋白(APP)在β 淀粉样前体蛋白裂解酶(BACE1)和r-分泌酶的连续作用下切产生Aβ。BACE-AS 通过增强BACE-1 的稳定性上调BACE-1 的表达水平,之后又证明了miR-485-5p 参与到对BACE-1 的转录后调节,在神经系统中miR-485-5p 与BACE-AS 形成调控网络调节BACE-1蛋白的表达水平[4-5]。在生物体内某些长非编码RNA 可以形成环状结构,如环状转录物ciRS-7 含有大量的miR-7 的结合位点,可以竞争性的结合miR-7,从而发挥调控作用[5]。通过体外的荧光素酶报告实验,可观察到miR-124 和Lmo4as 都具有通过作用于3'UTR 从而抑制性调节Lmo4,但这种类似的抑制性调节功能在神经系统发育中如何发挥功能,还需要进一步实验探索。
[1]John SM.The functional genomics of noncoding RNA[J].Science,2005,309:1527-1528.
[2]Modarresi F,Faghihi MA,Lopez-Toledano MA,et al.Inhibition of natural antisense transcripts in vivo results in gene-specific transcriptional upregula-tion[J].Nat biotechnol,2012,30:453-459.
[3]Amaral PP,Dinger ME,Mercer TR,et al.The eukaryotic genome as an RNA machine[J].Science,2008,319:1787-1789.
[4]Mishra PJ,Banerjee D,Bertino JR.MiRSNPs or miR-polymorphisms,new players in microRNA mediated regulation of the cell:introducing microRNA pharmacogenomics[J].Cell Cycle,2008,7:853-858.
[5]Hansen TB,Jensen TI,Clausen BH,et al.Natural RNA circles function as efficient microRNA sponges[J].Nature,2013,495:385-388.
[6]Bradley JM,Arlotta P,Menezes JR,et al.Neuronal subtype specification in the cerebral cortex[J].Nature,2007,8:427-437.
[7]李萍,张靖,李爱花,等.小鼠大脑皮层层次特异表达基因天然反义转录物的筛选与鉴定[J].中国医学科学院学报,2011,6:620-623.
[8]Kenny DA,Jurata LW,Saga Y,et al.Identification and characterization of LM04,an LMO gene with a novel pattern of expression during embryogenesis[J].Proc Natl Acad Sci U S A,1998,95:11257-11262.
[9]Lee SK,Jurata LW,Nowak R,et al.The LIM domain-only protein LMO4 is required for neuraltube closure[J].Mol Cell Neurosci,2005,28:205-214.
[10]Mercer TR,Dinger ME,Sunkin SM,et al.Specific expression of long noncoding RNAs in the mouse brain[J].Proc Natl Acad Sci U S A,2008,105:716-721.
[11]Li Q,Su ZJ,Xu XY,et al.AS1DHRS4,a head-to-head natural antisense transcript,silences the DHRS4 gene cluster in cis and trans[J].Proc Natl Acad Sci U S A,2012,109:14110-14115.

