拟南芥木聚糖合成关键酶基因的调控研究
王玉琪,贺俊波,吴蔼民
(1华南农业大学生命科学学院,广东广州 510642;2华南农业大学新能源与新材料研究所,广东广州 510642)
木质纤维素指任何纤维素和木质素等作为基本要素积累形成的材料,是地球上最丰富的生物质和可再生资源,近年来被开发成重要的可更新的生物质能源材料[1].现阶段,液体生物质燃料原料开发主要来源于糖、淀粉和油脂类.而从长期的角度出发,生物质能源原料应该主要来源于木质纤维素和藻类油脂等[2-3].虽然目前木质纤维素可以通过水解和酶解等一系列手段使纤维素降解成葡萄糖,并最终降解转化为乙醇,但其工艺较为复杂,成本较高[2-4].而通过了解木质纤维素形成的分子机理,特别是次生壁的形成机理,从而定向改变木质纤维素材料的生物结构和遗传品质,尤其是降低半纤维素和木质素的成分,改变其结构,可以简化工艺流程,降低多糖降解为单/寡糖的糖化成本,满足提高生物质能源产量的要求.这已成为全球能源植物基础研究的热点[2-6].木质纤维素的积累主要通过次生加厚生长完成,它主要由纤维素、半纤维素和木质素3部分组成[7].其中,半纤维素在初生壁中以木葡聚糖(Xyloglucan)存在,但在阔叶树的次生壁中木葡聚糖极少,主要以木聚糖(Xylan)形式存在[8].木聚糖主要由β-(1,4)-D-Xyl(木糖基)为主链、葡萄糖醛酸和/或甲基化的葡萄糖醛酸为侧链组成.另外,其还原末端由β-D-Xyl(木糖基)p-(1→3)-α-L-Rha(鼠李糖基)p-(1→2)-α-D-Gal(半乳糖基)p A-(1→ 4)-D-Xyl p构成[9-10].基于木聚糖的结构,估计需要多个酶来参与木聚糖的主链的延长、还原末端的合成和侧链的修饰.在拟南芥 Arabidopsis thaliana中,已报道基因FRA8(Fragile fiber 8)、F8H(Fragile fiber 8 homolog)、IRX9(Irregular xylem 9)、IRX9-L(Irregular xylem 9-like)、IRX14、IRX14-L、IRX10 和 IRX10-L 可能参与木聚糖主链的延长[10-15].其中,FRA8、IRX9、IRX14 和IRX10是主效基因,而 F8H、IRX9-L、IRX14-L和IRX10-L是次效基因.最近的研究结果也证明了IRX9基因和IRX14基因共同作用能将寡糖聚合更长[16].另外,基因 GUX1(Glucuronic acid substitution of xylan 1)、GUX2和GUX3负责将葡萄糖醛酸(GlcA)加到木聚糖的侧链上[17].
目前,已报道有众多转录因子参与次生细胞壁的加厚生长.研究表明SND1(Secondary wall-associated NAC domain protein 1,also called NST3)、NST1(NAC secondary wall thickening promoting factor 1)、NST2、VND6(Vascular-related NAC-domain 6)、VND7是调控植物次生细胞壁形成的关键转录因子[18-23].SND1基因和NST1基因促进纤维细胞次生壁的形成[20-22],而 VND6、VND7 基因参与导管次生壁的形成[24].研究表明NST1和NST2基因在调控花药内层细胞的次生壁加厚上存在功能冗余[20].转录因子SND1、NST1、NST2、VND6 和 VND7 能够调控 SND2、SND3、KNAT7(Knotted-like homeobox of Arabidopsis Thaliana 7)、MYB20(MYB domain protein 20)、MYB42、MYB43、MYB46、MYB52、MYB54、KMYB69、MYB85、MYB103等次生壁相关的转录因子基因的表达[21-22,24]. 其 中 基 因 SND3、KNAT7、MYB46 和MYB103 是转录因子 SND1、NST1、NST2、VND6 和VND7的直接靶标,显性抑制基因 SND2、SND3、MYB52、MYB54、MYB85和MYB103显著降低纤维细胞次生壁的厚度[24].此外,研究表明IFL1基因参与调控拟南芥束间纤维的分化[25].
木质素的生物合成关键酶基因的调控目前研究较为清楚.木质素生物合成有关的12个酶中,10个酶的基因的启动子区域中,在5'非翻译区100 bp以内的序列上,至少有1个相对保守的AC元件,一些MYB类的转录因子可以与AC元件结合[26-27].然而,目前为止鲜见专门研究调控木聚糖合成的转录因子的报道.我们从 SND1、NST1、NST2、VND7、KNAT7、ERF72(Ethylene response factor 72)、IFL1、MYB20、MYB46、MYB52、MYB69、MYB85 和 MYB103 等次生壁相关的转录因子着手,分析这些转录因子是否调控基因 FRA8、IRX9、IRX10、IRX14、F8H、IRX9-L、IRX10-L和IRX14-L的表达.这一研究有助于弄清木聚糖的合成网络,并且将为进一步把木质纤维素开发成生物燃料打下基础.
1 材料与方法
1.1 材料
用于制备原生质体的拟南芥植株为野生型,购自美国拟南芥种质资源中心(The Arabidopsis Biological Resource Center,ABRC),在 22 ℃,光周期为光照13 h黑暗11 h,50 ~75 μE ·m–2· s–1的弱光条件下培养.
1.2 方法
1.2.1 效应器和报告器的构建 通过PCR反应克隆转录因子基因 SND1、NST1、NST2、VND7、KNAT7、ERF72、IFL1、MYB20、MYB46、MYB52、MYB69、MYB85和MYB103的全长DNA或cDNA,基因 FRA8、IRX9、IRX10、IRX14、F8H、IRX9-L、IRX10-L和 IRX14-L 的启动子序列,再通过BP重组反应将这些序列接到入门载体pDONR207上,测序验证序列后通过LR重组反应将转录因子的序列连接到终载体pUGW2上,将调控序列连接到终载体pUGW35上.
1.2.2 拟南芥原生质体的制备 根据文献[28]介绍的方法制备拟南芥原生质体.将弱光条件下种植的拟南芥的叶片剪成约1 mm宽的细丝,放入含有纤维素酶和离析酶的酶液中消化.然后用75μm的尼龙膜过滤酶液,收集滤液并洗涤.
1.2.3 转染原生质体 用PEG4000-Ca2+溶液介导效应器、报告器和内参质粒转染拟南芥原生质体.每个样品用1×105个原生质体.
1.2.4 测定萤火虫荧光素酶和海参荧光素酶的活性 用Dual-Luciferase reporter assay system试剂盒(Promega公司)在GloMaxTM20/20发光检测仪(Promega公司)上分别测定样品中萤火虫荧光素酶和海参荧光素酶的活性.
1.2.5 显性抑制技术 将KNAT7基因的无终止密码子的全长DNA连接到载体pCAMBIA1304上,构建pCAMBIA1304-p35S∷KNAT7-SRDX表达载体.通过农杆菌 Agrobacterium tumefaciens花序浸染法将pCAMBIA1304-p35S∷KNAT7-SRDX表达载体转化到拟南芥中.通过潮霉素筛选获得转基因植株.同时种植转基因植株和野生型植株,并在相同条件下培养.
1.2.6 数据分析 利用Excel软件对数据进行统计分析.用t检验法分析处理组与对照组是否有显著差异.
2 结果与分析
2.1 转录因子和启动子的克隆
先前的研究发现,木聚糖合成酶关键基因主要在茎的维管组织(Vascular tissue)表达[14-15],而已有研究表明转录因子 SND1、NST1、NST2、VND7、KNAT7、ERF72、IFL1、MYB20、 MYB46、MYB52、MYB69、MYB85和MYB103参与拟南芥次生壁导管的加厚生长[24].为了研究木聚糖合成酶关键基因的调控网络,我们首先克隆这些已报道的转录因子基因来分析它们是否对木聚糖合成酶关键基因有调控作用.按照已发表的基因序列,利用聚合酶链式反应(Polymerase chain reaction,PCR)克隆这些转录因子基因(图1a、1b),连接到载体中并测序验证.同时克隆木聚糖合成关键酶基因的启动子序列(图1c),并连接到载体中测序验证.
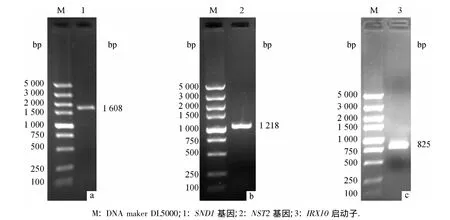
图1 部分转录因子和启动子的PCR扩增产物电泳图Fig.1 The electrophoretogram of partial promoters’and transcription factors’PCR products
2.2 转录因子对基因FRA8、IRX9、IRX10和IRX14的调控作用
将这些转录因子基因的全长序列连接到花椰菜花叶病毒(CaMV)35S的下游,构建成效应器,再将木聚糖合成酶的主效基因 FRA8、IRX9、IRX10和IRX14的启动子分别连接到报告基因萤火虫荧光素酶基因(Fire luciferase,FLUC)的上游,构建报告器(图2).克隆的 FRA8、IRX9、IRX10 和 IRX14 基因的启动子序列的范围依次是-2305~+196、-2011~+34、-663~+162和-2702~+153.将效应器、报告器和携带有位于CaMV35S下游的海参荧光素酶基因(Renilla Luciferase,RLUC)的内参质粒共同转染拟南芥原生质体,培养一段时间后测定FLUC蛋白和RLUC蛋白的活性.FLUC蛋白、RLUC蛋白的活性的比值代表FLUC报告基因的相对表达量,以只转染了报告器和内参质粒的拟南芥原生质体中的FLUC报告基因的相对表达量作为对照组(CK).
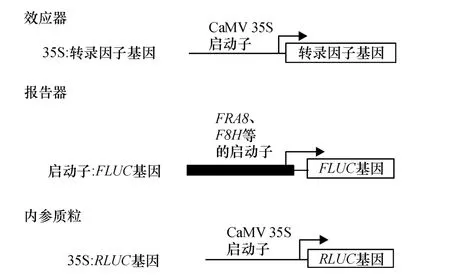
图2 效应器、报告器和内参质粒的结构Fig.2 The structure of effecter,reporter and internal reference plasmid
将克隆的所有次生生长相关的转录因子基因,分别转入上述的反式激活体系中,筛选FLUC报告基因的相对活性较高的处理组合,筛选出来的处理组合各设置3个重复.试验结果表明转录因子KNAT7、ERF72、MYB46、MYB103能促进 FRA8基因的表达,转录因子 SND1、KNAT7、MYB46、MYB85能促进IRX9基因的表达,转录因子 SND1、NST2、VND7、MYB85能促进IRX10基因的表达,转录因子NST2、MYB46能促进IRX14基因的表达(图3).

图3 转录因子对基因FRA8、IRX9、IRX10和IRX14的启动子驱动的报告基因的激活作用Fig.3 The activation of the transcription factors to the reporter gene driven by the promoters of FRA8,IRX9,IRX10 and IRX14
2.3 转录因子对基因 F8H、IRX9-L、IRX10-L和IRX14-L的调控作用
将木聚糖合成关键酶中的次效基因F8H、IRX9-L、IRX10-L和IRX14-L的启动子序列连接到FLUC报告基因的上游,构建报告器.克隆的 F8H、IRX9-L、IRX10-L和IRX14-L的启动子序列的范围依次是-2243~+259、-1212~ +220、-2277~ +237和-513~+28.将效应器、报告器和内参质粒共同转染拟南芥原生质体,培养一段时间后测定FLUC蛋白和RLUC蛋白的活性.FLUC蛋白、RLUC蛋白活性的比值代表FLUC报告基因的相对表达量,以只转染了报告器和内参质粒的拟南芥原生质体中的FLUC报告基因的相对表达量为对照组.
将克隆的所有次生生长相关的转录因子,分别转入上述的反式激活体系中,筛选FLUC蛋白相对活性较高的处理组合,筛选出来的处理组合各设置3个重复.未筛选出能促进IRX10-L基因表达的转录因子.瞬时转录激活分析试验表明转录因子SND1、NST2、MYB20、MYB46能促进F8H基因的表达,转录因子ERF72能促进IRX9-L基因的表达,转录因子VND7、KNAT7、ERF72、MYB46、MYB103 能促 进IRX14-L基因的表达(图4).
2.4 植物体内显性抑制KNAT7的表达
由于KNAT7转录因子能够显著地激活基因FRA8、IRX9和IRX14-L的表达,因此用显性抑制方法来进一步探究KNAT7基因在植物体内的功能.这一方法已经被成功地用于研究与次生壁合成有关的多个转录因子[18-22].将无终止密码子的KNAT7基因的全长DNA序列连接到CaMV35S下游和显性EAR抑制序列的上游,在拟南芥内显性抑制融合表达.KNAT7基因显性抑制的拟南芥转基因植株与野生型植株相比显著变矮,发育不正常(图5).
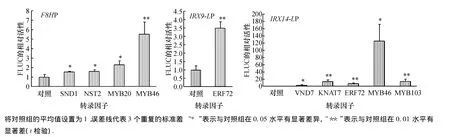
图4 转录因子对基因F8H、IRX9-L、和IRX14-L的启动子驱动的报告基因的激活作用Fig.4 The activation of the transcription factors to the reporter gene driven by the promoters of F8H,IRX9-L and IRX14-L

图5 KNAT7基因显性抑制的植株的表型Fig.5 The phenotype of the KNAT7 gene dominant repression plant
3 讨论与结论
近年来随着木质纤维素作为生物质能源被开发利用,木质纤维素相关的合成及降解机理成为近期的一个研究热点.其中木质素合成酶基因的调控已有一定的研究,发现启动子区有AC元件,并且MYB类转录因子可以与AC元件结合[26-27].而对木聚糖合成关键酶基因的调控研究目前还鲜见报道.这一研究不仅可以为了解木聚糖合成的调控网络打下基础,还可以为将来通过调控转录因子从上游来控制木聚糖合成提供理论基础.
我们通过初步筛选已经获得了多个可以调控木聚糖合成关键酶基因的转录因子,接下来我们准备对这些启动子进行缺失分析,研究这些转录因子与启动子的哪个区域结合.再进一步通过缺失、突变等分析鉴定这些转录因子结合的顺式作用元件.另外,虽然体外试验验证了多个转录因子对木聚糖合成关键酶基因的调控作用,而体内激活作用依旧需要进一步的验证.我们将通过T-DNA插入突变体以及过量表达这些基因,进一步分析在植物体内木聚糖合成关键酶基因的表达变化.
MYB46、KNAT7基因在木聚糖的合成中起着重要的调控作用.已有研究结果表明过量表达MYB46基因能激活木聚糖的合成途径,并且能够激活FRA8基因的表达[29].我们的试验结果表明转录因子MYB46不仅能够激活FRA8基因的表达,还可以激活基因IRX9、IRX14、F8H和IR14-L的表达.
[1]HIMMELM,DING S,JOHNSON D,et al.Biomass recalcitrance:Engineering plants and enzymes for biofuels production[J].Science,2007,315(5813):804-807.
[2]CARROLL A,SOMERVILLE C.Cellulosic Biofuels[J].Annu Rev Plant Biol,2009,60(4):165-182.
[3]PAULY M,KEEGSRRA K.Plant cell wall polymers as precursors for biofuels[J].Curr Opin Plant Biol,2010,13(3):1-8.
[4]SOUSA LD,CHUNDAWATS,BALANA V,etal.“Cradle-to-grave”assessment of existing lignocelluloses pretreatment technologies[J].Curr Opin Biotechnol,2009,20(3):339-347.
[5]PAULY M,KEEGSRRA K.Cell wall carbohydrates and theirmodification as a resource for biofuels[J].Plant J,2008,54(4):559-568.
[6]KO JH,KIM H T,HAN K.Biotechnological improvement of lignocellulosic feedstock for enhanced biofuel productivity and processing[J].Plant Biotechnol Rep,2011,5(1):1-7.
[7]MELLEROWICZ E J,SUNDBBERG B.Wood cell walls:Biosynthesis,developmental dynamics and their implications forwood properties[J].Curr Opin Plant Biol,2008,11(3):293-300.
[8]SCHELLER H,ULVSKOV P.Hemicelluloses[J].Annu Rev Plant Biol,2010,61(1):263-289.
[9]ANDERSSON S,SAMUELSON O,ISHIHARA M,et al.Structure of the reducing end-groups in spruce xylan[J].Carbohydr Res,1983,111(2):283-288.
[10]PENA M J,ZHONG Ruiqing,ZHOU G K,et al.Arabidopsis irregular xylem8 and irregular xylem9:Implications for the complexity of glucuronoxylan biosynthesis[J].Plant Cell,2007,19(2):549-563.
[11]BROWN D M,ZEEF L,ELLIS J,et al.Identification of novel genes in Arabidopsis involved in secondary cell wall formation using expression profiling and reverse genetics[J].Plant Cell,2005,17(8):2281-2295.
[12]BROWN D M,GOUBET F,VICKY W,et al.Comparison of five xylan synthesismutants reveals new insight into themechanisms of xylan synthesis[J].Plant J,2007,52(6):1154-1168.
[13]PERSSON S,CAFFALLH K,FRESHOUR G,etal.The Arabidopsis irregular xylem8 mutant is deficient in glucuronoxylan and homogalacturonan,which are essential for secondary cell wall integrity[J].Plant Cell,2007,19(1):237-255.
[14]WU Aimin,RIHOUEY C,SEVENOM,et al.The Arabidopsis IRX10 and IRX10-LIKE glycosyltransferases are critical for glucuronoxylan biosynthesis during secondary cellwall formation[J].Plant J,2009,57(4):718-731.
[15]WU Aimin,HORNBLAD E,VOXEUR A,etal.Analysis of the Arabidopsis IRX9/IRX9-L and IRX14/IRX14-L pairs of glycosyltransferase genes reveals critical contributions to biosynthesis of the hemicellulose glucuronoxylan[J].Plant Physiol,2010,153(2):542-554.
[16]LEE Chanhui,ZHONG Ruiqin,YE Zhenghua.Arabidopsis family GT43 members are xylan xylosyltransferases required for the elongation of the xylan backbone[J].Plant Cell Physiol,2012,53(1):135-143.
[17]MORTIMER JC,MILESG P,BROWN D M,et al.Absence of branches from xylan in arabidopsis gux mutants reveals potential for simplification of lignocellulosic biomass[J].PNAS(USA),2010,107(40):17409-17414.
[18]KUBO M,UDAGAWA M,NIISHIKUBO N,et al.Transcription switches for protoxylem and metaxylem vessel formation[J].Genes Dev,2005,19(16):1855-1860.
[19]MITSUDA N,SEKIM,SHINOZAKIK,et al.The NAC transcription factors NST1 and NST2 of Arabidopsis regulates secondary wall thickening and are required for anther dehiscence[J].Plant Cell,2005,17(11):2993-3006.
[20]MITSUDA N,IWASE A,YAMAMOTO H,et al.NAC transcription factors,NST1 and NST3,are key regulators of the formation of secondary walls in woody tissues of Arabidopsis[J].Plant Cell,2007,19(1):270-280.
[21]ZHONG R,DEMURA T,YE ZH.SND1,a NAC domain transcription factor,is a key regulator of secondary wall synthesis in fibers of Arabidopsis[J].Plant Cell,2006,18(11):3158-3170.
[22]ZHONG R,RICHARDSON E A,YE ZH.Two NAC domain transcription factors,SND1 and NST1,function redundantly in regulation of secondary wall synthesis in fibers of Arabidopsis[J].Planta,2007,225(6):1603-1611.
[23]KO JH,YANG SH,PARK A H,et al.ANAC012,a member of the plant-specific NAC transcription factor family,negatively regulates xylary fiber development in Arabidopsis thaliana[J].Plant J,2007,50(6):1035-1048.
[24]ZHONG Ruiqin,LEE Chanhui,ZHOU Jianli,et al.A battery of transcription factors involved in the regulation of secondary cell wall biosynthesis in Arabidopsis[J].Plant Cell,2008,20(10):2763-2782.
[25]ZHONG Ruiqin,YE Zhenghua.IFL1,a gene regulating interfascicular fiber differentiation in Arabidopsis,encodes a homeodomain-leucine zipper protein[J].Plant Cell,1999,11(11):2139-2152.
[26]RAES J,ROHDE A,CHRISTENSEN J H,et al.Genome-wide characterization of the lignification toolbox in Arabidopsis[J].Plant Physiol,2003,133(3):1051-1071.
[27]TAMAGNONEA L,MERIDA A,PARR A,et al.The AmMYB308 and AmMYB330 transcription factors from Antirrhinum regulate phenylpropanoid and lignin biosynthesis in transgenic tobacco[J].Plant Cell,1998,10(2):135-154.
[28]YOO Sangdong,CHO Younghee,SHEEN Jen.Arabidopsis mesophyll protoplasts:A versatile cell system for transient gene expression analysis[J].Nature Protocol,2007,2(7):1565-1572
[29]ZHONG Ruiqin,ELIZABETH A R,YE Zhenghua.The MYB46 transcription factor is a direct target of SND1and regulates secondary wall biosynthesis in Arabidopsis[J].Plant Cell,2007,19(9):2776-2792.
【责任编辑李晓卉】

