来源于葡萄球菌的N-乙酰神经氨酸裂合酶的基因克隆及性质
周传华,陈曦,冯进辉,肖冬光,吴洽庆,朱敦明
1 天津科技大学生物工程学院,天津 300457
2 中国科学院天津工业生物技术研究所 工业酶国家工程实验室,天津 300308
羟醛缩合反应是指含有活性α氢原子的化合物如醛、酮、羧酸和酯等,在催化剂的作用下与羰基化合物发生亲核加成,得到b-羟基醛酮或酸。羟醛缩合反应形成新的碳-碳键,是有机合成中极为重要的一类反应[1]。目前实现不对称羟醛缩合反应的化学方法主要有添加手性助剂[2]或用手性路易斯酸[3]、有机小分子[4-6]等进行催化。相对于化学催化法,应用生物催化剂实现羟醛缩合反应具有反应条件温和、绿色无污染、底物立体选择性强等优点,在工业上具有良好的应用前景。
N-乙酰神经氨酸 (Neu5Ac) 是某些复合糖(糖脂和糖蛋白) 中最常见的碳末端,在抗病毒和抗菌等多方面有生物学活性,是合成抗病毒试剂扎那米韦 (Zanamivir) 的重要中间体[7],在生物识别中有很重要的作用,具有极高的商业价值。N-乙酰神经氨酸裂合酶(N-Acetyl-D-neuraminic acid lyase,EC 4.1.3.3简称 NAL)是裂合酶(Lyase,EC 4.x.x.x.)的一种,在一定条件下,该酶既可以将 N-乙酰神经氨酸 (简称 Neu5Ac)裂解成 N-乙酰甘露糖胺 (ManNAc)和丙酮酸(Pyr),又能催化 ManNAc和 Pyr羟醛缩合生成N-乙酰神经氨酸[8](图1)。
NAL在微生物界的分布非常广泛,多种微生物中都曾检测到该酶的活性[9-11],其中来源于大肠杆菌 Escherichia coli[12-14]、梭菌属Clostridium perfringens[15]、嗜血杆菌属Haemophilus influenza[16]、阴道毛滴虫Trichomonas vaginalis[17]、巴氏杆菌属Pasteurella multocida[18]和 乳 杆 菌 属 Lactobacillus plantarum[19]的 N-乙酰神经氨酸裂合酶基因已经得到克隆和表达。目前国内有关Neu5Ac的制备,是通过乳酸氧化酶全细胞催化将乳酸转化成丙酮酸,再用NAL全细胞催化得到Neu5Ac[20]。在生物催化合成Neu5Ac的反应中,最为关键的是醛缩酶 (或裂解酶)的活性。为了获得更高活性、更有利于合成反应的 N-乙酰神经氨酸裂合酶,可以更高效地合成 Neu5Ac,我们通过基因挖矿选择了几个不同来源的基因作为研究对象,经过初步的研究发现来源于葡萄球菌 Staphylococcus hominis的N-乙酰神经氨酸裂合酶的活性较高,对该酶的性质进行了较详细的研究。
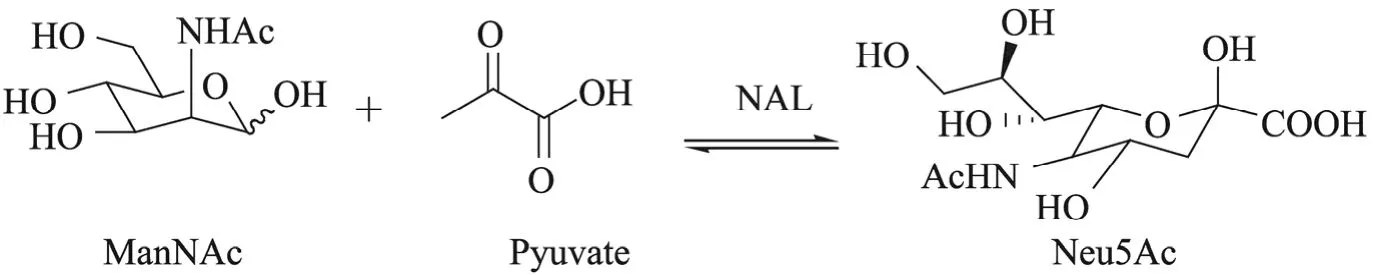
图1 酶法合成Neu5Ac的图示[8]Fig. 1 Enzymatic synthesis of N-acetylneuraminic acid (Neu5Ac)from N-acetyl-D-mannosamine (ManNAc)and pyruvate (Pyr)catalyzed by N-acetylneuraminate lyase (NAL)[8].
1 材料与方法
1.1 材料
1.1.1 菌株和质粒
菌株 E. coli Trans 5α由本实验室保存,pET28a(+)载体和 E. coli BL21 (DE3)均购自Novagen公司。
1.1.2 目的基因
通过基因挖矿获得候选基因,序列信息来源于 NCBI数据库 (GenBank Accession No.EFS20452.1),全基因序列由上海旭冠生物科技发展有限公司合成。
1.1.3 主要试剂
BCA蛋白浓度试剂盒购自康为世纪公司(CWBIO);用于蛋白电泳的试剂及N-乙酰神经氨酸和乳酸脱氢酶 (LDH)购自Sigma公司;考马斯亮蓝R250购自索莱宝公司 (Solarbio);限制性内切酶NdeⅠ和XhoⅠ以及T4 DNA连接酶均购自NEB公司;其他试剂均为分析纯或色谱纯。
1.2 序列分析
氨基酸序列的分析通过 BioEdit软件(CLUSTAL-W)完成,对蛋白的理化性质初步预测通过网站进行 (http://us.expasy.org/admin/users/login)。
1.3 工程菌的构建
质粒的提取、酶切、连接、转化、凝胶电泳、基因的诱导表达等实验操作均按照《分子克隆实验指南》[21]进行。DNA胶回收按照胶回收试剂盒 (Tiangen公司)说明书进行。目的基因shnal构建至pET-28a质粒的NdeⅠ和XhoⅠ两酶切位点之间,转化至E. coli BL21 (DE3)宿主菌中。
1.4 目的蛋白的表达及纯化
将工程菌接入800 mL LB培养基 (含终浓度50 mg/L的卡那霉素)中,37 ℃、200 r/min培养至OD600为0.8左右,添加终浓度为1 mmol/L的异丙基 β-D-硫代半乳糖苷 (IPTG)诱导表达4~6 h。菌液6 000 r/min离心10 min,生理盐水洗涤菌体2次,−20 ℃保存。
将细胞重悬到 100 mL含 5%甘油,500 mmol/L NaCl的 Tris-HCl缓冲液(20 mmol/L,pH 8.0) 中,通过高压匀浆破碎,12 000 r/min、4 ℃离心30 min,除去细胞碎片。上清用 0.45 μm 的滤膜过滤后通过蛋白纯化仪(Purifier 10,GE公司)在室温下用镍柱纯化 (填料购自GE Health Care),3 mL/min流速上样,用含50 mmol/L咪唑、500 mmol/L NaCl的Tris-HCl缓冲液 (20 mmol/L,pH 8.0)洗脱杂蛋白,含0~250 mmol/L咪唑、500 mmol/L NaCl的Tris-HCl缓冲液 (20 mmol/L,pH 8.0)线性洗脱目的蛋白。收集合并目的蛋白,经脱盐浓缩后,适当分装,−80 ℃保存。BCA试剂盒进行蛋白质浓度测定,牛血清白蛋白BSA作为标准蛋白。
ShNAL在溶液中的聚集状态通过凝胶层析以及 SDS-PAGE来进行确定。所用层析柱为Superdex 200 10/300 GL,Gel filtration calibration kit (GE)用于标准分子量测定。流动相为含150 mmol/L NaCl的 20 mmol/L磷酸缓冲液(pH 7.2),流速为 0.4 mL/min。蛋白的分子量大小通过和标准蛋白的保留体积标准曲线进行比较和计算得到。标准蛋白为:Ovalbumin(43.0 kDa),Conalbumin (75.0 kDa),Conalbumin(158.0 kDa),Ferritin (440.0 kDa),Thyroglobulin(669.0 kDa)。
1.5 活性的测定
1.5.1 裂解方向
裂解方向的活性通过酶标仪 (Spectra Max M2/M2e,Molecular Devices公司)测定,其原理是:N-乙酰神经氨酸裂解生成的丙酮酸能在NADH和乳酸脱氢酶存在的条件下被还原成乳酸,过程中消耗了NADH,引起340 nm波长下对 NADH的吸光度的下降[22-23]。一个酶活力单位定义为:在 pH 7.0、37 ℃下,1 min 裂解 1 μmol的 Neu5Ac生成 1 μmol的 N-乙酰-D-甘露糖胺和丙酮酸所需酶的量。反应体系包括 10 mmol/L Neu5Ac、0.25 mmol/L NADH、1 U/mL LDH、0.85 μg ShNAL,反应在常温下不同 pH 的100 mmol/L的缓冲液中进行。
1.5.2 合成方向
合成方向的活性通过 HPLC测定,其原理为:仅加入ManNAc、Pyr和酶,通过HPLC检测Neu5Ac的生成量确定活力大小。一个酶活力单位定义为:在pH 7.0、37 ℃下,1 min合成的1 μmol Neu5Ac所需要的酶量。反应体系包括30 mmol/L ManNAc,100 mmol/L Pyr和 10 μg 的酶,在100 mmol/L的缓冲液中进行30 min,添加10 μL浓H2SO4终止反应。
1.6 酶的性质分析
合成和裂解方向的最适反应 pH测定是在100 mmol/L的不同pH值的缓冲液中进行的,缓冲体系选用醋酸-醋酸钠缓冲液 (pH 4.0~6.0),磷酸盐缓冲液 (pH 6.0~8.0),Tris-HCl缓冲液(pH 8.0~9.0),碳酸盐缓冲液 (pH 9.0~11.0),裂解方向的反应温度为室温,合成方向的反应温度控制在37 ℃、pH 7.5。合成方向的最适反应温度的测定是将反应放入20 ~65 ℃℃的环境中进行反应,测定产物生成量,反应体系同上所述。
1.7 酶的稳定性研究和动力学参数测定
温度耐受性测定是将酶稀释到 pH 7.0的100 mmol/L的缓冲液中,4 ~60 ℃℃温浴0.5~2 h,检测裂解方向的残留活性。pH耐受性测定是将酶稀释到 pH 4.0~11.0的缓冲液中,4 ℃孵育24 h,测定裂解方向的残留活性。
在对Neu5Ac的动力学测定中,测定了不同Neu5Ac浓度下的活性,反应在pH 8.0的缓冲液中进行。在合成方向,反应在pH 7.5的缓冲液中进行,测定ShNAL对丙酮酸的Km值时,保持N-乙酰甘露糖胺的浓度为30 mmol/L,测定不同Pyr浓度下酶的活力;测定 N-乙酰甘露糖胺的Km值时,丙酮酸的浓度 (50 mmol/L)不变,测定N-乙酰甘露糖胺不同浓度下的酶活。
1.8 HPLC分析条件
Agilent公司1100型;分离柱:BioRad公司生产的型号为 Aminex HPX-87H糖分析柱(300 mm×7.8 mm,9 μm),流动相:5 mmol/L H2SO4,流速0.4 mL/min,柱温40 ℃。在上述条件下检测,Neu5Ac、Pyr和ManNAc的保留时间分别是11.9 min、14.2 min和16.2 min。
1.9 合成产物的分离和鉴定
20 mL的Tris-HCl (100 mmol/L,pH 8.0)缓冲液反应体系中加入 300 mmol/L Pyr、100 mmol/L ManNAc、4.5 g湿菌体,在37 ℃、200 r/min条件下反应20 h,通过离心除去菌体,100 ℃加热除去蛋白终止反应。反应的转化率通过HPLC检测ManNAc的减少量得出,产物是通过阴离子交换树脂 (201×7)分离方法[20]纯化。
2 结果
2.1 氨基酸序列分析
将来源于 Staphylococcus hominis与其他菌属来源的 N-乙酰胺神经氨酸裂合酶氨基酸序列进行比较 (图2),发现ShNAL与已知的N-乙酰胺神经氨酸裂合酶,与来源于 Haemophilus influenza[16]、 Clostridium perfringens[15]、Trichomonas vaginalis[17]和 Lactobacillus plantarum[19](GenBank Accession No.分别是P44539.1, Q9S4K9.2, AAB42182.1 和NP_786769.1)的同源性分别为 58%、56%、53%和 50%,而与来源于 Escherichia coli[11](Accession No. AAC76257.1)的同源性只有30%。

图2 ShNAL与已知的N-乙酰神经氨酸裂合酶序列的比对结果Fig. 2 Multiple sequence alignment for ShNAL and related N-acetylneuraminate lyases.
ShNAL拥有NAL亚家族活性位点的关键残基 (图2),包括K165、Y137 (仅指ShNAL的氨基酸位点编号,下同)位点、保守的 GxxGE底物专一性结合框架 (第 47~51个氨基酸)以及D191、E192和 S208[24-25]。GxxGE 是 α-酮酸的羧基结合位点,xx通常是S/T,而ShNAL中这两个氨基酸位点 (48、49位)是 S/S,这两个连续的氨基酸与Y137和水分子共同参与形成丙酮酸氢键网络,D191、E192和S208是底物糖的结合位点。保守区域外的位点有较大差异。
2.2 重组酶的纯化
通过镍柱亲和层析一步纯化得到电泳纯的目的蛋白酶 (图3和表1)。纯化总收率为88.5%,纯度提高了5.6倍。在pH 7.0、37 ℃的条件下,裂解方向的比活达到了 24.8 U/mg (表 1),在pH 7.5、37 ℃的条件下,合成反应方向的比活是3.1 U/mg。用胶过滤层析测定ShNAL的确定分子量为125 kDa,其单体分子量为33.8 kDa,因此该酶在溶液中以四聚体形式存在。
2.3 酶的最适反应pH和温度
裂解方向的最适反应pH是8.0,高于pH 8.0时酶的活性迅速下降,当pH升至9.0时活性下降到最高时的17.5% (图4)。合成方向的最适反应pH是7.5,升高pH对酶的活性影响不大,在pH 9.0的碳酸盐缓冲液中该酶仍然能保持最高活力的95%以上 (图5)。通过对ShNAL合成方向的最适反应温度研究发现,在20 ℃~45 ℃之间,随温度升高酶的活力不断提高,45 ℃时达到最大活力,当温度超过 50 ℃时酶活迅速下降(图 6)。温度对裂解方向酶活性影响的研究表明(数据未显示),直到65 ℃时,酶的活力都随温度升高而不断提高。

图3 SDS-PAGE分析ShNAL的纯化Fig. 3 SDS-PAGE analysis of the purified ShNAL. 1:protein marker; 2:cell-free extract without induction 3:crude cell-free extract; 4: purified ShNAL.

表1 ShNAL蛋白纯化表Table 1 Purification of ShNAL
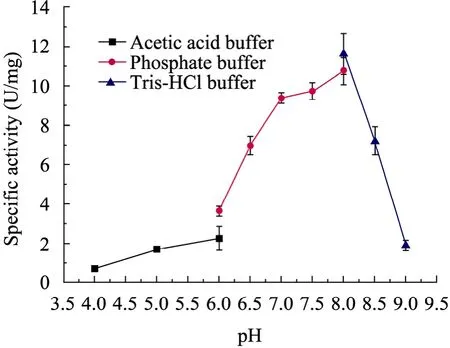
图4 pH对裂解方向酶活性的影响Fig. 4 Effect of pH on ShNAL activity for the cleavage direction. Acetic acid buffer (pH 4.0 to 6.0), phosphate buffer (pH 6.0 to 8.0)and Tris-HCl buffer (pH 8.0 to 9.0)were used.
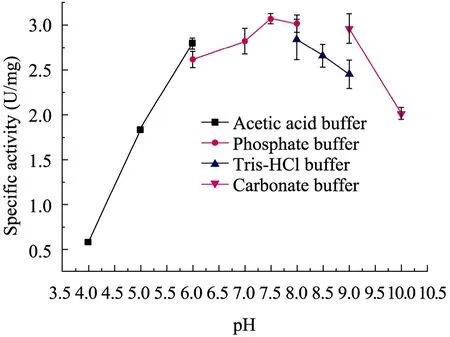
图5 pH对合成方向酶活性的影响Fig. 5 Effect of pH on ShNAL activity for the synthetic direction. Acetate buffer (pH 4.0 to 6.0), phosphate buffer (pH 6.0 to 8.0), Tris-HCl buffer (pH 8.0 to 9.0)and carbonate buffer (9.0 to 10.0)were used.
2.4 酶的稳定性
通过测定裂解反应活性变化来研究 ShNAL对pH的耐受性,结果发现,ShNAL可以耐受较宽的pH范围,在中性pH值范围内最稳定,在pH 5.0~10.0范围内 ShNAL的残留活性能保持70%以上,但pH 4.0的过酸环境和pH 11.0的过碱环境都能使酶失活 (图7)。

图6 温度对合成反应酶活性的影响Fig. 6 Effect of temperature on ShNAL activity for synthetic reaction.
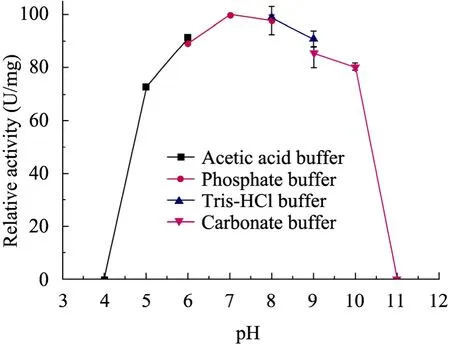
图7 pH的耐受性测定Fig. 7 pH stability of ShNAL. Acetate buffer (pH 5.0 to 6.0), phosphate buffer (pH 6.0 to 8.0), Tris-HCl buffer(pH 8.0 to 9.0)and carbonate buffer (pH9.0 to 11.0)were used.
通过测定裂解方向活性损失对ShNAL的热稳定性进行了研究,发现ShNAL具有较好的热稳定性,在4 ℃~45 ℃温度水浴0.5~2 h均无活性损失。在30 ℃下孵育210 h,ShNAL的活性没有下降,说明酶在30 ℃的稳定性良好,而当温度升到50 ℃时,随时间的延长活性不断下降,50 ℃温浴0.5 h活性约损失一半 (图8)。
2.5 动力学参数的测定
测定合成和裂解两个方向的动力学参数,裂解方向的Neu5Ac的Km值为 (4.0±0.2)mmol/L,合成方向的 ManNAc和 Pyr的 Km值分别为(131.7±12.1)mmol/L 和 (35.1±3.2)mmol/L。Neu5Ac、Pyr和 ManNAc的 kcat/Km值分别为1.9 L/(mmol·s)、0.08 L/(mmol·s)和 0.08 (L/mmol·s)。Neu5Ac、Pyr的Km值与目前报道的来源于其他菌属的 N-乙酰神经氨酸裂合酶相差不大,ManNAc的 Km值比其他来源的酶都要小。ShNAL与之前研究的动力学相关参数比较见表2,ShNAL的裂解方向的催化效率kcat/Km比已知的相关酶要低,而合成方向的催化效率kcat/Km与它们相当,可能更有利于合成反应进行。
2.6 合成产物的分离和鉴定
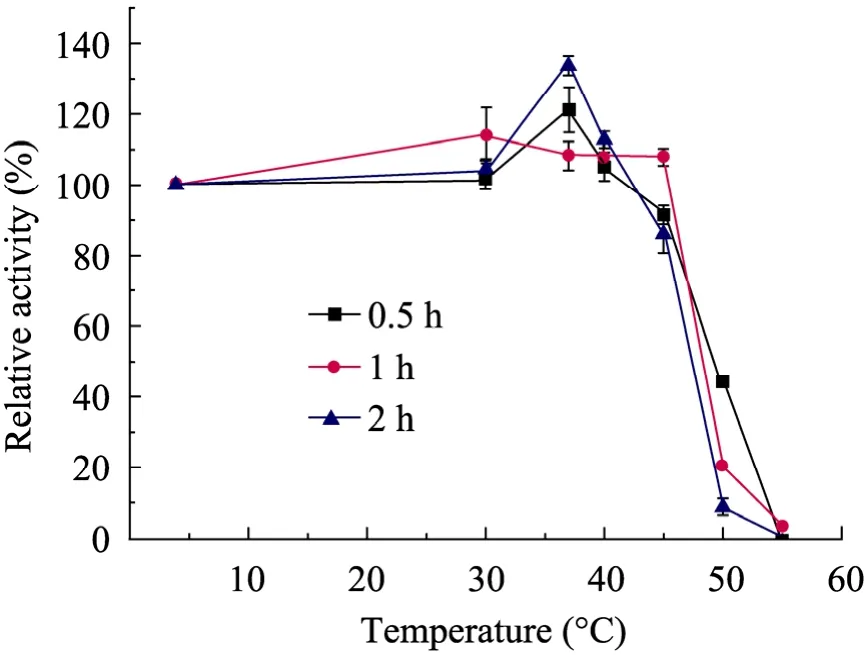
图8 温度耐受性研究Fig. 8 Thermal stability of ShNAL. Enzyme was incubated at certain temperature and maintain for three different times. Activity of enzyme stored in 4 °C was set as 100% relative activity.

表2 ShNAL与之前报道的不同来源的酶的动力学参数比较[18-19]Table 2 Kinetic parameters of recombinant ShNAL and other previously described

图9 Neu5Ac的HPLC图谱 (A:NeuAc标准样品;B:ShNAL催化的反应;C:NeuAc标样和反应物的混合物)Fig. 9 HPLC fingerprint spectrum of Neu5Ac. (A) NeuAc standard sample. (B)Reaction system. (C)Mixture of Neu5Ac standard sample and the reactant.
通过 HPLC检测反应转化率为 91.5%,Neu5Ac的产率达到了89.4% (图9)。反应液经处理上样0~0.2 mol/L甲酸洗脱的样品通过减压蒸馏除去甲酸和水,得到了纯度较高的产物。质谱检测结果:MS (ES−)计算值 C11H19NO9(M-H)308.10,实验值307.82。
3 讨论
目前,国内还没有利用分离纯化的 N-乙酰神经氨酸裂合酶合成Neu5Ac的工业化生产的报道,限制Neu5Ac工业化生产的因素很多,如生产原料 ManNAc非常昂贵外,醛缩酶 (或裂解酶)的活性低等也是重要的原因。有效的解决途径可能是通过异构酶将相对廉价的 N-乙酰葡萄糖胺转化成 ManNAc,再在高活性的 N-乙酰神经氨酸裂合酶催化下来合成Neu5Ac。
本研究克隆并表达了来自 Staphylococcus hominis的潜在 N-乙酰神经氨酸裂合酶基因shnal,研究了纯化酶的酶学性质和动力学参数,与其他来源的NAL相比,ShNAL对ManNAc的Km值最小,kcat/Km值为 0.08 L/(mmol·s),优于之前研究过的0.05 L/(mmol·s)的最大值;在pH 9.0的环境中,ShNAL在合成方向仍能保持合成反应最适pH下活力的95%,而裂解方向的活性降至最适pH下活力的17.5%,偏碱性条件更利于产物合成。综合以上特点,ShNAL在合成反应中具有一定的优势,但是酶的催化活性有待进一步提高,以满足工业化应用的要求。
[1]Palomo C, Oiarbide M, Garcia JM. Current progress in the asymmetric aldol addition reaction.Chem Soc Rev, 2004, 33(2): 65−75.
[2]Evans DA, Bartroli J, Shih TL. Enantioselective aldol condensations. 2. Erythro-selective chiral aldol condensations via boron enolates. J Am Chem Soc, 1981, 103(8): 2127−2129.
[3]Machajewski TD, Wong CH. The catalytic asymmetric aldol reaction. Angew ChemInt Edit,2000, 39(8): 1352−1374.
[4]Córdova A, Zou W, Dziedzic P, et al. Direct asymmetric intermolecular aldol reactions catalyzed by amino acids and small peptides. Chem Eur J, 2006, 12(20): 5383−5397.
[5]Dalko PI, Moisan L. In the golden age of organocatalysis. Angew ChemInt Edit, 2004,43(39): 5138−5175.
[6]Notz W, Tanaka F, Barbas CF. Enamine-based organocatalysis with proline and diamines: the development of direct catalytic asymmetric aldol,Mannich, Michael, and Diels−Alder reactions. Acc Chem Res, 2004, 37(8): 580−591.
[7]Von Itzstein M, Wu WY, Kok GB, et al. Rational design of potent sialidase-based inhibitors of influenza virus replication. Nature, 1993,363(6428): 418−423.
[8]Groher, A. and Hoelsch K. Mechanistic model for the synthesis of N-acetylneuraminic acid using N-acetylneuraminate lyase from Escherichia coli K12. J Mol Catal B: Enzym, 2012, 83(0): 1−7.
[9]Heimer R, Meyer K. Studies on sialic acid of submaxillary mucoid. Proc Natl Acad Sci USA,1956, 42(10): 728−734.
[10]Popenoe EA, Drew RM, Kee L. The action of an enzyme of Clostridium perfringens on orosomucoid. J Biol Chem, 1957, 228(2): 673−683.
[11]Yoshihir U, Yoji T, Tsunetake S. Distribution of N-acetylneuraminate lyase in bacteria and its production by Escherichia coli. Agric Biol Chem,1985, 49(1): 181−187.
[12]Aisaka K, Uwajima T. Cloning and constitutive expression of the N-acetylneuraminate lyase gene of Escherichia coli. Appl Environ Microbiol, 1986,51(3): 562−565.
[13]Ohta Y, Watanabe K, Kimura A. Complete nucleotide sequence of the E. coli N-acetylneuraminate lyase. Nucleic Acids Res,1985, 13(24): 8843−8852.
[14]Ohta Y, Shimosaka M, Murata K, et al. Molecular cloning of the N-acetylneuraminate lyase gene in Escherichia coli K-12. Appl Microbiol Biotechnol,1986, 24(5): 386−391.
[15]Traving C, Roggentin P, Schauer R. Cloning,sequencing and expression of the acylneuraminate lyase gene from Clostridium perfringens A99.Glycoconj J, 1997, 14(7): 821−830.
[16]Lilley GG, Barbosa JA, Pearce LA. Expression in Escherichia coli of the putative N-acetylneuraminate lyase gene (nanA)from Haemophilus influenzae: overproduction,purification, and crystallization. Protein Expr Purif,1998, 12(3): 295−304.
[17]Meysick KC, Dimock K, Garber GE. Molecular characterization and expression of a N-acetylneuraminate lyase gene from Trichomonas vaginalis. Mol Biochem Parasitol, 1996, 76(1/2):289−292.
[18]Li Y, Yu H, Cao H, et al. Pasteurella multocida sialic acid aldolase: a promising biocatalyst. Appl Microbiol Biotechnol, 2008, 79(6): 963−970.
[19]Sanchez-Carron G, Garcia-Garcia MI,Lopez-Rodriguez AB, et al. Molecular characterization of a novel N-acetylneuraminate lyase from Lactobacillus plantarum WCFS1. Appl Environ Microbiol, 2011, 77(7): 2471−2478.
[20]Xu P, Qiu JH, Zhang YN, et al. Efficient whole-cell biocatalytic synthesis of N-Acetyl-D-neuraminic acid. Adv Synth Catal, 2007, 349(10): 1614−1618.
[21]Sambrook J, William D, Russell. Molecular Cloning: A Laboratory Manual. 3rd ed. New York:Cold Spring Harbor Laboratory Press, 2001:99–125.
[22]Aisaka K, Igarashi A, Yamaguchi K, et al.Purification, crystallization and characterization of N-acetylneuraminate lyase from Escherichia coli.Biochem J, 1991, 276(2): 541−546.
[23]Wang TH, Lee WC. Production of 2-keto-3-deoxy-d-glycero-d-galacto-nonopyranulos onic acid (KDN)using fusion protein of N-acetyl-d-neuraminic acid aldolase. Biochem Eng J, 2006, 29(1/2): 75−80.
[24]Barbosa JA, Smith BJ, DeGori R, et al. Active site modulation in the N-acetylneuraminate lyase sub-family as revealed by the structure of the inhibitor-complexed Haemophilus influenzae enzyme. J Mol Biol, 2000, 303(3): 405−421.
[25]Lawrence MC, Barbosa JA, Smith BJ, et al.Structure and mechanism of a sub-family of enzymes related to N-acetylneuraminate lyase. J Mol Biol, 1997, 266(2): 381−399.