灰色链霉菌胰蛋白酶在变铅青链霉菌中的异源表达及酶学性质分析
马腾博,令桢民,康振,李江华,堵国成,陈坚
1 江南大学 工业生物技术教育部重点实验室,江苏 无锡 214122
2 江南大学生物工程学院,江苏 无锡 214122
3 江南大学 糖化学与生物技术教育部重点实验室,江苏 无锡 214122
4 江南大学 粮食发酵工艺与技术国家工程实验室,江苏 无锡 214122
胰蛋白酶 (Trypsin,EC 3.4.21.4)属于丝氨酸蛋白酶类,是一种通过专一作用于精氨酸和赖氨酸所形成的肽键来催化蛋白质水解的碱性蛋白酶。其最初是从牛胰腺泡中提取得到,后被发现广泛存在于猪、狗、鼠等哺乳动物、昆虫和海洋生物中[1]。目前,哺乳动物胰蛋白酶,尤其是牛胰蛋白酶,被广泛应用于诸多工业领域。Wahlby等在链霉菌中发现了胰蛋白酶,尤其以灰色链霉菌中胰蛋白酶的研究最为深入[2-4]。链霉菌源胰蛋白酶同哺乳动物胰蛋白酶具有高度同源性且作用机理相似[5],但因其成分相对单一,作用条件温和,纯化工艺简单而受到越来越多研究者的关注。
灰色链霉菌胰蛋白酶 (SGT) 由 sprT基因(GenBank Accession No. M64471)编码,其氨基酸序列由信号肽 (32个氨基酸)、前导序列 (4个氨基酸)和成熟酶 (223个氨基酸)三部分组成[6]。它是以无活性的酶原前体的形式合成、储存以及分泌的,和哺乳动物胰蛋白酶不同,SGT的分泌不需要外源激酶的切割,而是通过自催化作用产生有活性的成熟酶[6-7]。关于SGT野生酶的报道较早,但一直集中在酶学特性上,对产量的研究较少。Robert等在1975年就对其酶学和物理化学性质进行了研究[8],SGT的晶体结构也得到了进一步的阐释[9],并被广泛用作研究酶学动力学的模型。sprT基因GC含量高达74%,具有严重的密码子偏好性,在翻译过程中需要形成3对二硫键,因此其活性表达存在一定难度。在SGT异源表达方面,国内外报道较少,Page等将sprT成熟酶基因连接在pWB980载体上,以枯草芽胞杆菌Bacillus subtilis WB700为宿主实现了其异源表达[10]。Kim等以变铅青链霉菌Streptomyces lividans为宿主也实现了sprT基因的异源表达[11],但酶活相对较低,其表达以pWHM3为载体,采用sprT自身启动子,强度较弱。我们曾尝试以大肠杆菌 Escherichia coli和B. subtilis为宿主表达,结果都产生无活性包涵体。链霉菌宿主具有良好的生物安全性,同时能够解决重组胰蛋白酶限制修饰、酶原切割以及对宿主毒性的问题[12-13]。因此,本研究选用红霉素抗性基因启动子,以S. lividans为宿主,实现了胰蛋白酶的高效表达,并对重组酶酶学性质进行了系统的比较分析。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒
灰色链霉菌 Streptomyces griseus ATCC 10137来自美国典型菌种收藏所,变铅青链霉菌Streptomyces lividans TK24、大肠-链霉菌的穿梭载体pIJ86均来自英国John Innes研究所,克隆质粒pMDl8-T simple vector从TaKaRa公司购置。
1.1.2 培养基与抗生素
大肠杆菌采用 LB培养基,培养时加入100 μg/mL氨苄青霉素。变铅青链霉菌TK24液体生长培养基 (YEME)、原生质体转化培养基(R5)、固体平板生长培养基 (MS)配制方法参照文献[14],重组变铅青链霉菌发酵培养基 (XSH、C5[15]、R2YE[14]、TSB[14]、SELF)配制方法见表 1,重组变铅青链霉菌培养时加入 50 μg/mL安普霉素 (Apramycin)。
1.1.3 主要试剂和仪器
限制性内切酶SphⅠ和BglⅡ、SolutionⅠ连接酶、DNA标准分子量、DNA聚合酶均为TaKaRa公司产品。PCR产物纯化试剂盒为上海生工生物技术有限公司产品。质粒提取试剂盒为博凯思维公司产品。蛋白质分子量标准和考马斯亮蓝蛋白浓度测定试剂盒为碧云天公司产品。其余药品均为进口或国产分析纯化学试剂。本研究使用的主要仪器有:AKTA FPLC蛋白纯化系统(Aamersham pharmacia biotech);PCR仪 (Techne公司);SDS-PAGE系统 (Aamersham pharmacia biotech);UV-2450光谱分光光度计 (Shimadzu Co.,Kyoto,Japan);Hitrap benzamidine FF 纯化柱 (GE Healthcare)。
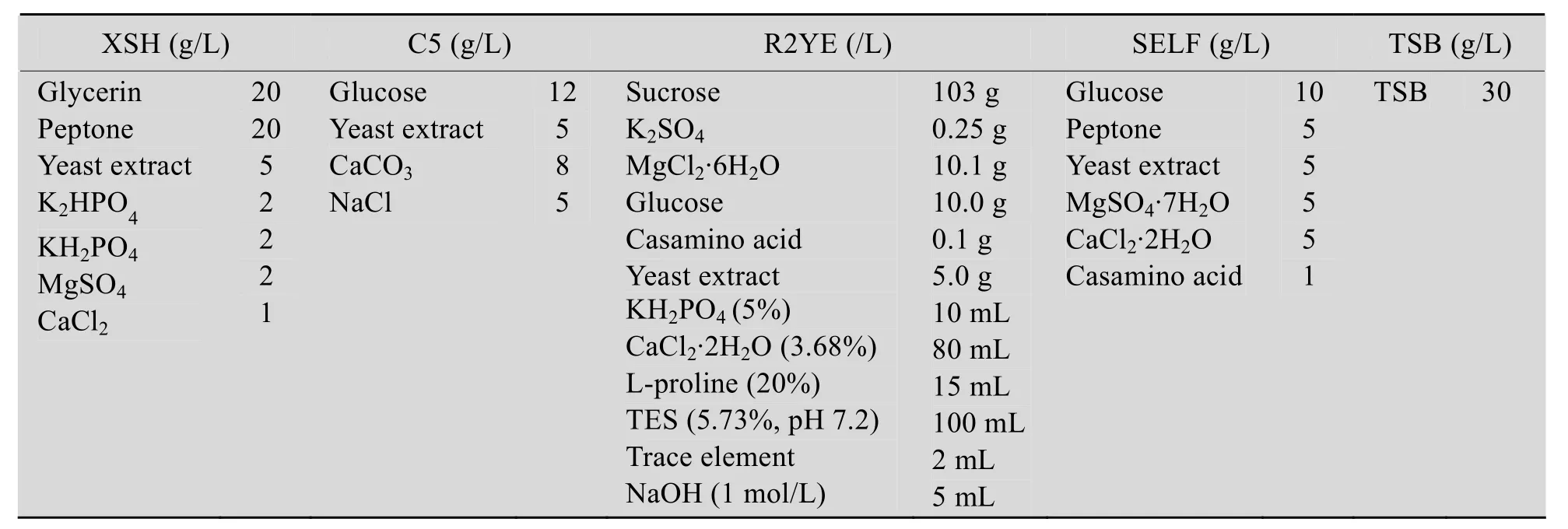
表1 重组链霉菌发酵培养基的组成Table 1 Composition of media for fermentation of recombinant strain
1.2 基因操作
1.2.1 S. griseus胰蛋白酶基因sprT的克隆
提取S. griseus ATCC 10137基因组,根据NCBI公布ATCC 10137胰蛋白酶CDS区基因序列 (sprT),分别以其5′和3′端设计正反引物序列(表 2),由上海生工生物工程技术服务有限公司合成,其上下游引物分别加入 SphⅠ和 BglⅡ酶切位点 (下划线标示)。PCR反应的总体系为50 μL,以1 μL的灰色链霉菌基因组为模板PCR扩增得到目的基因 sprT。反应条件为:95 ℃5 min;95 ℃ 1 min,60 ℃ 1 min,72 ℃ 40 s,30个循环;72 ℃ 10 min。
1.2.2 表达载体的构建
PCR产物经琼脂糖凝胶电泳鉴定、回收,采用TaKaRa公司PCR产物纯化试剂盒 (操作见试剂盒说明书)纯化后连接 pMDl8-T Simple Vector,转入大肠杆菌JM109中,提取重组质粒 pMDl8-T-sprT并用 SphⅠ、BglⅡ双酶切,经0.8%琼脂糖凝胶电泳鉴定、回收、纯化后,用SolutionⅠ连接到同样经过 SphⅠ、BglⅡ双酶切的pIJ86质粒上,构建重组表达载体pIJ86-sprT。转化后提取质粒经 PCR和双酶切鉴定,筛选阳性克隆进行测序分析。
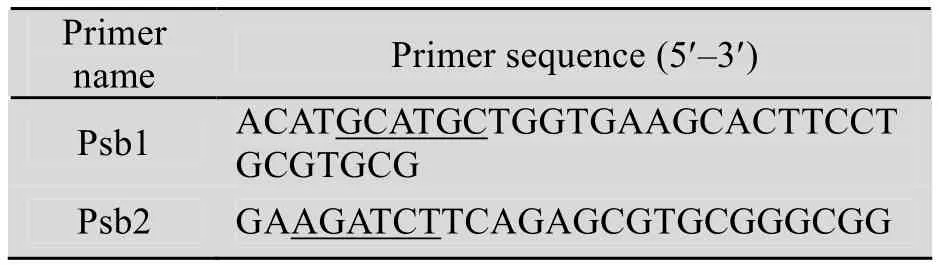
表2 克隆目的基因sprT的引物Table 2 Primers for cloning of target genes
1.2.3 重组S. lividans TK24/pIJ86-sprT的构建
将穿梭质粒 pIJ86和重组穿梭质粒pIJ86-sprT转化S. lividans TK24原生质体,TK24原生质体的制备与DNA转化参照Hopwood等链霉菌遗传实验手册[14]。转化后涂布R5再生固体培养基平板,培养 24 h后在平板表面均匀添加50 μg/mL的安普霉素,培养48 h筛选得到阳性菌落,挑选阳性转化子划线转接MS固体培养基,得到重组菌株TK24/ pIJ86和TK24/pIJ86-sprT。
1.3 重组胰蛋白酶在S. lividans TK24中表达
分别将重组菌株TK24/pIJ86和TK24/pIJ86-sprT链霉菌孢子接种于 100 mL含有安普霉素(50 μg/mL)的种子培养基R2YE中,于30 ℃摇床 (转速200 r/min)培养24 h后,按5%的接种量转接50 mL链霉菌发酵培养基XSH (50 μg/mL安普霉素,放置2个枪头用于搅拌),30 ℃摇床(转速200 r/min)培养。发酵第3天开始取样,之后每两天取样,采用BAPNA法测定酶活。样品处理后经SDS-PAGE检测,聚丙烯酰胺凝胶采用碧云天蛋白电泳试剂制备。
1.4 产酶发酵培养基的优化
将筛选出的重组菌株 TK24/pIJ86-sprT均匀涂布在MS固体培养基上,30 ℃培养3 d,对菌株进行活化,使其处于良好的生长状态,将活化后的重组菌株的孢子接种至种子培养基 R2YE(50 mL/250 mL)中,200 r/min、30 ℃培养24 h后,以 5%的接种量转接不同的发酵培养基R2YE、SELF、C5、TSB (50 mL/500 mL),第 3天开始取样,之后每两天取样,采用BAPNA法测定酶活。
1.5 酶活测定
1.5.1 BAPNA测定法
[16]的方法进行测定:N-苯甲酰-DL-精氨酸-硝基苯胺 (BAPNA)是一种含有酰胺键的合成物,胰蛋白酶对酰胺键有专一的水解能力,因此BAPNA测定法是一种通过检测胰蛋白酶对酰胺键的消化情况来测定胰蛋白酶活力的方法。底物为890 μL反应缓冲液 (50 mmol/L Tris-HCl,pH 8.0,20 mmol/L CaCl2)与 10 μL BAPNA (100 mmol/L)的混合物,将此底物在37 ℃下预热5 min后,加入100 μL酶液迅速混合均匀,37 ℃反应2 min,测其在410 nm处吸光值的变化情况。在上述条件下,每分钟A410升高0.1为一个酶活。胰蛋白酶酰胺键水解活力计算公式如下:
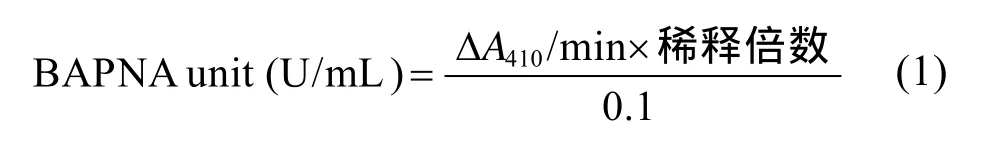
1.5.2 BAEE测定法
参考文献[16]的方法进行测定:N--苯甲酰-L-精氨酸盐酸乙酯是一种含有酯键的合成物,胰蛋白酶能够专一地水解酯键,BAEE测定法就是一种通过酯键的水解来检测吸光值的变化从而计算出胰蛋白酶活力的方法。底物为3 mL BAEE缓冲溶液 (67 mmol/L的磷酸钠缓冲液,pH 7.6,0.25 mmol/L BAEE),将此底物在25 ℃预热5 min后,加入200 μL酶液迅速混合均匀,25 ℃反应2 min,测其在253 nm处吸光值的变化情况。在上述条件下,每分钟A253升高0.001为一个酶活。胰蛋白酶酯键水解活力计算公式如下:
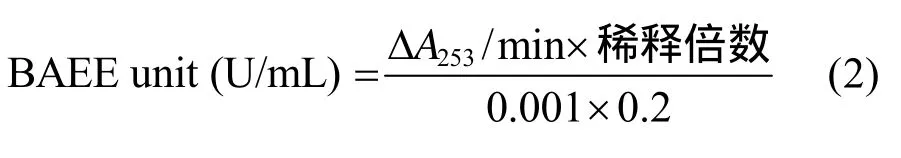
1.6 重组胰蛋白酶的分离与纯化
参照文献[16]和[17]的方法:取500 mL重组菌株发酵产物,4 ℃、10 000 r/min离心10 min,将所得发酵上清液用0.22 μm的PVDF水膜过滤后,流经Hitrap benzamidine FF纯化柱 (Φ1.6 cm×2.5 cm,CN17-5144-01,GE公司),先用缓冲液A (50 mmol/L Tris-HCl,pH 7.4,0.5 mol/L NaCl)洗去未特异结合于柱上的杂蛋白,再用缓冲液B(50 mmol/L甘氨酸,10 mmol/L HCl,pH 3.0)洗脱,收集洗脱下来的色谱峰,得到纯化后样品。12% SDS-PAGE分析纯化后样品条带,考马斯亮蓝试剂盒 (碧云天P0006)测定纯化后蛋白浓度。
1.7 重组胰蛋白酶rSGT与牛胰蛋白酶BT的酶学性质比较
1.7.1 rSGT和BT最适温度及温度稳定性比较
选用 BAPNA测定法,在不同温度条件下(10 ℃ ~ 70 ℃ )分别测定rSGT和BT活力;将纯化后的rSGT和BT纯酶液分别在不同温度下保温不同的时间 (10、20、30、40、50、60 min),检测剩余的酶活力。
1.7.2 rSGT和BT最适pH及pH稳定性比较
选用BAPNA测定法,在不同pH反应条件下 (pH 3.0~12.0)分别测定rSGT和BT的活力;将纯化后的rSGT和BT纯酶液分别在不同pH值的缓冲液中37 ℃保温1 h,检测剩余的酶活力。
1.7.3 rSGT和BT酶反应动力学参数比较
选用BAPNA测定法和BAEE测定法,分别选择合适的底物浓度,以系列稀释的底物溶液测定酶动力曲线,根据双倒数作图法求得在两种底物反应条件下 rSGT和 BT的 Km、Vmax、kcat和kcat/Km数值。
1.7.4 金属离子对rSGT和BT活力的影响比较
分别在BAPNA和BAEE两种底物的反应体系中加入不同浓度的金属离子,使 Ca2+和 Mg2+的最终浓度为1 mmol/L,其余金属离子的终浓度为0.5 mmol/L,以不加金属离子的反应体系的酶活定义100%,在两种底物反应条件下分别测定加入金属离子后各反应体系中rSGT和BT的相对酶活。
1.7.5 抑制剂和有机溶剂对rSGT和BT活力的影响比较
在两种底物和酶的反应体系中,分别加入不同的蛋白抑制剂和有机溶剂,以不加任何蛋白抑制剂和有机溶剂的反应体系的酶活定义100%,测定rSGT和BT的相对酶活。
2 结果与分析
2.1 重组质粒pIJ86-sprT的构建
以 ATCC 10137基因组为模板,利用引物Psb1、Psb2,PCR扩增出sprT,产物经1.0%琼脂糖凝胶电泳检测,可见在780 bp附近的扩增条带。纯化后的 PCR 产物和 pMD18-T Simple Vector 连接后,与 pIJ86质粒同时用 SphⅠ和BglⅡ双酶切,纯化后连接,得到重组穿梭质粒pIJ86-sprT (图1)。酶切鉴定表明重组载体构建成功。测序结果与NCBI登记序列相一致。本研究选用携带红霉素抗性基因启动子 (PermE)的pIJ86质粒为载体,成功构建了重组表达质粒pIJ86-sprT,与sprT自身启动子 (Ptry[6])相比,PermE具有更高的强度 (图2)。
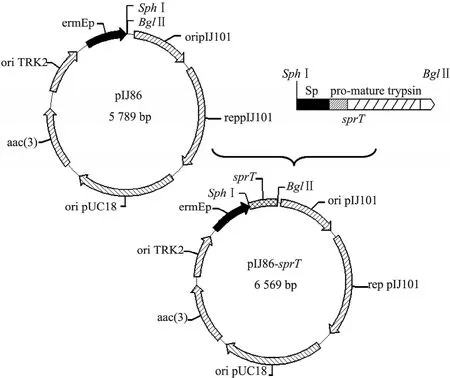
图1 重组穿梭质粒pIJ86-sprT的构建Fig. 1 Construction of the recombinant shuttle plasmid pIJ86-sprT.
2.2 pIJ86-sprT在TK24中的表达
分别将质粒pIJ86和重组质粒pIJ86-sprT转化至原生质体S. lividans TK24挑取阳性转化子,命名为TK24/pIJ86和TK24/pIJ86-sprT。将重组菌株接种至 100 mL种子培养基 R2YE中,200 r/min、30 ℃摇床培养24 h,以5%接种量转接链霉菌发酵培养基XSH,从第3天开始取样,之后每隔一天取样,将1 mL样品5 000 r/min离心5 min,得发酵上清液。根据BAPNA法测定酶活,对照重组菌TK24/pIJ86基本检测不到酶活,携带重组质粒的TK24/pIJ86-sprT菌株第3天可以检测到酶活,到第9天酶活最高可达3.56 U/mL(图 3)。为进一步确定重组蛋白表达,利用SDS-PAGE电泳检测蛋白条带,在预测的28 kDa处有明显的特异性表达条带,如图4所示。
2.3 发酵培养基的优化
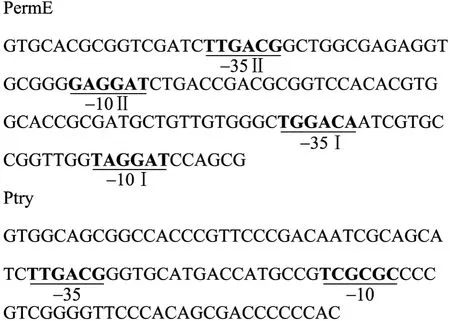
图2 PermE和Ptry的序列比较Fig. 2 Comparison of the sequences of PermE and Ptry. The putative −10 and −35 regions are underlined and in bold.
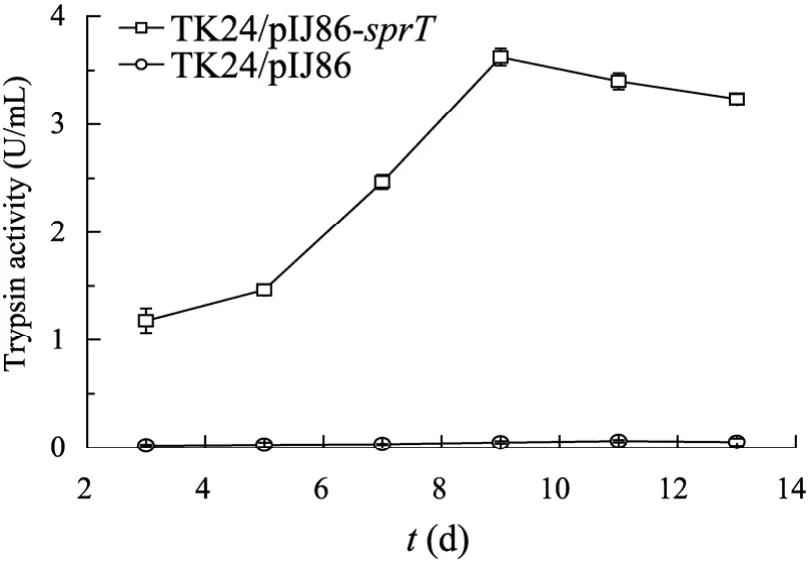
图3 重组TK24/pIJ86-sprT发酵过程中的胰蛋白酶酶活曲线Fig. 3 The curve of trypsin activity during fermentation of recombinant TK24/pIJ86-sprT.
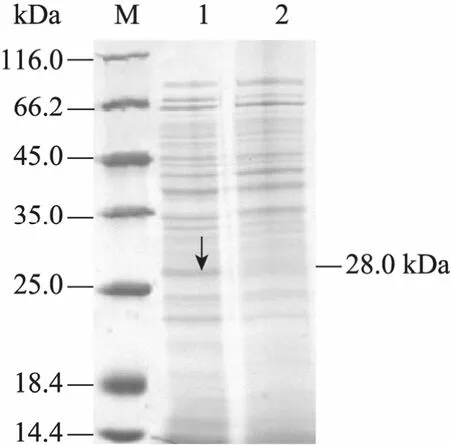
图4 重组TK24/pIJ86-sprT蛋白表达的SDS-PAGE分析Fig. 4 SDS-PAGE analysis of expression proteins in recombinant TK24/pIJ86-sprT. M: protein marker; 1:recombinant TK24/pIJ86-sprT; 2: recombinant TK24/pIJ86.
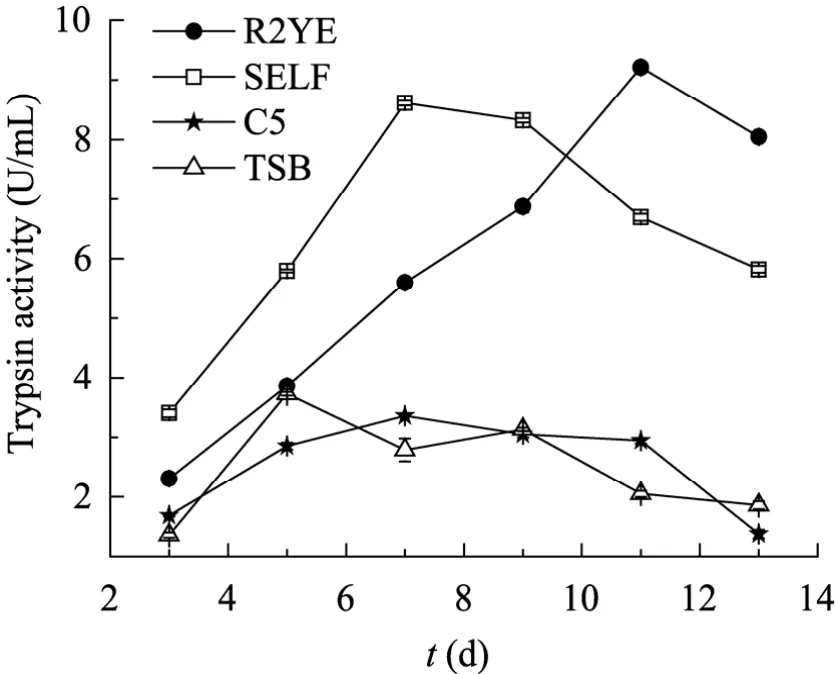
图5 不同发酵培养对重组TK24/pIJ86-sprT产胰蛋白酶的影响Fig. 5 Effects of different media on production of trypsin from TK24/pIJ86-sprT.
将筛选出的重组菌株 TK24/pIJ86-sprT涂布于MS固体培养基上活化,使其处于良好的生长状态,将活化后重组菌株的孢子接种至种子培养基 R2YE (50 mL/250 mL)中,200 r/min、30 ℃培养24 h,以5%的接种量转接不同的发酵培养基 R2YE、SELF、C5、TSB (50 mL/500 mL),第3天开始取样,之后每隔一天取样,测定酶活,结果如图5所示。从图中可以看出,R2YE在第11天酶活最高达9.21 U/mL,SELF在第7天酶活最高达 8.61 U/mL,C5在第 7天酶活最高达3.36 U/mL,TSB在第5天酶活最高达3.73 U/mL。以上结果表明,以R2YE作为发酵培养基可获得最高酶活9.21 U/mL,但发酵周期较长;以SELF为发酵培养基,虽然最高酶活为8.61 U/mL,但在第7天便得到最高值,相比以R2YE作为发酵培养基,发酵周期缩短。因此选取SELF培养基为重组菌发酵培养基。
2.4 重组胰蛋白酶的分离与纯化
将纯化的重组胰蛋白酶进行 SDS-PAGE电泳,如图6所示,其蛋白条带显示其相对分子质量约为28 kDa,与报道基本一致,且蛋白带纯度可以用于酶学性质检测。
2.5 rSGT与BT的酶学性质比较
2.5.1 rSGT和BT最适温度及温度稳定性比较
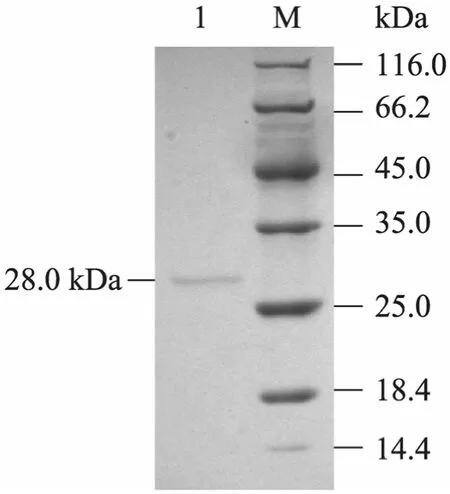
图6 纯化后rSGT的SDS-PAGE分析Fig. 6 SDS-PAGE analysis of the purified rSGT. 1:purified rSGT; 2: protein marker.
rSGT和BT的最适温度和温度稳定性见图7。由图7A可知,两者的最适温度都在50 ℃左右,在10 ~50 ℃℃范围内,随着温度的升高,rSGT和BT的活力均成上升趋势,当温度高于50 ℃,两种酶活力迅速降低,70 ℃时催化活性趋近于零;在图7B和7C中,BT在30 ℃~40 ℃均能保持较高活力 (>80%),且在30 ℃几乎没有酶活损失,50 ℃处理60 min仍能保持42.45%的酶活力;rSGT在30 ℃也能保持较高活力,但在40 ℃时活力明显下降,但在40 min内仍能保持50%以上的活力,50 ℃~70 ℃活力迅速下降,10 min后基本检测不到酶活。表明虽两者最适温度均在50 ℃左右,但从稳定性来看,作用温度在30 ℃~40 ℃之间更为合理,且相对于BT,rSGT在40 ℃~50 ℃稳定性更差。
2.5.2 rSGT和BT最适pH 及pH 稳定性比较
图8A中,在不同pH反应条件下分析rSGT和BT活力,发现两者均在中性偏碱性环境下有较高的催化活性,最适反应pH为7.5~10,与BT相比,rSGT具有较高的酸度耐受性,在pH 5.0的条件下也能保持 60%以上的催化活性。但从稳定性上来看 (图8B),rSGT和BT在酸性和中性条件下放置1 h后活力几乎没有受到影响,在pH 8.0条件下测定酶活力均保持在 90%以上,而在 pH>9.0条件下放置,rSGT和BT活力都有不同程度的降低,pH 12.0条件下,rSGT活力降低到40%以下。
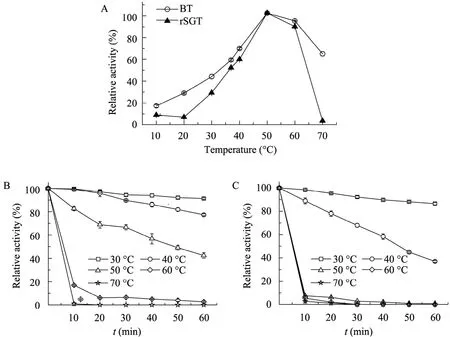
图7 rSGT和BT的最适反应温度和温度稳定性的比较分析Fig. 7 Comparative analysis of the optimal reaction temperature and thermostability of rSGT and BT. (A)The optimal reaction temperature of rSGT and BT. (B)The thermostability of BT. (C)The thermostability of rSGT.
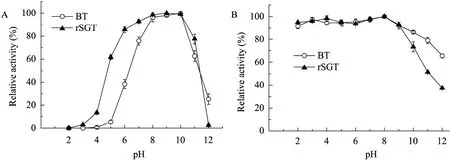
图8 rSGT和BT的最适反应pH和pH稳定性的比较分析Fig. 8 Comparative analysis of the optimal reaction pH and pH stability of rSGT and BT. (A)The optimal reaction pH of rSGT and BT. (B)The pH stability of rSGT and BT.
2.5.3 rSGT和BT酶反应动力学参数比较
分别选用BAPNA测定法和BAEE测定法,分析 BT和 rSGT在这两种底物反应条件下的Km、Vmax、kcat和 kcat/Km(表 3)。rSGT 与 BAPNA和BAEE两种底物反应的Km相近,但水解酯键(BAEE) 的最大反应速率、转换常数 (Kcat)和催化效率 (Kcat/Km)则分别是水解酰胺键(BAPNA)的8倍、13倍和15倍;BT对两种底物的特异性有明显差别,与 BAPNA反应的 Km是与BAEE反应Km的30倍,其对酯键的水解活力远远高于酰胺键,与报道的大多数鱼类及甲壳类动物相近[18]。选用BAPNA做底物时,BT的Km是rSGT的10倍,由此可见rSGT对酰胺键的特异性远远大于 BT,其最大反应速率、转化常数和催化效率分别是BT的2倍、3倍和27倍,而采用 BAEE作为底物,BT的特异性略高于rSGT,催化效率相同,但反应速率和转换常数均小于rSGT。
2.5.4 金属离子对rSGT和BT活力的影响比较
各种金属离子对rSGT和BT活力的影响如表4所示,在两种底物反应条件下,rSGT和BT均能被1 mmol/L的Ca2+激活[19],被0.5 mmol/L的 Cu2+、Pb2+、Co2+、Ag+、Hg2+抑制,其中 Cu2+、Pb2+、Co2+对rSGT和BT与两种底物反应的抑制作用均较弱,Ag+对rSGT和BT与BAPNA反应的抑制作用不明显,而对rSGT和BT与BAEE反应的抑制作用较强,在 Hg2+存在条件下,BT与BAEE反应,酶活力被完全抑制,而rSGT与BAEE作用只有较弱的抑制作用。Mg2+对两种底物的作用效果不明显,Zn2+对 rSGT和 BT与BAPNA的反应有一定的抑制作用,但对 rSGT和BT与BAEE的反应却有一定的激活作用。由于rSGT和BT在结构和功能上的相似性,不同金属离子对rSGT和BT活力的影响也表现出一定的相似性,但在与两种底物的反应中,Ag+、Hg2+和Zn2+对rSGT和BT酰胺酶和酯酶的催化活力表现出了一定的差异。

表3 rSGT和BT在两种底物反应条件下的动力学参数的比较Table 3 Comparison of kinetic properties of rSGT and BT with two kinds of substrates
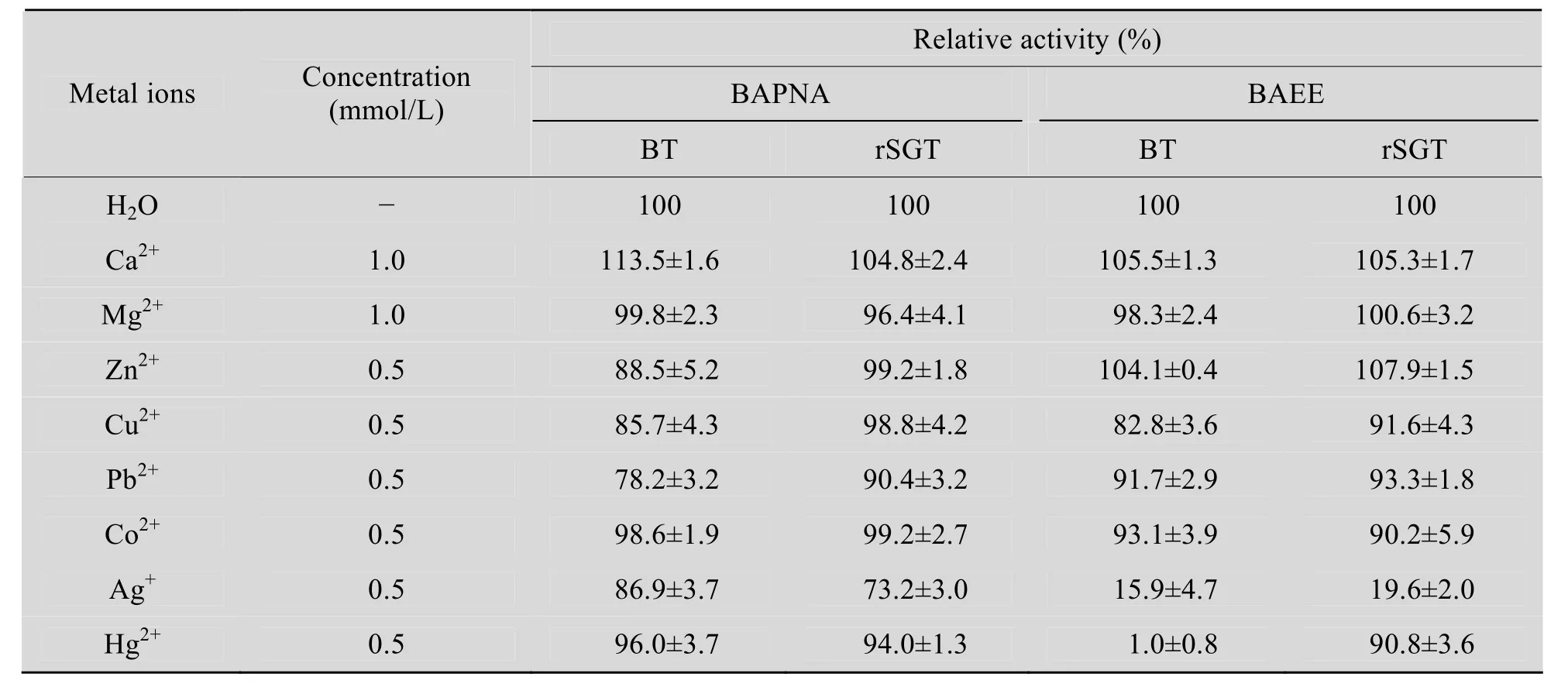
表4 在两种底物反应条件下各种金属离子对rSGT和BT活力的影响Table 4 Effect of different metallic ions on rSGT and BT activity with two kinds of substrates
2.5.5 抑制剂和有机溶剂对rSGT和BT活力的影响比较
抑制剂和有机溶剂对rSGT和BT活力的影响见表5,在两种底物反应条件下,CEOM、SBTI和Benzamidine都能强烈抑制rSGT和BT的活力,与 CEOM、SBTI和 Benzamidine能抑制甲壳类动物、鱼和高等脊椎动物的胰蛋白酶活力的报道相符[18]。PMSF是一种常见的丝氨酸蛋白酶抑制剂,能够一定程度地抑制rSGT和BT的活力,对酯键 (BAEE)的抑制作用明显大于对酰胺键 (BAPNA)的抑制作用。TPCK对rSGT和BT与两种底物的反应均有抑制作用,但抑制作用较弱。在以BAPNA为底物时,25%的甲醇、乙醇、甘油、丙酮和DMSO对BT均有一定的抑制作用,但对rSGT却几乎没有抑制,反而使其活力增强,表明当作用于酰胺键时,rSGT的有机溶剂耐受性要强于BT,甚至有机溶剂的存在有利于rSGT活力的提高。在以BAEE为底物时,不同有机溶剂对rSGT和BT活力的影响不同,甲醇、DMSO对rSGT和BT活力有促进作用,乙醇和丙酮对rSGT和BT有抑制作用,在丙酮存在的条件下,两者的活力被完全抑制。
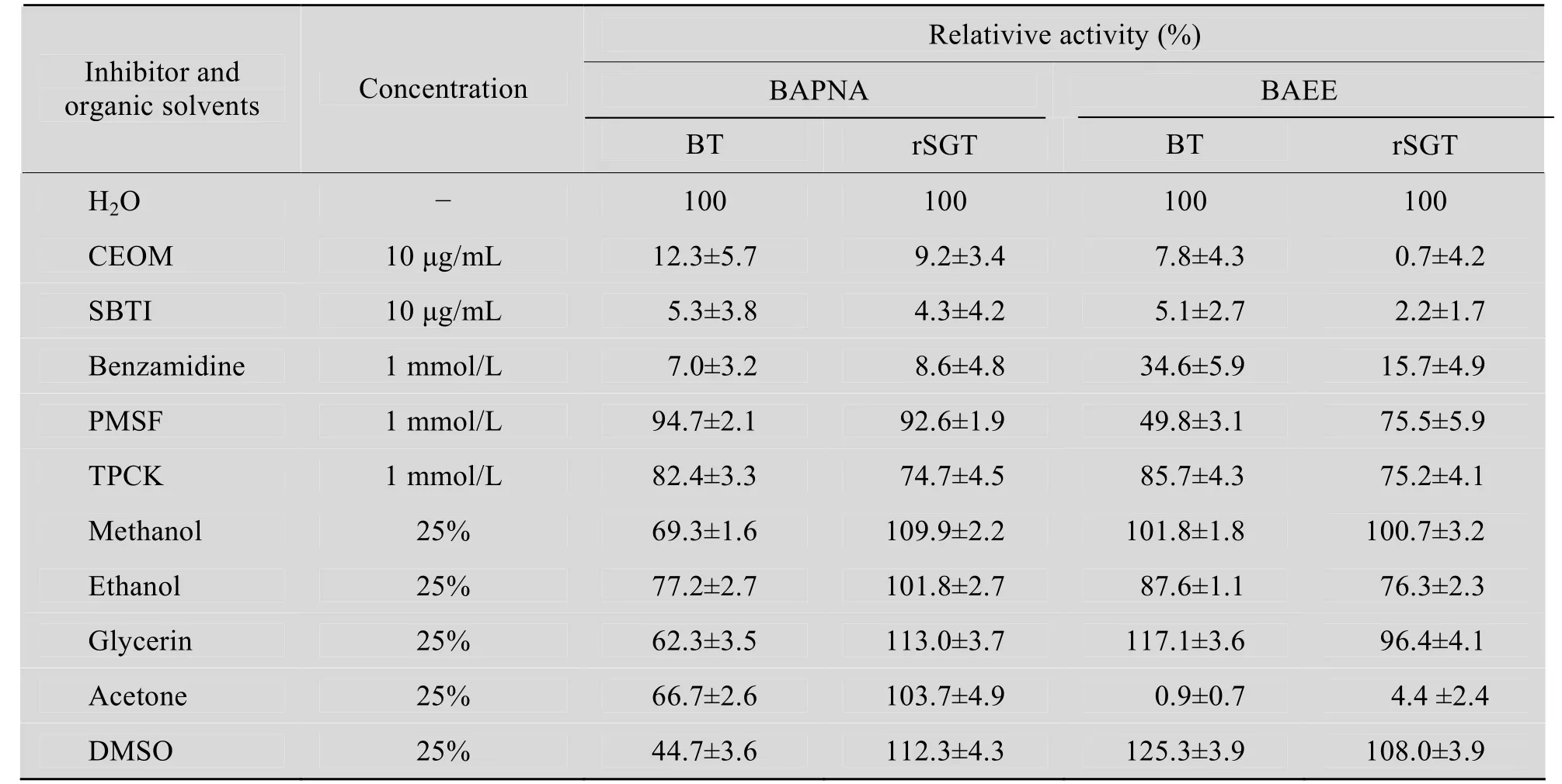
表5 在两种底物反应条件下各种抑制剂和有机溶剂对rSGT和BT活力的影响Table 5 Effect of different inhibitors and organic solvents on rSGT and BT activity with two kinds of substrates
3 讨论
本研究应用红霉素抗性基因启动子,以S. lividans为表达宿主,成功实现了胰蛋白酶的高效表达,在此基础上,系统对重组酶酶学性质进行了分析。关于灰色链霉菌胰蛋白酶的异源表达研究,国内外报道相对较少。在2003年,Page等将 sprT基因克隆至质粒 pWB980[20],并采用来自 B. subtilis的 sacB信号肽,在 B. subtilis WB700[21]中实现了其异源表达[10]。但是,在B. subtilis中的相关研究报道仅此一篇。此前,我们根据其叙述方法进行了构建并应用 B. subtilis WB700进行了表达验证。实验结果证实,采用B. subtilis并不能实现sprT的异源活性表达 (获得大量包涵体,数据未显示)。此外,我们也尝试了在E. coli 中表达,和B. subtilis中的结果类似,也没有得到活性表达。显然,具有3对二硫键灰色链霉菌胰蛋白酶并不适合在B. subtilis和E. coli中表达。近日,令桢民等应用真核表达系统,成功实现了灰色链霉菌胰蛋白酶在毕赤酵母Pichia pastoris中的活性表达[16],进一步说明胰蛋白酶中二硫键的形成可能是制约其活性表达的关键。
国内外关于胰蛋白酶的相关研究主要集中在 S. griseus以及同一属的其他菌株,如S. lividans。Kim等将含有sprT自身启动子 (Ptry)和编码基因的全基因片段连接到链霉菌克隆质粒pWHM3上,在S. lividans中实现了胰蛋白酶的表达,最高酶活达到0.74 U/mL[11]。而本研究选用pIJ86为表达质粒,同样以菌株 S. lividans TK24为宿主,采用 SELF发酵培养基,最高酶活达8.61 U/mL,比报道结果提高了11.6倍。pIJ86表达质粒携带红霉素抗性基因启动子 (PermE),该启动子来源于红色糖多孢菌的红霉素合成基因簇,位于ermE和eryC之间,是链霉菌组成型表达的强启动子。它有2个启动区域PermEⅠ和Ⅱ,含有2对相同方向的−10、−35区和 2个转录起始位点 (图 2)。而 Kim 等[6]所采用的 sprT自身启动子 (Ptry)只含有一个启动区域,其−35区与PermE Ⅱ的−35区相同,且Ptry的−35区和−10区保守序列的间隔也同PermEⅡ相似 (图2)。比较发现,与Ptry相比,PermE多出一个启动区域PermEⅠ,从而使启动子强度增加。此外,在S. griseus中,胰蛋白酶参与其生理生化的多种调节[22],sprT基因在转录水平上的表达调控复杂[23-24],因此,在同属的S. lividans中采用sprT基因的自身启动子同样不利于 sprT的表达。由此可见,以菌株S. lividans为表达宿主,启动子的强度是影响胰蛋白酶表达的重要因素。
此外,为进一步提高产量,本研究比较了5种链霉菌常用培养基,结果表明以R2YE和SELF为发酵培养基时酶活最高,分别为9.21 U/mL和8.61 U/mL。与其他 3种培养基相比,R2YE和SELF均含有少量游离氨基酸,且R2YE中还含有一些微量元素和缓冲盐成分。可见,其高效表达需要多种成分的相互协作,一些微量成分也是影响链霉菌菌丝生长以及高效表达的重要因素。
关于野生链霉菌胰蛋白酶 SGT酶学性质的报道较多,但对重组酶酶学性质则缺少系统详尽的报道。Page等对重组酶和野生酶的底物特异性进行了比较,发现两者降解精氨酸和赖氨酸的最佳比例均为4:1[10]。Kim等研究了温度及pH对野生酶和重组酶活力的影响,发现两者并无明显差别[15]。本文基于纯化的重组链霉菌胰蛋白酶(rSGT),对其酶活性质进行了系统的研究,结果表明其酶学性质与SGT以及BT在最适温度、最适pH以及抑制剂的影响等方面表现出极大的相似性[8,25-26]。但在耐酸性上,rSGT具有更大的潜力,在pH 5.0时还能保持60%以上的活力,而其他报道的胰蛋白酶在pH≤6.0时,稳定性就急剧下降[8,15,18,25]。同时,我们还测定了rSGT在两种常用底物下Km值,虽与报道的SGT相比,rSGT对两种底物的特异性都有所降低,但其对酰胺健底物的特异性仍远远高于 BT。此外,我们研究了不同金属离子对rSGT酰胺酶和酯酶活力的影响,基本与SGT的报道相符,但发现Zn2+对rSGT的酯酶活力有促进作用而对其酰胺酶活力有抑制作用。和其他胰蛋白酶相关报道不同[25],rSGT的酰胺酶活力在添加了终浓度为 25%的有机溶剂后没有受到抑制反而有所提高 (表5)。
综上所述,本研究结果为胰蛋白酶在食品、医药、皮革等诸多工业领域的应用提供了科学依据和选择思路,并为后期链霉菌胰蛋白酶分子水平上的突变和性质改造 (提高耐热性能)明确了方向。
[1]Walsh KA. Trypsinogens and trypsins of various species. Serine Proteases, 1970, 4: 41−63.
[2]Wahlby S, Zetterqvist O, Engstrom L. Reaction of two enzyme fractions from Streptomyces griseus protease with diisopropylphosphoro-fluoridate.Acta Chem Scand, 1965, 19: 1247−1248.
[3]Trop M, Birk Y. The trypsin-like enzyme from Streptomyces griseus (pronase). Biochem J, 1968,109: 475−476.
[4]Trop M, Birk Y. The specificity of proteinases from Streptomyces griseus (pronase). Biochem J, 1970,116: 19−25.
[5]Olafson RW, Jurasek L, Carpenter MR, et al.Amino acid sequence of Streptomyces griseus trypsin. Cyanogen bromide fragments and complete sequence. Biochemistry, 1975, 14: 1168−1177.
[6]Kim JC, Cha SH, Jeong ST, et al. Molecular cloning and nucleotide sequence of Streptomyces griseus trypsin gene. Biochem Biophys Res Commun, 1991, 181: 707−713.
[7]Stroud RM, Kossiakoff AA, Chambers JL.Mechanisms of zymogen activation. Annu Rev Biophys Bioeng, 1977, 6: 177−193.
[8]Olafsoni RW, Smillie LB. Enzymic and physicochemical properties of Streptomyces griseus trypsin. Biochemistry, 1975, 14: 1167−1172.
[9]Read RJ, James MN. Ref i ned crystal structure of Streptomyces griseus trypsin at 1.7 A˚ resolution. J Mol Biol, 1988, 200: 523−551.
[10]Page MJ, Wong SL, Hewitt J, et al. Engineering the primary substrate specificity of Streptomyces griseus trypsin. Biochemistry, 2003, 42: 9060−9066.
[11]Kim, Jong H, Soon KH. Overproduction of bacterial trypsin in Streptomyces-Optimization for Streptomyces griseus trypsin production by recombinant Streptomyces. Kor J Microbiol Biotechnol, 2008, 36: 28−33.
[12]Zhu GP, Zhang Y, Xu Y, et al. Overexpression and detection of the mutated glucose isomerase GIG138P and GIG138P-G247D in Streptomyces lividans. Chin J Biotech, 2002, 18(3): 304−307 (in Chinese).朱国萍, 张颖, 徐旸, 等. 葡萄糖异构酶突变体GIG138P和GIG138P-G247在变铅青链霉菌中的高效表达及检测. 生物工程学报, 2002, 18(3):304−307.
[13]Lee WS, Park CH, Byun SM. Streptomyces griseus trypsin is stabilized against autolysis by the cooperation of a salt bridge and cation-π interaction. J Biochem, 2004, 135: 93−99.
[14]Hopwood DA, Bibb MJ, Chater KF, et al .Genetic Manipulation of Streptomyces. A Laboratory Manual. Norwich: John Innes Foundation Press, 2000.
[15]Koo BJ, Kim JM, Byun SM, et al. Articles-optimal production conditions of Streptomyces griseus trypsin (SGT)in Streptomyces lividans. J Biochem Mol Biol, 1999, 32: 86−91.
[16]Ling ZM, Ma TB, Li JH, et al. Functional expression of trypsin from Streptomyces griseus by Pichia pastoris. J Ind Microbiol Biotechnol, 2012,39: 1651−1662.
[17]Chi WJ, Song JH, Oh EA, et al. Medium optimization and application of an aff i nity column chromatography for Streptomyces griseus trypsin production from the recombinant Streptomyces griseus. J Microbiol Biotechnol, 2009, 19:1191−1196.
[18]Wu ZQ, Jiang GL, Xiang P, et al. Anionic trypsin from North Pacific krill (Euphausia pacifica):purification and characterization. Int J Peptide Res Ther, 2008, 14: 113−120.
[19]Caldwell RA. Effect of calcium and phytic acid on the activation of trypsinogen and the stability of trypsin. J Agric Food Chem, 1992, 40: 43−46.
[20]Wu SC, Wong SL. Development of improved pUB110-based vectors for expression and secretion studies in Bacillus subtilis. J Biotechnol, 1999, 72:185−195.
[21]Wu SC, Yeung JC, Duan YJ, et al. Functional production and characterization of a fibrin-specif i c single-chain antibody fragment from Bacillus subtilis: effects of molecular chaperones and a wall-bound protease on antibody fragment production. Appl Environ Microbiol, 2002, 68:3261−3269.
[22]Horinouchi S. A microbial hormone, A-factor, as a master switch for morphological differentiation and secondary metabolism in Streptomyces griseus.Front Biosci, 2002, 7: 2045−2057.
[23]Kato JY, Chi WJ, Ohnishi Y, et al.Transcriptional control by A-factor of two trypsin genes in Streptomyces griseus. J Bacteriol, 2005,187: 286−295.
[24]Eun AO, Kim MS, Chi WJ, et al. Characterization of the sgtR1 and sgtR2 genes and their role in regulating expression of the sprT gene encoding Streptomyces griseus trypsin. FEMS Microbiol Lett, 2007, 276: 75−82.
[25]Outzen H, Berglund GI, Smalds AO, et al.Temperature and pH sensitivity of trypsins from Atlantic salmon (Salmo salar)in comparison with bovine and porcine trypsin. Comp Biochem Physiol, 1996, 115: 33−45.
[26]Sekizaki H, Itoh K, Murakami M, et al. Anionic trypsin from chum salmon: activity with p-amidinophenyl ester and comparison with bovine and Streptomyces griseus trypsins. Compar Biochem Physiol Part B, 2000, 127: 337−346.