短乳杆菌天然质粒分类
孙大庆,李洪飞,杨 健,宋大巍,3
(1.黑龙江八一农垦大学 国家杂粮工程技术研究中心,黑龙江 大庆 163319;2.黑龙江八一农垦大学 牡丹江食品与生物技术创新研究院,黑龙江 牡丹江 157000;3.黑龙江八一农垦大学食品学院,黑龙江 大庆 163319)
短乳杆菌是乳杆菌属重要的菌种之一,它们广泛分布于植物、动物及人体多种复杂环境中,是乳杆菌中环境适应能力很强的菌种之一。短乳杆菌具有发酵能力强、耐酸、产细菌素等多种优良表型[1-3],至今已广泛应用于食品、医疗和养殖等行业,与人们饮食、卫生和健康息息相关[4-6],因此开展短乳杆菌相关研究具有重要的经济和社会应用价值。
目前,NCBI的Genome数据库已收录60 个短乳杆菌天然质粒基因组序列,从质粒大小、数量和复制类型而言,短乳杆菌是乳杆菌属中携带天然质粒最丰富的菌种之一[7],这一现象表明,质粒在短乳杆菌生长、代谢、遗传和进化等生物学过程中可能扮演着特殊且重要的角色,此外,众多天然质粒的发现和测定为短乳杆菌基因工程改造和生物技术应用提供了必要的研究和材料基础。然而,目前对短乳杆菌天然质粒的了解和研究比较有限,现有研究主要集中于载体构建[8-10]和特殊功能质粒[11-12]。本研究以短乳杆菌天然质粒为研究对象,在原有质粒复制起始蛋白(replication initiation protein,Rep)进化树分类方法[13-14]基础上,进一步结合质粒基因组共线性分析方法,探索建立一种更加准确、有效的天然质粒的分类方法,从而为今后短乳杆菌天然质粒的科学分类和理性应用提供有益的探索和研究基础。
1 材料与方法
1.1 质粒基因组数据来源
短乳杆菌质粒基因组由NCBI Genome数据库下载获得,质粒详细信息见表1。质粒pC194、pMV158、pUCL287基因组登录号分别为NC_002013.1、NC_010096.1、X75607.1。质粒pAD1和pIP501基因组序列来自文献[15-16]。
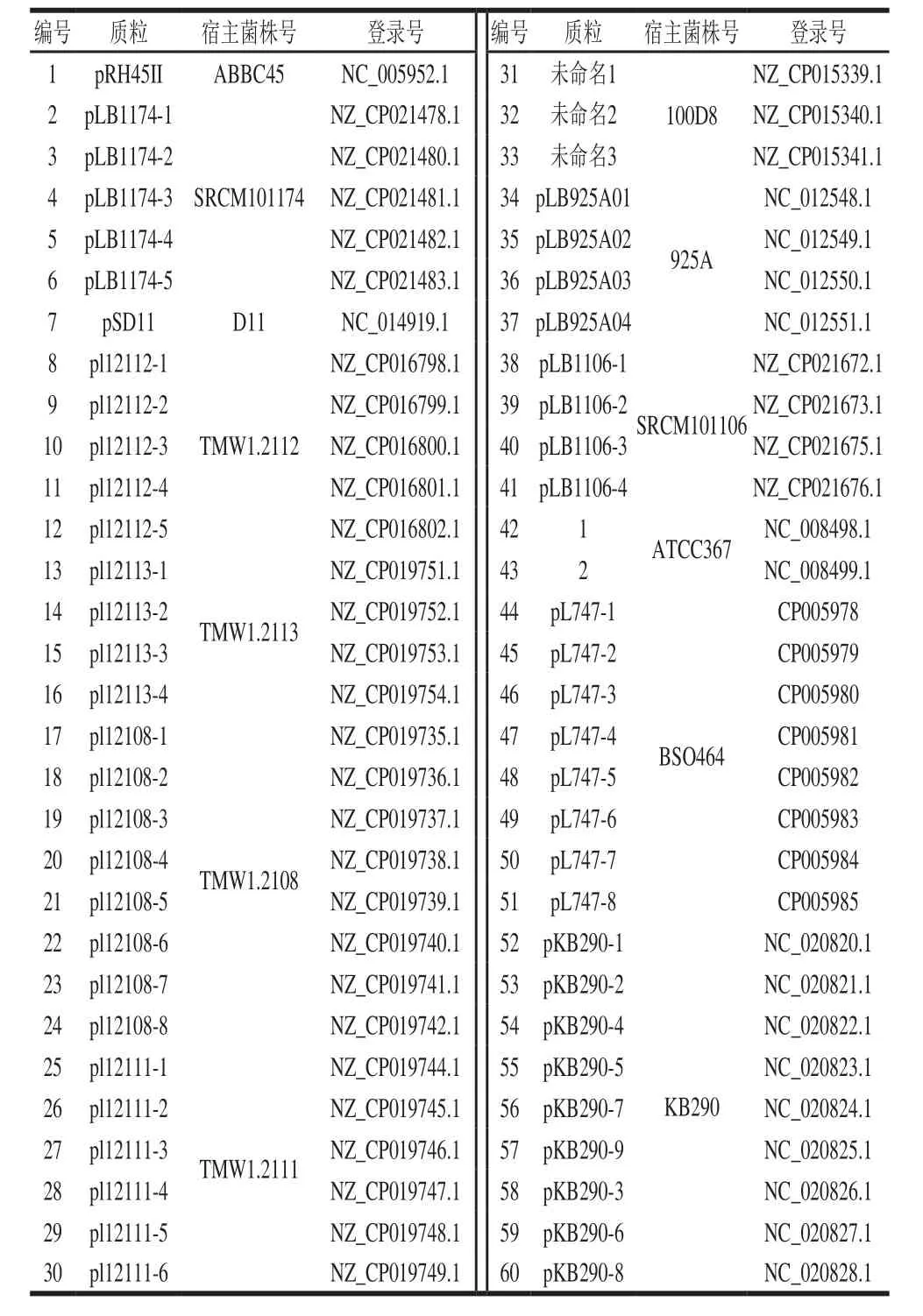
表1 短乳杆菌质粒信息Table 1 Information about plasmids in Lactobacillus brevis
1.2 质粒基因组序列的收集、整理和初步分析
筛查Genome数据库中所有短乳杆菌质粒基因组序列完整性和每个质粒Rep编码情况,对注释或疑似的Rep进行BLAST比对分析,鉴定其保守结构域,确定其所属蛋白家族。
1.3 方法
1.3.1 质粒Rep进化树构建及分类
利用MEGA 6.0软件[17]中Muscle法进行Rep氨基酸多序列比对,之后利用Neighbor-Joining法进行质粒Rep进化树构建。进化树检验采用Bootstrap法进行,自展值设定为1 000。
1.3.2 质粒基因组共线性分析及分类
利用Mauve 2.4.0软件[18]进行质粒基因组共线性分析,所有参数为默认设置。DNA多序列对比分析利用DNAMAN 5.2.2软件进行。DNA重复序列分析利用DNASTAR 7.1.0 GeneQuest软件包进行。
2 结果与分析
2.1 质粒基因组基本特征

表2 短乳杆菌质粒基因组和Rep基本特征Table 2 Basic characteristics of plasmid genome and Rep in Lactobacillus brevis
经检索、比对和统计分析,Genome数据库收录的60 个质粒,有9 个质粒基因组序列不完整或存在拼接错误,其余51 个质粒为完整基因组序列,完整质粒基因组特征及编码Rep情况见表2。由表2可知,短乳杆菌51 个完整质粒基因组大小为1.81~107.03 kb,GC含量为34.40%~45.63%,其中39 个质粒编码了Rep,35 个质粒含有1 个Rep(质粒pRH45II的Rep为本研究推定),4 个质粒含有2 个Rep,12 个质粒不含有已知Rep。根据Rep含有的保守结构域,所有Rep可划分为5 个Rep蛋白家族(表1)。
2.2 质粒Rep进化树分类
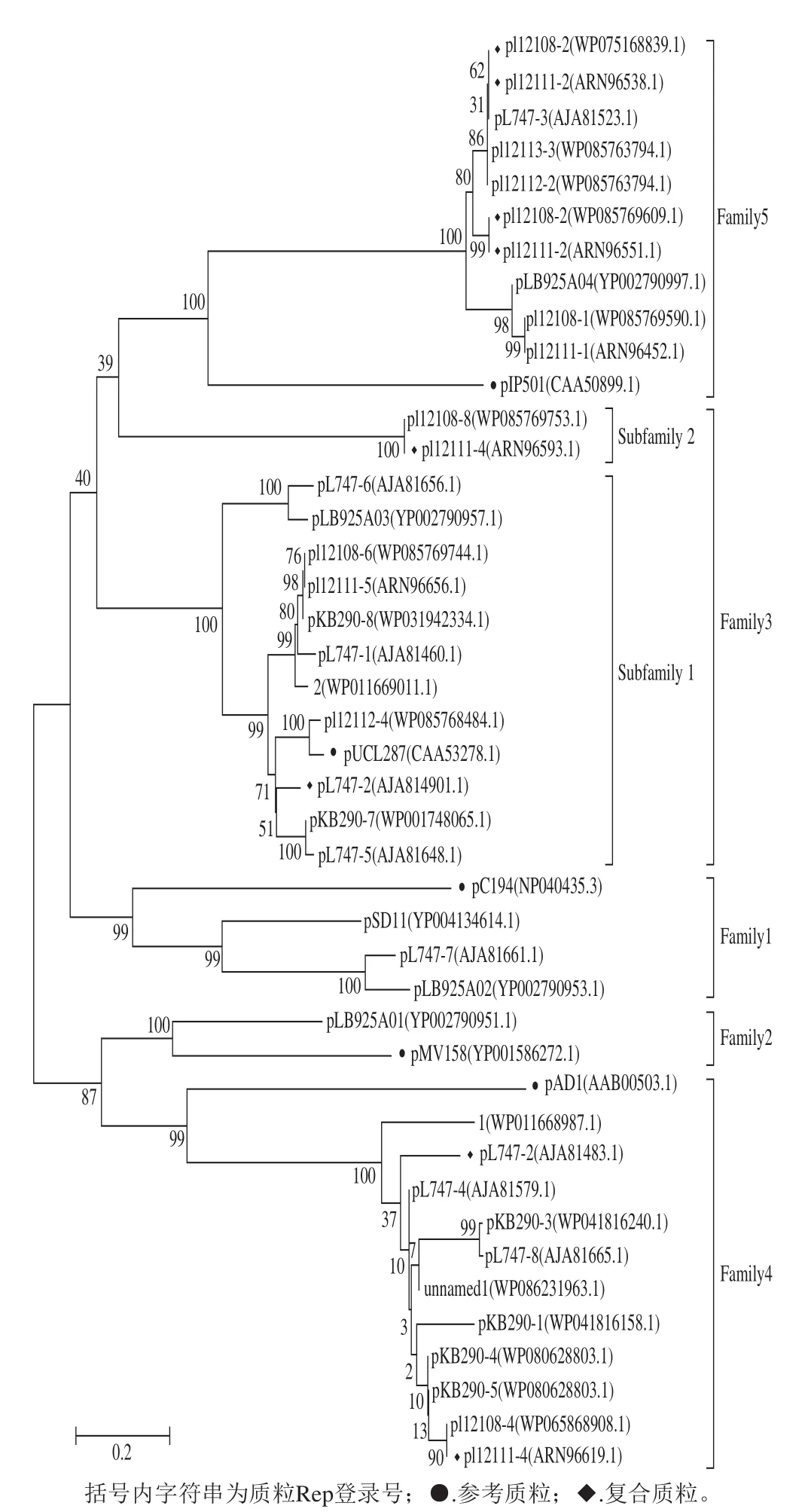
图1 短乳杆菌质粒Rep氨基酸序列进化树Fig. 1 Phylogenetic tree based on amino acid sequences of plasmid Rep in Lactobacillus brevis
基于Rep氨基酸序列同源性,构建了短乳杆菌质粒和5 个蛋白家族参考质粒Rep进化树(图1)。进化树没有包括假基因编码和推定的Rep,这些质粒将在后文论述。由图1可以看出,短乳杆菌34 个质粒编码的38 个Rep和5 个参考质粒编码的Rep,根据各自含有的保守结构域不同,能够明显聚类为5 个不同的质粒家族,因此短乳杆菌30 个含有1 个Rep质粒可以有效地划分为5 个类型,即5 个质粒家族。
家族3质粒pl12108-8和pl12111-4的Rep(WP085769753.1和ARN96593.1)虽然都含有Rep_3家族蛋白共有的保守结构域(pfam01051),但它们与该家族其他质粒Rep存在很远的进化距离和较低的自展值(40),进一步序列比对发现,这2 个Rep不仅序列较短,而且与该家族其他质粒Rep序列一致性较低,因此将家族3质粒划分为2 个分类亚家族。
由图1可以看出,短乳杆菌4 个复合质粒(2 个及以上复制子融合形成的质粒)的8 个Rep与各自同源Rep明显聚类。复合质粒pl12108-2和pl12111-2含有2 个家族5 Rep,且聚类在一个分支的2 个Rep氨基酸序列一致性均为100%,这表明两个复合质粒由2 个不同的家族5质粒融合形成,它们具有非常近的进化关系,由于融合前两个质粒都属于家族5,因此这两个复合质粒也属于家族5。由于复合质粒pL747-2和pl12111-4分别含有1 个家族3 Rep和1 个家族4 Rep,因此它们不能简单归属于一个家族,只能暂时归属于两个家族(家族3和4)。
综上可知,根据Rep进化树,32 个短乳杆菌质粒可以分类为5 个不同的质粒家族,2 个复合质粒pL747-2和pl12111-4同时归属于家族3和家族4,由参考质粒可知,家族1和2质粒属于滚环型复制质粒,家族3~5属于theta型复制质粒。
2.3 质粒基因组共线性分类
复制子是质粒复制和生存必不可少的保守区域,因此本实验利用基因组共线性分析结果进行质粒分类时,将是否具有同源性复制子作为分类的优先原则,其次根据质粒基因组共线性程度大小进行分类。根据质粒基因组的同源性和差异性,对短乳杆菌所有51 个质粒进行了基因组共线性分析,并按照上述分类原则进行质粒分类,结果见图2。
由图2可知,短乳杆菌质粒家族1含有3 个成员,它们都含有RCR质粒家族pC194同源复制子,该复制子由单链起点、rep和双链起点组成[19],是家族1质粒共有序列,因此可以作为该家族质粒分类的遗传标记。进一步序列分析显示,质粒pSD11和pLB925A02均编码一个高度同源的转移蛋白,这使质粒在一定条件下可以发生接合转移,而质粒pL747-7没有编码任何功能基因,因此家族1质粒可以进一步划分为2 个亚家族类群。
由图2可知,短乳杆菌质粒家族2只含有1 个成员,它与RCR质粒家族pMV158具有同源的复制子序列,该复制子由拷贝数调控基因、单链起点、rep和双链起点组成[20],是它们的共有序列,同样可以作为该家族质粒分类的遗传标记。
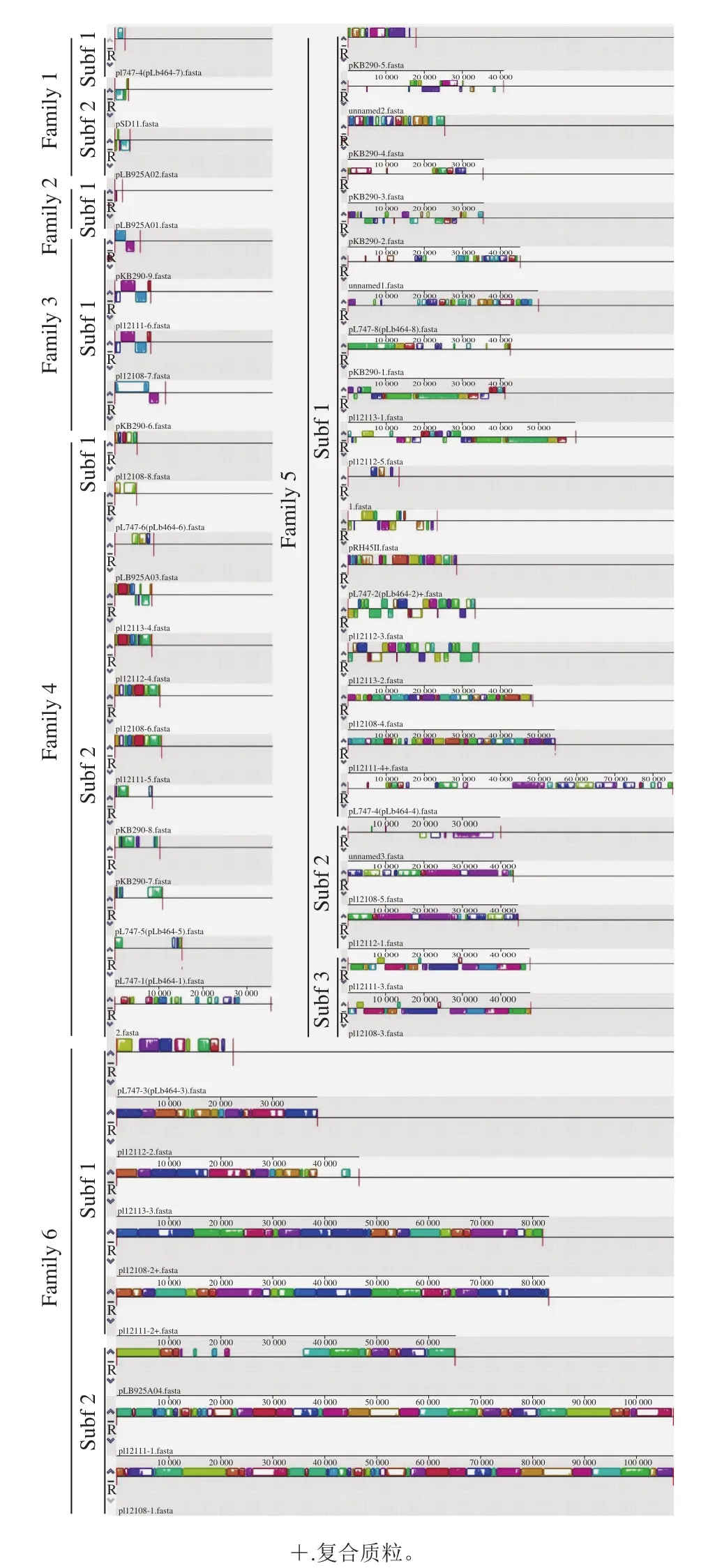
图2 短乳杆菌质粒基因组共线性Fig. 2 Plasmid genome colinearity in Lactobacillus brevis
由图2可知,短乳杆菌质粒家族3含有4 个成员,均未编码Rep,含有大于4 kb共有序列,共有序列注释了2 个共有基因,分别编码推定蛋白和移动蛋白。这些分析结果表明,家族3质粒均为可转移质粒,很可能属于完全依赖宿主酶系进行theta复制的质粒,如著名的ColE1家族质粒[21-23]。
由图2可知,短乳杆菌质粒家族4含有12 个成员,均含有theta复制质粒pUCL287[24-25]同源的Rep(属于Rep_3家族),需要说明的是,质粒pl12113-4 Rep注释为假基因,经序列比对,该基因提前出现终止密码子,因此编码的Rep不完整(290 aa)。家族4质粒基因组整体共线性较差,除了Rep编码序列,12 个质粒没有发现共有序列,因此Rep编码序列可以作为家族4质粒分类的遗传标记。由参考质粒pUCL287可知,rep基因上游重复序列(即复制起点)具有该家族复制子典型特征[26],因此对所有14 个Rep_3家族Rep编码序列进行了比对分析,分析结果见图3。由图3可知,家族4质粒复制起点可以分为2 种类型,其中11 个质粒与pUCL287具有相同或相近的复制起点(4×11 bp+4.5×22 bp)(图3A),而家族4质粒pl12108-8和复合质粒pl12111-4具有另外一种类型的复制起点(3.5×21 bp)(图3B),这一分析结果与Rep进化树(图1)对该家族质粒的分类结果一致,这进一步证明家族4质粒可以划分为2 个亚家族类型。
由图2可知,短乳杆菌质粒家族5含有23 个成员,包括15 个编码Rep(属于RepA_N家族[27-28])质粒和8 个非编码Rep质粒。根据基因组共线性和序列比对,家族5质粒可以进一步分为3 个亚家族。亚家族1含有18 个质粒,其中3 个质粒没有发现Rep,大多数质粒编码了接合转移相关基因,因此推测这些质粒起源于一个接合质粒。亚家族2含有3 个质粒,均为非编码Rep质粒,它们共有一个约20 kb噬菌体相关基因编码区域,因此推测它们可能起源于一个编码噬菌体质粒。亚家族3含有2 个同源性很高的质粒,它们没有编码Rep,与家族5其他质粒具有少量的同源性区域,但都编码的是一些未知蛋白。
由图2可知,短乳杆菌质粒家族6含有8 个成员,均含有theta复制质粒pIP501同源的Rep(属于RepR家族[29]),多数为大型质粒。根据基因组共线性差异,8 个质粒可以明显划分为2 个亚家族。此外,由基因组共线性可知,虽然家族6质粒普遍较大,编码了众多功能基因,但与家族4、5质粒相比,家族6质粒基因组普遍显示了良好共线性,这一结果表明,家族6质粒之间可能具有亲密的进化关系,并且/或者它们可能较少发生基因水平转移、转座重组等进化事件。

图3 短乳杆菌家族3质粒rep基因上游重复序列Fig. 3 Repeat sequences of rep gene flank of Family 3 plasmids in Lactobacillus brevis
3 讨 论
乳杆菌属菌种数量和菌种多样性是乳酸菌中最丰富的菌属[30],同时乳杆菌属携带的天然质粒数量和多样性也是乳酸菌中最丰富的菌属[31],乳杆菌属这两种特性是一种巧合,还是存在某种联系,目前还是未知的。为了探究质粒多样性与乳杆菌生理、遗传等生物多样性之间是否存在联系,本课题组对乳杆菌属中质粒数量最多的植物乳杆菌进行了质粒进化、分类和起源研究,建立了一种准确、有效的分析方法,揭示了植物乳杆菌天然质粒系统进化、分类和起源[13-14]。本研究以短乳杆菌(乳杆菌属中质粒多样性仅次于植物乳杆菌)天然质粒为研究对象,在以往质粒Rep进化树分类方法研究基础上,进一步结合质粒基因组共线性分析方法,建立了一种更加准确、有效的短乳杆菌天然质粒分类方法,清晰、准确的揭示了短乳杆菌天然质粒的分类关系,为今后短乳杆菌天然质粒科学分类和理性应用奠定了理论研究基础。
本研究经多序列比对和重复序列分析发现,短乳杆菌家族4质粒复制子可以分为明显不同的2 个亚家族类型(图3),亚型1质粒与参考质粒pUCL287复制子的重复序列相似,均由两组不同长度的串联重复序列组成,该特征由Benachour等[26]1997年首次鉴定,而亚型2质粒重复序列仅由一组串联重复序列组成,这在以往有关乳杆菌质粒的文献中未发现报道,因此这个新的Rep_3家族复制子是否具有复制功能仍需后续实验验证。此外,亚型1质粒复制子重复序列也并非完全一致,质粒pL747-2、pLB925A03和pL747-6与其他质粒重复序列也存在不同程度的差异性(图3A)。这些分析结果表明,短乳杆菌家族4质粒Rep及其结合位点(即重复序列)并非严格保守,显示出较丰富的多样性,因此短乳杆菌家族4质粒的复制起始机制很可能因此存在一定的灵活性和鲁棒性。
4 结 论
本研究通过质粒Rep进化树分析,将短乳杆菌34 个编码Rep质粒划分为5 个家族类型,之后通过质粒基因组共线性分析,将短乳杆菌所有51 个质粒划分为6 个家族类型,以及更详细的11 个亚家族类型,推测家族1、2质粒属于滚环型复制质粒,家族3~6质粒属于theta型复制质粒。基因组共线性质粒分类方法克服了Rep进化树分类方法对质粒编码Rep的依赖和不足,Rep进化树和基因组共线性两种分析方法对编码Rep质粒的分类结果一致,两种方法互为补充和证明,因此本研究表明,Rep进化树和基因组共线性相结合的分析方法可以准确、有效的分析和分类短乳杆菌天然质粒,该方法是短乳杆菌天然质粒系统分类的一种有效方法。
参考文献:
[1]GUO T, ZHANG L, XIN Y, et al. Oxygen-inducible conversion of lactate to acetate in heterofermentative Lactobacillus brevis ATCC367[J]. Applied and Environmental Microbiology, 2017, 83(21):1659. DOI:10.1128/AEM.01659-17.
[2]NODA M, MIYAUCHI R, DANSHIITSOODOL N, et al.Characterization and mutational analysis of a two-polypeptide bacteriocin produced by citrus lyo-derived Lactobacillus brevis 174A[J]. Biological and Pharmaceutical Bulletin, 2015, 38(12): 1902-1909. DOI:10.1248/bpb.b15-00505.
[3]BANERJEE S P, DORA K C, CHOWDHURY S. Detection,partial purification and characterization of bacteriocin produced by Lactobacillus brevis FPTLB3 isolated from freshwater fish[J]. Journal of Food Science and Technology, 2013, 50(1): 17-25. DOI:10.1007/s13197-011-0240-4.
[4]KIM S Y. Production of fermented kale juices with Lactobacillus strains and nutritional composition[J]. Preventive Nutrition and Food Science, 2017, 22(3): 231-236. DOI:10.3746/pnf.2017.22.3.231.
[5]OGAWA M, SAIKI A, MATSUI Y, et al. Effects of oral intake of heat-killed Lactobacillus brevis SBC8803 (SBL88™) on dry skin conditions: a randomized, double-blind, placebo-controlled study[J].Experimental and Therapeutic Medicine, 2016, 12(6): 3863-3872.DOI:10.3892/etm.2016.3862.
[6]ACOSTA M P, RUZAL S M, CORDO S M. S-layer proteins from Lactobacillus sp. inhibit bacterial infection by blockage of DC-SIGN cell receptor[J]. International Journal of Biological Macromolecules,2016, 92: 998-1005. DOI:10.1016/j.ijbiomac.2016.07.096.
[7]FUKAO M, OSHIMA K, MORITA H, et al. Genomic analysis by deep sequencing of the probiotic Lactobacillus brevis KB290 harboring nine plasmids reveals genomic stability[J]. PLoS One, 2013,8(3): 60521. DOI:10.1371/journal.pone.0060521.
[8]PARK J Y, JEONG S J, SA H D, et al. Construction of a shuttle vector based on the small cryptic plasmid pJY33 from Weissella cibaria 33[J]. Plasmid, 2015, 79: 30-36. DOI:10.1016/j.plasmid.2015.03.008.
[9]WADA T, NODA M, KASHIWABARA F, et al. Characterization of four plasmids harboured in a Lactobacillus brevis strain encoding a novel bacteriocin, brevicin 925A, and construction of a shuttle vector for lactic acid bacteria and Escherichia coli[J]. Microbiology, 2009,155(5): 1726-1737. DOI:10.1099/mic.0.022871-0.
[10]JEONG S J, PARK J Y, LEE H J, et al. Characterization of pFMBL1, a small cryptic plasmid isolated from Leuconostoc mesenteroides SY2[J]. Plasmid, 2007, 57(3): 314-323. DOI:10.1016/j.plasmid.2006.09.003.
[11]BERGSVEINSON J, ZIOLA B. Comparative genomic and plasmid analysis of beer-spoiling and non-beer-spoiling Lactobacillus brevis isolates[J]. Canadian Journal of Microbiology, 2017, 63(12): 970-983.DOI:10.1139/cjm-2017-0405.
[12]BERGSVEINSON J, BAECKER N, PITTET V, et al. Role of plasmids in Lactobacillus brevis BSO 464 hop tolerance and beer spoilage[J]. Applied and Environmental Microbiology, 2015, 81(4):1234-1241. DOI:10.1128/AEM.02870-14.
[13]孙大庆, 李洪飞, 宋大巍, 等. 植物乳杆菌天然质粒分类[J]. 食品科学, 2017, 38(12): 69-74. DOI:10.7506/spkx1002-6630-201712011.
[14]孙大庆, 李洪飞, 杨健. 植物乳杆菌编码复制起始蛋白天然质粒的系统进化分析[J]. 微生物学通报, 2017, 44(5): 1047-1055.DOI:10.13344/j.microbiol.china.160940.
[15]FRANCIA M V, HAAS W, WIRTH R, et al. Completion of the nucleotide sequence of the Enterococcus faecalis conjugative virulence plasmid pAD1 and identification of a second transfer origin[J].Plasmid, 2001, 46(2): 117-127. DOI:10.1006/plas.2001.1533.
[16]THOMPSON J K, COLLINS M A. Completed sequence of plasmid pIP501 and origin of spontaneous deletion derivatives[J]. Plasmid,2003, 50(1): 28-35. DOI:10.1016/S0147-619X(03)00042-8.
[17]TAMURA K, STECHER G, PETERSON D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197.
[18]DARLING A C E, MAU B, BLATTNER F R, et al. Mauve: multiple alignment of conserved genomic sequence with rearrangements[J].Genome Research, 2004, 14(7): 1394-1403. DOI:10.1101/gr.2289704.
[19]SEERY L T, NOLAN N C, SHARP P M, et al. Comparative analysis of the pC194 group of rolling circle plasmids[J]. Plasmid, 1993, 30(3):185-196. DOI:10.1006/plas.1993.1051.
[20]MOSCOSO M, ERITJA R, ESPINOSA M. Initiation of replication of plasmid pMV158: mechanisms of DNA strand-transfer reactions mediated by the initiator RepB protein[J]. Journal of Molecular Biology, 1997, 268(5): 840-856. DOI:10.1006/jmbi.1997.1012.
[21]WANG Z, YUAN Z, HENGGE U R. Processing of plasmid DNA with ColE1-like replication origin[J]. Plasmid, 2004, 51(3): 149-161.DOI:10.1016/j.plasmid.2003.12.002.
[22]CESARENI G, HELMER-CITTERICH M, CASTAGNOLI L. Control of ColE1 plasmid replication by antisense RNA[J]. Trends in Genetics,1991, 7(7): 230-235. DOI:10.1016/0168-9525(91)90370-6.
[23]JUNG Y H, LEE Y. RNases in ColE1 DNA metabolism[J]. Molecular Biology Reports, 1995, 22(2/3): 195-200. DOI:10.1007/BF00988728.
[24]BENACHOUR A, FRÈRE J, NOVEL G. pUCL287 Plasmid from Tetragenococcus halophila (Pediococcus halophilus) ATCC 33315 represents a new theta-type replicon family of lactic acid bacteria[J].FEMS Microbiology Letters, 1995, 128(2): 167-175. DOI:10.1111/j.1574-6968.1995.tb07518.x.
[25]BENACHOUR A, FRÈRE J, BOUTIBONNES P, et al.Characterization and replication mode determination of the minimal replicon of Tetragenococcus halophila ATCC33315 plasmid pUCL287[J]. Biochimie, 1995, 77(11): 868-874. DOI:10.1016/0300-9084(95)90005-5.
[26]BENACHOUR A, FRÈRE J, FLAHAUT S, et al. Molecular analysis of the replication region of the theta-replicating plasmid pUCL287 from Tetragenococcus (Pediococcus) halophilus ATCC33315[J].Molecular and General Genetics MGG, 1997, 255(5): 504-513.DOI:10.1007/s004380050523.
[27]WEAVER K E, KWONG S M, FIRTH N, et al. The RepA_N replicons of Gram-positive bacteria: a family of broadly distributed but narrow host range plasmids[J]. Plasmid, 2009, 61(2): 94-109.DOI:10.1016/j.plasmid.2008.11.004.
[28]SCHUMACHER M A, TONTHAT N K, KWONG S M, et al.Mechanism of staphylococcal multiresistance plasmid replication origin assembly by the RepA protein[J]. Proceedings of the National Academy of Sciences, 2014, 111(25): 9121-9126. DOI:10.1073/pnas.1406065111.
[29]BRANTL S. Antisense-RNA mediated control of plasmid replicationpIP501 revisited[J]. Plasmid, 2015, 78: 4-16. DOI:10.1016/j.plasmid.2014.07.004.
[30]SUN Z, HARRIS H M B, MCCANN A, et al. Expanding the biotechnology potential of Lactobacilli through comparative genomics of 213 strains and associated genera[J]. Nature Communications, 2015,6: 8322. DOI:10.1038/ncomms9322.
[31]孙大庆, 李洪飞, 宋大巍, 等. 乳杆菌属天然质粒研究进展[J]. 食品科学, 2015, 36(11): 251-255. DOI:10.7506/spkx1002-6630-201511047.
——以多重共线性内容为例

