CODEHOP RT-PCR方法在汉坦病毒基因型别鉴定和分子溯源中的应用
胡 群,马思杰,童淑梅
汉坦病毒(Hantavirus,HV)属于布尼亚病毒科(Bunyaviridae)汉坦病毒属(geneus hantavirus)[1]。根据汉坦病毒基因分子结构和抗原性的不同,将汉坦病毒至少分为34个血清型/基因型[2-3],包括汉滩型病毒(Hantaan Virus,HTNV),汉城型病毒(Seoul Virus,SEOV),普马拉型病毒(Puumala Virus,PUUV),多 不 拉 伐 型 病 毒 (Dobrava Virus,DOBV),图拉型病毒(Tula virus,TULV),索托帕拉雅型病毒(Thottapalayam virus,THOV)和安第斯型病毒(Andes virus,ANDV)等。对于汉坦病毒的基因分型目前主要采用的方法为通过设计各种基因型别病毒的特异性引物进行PCR扩增检测[4-5],但是这种方法,只能针对性的检测已知的某种或者几种病毒,对于存在的未知基因型别病毒,可能会漏检。随着PCR技术的发展,依据同一个病毒家族中共有的保守基因序列所设计的简并引物,可以实现对不同基因型别病毒的同时扩增检测。CODEHOP是一种最新的简并引物设计方法,解决了传统方法设计的简并引物灵敏度不高和特异性差的缺点,已被逐步得到应用[6-7]。本文探讨了针对汉坦病毒属病毒设计的一对CODEHOP简并引物,结合RTPCR技术所建立的汉坦病毒属CODEHOP RTPCR检测方法对汉坦病毒基因型别鉴定和分子溯源的应用效果。
1 材料和方法
1.1 病毒样品 汉滩型汉坦病毒(HTNV)阳性鼠肺样品DX0901,由本实验室在2009年大榭港区媒介监测捕获的社鼠中分离。汉城型汉坦病毒(SEOV)阳性鼠肺样品DX1101,由本实验室在2011年从入境集装箱中捕获的褐家鼠中分离。
1.2 试剂 Ribounclease inhibition、100bp ladder marker、琼脂糖均购自宝生物工作(大连)有限公司,病毒RNA提取试剂盒、PCR产物胶中回收试剂盒、一步法RT-PCR试剂盒购自德国Qiagen公司。
1.3 方法
1.3.1 病毒RNA提取 取约50mg鼠肺样品,研磨匀浆后参照Qiagen公司RNA提取试剂盒使用说明书提取病毒RNA,用40μL无RNA酶的超纯水溶解,于-80℃保存备用。
1.3.2 引物合成 从GenBank中下载不同基因型别汉坦病毒病毒L基因组片段蛋白质氨基酸序列,用CODEHOP引物专业设计软件设计引物,引物序列如下:上游引物 G1:5′GCA ACA GCA ACA TGG TTT car tay tay ac-3′;下游引物 G2:5′CTT CTT CAT TCAT ATT TCC ATG Car ncc ytt ytc-3′。引物由上海英骏生物工程公司合成。
1.3.3 CODEHOP RT-PCR反应体系 采用一步法RT-PCR试剂盒进行病毒模板目标基因片段扩增,反应体系(50μL):其中 RNase Free Water 19 μL ,5×RT-PCR Buffer 10μL,10mmol/L dNTP Mixture 2μL,Enzyme Mix 2μL,Ribonuclease Inhibitor 1μL,20μmol/L上游引物G1 4μL,下游引物G2 2μL,RNA模板10μL。反应程序为60℃1min,42℃10min,50℃30min,95℃15min反转录结束后,94℃30s,52℃30s,72℃1min,35个循环,72℃7min,4℃保存。
1.3.3 扩增产物序列分析 取5μL PCR扩增产物用1.5%的琼脂糖凝胶电泳,凝胶成像系统观察结果。剩余扩增产物送上海英俊生物工程公司进行测序,测序后得到的片段序列登录NCBI进行同源性比对,判定基因型别。选择在GenBank上已经公开发表的11种基因型别的35个汉坦病毒株,见表1,利用Mega5.0软件构建系统发生树,并计算遗传距离。

表1 研究中使用的汉坦病毒毒株及来源Tab.1 Hantavirus strains and their origination in research
2 结 果
2.1 PCR产物电泳分析结果 汉滩病毒(HTNV)DX0901和汉城病毒(SEOV)DX1101,经 CODEHOP RT-PCR扩增后得到的电泳结果显示(图1),均获得预期大小的目的片段扩增产物(478bp)。
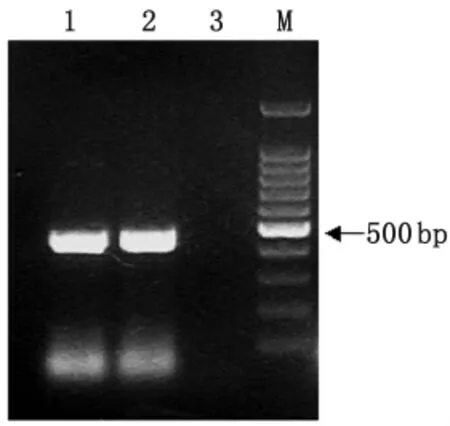
图1 汉坦病毒PCR产物电泳图1:汉滩病毒DX0901扩增产物;2汉城病毒DX1101扩增产物;3:阴性对照 M:100bp ladder markerFig.1 Electrophoresis of hantavirus PCR products1:PCR product of hantaan virus DX0901;2:PCR product of Seoul virus DX1101;3:Negative control;4:100bp DNA ladder marker
2.2 PCR产物序列分析结果 DX0901,DX1101病毒扩增产物测序后,用软件Mega 5.0进行分析,去除引物部分序列,均获得大小415bp的序列片段(表2),通过Blast检索GenBank进行同源性比对。结果显示,病毒株DX0901与汉滩型病毒Nc167同源性最为接近,达87%。病毒株DX1101与汉城型病毒ZT10,ZT71,Z37同源性最为接近,均为95%。
2.3 汉坦病毒基因进化分析 以不同汉坦病毒株L基因G1G2片段核苷酸序列为依据,构建的系统进化树见图2所示,汉坦病毒DX0901与汉滩型(HTN)病毒株Nc167位于同一分支,DX1101与5株汉城型病毒株处于同一分支。从软件计算的遗传距离来看(表3),DX0901与Nc167的遗传距离最为接近值为0.142,DX1101与ZT10,L99,Z37的遗传距离最为接近值为0.049。
3 讨 论
CODEHOP方法所设计的引物称之为一致简并杂合寡核苷酸引物(consensus-degenerate hybrid oligonucleotide primer,CODEHOP),该引物由3′端简并的核心区和5′端为非简并夹板区2个部分组成,与常规方法设计的简并引物相比具有简并度低、退火温度高、特异性更强等优势,目前已经被应用于克隆未知病毒或基因的检测[8-11]。本研究团队已经将该CODEHOP应用于汉坦病毒属病毒简并引物的设计,将所获得的CODEHOP引物与RT-PCR相结合,建立了汉坦病毒属CODEHOP RT-PCR检测体系,可以实现对汉坦病毒属内已知病毒和未知病毒的检测[12]。

表2 2个病毒PCR产物测序结果Tab.2 Sequences of PCR products of 2viruses
本文证实了所建立的CODEHOP RT-PCR方法可以对大榭港区分离的两个不同基因型别的汉坦病毒进行扩增,并获得目的片段大小(478bp)的扩增产物。通过电泳进行观察,可以用于判定样品感染病毒情况。将扩增产物序列登录NCBI进行Blast同源性比对,可以进行基因型别鉴定,CODEHOP RT-PCR方法所获得的型别鉴定结果与已知的结果一致。用 Mega 5.0软件建立系统进化树,可以获得两种汉坦病毒与GenBank上公布的已知汉坦病毒株的亲源关系方面的信息。

表3 汉坦病毒DX0901DX1101与不同基因型别汉坦病毒遗传距离Tab.3 Genetic distance of the hantavirus DX0901and DX1101with different hantavirus strain
本文从GenBank里选择汉坦病毒属的11种基因型别的35个汉坦病毒株进行了相应L基因核序列的分析,发现都存在G1,G2引物的结合位点,因此理论上这些型别的汉坦病毒均可以通过本文建立的CODEHOP RT-PCR体系进行扩增,进而实现对不同汉坦病毒的基因型别鉴定和分子溯源。
[1]Plyusnin A,Vapalahti O,Vaheri A.Hantaviruses:genome structure,expression and evolution[J].J General Virol,1996,77 (11):2677-2687.DOI:10.1099/0022-1317-77-11-2677
[2]Zhou JH,Zhang HL.Research progress on Hantavirus type and pathogenic[J].Int J Virol,2008,15(2):37-42.(in Chinese)周济华,张海林.汉坦病毒分型及致病性的研究进展[J].国际病毒学杂志,2008,15(2):37-42
[3]Jonsson CB,Moraes Figueiredo LT,Vapalahti O.A global perspective on hantavirus ecology,epidemiology,and disease[J].Clin Microbiol Rev,2010,23(4):412-441.DOI:10.1128/CMR.00062-09
[4]Guo WP,Lin XD,Wang W,etal.A new subtype of Thottapalayam virus carried by the Asian house shrew (Suncus murinus)in China[J].Infect Gen Evol,2011,11(8):1862-1867.DOI:10.1016/j.meegid.2011.07.013
[5]Wu SL,Yan YS,Li SQ,etal.Genotyping and DNA sequence analysis of Hantaviruses[J].Chin J Zoonoses,2005,21(9):804-808.(in Chinese)吴守丽,严延生,李世清,等.汉坦病毒基因分型方法的建立及其核苷酸序列特征分析[J].中国人兽共患病杂志,2005,21(9):804-808.
[6]Rose TM,Henikoff JG,Henikoff S,etal.CODEHOP(Consensus-Degenerate Hybrid Oligonucleotide Primer)PCR primer design[J].Nucleic Acids Res,2003,31(13):3763-3766.DOI:10.1093/nar/gkg524
[7]Rose TM.CODEHOP-mediated PCR-A powerful technique for the identification and characterization of viral genomes[J].Virol J,2005,2:20.DOI:10.1093/nar/gkp379
[8]Hermouet S,Sutton CA,Rose TM,etal.Qualitative and quantitative analysis of human herpesviruses in chronic and acute B cell lymphocytic leukemia and in multiple myeloma[J].Leukemia,2003,17(1):185-195.DOI:10.1038/sj.leu.2402748
[9]Watanabe S,Ueda N,Iha K,etal.Detection of a new bat gammaherpesvirus in the Philippines[J].Virus Genes,2009,39:90-93.DOI:10.1007/s11262-009-0368-8
[10]Zlateva KT,Crusio KM,Leontovich AM,etal.Design and validation of consensus-degenerate hybrid oligonucleotid primers for broad and sensitive detection of corona-and toroviruses[J].J Virol Methods,2011,177(2):174-183.DOI:10.1016/j.jviromet.2011.08.005
[11]Baines JE,McGovern RM,Persing D,etal.Consensus-degenerate hybrid oligonucleotide primers(CODEHOP)for the detection of novel papillomaviruses and their application to esophageal and tonsillar carcinomas[J].J Virol Methods,2005,123(1):81-87.
[12]Hu Q,Ma SJ,Wang J,etal.Development of CODEHOP RTPCR to detect hantavirus[J].J Pathog Biol,2012,7(6):414-417.(in Chinese)胡群,马思杰,王静,等.汉坦病毒群CODEHOP RT-PCR检测方法的建立[J].中国病原生物学杂志,2012,7(6):414-417.

