基于主元分析与近邻距离的特征基因选择与去噪
吕江婷,陈少斌,黄宴委
(福州大学电气工程与自动化学院,福建福州 350116)
0 引言
基因芯片技术对于在基因级别上研究疾病的发病机理、肿瘤的诊断及分类等具有重要的应用价值[1].由于基因芯片数据存在样本数量小、基因维数高以及样本噪声大等问题,增加了肿瘤诊断的难度,并且其中还有大量冗余信息,不仅降低了分类器的性能,还增大了算法的复杂度.因此,应用特征选择及去噪方法挑选出具有稳定高效分类性能的特征基因十分必要[2-3].
传统的聚类和分类方法直接对基因表达数据矩阵进行分析,往往存在维数灾难问题,因而首先要对其进行降维.主元分析(PCA)是一种经典的降维方法,被广泛应用于多元数据分析中,同时也成为基因选择的一种有用工具.迄今为止,人们已经提出很多基于PCA的基因选择或特征提取方法来对基因表达数据进行分析.如:文献[4]直接将Krzanowski[5]基于主成分的变量选择方法应用于基因选择问题;文献[6]提出一种融合PCA与线性判别分析(LDA)的鉴别主成分分析方法来对基因表达数据进行特征提取;文献[7]提出一种基于PCA主元的内积值选择成对特征基因的方法,结合SVM 分类器对基因表达数据进行分类.但这些方法仅考虑了基因变量的冗余及噪声,而在数据样本的噪声较大时,无法获得稳定高效的分类精度.在数据挖掘中,基于k-近邻距离(k-DNN)的样本集去噪是一种高效的非参数噪声监测方法[8-9],它能快速有效地检测出样本集中的野值点噪声.由此,本文提出一种基于PCA+k-DNN的特征基因选择及去噪方法.
1 基于PCA和k-DNN的特征基因选择及去噪
1.1 主元分析(PCA)
设样本数据矩阵为X=[x1,…,xm]∈Rn×m,其中,m为基因个数,n为样本个数,xi∈Rn×1为n个样本中基因i的表达值.PCA的目的是通过协方差矩阵C,寻找一个新的投影轴.

C可转换为一个有m个特征值λi(1≤i≤m)的对角阵,并得到其对应的特征向量Pi∈Rm×1(1≤i≤m).按降序排列特征值,选出前q个主元使相应特征值满足:

其中:β为一个设定的阈值.β值越大,相应q就越大.当1≤i≤q时,主元Pi∈Rm×1代表具有最大方差的方向,其余的Pi在q≤i≤m时则被视为冗余.
主元P=[P1,…,Pq]选定后,通过公式(3)计算主成分t:

其中:x∈Rm×1为原始变量的向量,t∈Rq×1为相应的主成分.主成分t是所有原始基因变量的线性组合,要使基因个数减少还需进行基因选择.
1.2 特征基因选择
若PCA模型有q个主元,将每个主元Pi(1≤i≤q)改写成为:

其中:系数pi,j表示第i个主元中第j个基因的得分.pi,j越大,则第i个主元中第j个基因越重要.
定义基因的贡献率为:

其中:1≤j≤m,gcj值越大,基因j越重要.按降序排列gcj,选出gcj值大的基因,即为特征基因(FG).
1.3 k-近邻距离去噪
定义(k-近邻距离)给定m维样本空间Rm和空间点xi,J(i)为距离点xi最近的前k个点的集合,则点xi的k-近邻距离d(i)定义为:

其中:N为样本个数,dij表示点xi和点xj之间的欧氏距离.
利用k-近邻距离模型,样本i的类内距离dW(i)和类间距离dB(i)可以通过下式求出:
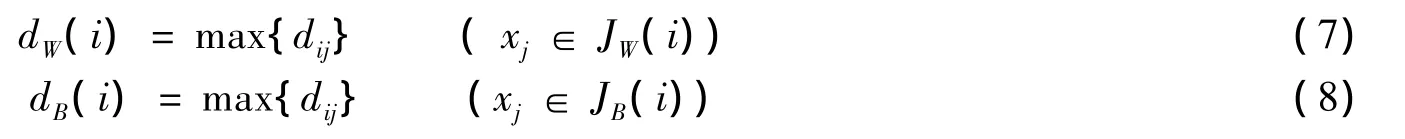
其中:JW(i)为样本i的k个同类最近邻样本的集合;JB(i)为样本i的k个异类最近邻样本的集合.
若样本i的类内距离大于其类间距离,即:dW(i)>dB(i),则为野值噪声点.本文利用k-DNN方法来消除FG集合中的野值噪声样本,来获得稳定高效的分类精度.
2 实验及结果分析
为了验证所提出的特征基因选择及去噪方法的性能,分别对表1中的三个数据集:Leukemia、Prostate Cancer及 Colon Cancer(来自 http://datam.i2r.a-star.edu.sg/datasets/krbd 和 http://www.biolab.si/supp/bi-cancer/projections)进行测试,并利用线性支持向量机来评估特征基因的分类性能.
Leukemia数据集由ALL与AML两类样本组成,每个样本包含7 129个基因数据.其训练集有38个样本(27个ALL类和11个AML类),测试集有34个样本(20个ALL类和14个AML类).使用训练集建立相应的PCA模型,设定阈值β=0.95,计算出主元个数q=16,由式(5)计算各基因贡献率的大小,选择排列靠前的21个基因为FG(见表2).

表1 数据集的说明Tab.1 Descriptions of the datasets

表2 各数据集选出的FGTab.2 The selected FGs of the datasets
与之类似的,Prostate Cancer数据集由Prostate Cancer组织与正常组织两类样本组成,每个样本包含12 600个基因.其训练集有102个样本(52个Prostate Cancer组织和50个正常组织),测试集有34个样本(25个Prostate Cancer组织和9个正常组织).使用训练集建立相应的PCA模型,设定阈值β=0.90,计算出主元个数q=36,由式(5)计算各基因贡献率的大小,选择排列靠前的18个基因为FG(见表2).
Colon Cancer数据集包含62个组织样本(40个Colon Cancer组织样本和22个正常组织样本),每个样本包含2000个基因.由于此样本集未拆分为训练集与测试集,因此将所有样本用于基因选择过程.首先,建立PCA模型,设定阈值β=0.95,计算出主元个数q=26,由式(5)计算各基因贡献率的大小,选择排列靠前的14个基因为FG(见表2).
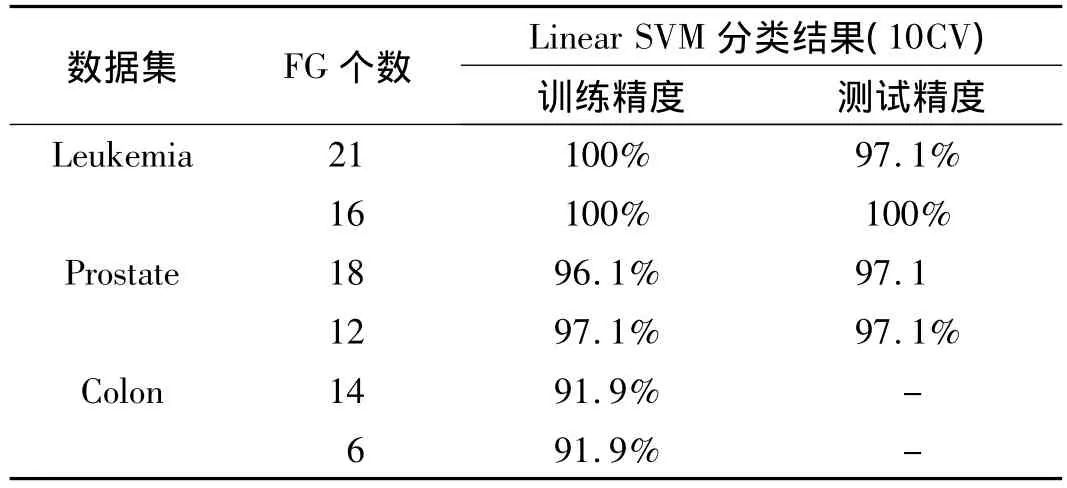
表3 线性SVM分类结果Tab.3 Results by the Linear SVM classifer
利用Linear SVM分类器来分别验证以上3类FG的分类性能,SVM参数为10CV的分类结果如表3所示,从FG集合中选择合适的子集,不仅可降低数据量,还可提高分类精度.
对选出的FG做筛选时发现,由于野值噪声样本的存在,严重影响了FG分类精度的提高.以Leukemia数据集为例,从已得到的21个FG集合中选择12个基因(编号为:M31523,U05259,U22376,M92287,L47738,M31303,X74262,HG1612,X59417,Z15115,J05243,D38073)来进行分类性能测试.
在Linear SVM分类器参数为10CV时,训练集精度为97.3%,测试集精度为100%.利用k-DNN方法对训练集进行野值去噪,检测出第12个ALL类样本为野值噪声点.从训练集中删除该样本,得到去噪后的训练集.再利用Linear SVM分类器对去噪后的训练集及原始测试集进行分类,结果如表4所示,删除野值样本后,训练集的分类精度提升为100%,测试集的分类精度仍保持100%.表明基于k-DNN的去噪方法,可以进一步提升FG的分类性能,获得更加稳定高效的分类精度.

表4 k-DNN去噪结果对比Tab.4 Results comparison after denoising by k-DNN
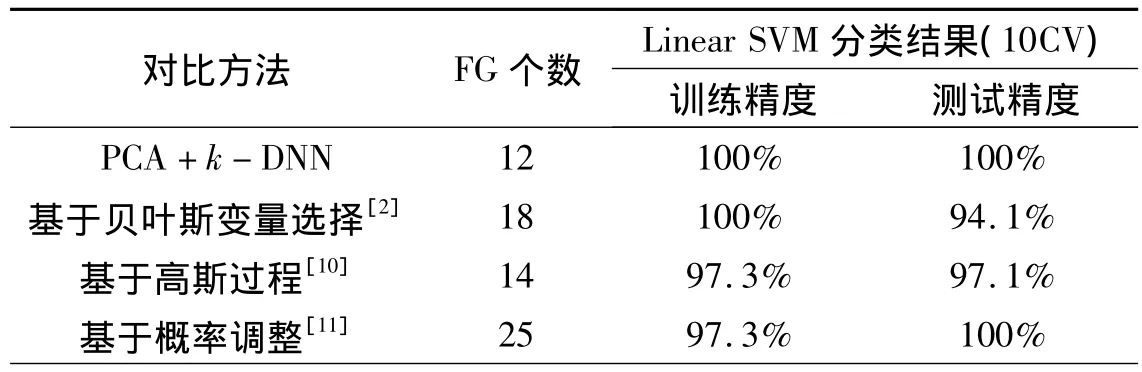
表5 不同方法所得FG分类结果对比Tab.5 Classification results comparison for FGs obtained by different approaches
应用Linear SVM分类器,对比本文及其他几种不同特征基因选择方法得到的FG的分类性能.实验结果如表5所示.由表5可知:基于PCA+k-DNN方法进行特征基因选择及去噪,再用SVM分类器对Leukemia样本数据的12个基因进行分类,训练集及测试集精度均为100%;基于贝叶斯变量选择的基因选择方法得到18个基因,训练集精度为100%,测试集精度为94.1%;基于高斯过程发现一致基因表达模式的基因选择方法得到的14个基因,训练集精度为97.3%,测试集精度为97.1%;基于概率调整的基因选择方法得到25个基因,训练集精度为97.3%,测试集精度为100%.
由此可见,提出的基于PCA+k-DNN的特征基因选择及去噪方法,从基因芯片数据中为SVM分类提供了重要信息,不仅简单易实现,还可使选得的FG达到稳定高效的分类精度.
3 结论
提出一种基于PCA+k-DNN的特征基因选择及去噪方法.首先,利用主元分析法获取低维投影空间中的模式特征,依据各个基因贡献率大小排序,选择贡献率大的基因为特征基因;进而利用k-DNN方法消除野值噪声以获得稳定高效的分类精度.该方法既解决了高维小样本问题,避免了维数灾难问题,又结合了样本集去噪,降维的同时,保证了特征基因获得稳定高效的分类精度.实验结果表明:利用该方法进行特征基因选择及去噪,不仅简单易实现,且稳定高效.实验中还发现,从FG集合中选择合适的子集不仅可降低数据量,还可提高分类精度.如何选取最佳的FG子集是今后研究的内容.
[1]刘全金,李颖新,阮晓钢.基于BP网络灵敏度分析的肿瘤亚型分类特征基因选取[J].中国生物医学工程学报,2008,27(5):710-715.
[2]Yang Ai-jun,Song Xin-yuan.Bayesian variable selection for disease classifcation using gene expression data[J].Bioinformation,2010,26(2):215-222.
[3]游伟,李树涛,谭明奎.基于SVM-RFE-SFS的基因选择方法[J].中国生物医学工程学报,2010,29(1):93-99.
[4]Wang An-tai,Gehan E A.Gene selection for microarray data analysis using principal component analysis[J].Statistics in Medicine,2005,24(13):2 069-2 087.
[5]Krzanowski W J.Selection of variables to preserve multivariate data structure,using principal components[J].Applied Statistics,1987,36(1):22–33.
[6]廖海斌,徐鸿章.基于鉴别主成分分析的基因表达数据特征提取[J].燕山大学学报,2010,34(5):426-430.
[7]Sohn K,Lim S H.A new gene selection method based on PCA for molecular classification[C]//Fourth International Conference on Fuzzy System and Knowledge Discovery.Haikou:[s.n.],2007,4:275-279.
[8]Ghoting A,Parthasarathy S,Otey M E.Fast mining of distance-based outliers in high-dimensional datasets[J].Data Mining and Knowledge Discovery,2008,16(3):349-364.
[9]陈圣兵,李龙澍.基于近邻距离的大规模样本集去噪及减样[J].计算机工程,2011,37(5):184-186.
[10]Chu Wei,Ghahramani Z,Falciani F,et al.Biomarker discovery in microarray gene expression data with Gaussian processes[J].Bioinformatics,2005,21(16):3 385-3 393.
[11]Wang Hong-qiang,Huang De-shuang.Regulation probability method for gene selection[J].Pattern Recognition Letters,2006,27(2):116-122.

