A组轮状病毒的基因组及其蛋白研究进展
杨 盟,周建国,马海利
(1.山西农业大学动物科技学院,太谷 030801;2.河北省迁安市畜牧水产技术中心,迁安 064400)
轮状病毒(Rotavirus,RV)是呼肠孤病毒科(Reoviridae)、轮状病毒属(Rotavirus)的典型代表。自1963年被Adams等[1]证实可引起小鼠腹泻以来,现已成为引起哺乳类和禽类动物病毒性腹泻的主要病原微生物之一。轮状病毒感染引起的疾病广泛分布于世界各地,病人、患病动物和隐形感染的动物为主要传染源,粪—口途径是RV感染的最主要传播途径。
完整的RV颗粒为球形,直径大约60~80 nm,也有直径为50 nm左右的缺损病毒。RV无包膜,具有多层核衣壳形态学特征,同时还具有成节段的双链RNA基因组和在宿主细胞浆内复制等理化特性。RV根据其基因组结构和抗原性分为A~G等7个组。A组、B组、C组可引起人类和动物感染,而D组、E组、F组、G组主要引起动物感染[2]。其中A组RV在人类和动物中感染最为普遍。
1 基因组结构
轮状病毒的基因组为dsRNA,全长介于0.6~3.3 kb之间。基因组由11个片段组成,编码6种结构蛋白和6种非结构蛋白。根据基因在聚丙烯酰胺凝胶电泳上的位置,由大到小依次命名为基因1至基因11[3]。A组RV有特异性电泳图形,包括4个高分子量基因,5个中分子量基因和2个小分子量基因,此为A组RV特异性图形。但这不能作为RV分组的绝对标准[4]。
组成RV的每个基因片段具有共同特征,即5′末端和3′末端序列高度保守。在5′末端有一个鸟嘌呤核苷酸,紧随其后为5′末端保守序列,再后为开放读码框,编码一种蛋白质产物,在终止密码之后为3′末端保守序列[5]。另外,RV基因中富含A和T,其比值高达65%左右,这也是与大多数病毒的相异之处[6]。
RV在同一组内可以进行基因重组,这种现象曾经在许多国家及地区发生。在斯洛文尼亚,Steyer等[7]收集猪、牛和人的粪便,采用RT-PCR对A组轮状病毒进行测序分析发现,在人轮状病毒G3P[6]基 因 型 SI-MB6株 中 编 码 VP6、VP7、 VP8*和NSP4蛋白的基因核苷酸序列和猪的核苷酸序列具有同一性。在印度东部,Mukherjee等[8]对3岁儿童感染的A组轮状病毒G9P[6]株进行全基因序列分析发现,VP1~VP3、NSP1~NSP3和NSP5与猪轮状病毒P[6]株高度相似。Chitambar等[9]通过对罕见的G1P轮状病毒株研究发现,人类VP7/VP6基因和猪VP4/NSP4基因之间发生重组。在厄瓜多尔[10]、韩国[11]、中国[12]、中国台湾[13]等地也有A组轮状病毒组内重组的报道。此外,RV尚可在基因内重组。目前尚无组与组之间发生基因重组的报道。
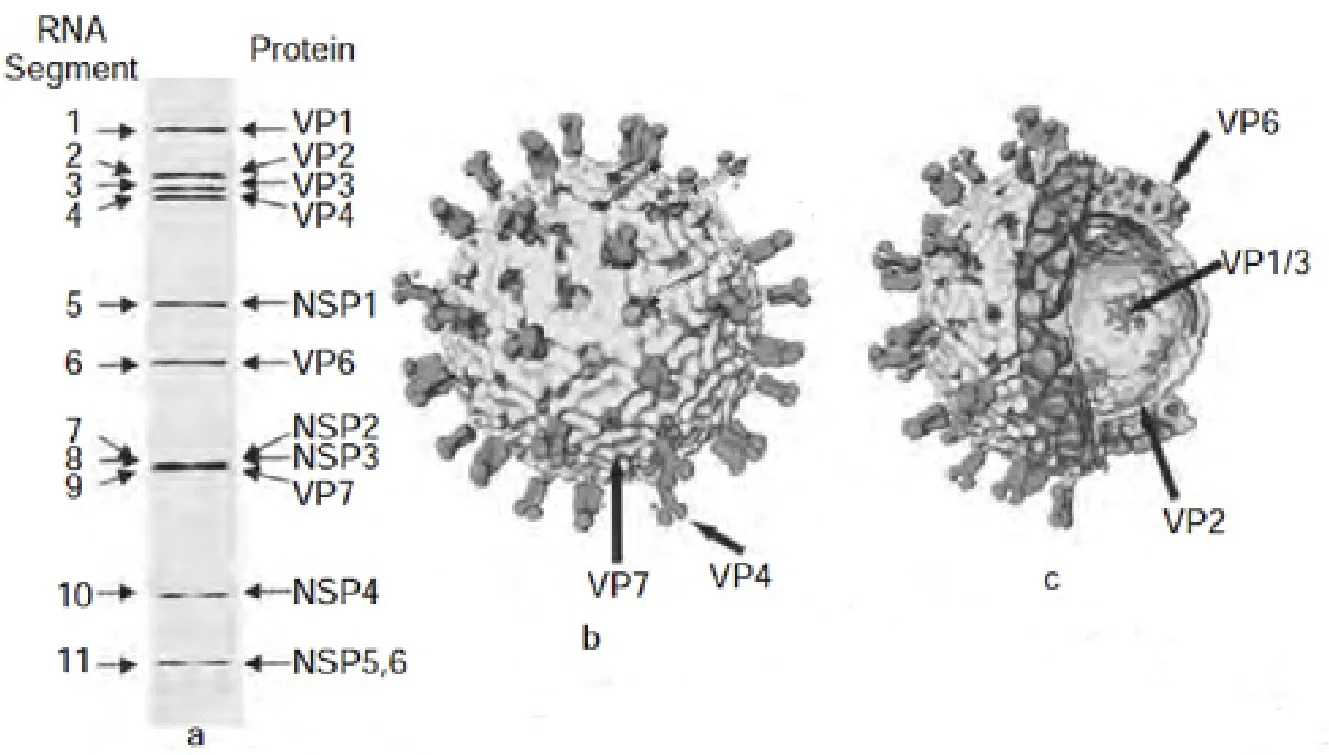
图1 轮状病毒结构图Fig.1 Structural organization of Rotavirus
2 主要的病毒蛋白
RV有11个基因片段,编码6种结构蛋白(VP1~VP4和VP6及VP7)和6种非结构蛋白(NSP1~NSP6)。其中VP1、VP2、VP3为病毒核心的结构成分,在基因转录和复制中起重要作用;VP4、VP6、VP7是病毒衣壳的主要结构蛋白,在病毒的生物学和免疫学等方面有着非常重要的功能。NSP1~NSP6在病毒复制、翻译、出芽等过程中具有重要作用。
2.1 结构蛋白 结构蛋白是形成病毒颗粒、维持病毒形态、影响病原性及免疫原性的重要蛋白。成熟的RV粒子具有3层同心的蛋白壳层:外层由糖蛋白VP7和由VP4组成的钉状物构成;中间层由260个VP6三聚体形成T=13的二十面体晶格构成;最内层主要由RNA结合蛋白VP2组成,VP2与VP1和VP3蛋白一起包装轮状病毒基因组,形成核心颗粒。
A组轮状病毒VP4是一种非糖基化的胰酶敏感蛋白,位于病毒颗粒表面,形成穗状突起,由基因组第4基因编码,分子量约为87 kDa。VP4约占病毒蛋白量的1.5%,由775个氨基酸组成,但动物RV分离株的VP4常为776个氨基酸[14]。VP4在细胞吸附和穿透中具有重要作用,使病毒具有致病性;VP4是病毒主要交叉中和抗原,自然感染时能够刺激机体产生中和抗体;VP4还是病毒结合宿主细胞受体的病毒蛋白,直接决定了病毒的毒力[15]。VP4由蛋白水解酶和糜蛋白酶连续分解产生了同质的VP8*和VP5核心(VP8CT、VP5CT),分子量分别为60 kDa和28 kDa,二者在提高病毒传染性,诱导构象变化和稳定穗状结构中起到重要作用[16]。其中VP8*靠近氨基端(247AA),拥有VP4蛋白的主要抗原位点,且负责VP4血清特异性中和反应,VP8*的抗原表位位于穗状结构远端的头部。VP5*靠近羧基端(529AA),包含了病毒融合作用的区域,负责病毒穿入过程[17]。VP8*抗体具有高度型特异性,这可能与VP8*的血凝素活性和半乳糖凝集素样折叠结构有关。而VP5*片段则在各型之间具有交叉反应[18]。VP8CT是可溶性的,主要包括β片层的单体;VP5CT形成了耐十二烷基硫酸钠单体,为交叉反应部位,这表明RV的蛋白消化作用激发了在VP4区中VP5*的重新分配[19]。基于RV外层蛋白VP4的抗原特性建立了血清分型系统,人类和动物A组轮状病毒可分为28个P型[20]。随着分子生物学在分类学中的应用,A组RV的血清型可能会进一步增加。VP4也是人与动物RV之间发生重组的主要部位之一。
VP6位于RV三层核衣壳的中层,是维持病毒颗粒稳定的主要结构蛋白。A组RV的VP6由病毒基因组6编码,分子量约45 kDa,由387个氨基酸组成,占病毒蛋白的5l%[21]。VP6是RV主要的内衣壳蛋白,由寡聚体形成三聚体而构成RV单层壳颗粒的二十面体表面结构。VP6的功能性区域为105~328位氨基酸,其中122~147位氨基酸为主要的聚合区域。VP6无多聚酶活性,但病毒缺乏VP6时可使病毒丢失转录酶活性[22]。VP6是RV组共同抗原和亚组特异性抗原,这也是RV分组依据所在[23]。
VP7占病毒蛋白总量的30%。依毒株不同,VP7可由病毒基因组的基因7、8或9编码。分子量36 kDa左右,由297或326个氨基酸组成[24]。VP7编码区有978个碱基,含有2个翻译起点,上游为弱启动子,下游为强启动子[25]。VP7是一种糖基化蛋白,含2个左右N型糖基化位点,是一种膜内蛋白,通过其第51~61和61~111位氨基酸固定于内质网膜上,并与VP4和NSP4形成多聚体,在病毒核心出芽时装配到病毒表面[26]。VP7构成病毒三层的最外光滑层,是轮状病毒最主要的外衣壳蛋白和中和抗原,具有CTL识别的抗原决定簇,对于保护性免疫尤为重要,且VP7基因与致病性无关。Dyall-Smith等[27]通过抗单克隆抗体突变和测序技术在轮状病毒SA11株VP7蛋白上鉴定出3个抗原区域A(87~96 aa),B(145~150 aa)和C(211~223 aa),同时还指出抗原区A和C虽然在线性序列上距离较远,但在折叠后的VP7蛋白上的空间距离很近。进一步研究发现中和抗原区域位于两个可变区87~101aa和208~221aa序列上[28]。VP7还是Ca2+结合蛋白,Ca2+对轮状病毒外壳蛋白的稳定和病毒的成熟至关重要。完整的病毒颗粒倾向形成三层颗粒,螯合剂剥夺Ca2+后,可导致成熟的轮状病毒颗粒失去VP4和VP7而形成具有转录活性的双层颗粒[29]。VP7能通过与VP4相互作用而影响VP4与细胞受体的结合效率,从而影响轮状病毒的复制效率。根据VP7抗原特性可将RV分为20个G血清型,这与P血清型相独立。同一种血清型不同株之间的VP7氨基酸序列同源性高达95%左右[30]。
2.2 非结构蛋白 RV共有6种非结构蛋白(NSP1~NSP6),分别由5、8、10、11基因片段编码,具有影响RV复制、参与其转录、组成病毒复制酶、介导病毒与靶细胞结合及形成病毒外衣壳等功能。NSP1由RV第5基因片段编码,基因片段的总长度以及开放阅读框的大小,在不同毒株间有所不同。NSP1是RV蛋白中变化最为活跃的部分,在A组RV中其序列多样性在65%以上。虽然在整体上其具有高度可变性,但在序列末端都有一个富含半胱氨酸的基序。因此NSP1被认为是可能与RV种属限制性关系最密切一种蛋白质,同时NSP1的活性会依宿主的不同而发生改变[31]。NSP1从功能上可以分为RNA结合结构域(1~81 aa)、细胞内定位结构域(82~177 aa)和效应结构域(328 aa~C末端)。其N端的RNA结合结构域中还包括一个锌指结构域(RING,42~79 aa),其可以与RV的dsRNA结合,在蛋白酶依赖的IRF3等的降解过程和NSP1稳定性的自动调节方面发挥重要作[32]。N-末端是保守区域,当锌指结构域中组氨酸和半胱氨酸发生点突变时,IRF3等的降解将发生中断,因此锌指结构的完整性对于蛋白酶依赖的IRF3等的降解过程至关重要[33]。细胞内定位结构域决定NSP1在宿主细胞的细胞骨架中聚集的位置[34],而效应结构域可以和干扰素调控因子家族结合[35]。NSP1具有E3泛素连接酶的功能,它能诱导蛋白酶介导的干扰素调节因子IRF3、IRF5和IRF7降解,抑制Ⅰ型干扰素的反应[36]。另外,NSPl蛋白还可通过降解β-TrCP进而抑制核转录因子(NF-κB)的活化[37],以及抑制信号转导和转录激活因子STAT1和STAT2核转位等机制对抗IFN信号传导通路[38],从而起到抵抗宿主天然免疫反应的功能。此外,Bagchi等[39]还发现,NSP1可通过抑制感染细胞过早凋亡而促进感染后病毒生长,这也体现了RV的生存策略。
NSP3是一种多功能蛋白,大多由第7基因片段编码,通常由313个氨基酸残基组成,相对分子质量约34 kDa,呈弱酸性,为非糖基化蛋白。其N端为RNA结合区,中间为寡聚化区,C端为真核翻译起始因子eIF4GI结合区。NSP3的羧基末端可与真核生物翻译起始因子eIF4GI相互作用,寡聚化区可与RoXaN(轮状病毒NSP3相关X蛋白)结合[40]。在感染最初阶段,NSP3在轮状病毒引起的细胞蛋白质合成关闭现象中发挥重要作用。这一抑制作用可能与NSP3作为真核细胞PABP [poly(A)结合蛋白]的结构类似物而与eIF4GI竞争性结合有关,表明NSP3可能在RV复制和形态发生中起重要作用。Mentero等[41]应用RNAi抑制了NSP3在RV感染细胞中的表达,结果发现病毒蛋白的合成没有降低,病毒mRNA合成增加,并且病毒子代产量增加,因此认为NSP3对于病毒mRNA的翻译不是必需的。
NSP5蛋白由基因片段11编码,位于病毒质粒中,分子量为26 kDa。NSP5氨基酸序列高度保守,C末端是最保守的区域,这一区域也是NSP5多聚化结构域。NSP5多聚化结构域富含丝氨酸和苏氨酸,翻译后经过磷酸化和糖基化修饰。不同翻译后修饰使NSP5具有不同的分子量(26~35 kDa),但在病毒感染的细胞中NSP5主要以26 kDa或28 kDa两种形式存在[42]。在被感染的细胞中可发现多磷酸异构体,有研究证明NSP5具有自磷酸化酶活性。Afrikanova等[43]的研究发现在细胞中瞬时表达NSP2和NSP5可以使NSP5获得高磷酸化状态,而NSP5单独在细胞中表达时无法实现高磷酸化。有些研究还显示NSP5的磷酸化与细胞的激酶和磷酸酶有关[44]。NSP5形成较早,出现在病毒质内。采用小分子RNA干扰技术研究发现,阻断NSP5的形成,即可抑制病毒质的形成和病毒的复制[45]。另外,NSP5也可与其他NSP相互作用。NSP2和NSP5已被证明是毒质体形成所必须的,并共同表达于未受感染的细胞,形成毒质体样结构。RV基因组的复制和包装,在毒质体样结构中进行。毒质体样结构在感染过程中形成,涉及多个组成部分并相互协调作用,其中NSP2和NSP5起重要作用[46]。
对轮状病毒的研究始于20世纪60年代,随着病毒检测技术和分子生物学的不断进步,我们对轮状病毒的认识亦逐渐明晰。从微观形态的观察到病毒基因组结构功能的测定,从病毒蛋白抗原特性到病毒逐个结构蛋白和非结构蛋白的研究,无不体现出科学研究从感性认识到理性认识的发展过程。随着实践的不断深入,轮状病毒的本质也将进一步为人们所揭示。
[1] Adams W R, Kraft L M.Epizootic diarrhea of infant mice:identification of the etiologic agent [J].Science, 1963,141(3578): 359-360.
[2] van Regenmortel M H, International, Fauquet C M.Virus taxonomy (classification and nomenclature of Viruses)[M].Academic Press of Elsevier, 2012: 603-613.
[3] Zuckerman A J, Banatvala J E, Schoub B,et al.Principles and Practice of Clinical Virology[M].6th ed.John Wiley& Sons Ltd, 2009: 337-353.
[4] Greig S L, Berriman J A, O’Brien J A,et al.Structural determinants of rotavirus subgroup specificity mapped by cryo-electron microscopy [J].J Mol Biol, 2006, 356(1):209-221.
[5] Matthijnssens J, Ciarlet M, Heiman E,et al.Full genomebased classification of rotaviruses reveals a common origin between human Wa-like and porcine rotavirus strains and human DS-1-like and bovine rotavirus strains[J].J Virol,2008, 82(7): 3204-3219.
[6] Aurisicchio L, De Tomassi A, La Monica N,et al.Regulated and liver-specific tamarin alpha interferon gene delivery by a helper-dependent adenoviral vector [J].J Virol, 2005, 79(11): 6772-6780.
[7] Steyer A, Poljsak-Prijatelj M, Barlic-Maganja D,et al.Human, porcine and bovine rotaviruses in Slovenia:evidence of interspecies transmission and genome reassortment [J].J Gen Virol, 2008, 89(Pt 7):1690-1698.
[8] Mukherjee A, Dutta D, Ghosh S,et al.Full genomic analysis of a human group A rotavirus G9P[6] strain from Eastern India provides evidence for porcine-to-human interspecies transmission[J].Arch Virol, 2009: 154(5):733-746.
[9] Chitambar S D, Arora R, Chhabra P.Molecular characterization of a rare G1P [19] rotavirus strain from India: evidence of reassortment between human and porcine rotavirus strains[J].J Med Microbiol, 2009, 58(Pt 12):1611-1615.
[10] Bányai K, Esona M D, Kerin T K,et al.Molecular characterization of a rare, human-porcine reassortant rotavirus strain, G11P[6], from Ecuador[J].Arch Virol,2009, 154(11):1823-1829.
[11] Park S I, Matthijnssens J, Saif L J,et al.Reassortment among bovine, porcine and human rotavirus strains results in G8P[7] and G6P[7] strains isolated from cattle in South Korea[J].Vet Microbiol, 2011, 152(1-2): 55-66.
[12] Wang Y H, Kobayashi N, Nagashima S,et al.Full genomic analysis of a porcine-bovine reassortant G4P[6]rotavirus strain R479 isolated from an infant in China[J].J Med Virol, 2010, 82(6): 1094-1102.
[13] Wu F T, Bányai K, Lin J S,et al.Putative canine origin of rotavirus strain detected in a child with diarrhea, Taiwan[J].Vector Borne Zoonotic Dis, 2012, 12(2): 170-173.
[14] Pesavento J B, Crawford S E, Estes M K,et a1.Rotavirus proteins: structure and assembly[J].Curt Top Microbiol Immunol, 2006, 309: 189-219.
[15] Estes M K, Kapikian A Z.Fields Virology[M].5th ed Wolters Kluwer Health/Lippincott Williams& Wilkins,Philadelphia, 2007: 1917-1974.
[16] Crawford S E, Mukherjee S K, Estes M K,et al.Trypsin cleavage stabilizes the rotavirus VP4 spike[J].J Virol,2001,75(13): 6052-6061.
[17] Padilla-Noriega L, Dunn S J, Lopez S,et al.Identification of two independent neutralization domains on the VP4 trypsin cleavage products VP5* and VP8* of human rotavirus ST3[J].Virology, 1995, 206(1): 148-154.
[18] Zade J K, Chhabra P, Chitambar S D.Characterization of VP7 and VP4 genes of rotavirus strains: 1990-1994 and 2000-2002[J].Epidemiol Infect, 2009, 137(7): 936-942.
[19] Dormitzer P R, Greenberg H B, Harrison S C.Proteolysis of monomeric recombinant rotavirus VP4 yields an oligomeric VP5* core [J].J Viro1, 2001, 75(16): 7339-7350.
[20] Solberg O D, Hasing M E, Trueba G,et a1.Characterization of novel VP7, VP4, and VP6 genotypes of a previously untypeable group a rotavirus [J].Virology,2009, 385(1):58-67.
[21] Ghosh S, Varghese V, Samajdar S,et a1.Evidence for independent segregation of the VP6- and NSP4-encoding genes in porcine group A rotavirus G6P [13] strains [J].Arch Virol, 2007, 152(2): 423-429.
[22] Kohli E, Pothier P, Tosser G.In vitro reconstitution of rotavirus transcriptional activity using viral cores and recombinant baculovirus expressed VP6[J].Arch Virol,1993,133(3-4): 451-458.
[23] Santos N, Hoshino Y.Global distribution of rotavirus serotypes/genotypes and its implication for the development and implementation of all effective rotavirus vaccine [J].Rev Med Virol, 2005,15(1): 29-56.
[24] Ghosh S, Kobayashi N.Whole-genomic analysis of rotavirus strains: current status and future prospects [J].Future Microbiol, 2011, 6(9): 1049-1065.
[25] Martella V, Ciarlet M, Baselga R,et a1.Sequence analysis of the VP7 and VP4 genes identifies a novel VP7 gene allele of porcine rotaviruses, sharing a common evolutionary origin with human G2 rotaviruses [J].Virology, 2005, 337(1): 111-123.
[26] Rao C D, Gowda K, Reddy B S Y.Sequence analysis of VP4 and VP7 genes of nontypeable strains identifies a new pair of outer capsid proteins representing novel P and G genotypes in bovine rotaviruses[J].Virology, 2000,276(1):104-113.
[27] Dyall-Smith M L, Lazdins I, Tregeart G W,et al.Location of the major antigenic sites involved in rotavirus serotype-specific neutralization[J].Proc Natl Acad Sci USA, 1986, 83(10): 3465-3468.
[28] Green K Y, Midthun K, Gorziglia M,et al.Comparison of the amino acid sequences of the major neutralization protein of four human rotavirus serotypes[J].Virology,1987, 161(1): 153-159
[29] Dormitzer P R, Greenberg H B.Purified recombinant rotavirus VP7 forms soluble, calcium-dependent trimers[J].Virology, 2000, 277(2): 420-428.
[30] Ghosh S, Kobayashi N, Nagashima S,et al.Full genomic analysis and possible origin of a porcine G12 rotavirus strain RU172[J].Virus Genes, 2010, 40(3): 382-388.
[31] Chung K T, McCrae M A.Regulation of gene expression by the NSP1 and NSP3 non-structural proteins of rotavirus[J].Arch Virol, 2011, 156(12): 2197-203.
[32] Graff J W, Ewen J, Ettayebi K,et al.Zinc-binding domain of rotavirus NSP1 is required for proteasomedependent degradation of IRF3 and autoregulatory NSP1 stability [J].J Gen Virol, 2007, 88(Pt 2): 613-620.
[33] Barro M, Patton J T.Rotavirus nonstructural protein 1 subverts innate immune response by inducing degradation of IFN regulatory factor 3[J].Proc Natl Acad Sci USA,2005, 102(11):4114-4119.
[34] Taniguchi K, Kojima K, Kobayashi N,et al.Structure and function of rotavirus NSP1[J].Arch Virol Suppl,1996, 12:53-58.
[35] Sen A, Feng N, Ettayebi K,et al.IRF3 inhibition by rotavirus NSP1 is host cell and virus strain dependent but independent of NSP1 proteasomal degradation[J].J Virol,2009, 83(20): 10322-10335.
[36] Azim T, Zaki M H, Podder G,et al.Rotavirus-specific subclass antibody and cytokine responses in Bangladeshi children with rotavirus diarrhoea[J].J Med Virol, 2003,69(2): 286-295.
[37] Graff J W, Ettayebi K, Hardy M E.Rotavirus NSP1 inhibits NFkappaB activation by inducing proteasomedependent degradation of beta-TrCP: a novel mechanism of IFN antagonism [J].PLoS Pathog, 2009, 5(1):1-12.
[38] Holloway G, Truong T T, Coulson BS.Rotavirus antagonizes cellular antiviral responses by inhibiting the nuclear accumulation of STAT1, STAT2, and NF-kappaB[J].J Virol, 2009, 83(10): 4942-4951.
[39] Bagchi P, Dutta D, Chattopadhyay S,et a1.Rotavirus nonstructural protein l suppresses virus-induced cellular apoptosis to facilitate viral growth by activating the cell survival pathways during early stages of infection[J].J Virol, 2010, 84(13): 6834-6845.
[40] Piron M, Delaunay T, Grosclaude J,et al.Identification of the RNA-binding , dimerization,and elF4GI-binding domains of rotavirus nonstructural protein NSP3[J].J Virol,1999,73(7): 5411-5421.
[41] Montero H, Arias C F, Lopez S.Rotavirus nonstructural protein NSP3 is not required for viral protein synthesis[J].J Virol, 2006, 80(18): 9031-9038.
[42] Martin D, Ouldali M, M é n é trey J,et al.Structural organization of the rotavirus nonstructural protein NSP5[J].J Mol Biol, 2011, 413(1): 209-211.
[43] Afrikanova I, Fabbretti E, Miozzo M C,et al.Rotavirus NSP5 phosphorylation is up-regulated by interaction with NSP2 [J].J Gen Virol, 1998, 79(Pt 11): 2679-2686.
[44] Eichwald C, Vascotto F, Fabbretti E,et al.Rotavirus NSP5: mapping phosphorylation sites and kinase activation and viroplasm localization domains [J].J Virol, 2002,76(7): 3461-3470.
[45] Contin R, Arnoldi F, Gampagna M,et al.Rotavirus NSP5 orchestrates recruitment of viroplasmic proteins [J].J Gen Virol, 2010, 91(Pt7):1782-1793.
[46] Jiang X, Jayaram H, Kumar M,et a1.Cryoelectron microscopy structures of rotavirus NSP2-NSP5 and NSP2-RNA complexes: implications for genome replication[J].J Virol, 2006, 80(21):10829-10835.

