禽肉中弯曲菌的分离、PFGE和DGGE分型*
孟赫诚 毕水莲 闫鹤,3 石磊†
(1.华南理工大学轻工与食品学院,广东广州510640;2.广东药学院食品科学学院,广东中山528435;3.辽宁省食品安全重点实验室,辽宁锦州121013)
近年来,弯曲菌感染率在世界各地普遍呈上升趋势.美国、加拿大、日本等国家正在全力开展有关弯曲菌的研究工作,但在我国,对弯曲菌的研究相对较少且不够深入.弯曲菌属于革兰氏阴性短杆菌,它在自然界广泛存在,尤其在鸡、鸭等家禽肠道内分布最多.弯曲菌是最常见的腹泻致病菌之一[1-2],可以引发格林巴氏综合症等严重并发症[3],其中,空肠弯曲菌(Campylobacter jejuni)和结肠弯曲菌(Campylobacter coli)是弯曲菌感染最常见的两个种,约占99%以上[4].弯曲菌培养条件比较苛刻,需要5%O2、10%CO2和 85%N2的微需氧环境.多采用增菌肉汤预增菌,然后使用选择性培养基或者膜过滤的方法对食品中弯曲菌进行分离.增菌培养基以Bolton和Preston肉汤最为常见.选择性培养基可分为含血和不含血两种类型,Skirrow和mCCDA分别是这两种类型的主要代表.
变性梯度凝胶电泳(DGGE)是一种广泛应用于不同领域微生物群落多样性分析和种群动态监测的分子生物学技术,由Fischer和Lerman于1979年提出[5-6],现已逐步应用于发酵食品的微生物研究、食源性致病微生物的快速检测和分型[7-8].由于被弯曲菌污染的肉类样品的污染源可能来源于弯曲菌属的不同种,也可能是同一种弯曲菌的不同型别,因此,在调查肉类样品弯曲菌污染情况时,除了对弯曲菌进行分离鉴定外,还需要通过分型技术对样品中分离的弯曲菌进行分型研究,以掌握弯曲菌污染的总体情况.目前,毕水莲等[9]应用DGGE技术对食品中C.jejuni和C.coli进行了同时检测和分型研究,但这种方法只能区别不同的型别,并不能鉴定是C.jejuni或C.coli.本研究针对此缺陷,采集市售生鲜鸡、鸭肉样品,使用Bolton肉汤和Preston肉汤增菌、Skirrow、mCCDA选择培养基和膜过滤的方法分离样品中的弯曲菌,并对分离的弯曲菌进行脉冲场凝胶电泳(PFGE)和DGGE基因分型.
1 材料与方法
1.1 材料
1.1.1 样品来源
2009年3月,从广州市天河区某肉菜市场随机采集生鲜肉样品8份,其中鸡肉5份,鸭肉3份(分别来源于5只鸡和3只鸭).样品在2 h内运送至实验室进行后续操作.
1.1.2 菌株
标准菌株 C.jejuni LMG8841、C.coli JCM2529 和胎儿弯曲菌(C.fetus)ATCC27374由日本大阪府立大学提供.菌种保存在17%(体积分数)甘油的脑心浸液培养基中,-80℃冻存.
1.1.3 培养基与试剂
Bolton、Preston、Skirrow 和 mCCDA 培养基购自英国Oxoid公司,脑心浸液培养基购自广州环凯生物技术有限公司.0.45 μm无菌醋酸纤维素滤膜购自日本Advantec公司.SmaI限制性内切酶和DNA Taq聚合酶购自大连宝生物工程有限公司.蛋白酶K、低熔点琼脂糖、电泳级琼脂糖购自美国Bio-Rad公司;DL2000 Marker购自广州东盛生物科技有限公司.弯曲菌PCR检测试剂盒购自日本TaKaRa公司;弯曲杆菌生化反应管购自北京友康基业生物科技有限公司.
1.1.4 仪器
主要仪器如下:Icycler PCR仪、Universal HoodⅡ凝胶成像系统和DCode Apparatus DGGE电泳仪,Smartspec plusTM分光光度计,Chef Mapper XA脉冲场电泳仪,美国 Bio-Rad 公司产品;CHB-202 ThermoCell恒温金属浴,杭州博日科技有限公司产品;BagMixer lab blender 400拍打仪,法国Interscience公司产品;Multi-gas incubator MCO-5M,日本 Sanyo 公司产品.
1.2 方法
1.2.1 样品中弯曲菌的分离与鉴定
采用无菌操作切取约10 g肉品放至无菌Stomacher袋中,加入10 mL 0.1%蛋白胨水,使用拍打仪拍打30 s,透过滤膜吸取1 mL拍打液分别加入到4mL Bolton和4 mL Preston增菌肉汤中,37℃培养24 h,然后分别取一接种环培养液在 Skirrow和mCCDA选择培养基上划线.此外,在血平板上放置0.45μm无菌醋酸纤维素滤膜,在膜上滴入6-8滴(每滴约5 μL)增菌培养液,静置30 min后将膜取出,将该平板与前面已划线的Skirrow和mCCDA平板37℃培养48 h.以上所有培养均在三气培养箱中进行,气体设置参数为5%O2、10%CO2和85%N2.
每个平板挑取2-3个可疑菌落进行25℃和42℃生长测定,通过革兰氏染色,用氧化酶、吲哚乙酸酯、马尿酸盐和过氧化氢酶水解,采用弯曲菌聚合酶链式反应(PCR)检测试剂盒进行鉴定,对使用两种方法检测结果均为阳性的菌落进行统一保存.
1.2.2 PFGE分型
参照美国疾病控制与预防中心(CDC)推荐的C.jejuni脉冲场电泳标准方法[10]操作,具体如下:
(1)胶块的制备
从冻存管挑取一环菌接种到脑心浸液培养基加入5%马血制成的固体培养基上,在上述微需氧条件下,37℃培养48h,刮取培养皿上所有菌落至2mL细胞稀释液(10mmol/L三乙丙基乙磺酰(Tris-HCl),50mmol/L 乙二胺四乙酸(EDTA),pH 8.0,20mmol/L NaCl)中,冲洗3次后再用1mL细胞稀释液混匀.测其光密度(OD)值,并将OD值调整为1.5~1.6.取150μL菌悬液与等体积的2%的低熔点琼脂糖混匀,55℃温育.将混合物注入模具中,4℃凝固15min,加入溶菌酶、细胞裂解液(含蛋白酶K)作用.加入2mL 54℃预热的灭菌水,54℃摇动反应10min,冲洗2次,每次20min.再加入2mL 54℃预热的三乙丙基乙磺酰-乙二胺四乙酸缓冲液(TE),54℃摇动反应15min,冲洗4次,每次20min.
(2)酶切及电泳
切取胶栓,浸入300 μL内切酶缓冲液中,室温静置30min.吸出内切酶缓冲液,浸入300μL内切酶反应体系中(其中含SmaI酶50U),37℃酶切过夜.将胶块在0.5×TBE(三乙丙基乙磺酰-硼酸)中平衡15min,以终止消化作用.然后将消化好的胶块贴附于制胶梳子末端,置于水平放置的倒胶模具上,将50℃已熔化的1%脉冲场琼脂糖倒入电泳槽,室温凝固1h.将电泳胶块放入电泳槽中电泳,电泳条件为:2L 0.5×TBE电泳缓冲液、电泳温度14℃、电场强度6V/cm、脉冲角度120°、脉冲时间6.76 ~35.38s、电泳时间18 h.电泳结束后,取出凝胶放入溴化乙锭(EB)溶液(0.5 μg/mL)染色30 min,去离子水脱色30min.通过凝胶成像系统拍照并保存图像.
1.2.3 DGGE分型
(1)DNA制备
取1.5mL收集的滤液,10000r/min离心5min,沉淀重悬于567μL的TE缓冲液,加入30μL 10%SDS和3μL 20mg/mL蛋白酶 K,混匀,37℃保温1 h,加入 100 μL 5 mol/L NaCl充分混匀,再加入 80 μL CTAB/NaCl溶液混匀,65℃保温30min,加入750μL氯仿/异戊醇(体积比为24∶1)混匀,12000 r/min离心5min,吸上清,转移到新1.5mL离心管中,加入等体积的酚/氯仿/异戊醇(体积比为25∶24∶1)混匀,12000r/min离心5min,将上清转移到新1.5 mL离心管,加50μL 3mol/L NaOAc和1mL冰无水乙醇,混匀,-20℃放置1 h,12000 r/min离心5 min,弃上清,加入1 mL 70%冰乙醇混匀,12000 r/min离心5min,弃上清,室温放置使乙醇挥发干净,加入50μL灭菌水和5μg Rnase A酶,-20℃保存备用.
(2)PCR扩增
引物参照文献[11]设计,引物CF02为:5'AAGCAAGAAGTGTTCCAAGTTT3',CF03 为:5'CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGCTCAAAGTGGTTCTTATGCNATGG3',由广州英骏生物技术有限公司合成.
反应体系:50μL 1.25U rTaq聚合酶,5μL 10×缓冲液,1μL 10mmol/mL脱氧核糖核苷三磷酸(dNTP),2μL DNA ,引物CF03和CF02各3μL(10μmol/L),灭菌双蒸水补足至50μL.
反应条件:预变性94℃ 5 min,复性94℃ 30 s,退火56℃ 30s,延伸72℃ 30s,循环35次;延伸72℃7min.PCR产物经1.2%琼脂糖凝胶电泳检测后切胶回收,回收程序按照Wizard SV Gel and PCR Cleanup System使用说明操作,最后使用50 μL双蒸水溶解DNA.
(3)产物的变性梯度凝胶电泳
PCR产物在变性剂梯度为5% ~35%(100%变性剂含有7mol/L尿素和40%去离子甲酰胺),8%聚丙烯酰胺凝胶,1×TAE(三乙丙基乙磺酰-乙酸)缓冲液中进行DGGE电泳.每个样本的上样量为10μL,电泳条件为60℃,20 V 预电泳15 min,200 V电泳3h.EB染色30min,水洗10min后用凝胶成像系统拍照.
2 结果与讨论
2.1 弯曲菌的分离结果
生化试验和多重PCR检测的结果完全一致.在随机采集的8份样品中,6份样品显示弯曲菌阳性,分离得到4株C.jejuni和7株C.coli(见表1).根据后续PFGE和DGGE分型的结果,排除来源于同一份样品的重复克隆菌株,共分离到9株弯曲菌(3株C.jejuni和6 株 C.coli),其中 C.coli阳性的样本和菌株数量远多于C.jejuni阳性样本和菌株数量(前者是后者的2倍).本次分离鸡肉和鸭肉中弯曲菌的结果表明,Preston肉汤作为增菌培养基的效果远优于Bolton肉汤,在使用Preston肉汤增菌时,mCCDA作为选择培养基的效果最好.样品C1和D6中分别分离到2株C.jejuni和3株C.coli,其它4份样品中均分离到1株弯曲菌.C.jejuni均分离自鸡肉样品,鸭肉样品只分离出了C.coli.

表1 禽肉样品中的弯曲菌1)Table 1 Campylobacter spp.of poultry product samples
2.2 PFGE分型结果
根据 PFGE分型结果(见图1和表1),4株C.jejuni和7株C.coli各包含 3种 PFGE 型.C1样品中分离的2株C.jejuni的PFGE和DGGE型别均相同,D8样品中分离的 2株 C.coli的 PFGE和DGGE型别也相同,推测可能为相同克隆菌株.C1样品分离的3株C.jejuni分为2个型,D6样品分离的3株C.coli分为3个型.由图1和表1可见此次禽肉制品中不同PFGE型别和相应的菌株,其中包括Cj-P1、Cj-P3、Cc-P2 和 Cc-P3 型各1 株,Cj-P2 型2 株,Cc-P1型5 株.C4、C5 样品(鸡肉)和 D6、D8 样品(鸭肉)同时污染了Cc-P1型菌株.
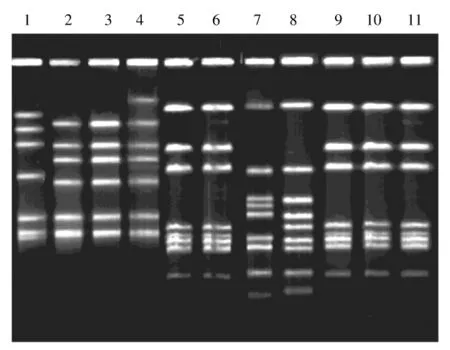
图1 11株弯曲菌的PFGE图谱Fig.1 PFGE profiles of 11 Campylobacter spp.isolates
2.3 DGGE分型结果
根据DGGE分型结果,4株C.jejuni包含3种DGGE型,7株C.coli包含2种 DGGE型.图2是共计分离所得11株弯曲菌的DGGE图谱.图2和表1表明,C1样品中分离的2株C.jejuni的DGGE型别相同,D8样品中分离的2株C.coli的DGGE型别相同.C1和D6样品分别被2株C.jejuni和3株C.coli污染.其它4份样品只被1种弯曲菌污染.与PFGE分型结果不同的是,D6样品中 BS6b和 PC6b的DGGE型别一致.为确保DGGE分型方法的可重复性,试验重复操作3次.3次试验图谱一致,表明该DGGE方法具有很好的可重复性.

图2 11株弯曲菌的DGGE图谱Fig.2 DGGE profiles of 11 Campylobacter spp.isolates
2.4 讨论
Bolton肉汤和Preston肉汤是分离弯曲菌最常用的增菌培养基.Baylis等[12]的研究表明,Bolton肉汤对于分离禽肉中弯曲菌效果很好.Ng等[13]研究发现,Preston肉汤中抗生素抑制某些C.coli的生长,从而影响了C.coli的分离效果.然而,在本研究中,Preston肉汤对C.jejuni和C.coli的分离率均远高于Bolton肉汤.任何一种分离方法都不能够分离到样品中所有弯曲菌,所以在分离食品中弯曲菌时,应尽可能同时采用多种分离方法以保证结果的准确性,减少漏检的可能.
目前在国外,在使用DGGE技术对弯曲菌分型方面仅有少数几篇文献报道[14-16].在国内,仅毕水莲等[9]于2010年应用 fla-DGGE 技术实现了鸡、鸭肉样品中的C.jejuni和C.coli的同时检测和分型.这些研究中,毕水莲和 Najdenski等[9,16]对样品中C.jejuni和C.coli进行了DGGE分型研究,其采用直接提取样品中细菌总DNA的方法,继而进行半巢式PCR、DGGE分型;其它文献中仅仅对C.jejuni做了DGGE分型研究.本研究中涉及的DGGE分型研究有别于以上相关研究,首先,本研究对弯曲菌属中最常见的C.jejuni和C.coli均进行了 DGGE分型.其次,先对样品中弯曲菌进行了分离,然后提取弯曲菌基因组DNA进行PCR、DGGE分型,与毕水莲和Najdenski等[9,16]的研究相比具有许多优点:①由于没有杂菌干扰,DGGE分型的准确性和重复性提高;②本研究用常规PCR取代了半巢式PCR(因毕水莲和 Najdenski等[9,16]选用的模板为细菌总基因组DNA,半巢式PCR第1轮扩增起特异性扩增C.jejuni或C.coli和富集特异性DNA产物的作用),时间有所缩短;③先对样品中分离的菌株进行了鉴定,弥补了毕水莲和 Najdenski等[9,16]对 C.jejuni和 C.coli的DGGE分型只能区别不同的型别,而不能确定弯曲菌是 C.jejuni还是 C.coli的缺陷.
DGGE的原理是,双链DNA分子由于碱基组成的差异,在变性凝胶电泳时具有不同的解链温度而滞留于凝胶的不同位置,形成相互分开的谱带[17].理论上,只要选择合适的电泳条件,如变性剂梯度、电泳时间等,DGGE均能够将大小相同、碱基组成不同,甚至只有一个碱基差异的DNA片段分开[18].DGGE分型具有重现性好、准确度高的特点,与弯曲菌基因分型的金标准PFGE相比,它的灵敏度略低,但耗时更短,成本更低.通常采用PFGE方法对弯曲菌分型大约需要5~6d,而采用DGGE分型仅需5~6h,极大地缩短了时间.同时DGGE技术不需要使用蛋白酶K、限制性内切酶等昂贵试剂,因此成本较低.由此可知,DGGE技术在弯曲菌的基因分型方面具有较高的应用价值.
3 结语
本研究发现,从广州市天河区某肉菜市场随机采集的8份鸡肉和鸭肉样品中,6份样品带有弯曲菌,且2份鸡肉和2份鸭肉样品均被同一种C.coli污染.可见,该市场肉品中弯曲菌的带菌率非常高,且存在水平传播.在后续的研究中,笔者将针对广州市肉菜市场弯曲菌污染情况展开全面的大样本量的调查,也期望今后有关部门能够对弯曲菌的研究给予足够的重视.
[1] Robort V T.Incidence,trends and source of campylobacteriosis in developed countries[C]∥The Creasing Incidence of Human Campylobacteriosis.Copenhagen:WHO,2000:42-43.
[2] CokerA O,IsokpehiR D,ThomasB N,etal.Campylobacter enteritis in Lagos,Nigeria[C]∥ The Creasing Incidence of Human Campylobacteriosis.Copenhagen:WHO,2000:117.
[3] Zilbauer M,Dorrell N,Wren B,et al.Campylobacter jejuni-mediated disease pathogenesis:an update [J].Transactions of the Royal Society of Tropical Medicine and Hygiene,2008,102(2):123-129.
[4] Tauxe R V.Epidemiology of Campylobacter jejuni infections in the United States and other industrialized nations[C]∥Nachamkin.Campylobacter jejuni:Current and Future Trends.Washington:American Society for Microbiology,1992:9-12.
[5] Fischer S G,Lerman L S.Length-independent separation of DNA restriction fragments in two-dimensional gel electrophoresis[J].Cell,1979,16(1):191-200.
[6] Muyzer G,Smalla K.Application of denaturing gradient gel electrophoresis(DGGE)and temperature gradient gel electrophoresis(TGGE)in microbial ecology [J].International Journal of General and Molecular Microbiology,1998,73(1):127-141.
[7] Cocolin L,Rantsiou K,Iacumin L,et al.Direct identification in food samples of Listeria spp and Listeria monocytogenes by molecular methods [J].Applied and Environmental Microbiology,2002,68(12):6273-6282.
[8] Cocolin L,Comi G.Use of a culture-independent molecular method to study the ecology of Yersinia spp in food[J].International Journal of Food Microbiology,2005,105(1):71-82.
[9] 毕水莲,陈妙瑞,张志刚,等.Fla-DGGE方法对食品中空肠弯曲菌和结肠弯曲菌的检测和分型[J].现代食品科技,2010,26(10):1148-1152.Bi Shui-lian,Chen Miao-rui,Zhang Zhi-gang,et al.Detection and genotyping of Campylobacter jejuni and Campylobacter coli in food samples by Fla-DGGE [J].Modern Food Science and Technology,2010,26(10):1148-1152.
[10] Ribot E M,Fitzgerald C,Kubota K,et al.Rapid pulsedfield gelelectrophoresis protocolfor subtyping of Campylobacter jejuni[J].Journal of Clinical Microbiology,2001,39(5):1889-1894.
[11] Yu Z T,Morrison M.Improved extraction of PCR-quality community DNA from digesta and fecal samples[J].BioTechniques,2004,36(5):808-812.
[12] Baylis C L,MacPhee S,Martin K W,et al.Comparison of three enrichment media for the isolation of Campylobacter spp.from foods [J].Journal of Applied Microbiology,2000,89(5):884-891.
[13] NgL K,StilesM E,TaylorD E.Inhibition of Campylobacter coli and Campylobacter jejuni by antibiotics used in selective growth media[J].Journal of Clinical Microbiology,1985,22(4):510-514.
[14] Nielsen E M,Engberg J,Fussing V,et al.Evaluation of phenotypic and genotypic methods for subtyping Campylobacter jejuni isolates from humans,poultry,and cattle [J].Journal of Clinical Microbiology,2000,38(10):3800-3810.
[15] Heina I,Machb R L,Farnleitnerb A H,et al.Application of single-strand conformation polymorphism and denaturing gradient gel electrophoresis for fla sequence typing of Campylobacter jejuni[J].Journal of Microbiological Methods,2003,52(3):305-313.
[16] Najdenski H,Heyndrickx M,Herman L,et al.Fla-DGGE analysis of Campylobacter jejuni and Campylobacter coli in cecal samples of broilers without cultivation [J].Veterinary Microbiology,2008,127(1):196-202.
[17] Fischer S G D,Lerman L S.DNA fragments differing by single base-pair substitutions are separated in denaturing gradient gel:correspondence with melting theory[J].Proceedings of the National Academy of Sciences of the United States of America,1983,80(6):1579-1583.
[18] Myers R M,Fischer S G,Lerman L S,et al.Nearly all single base substitution in DNA fragments joined to a GC-clamp can be detected by denaturing gradient gel electrophoresis[J].Nucleic Acids Research,1985,13(9):3131-3145.

