杜氏盐藻S腺苷高半胱氨酸水解酶基因的克隆及功能分析*
阎赟梦,李庆华,李 杰,柴丹丹,薛乐勋
郑州大学生物工程系细胞生物学研究室郑州 450001
#通讯作者,男,1944年6月生,教授,研究方向:生物工程,E-mail:xuelx@zzu.edu.cn
杜氏盐藻S腺苷高半胱氨酸水解酶基因的克隆及功能分析*
阎赟梦,李庆华,李 杰,柴丹丹,薛乐勋#
郑州大学生物工程系细胞生物学研究室郑州 450001
#通讯作者,男,1944年6月生,教授,研究方向:生物工程,E-mail:xuelx@zzu.edu.cn
杜氏盐藻;S腺苷高半胱氨酸水解酶;鞭毛
目的:探讨杜氏盐藻S腺苷高半胱氨酸水解酶(SAHase)基因在鞭毛再生中的作用。方法:通过设计简并引物及RACE法克隆了杜氏盐藻SAHase基因全长,使用pH休克法对杜氏盐藻进行鞭毛去除及再生,通过实时荧光定量PCR研究在转录水平上SAHase基因的变化。结果:所克隆的杜氏盐藻SAHase基因cDNA全长1 926 bp,包含ORF 1 458 bp、3’UTR 61 bp和5’UTR 407 bp。实时荧光定量PCR结果显示,在鞭毛再生的过程中,SAHase mRNA转录量升高,其水平在3 h时达到了未处理组的3倍左右。结论:杜氏盐藻的SAHase基因与鞭毛再生有关。
S腺苷高半胱氨酸水解酶(SAHase)参与真核生物最主要的甲基化反应——以S-腺苷甲硫氨酸(S-Met)为甲基供体的甲基化反应[1]。S-Met在甲基转移酶的作用下,将甲基转移给供体,生成S-腺苷高半胱氨酸(S-Hcy),后者经SAHase催化生成高半胱氨酸和腺苷。抑制SAHase将导致S-Hcy积累,S-Hcy与S-Met竞争结合甲基转移酶,进而反馈抑制甲基化作用[2]。同时,作为腺苷结合蛋白,SAHase还调控着胞内游离腺苷浓度[3]。
鞭毛是广泛存在于各类细胞表面的细胞器,结构高度保守,参与细胞运动、感知外界刺激和细胞信号转导,在维持细胞正常生命活动中发挥重要作用。虽然对鞭毛内运输、蛋白组装研究得比较多,但到目前为止,鞭毛长度的调控机制依然不清楚[4]。最近,有研究[5]报道甲基化作用可能参与了鞭毛再生过程的调控。杜氏盐藻是一种高度耐盐的单细胞真核生物,具有双鞭毛,鞭毛长度约10 μm,便于观察。作者克隆杜氏盐藻SAHase基因全长,采用实时荧光定量PCR方法观察分析转录水平上SAHase与杜氏盐藻鞭毛再生的关系,初步探讨甲基化作用在鞭毛再生过程中的意义。
1 材料与方法
1.1 藻株与培养 杜氏盐藻UTEX LB-1644购自美国德州大学,采用改良PKS盐藻培养基进行液体培养,培养条件:温度26℃,光照强度4 500 Lux,光培养和暗培养各12 h。
1.2 试剂 LaTaq酶、限制性内切酶、反转录试剂盒和RACE试剂盒为TaKaRa公司产品,实时荧光定量染料购自天根公司。
1.3 杜氏盐藻SAHase基因全长的克隆
1.3.1 总RNA的提取及反转录 取对数生长期杜氏盐藻1 mL,2 000 r/min离心5 min,收集沉淀,培养基洗沉淀1次,再次离心收集沉淀。Trizol法裂解细胞,提取总RNA。总RNA经变性琼脂糖凝胶电泳和吸光度检测,纯度符合要求,按反转录试剂盒说明进行反转录。
1.3.2 简并引物扩增SAHase基因片段 根据莱茵衣藻等物种SAHase基因的保守序列设计一对简并引物:上游5’-AA(A/G)ATGTA(T/C)(C/A) GNATGG-3’,下游5’-TCNAC(G/A)CA(G/A) TANGG(G/A)TC(T/G/A)AT-3’。PCR扩增条件: 95℃预变性4 min;95℃变性40 s,55℃退火30 s,72℃延伸1 min,30个循环;最后72℃延伸10 min,4℃终止反应。PCR产物经琼脂糖电泳后胶回收,与pMD18-T载体 16℃过夜连接,转化 E.coli DH5α,进行蓝白斑筛选,挑取单克隆,提取质粒,EcoRⅠ、HindⅢ双酶切鉴定,取鉴定插入片段大小正确的单克隆送样测序,进行进一步鉴定。
1.3.3 RACE法扩增基因全长 根据巢式PCR原理,设计 2个上游引物 5’-TGTATGGCTGCCGT CACTCT-3’和5’-CGTGTTGTGGTGTCGGAGAT-3’。下游引物为3’-RACE试剂盒中自带的3’-RACE Inner Primer 5’-CGCGGATCCTCCACTAGTGATTTCAC TATAGG-3’和3’-RACE Outer Primer 5’-TACCG TCGTTCCACTAGTGATTT-3’。按照3’-RACE说明书上的反应条件设置反应程序。提取总RNA,按照5’-RACE试剂盒说明书去帽进行反转录,设计5’-Race引物5’-CACAATGGCGTCGTTCTTCA和TCTC CGACACCACAACACGA-3’,与试剂盒中自带引物的内外侧配对,按照5’RACE说明书上的反应条件设置反应程序。PCR产物进行琼脂糖电泳,回收产物。蓝白斑筛选,挑取阳性单克隆,提取质粒酶切鉴定,鉴定正确的单克隆送样测序。
1.4 实时荧光定量PCR 使用pH休克法去除杜氏盐藻鞭毛[6],参照王翠等[7]的研究,在去除鞭毛后的420 min内每30 min取样1次,用Trizol法提取总RNA,反转录为cDNA。设计一对引物5’-AAC ATCGTGGGCGTGTCTGA-3’和5’-ATGCGGTCTTGC CAGAAATC-3’,以GAPDH作为内参,进行实时荧光定量PCR。
2 结果
2.1 SAHase基因片段的RT-PCR产物鉴定结果
PCR扩增得到长度为327 bp的片段(图1)。使用NCBI数据库对测序结果进行Blast分析,结果显示所得片段与莱茵衣藻 SAHase基因的同源性为84%,所得片段为杜氏盐藻SAHase基因的一部分。
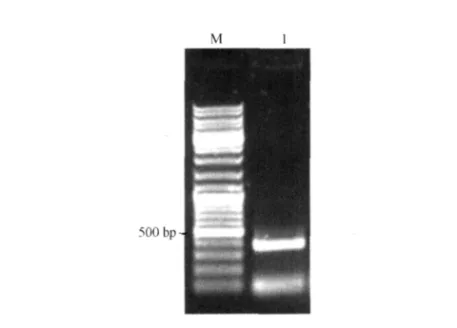
图1 杜氏盐藻SAHase cDNA片段RT-PCR扩增结果
2.2 RACE扩增及全长分析结果 3’-RACE PCR扩增得到长度为982 bp的片段,测序结果显示这部分片段包含完整的polyA尾。5’-RACE扩增得到长度为921 bp的片段(图2)。经拼接后得到SAHase cDNA全长为1 926 bp,其中包括3’UTR 61 bp,5’UTR 407 bp,ORF 1 458 bp,编码604个氨基酸,与莱茵衣藻同源性约为83%。

图2 RACE扩增结果
2.3 实时荧光定量PCR结果 结果显示,与未去除鞭毛时比较,在去除鞭毛后的420 min内,SAHase基因的转录量升高,其中,第180 min时甚至达到了对照组的3倍(图3)。
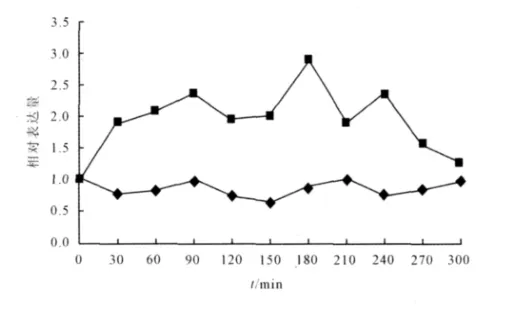
图3 实时荧光定量PCR结果
3 讨论
因为杜氏盐藻基因组序列尚未公布,所以首先比对多个物种的SAHase基因序列,选择保守区域,设计简并引物,通过PCR与RACE法相结合扩增了杜氏盐藻SAHase cDNA全长,经测序得到了杜氏盐藻SAHase cDNA序列,并提交GenBank。这为下一步研究SAHase基因与鞭毛长度调控之间的关系做好了准备工作。与其他物种的比对结果显示,扩增得到的SAHase cDNA与其他物种序列同源性很高,说明在不同物种中,SAHase基因结构高度保守。序列扩增过程中用到了RACE法,RACE法的主要目的是为了扩增基因的3’末端与5’末端。RACE结果的好坏主要取决于提取的RNA的质量和制备的模板的质量。一方面需要高质量的RNA,保证无降解,另一方面模板制备过程中也要小心操作,避免RNA降解。
SAHase蛋白广泛存在于各种生命体内,调控细胞内甲基化作用,直接或间接参与胞内多种重要的生命活动,如DNA甲基化、RNA加帽、蛋白甲基化及cAMP信号通路等[8]。鞭毛蛋白质组学分析证实SAHase蛋白是鞭毛蛋白组分之一,其催化底物AdoHcy也在鞭毛中存在[9]。Schneider等[5]证实甲基化途径组分甲硫氨酸合酶(MetE)在鞭毛顶端存在,并且MetE在完整鞭毛中含量比较低,在再生鞭毛中含量高,在重吸收鞭毛中含量最高。作者采用PCR法扩增了杜氏盐藻SAHase cDNA全长,实时荧光定量PCR结果显示鞭毛再生过程中SAHase的转录量明显升高,证明SAHase在鞭毛再生过程中发挥重要作用,提示甲基化作用参与了鞭毛再生的调控。SAHase基因可能是通过以下途径来调控鞭毛的再生:一方面,SAHase基因表达量的高低直接影响到甲基化途径的效率[10],DNA高甲基化状态不利于基因的表达,低甲基化状态有利于基因表达。因此,在鞭毛再生过程中SAHase大量转录,从而间接调控鞭毛组装相关基因的表达。另一方面,在鞭毛内可能存在完整的甲基化通路[5]。Schneider等[5]在重吸收的鞭毛中分离出一些高度甲基化的蛋白,这些蛋白大部分与轴丝相结合,且其甲基化形式在完整鞭毛和再生鞭毛中不存在,提示这些蛋白可能通过甲基化与非甲基化2种构象的转换来调控鞭毛再生。Sloboda等[11]也从鞭毛中分离出了3个与轴丝相结合的甲基化蛋白。以上研究均证实SAHase蛋白作为甲基化途径的组分参与了鞭毛长度的调控。
在莱茵衣藻中,SAHase蛋白在鞭毛和胞体内都存在。作者的研究结果显示SAHase与鞭毛再生有关,至于鞭毛内的SAHase蛋白是否参与了鞭毛内某些蛋白的甲基化,以及这些蛋白是在胞体内被甲基化后转运至鞭毛还是在鞭毛内发生了甲基化,尚有待进一步证实。
[1]田铃,嵇保中,刘曙雯,等.甲基转移酶的功能与分类[J].生命的化学,2007,27(5):425
[2]李爱秀,刘兴太,蔡德海.S-腺苷高半胱氨酸水解酶的晶体结构及其催化机制[J].生命的化学,2007,27(6):521
[3]Hermes M,Osswald H,Kloor D.Role of S-adenosylhomocysteine hydrolase in adenosine-induced apoptosis in HepG2 cells[J].Exp Cell Res,2007,313(2):264
[4]Pedersen LB,Rosenbaum JL. Intraflagellar transport (IFT)role in ciliary assembly,resorption and signalling[J].Curr Top Dev Biol,2008,85:23
[5]Schneider MJ,Ulland M,Sloboda RD.A protein methylation pathway in Chlamydomonas flagella is active during flagellar resorption[J].Mol Biol Cell,2008,19(10):4319
[6]Cheshire JL,Evans JH,Keller LR.Ca2+signaling in the Chlamydomonas flagellar regeneration system:cellular and molecular responses[J].J Cell Sci,1994,107(Pt 9):2491
[7]王翠,李杰,柳丽平,等.杜氏盐藻寡糖基转移酶亚基STT3a功能结构域的克隆与表达分析[J].生物工程学报,2010,26(6):760
[8]Kloor D,Osswald H.S-Adenosyl homocysteine hydrolase as a target for intracellular adenosine action[J].Trends Pharmacol Sci,2004,25(6):294
[9]Pazour GJ,Agrin N,Leszyk J,et al.Proteomic analysis of a eukaryotic cilium[J].J Cell Biol,2005,170(1):103
[10]Hermes M,Osswald H,Riehle R,et al.S-Adenosyl homocysteine hydrolase overexpression in HEK-293 cells:effect on intracellular adenosine levels,cell viability,and DNA methylation[J].Cell Physiol Biochem,2008,22(1/4):223
[11]Sloboda RD,Howard L.Protein methylation in full length Chlamydomonas flagella[J].Cell Motil Cytoskeleton,2009,66(8):650
Cloning and functional analysis of S-Adenosyl homocysteine hydrolase gene of Dunaliella salina
YAN Yunmeng,LI Qinghua,LI Jie,CHAI Dandan,XUE Lexun
Laboratory for Cell Biology,Department of Bioengineering,Zhengzhou University,Zhengzhou 450001
Dunaliella salina;SAHase;flagellar
Aim:To investigate the function of the SAHase gene in flagellar regeneration of Dunaliella salina(D.salina).Methods:A pair of degenerate primers was designed to obtain a fragment of SAHase cDNA,and the RACE method was used to amplify the full length of the SAHase cDNA.The transcriptional level of the SAHase gene from D.salina was monitored by real-time fluorescence quantitative PCR during flagellar regeneration.Results:The results of PCR and RACE showed that the full length of the SAHase cDNA cloned in this study was 1 926 bp,consisting of ORF 1 458 bp,3’UTR 61 bp and 5’UTR 407 bp.The result of real-time fluorescence quantitative PCR indicated that abundance of the SAHase mRNA was induced during the process of the flagellar regeneration,reaching to about 3-fold higher than that without the treatment of pH shock.Conclusion:The SAHase gene is related to regeneration of the flagella of D.salina.
Q781
*国家自然科学基金资助项目 3070014;科技部国际科技合作基金资助项目 2007DFA01240
(2010-10-11收稿 责任编辑王 曼)

