ISSR技术用于三种大米的鉴定
陈国培 赖心田 林霖 杨国武
(深圳市计量质量检测研究院 广东深圳 518131)
1 前言
增城丝苗米、马坝油粘米、盘锦大米等地理标志大米具有良好的市场知名度和极高的市场价格,但是由于缺乏有效的真伪鉴定方法,导致一些不法企业或者假冒牌子,以假乱真,或者以次充好,大大地损害了地理标志大米的市场信誉度和市场销售。因此,建立起一套行之有效的鉴定体系,对于维持地理标志大米产品的保护和市场秩序的维护非常必要。
广东省地方标准《DB44/303-2006马坝油粘米》,以及国家标准《GB/T 23402-2008地理标志产品 增城丝苗米》和《GB/T 18824-2008地理标志产品盘锦大米》中对马坝油粘米、增城丝苗米、盘锦大米的感官指标(色泽、气味、口感、组织形态)以及加工质量指标、理化指标进行了规定,但是单从这些规定无法区分。
ISSR分子标记技术是由加拿大蒙特利尔大学Zietkiewicz等于1994年创建的一种分子标记技术,其基本原理是在SSR的3’或5’端锚定1-4个碱基作引物,对两侧具有反向排列SSR的一段序列进行扩增,而不是扩增SSR本身。ISSR分子标记技术兼具SSR、RAPD、RFLP、AFLP等分子标记的优点,与SSR相比,ISSR不需要预先获知序列信息而使成本降低,且多态性更丰富;与RAPD相比,ISSR重复性高,稳定性好,同时具备RAPD的简便、易操作等特点;与RFLP、AFLP相比,ISSR更快捷、成本较低、DNA用量小、安全性较高。利用ISSR分子标记技术构建DNA指纹图谱已经在植物分子生物学研究中得到广泛的应用。2009年欧力军等从90个ISSR引物中筛选出条带清晰、多态性高的引物37个,对水稻籼粳亚种进行遗传差异分析,通过UPGMA聚类分析法把籼稻和粳稻各划为1支,并认为ISSR分子标记法适用于水稻籼粳性和广亲和基因的判断;2010年范付华等为开展水稻新品种亲缘关系的鉴定,拓展水稻种质资源创新与利用途径,以筛选出的12条ISSR引物对23个水稻品种(系)进行PCR扩增,建立了23份水稻品种(系)的ISSR指纹图谱,经聚类分析,可将所有材料严格地按粳、籼稻分为2类。
本研究采集了增城丝苗米7个品种、马坝油粘米3个品种、盘锦大米5个品种,通过构建ISSR分子标记图谱并进行分析,为马坝油粘米、增城丝苗米、盘锦大米的品种鉴别提供技术支持。
2 材料与方法
2.1 材料
2.1.1 样品
采集了增城丝苗米7个品种、马坝油粘米3个品种、盘锦大米5个品种。采样时优先采集市场占有率高的优势品种,其中象芽香占和香丝苗A为近几年增城市挂绿米业有限公司栽种的主要品种,美香粘和马坝银粘为马坝油粘米在市面上的优势品种,盘锦大米样品采集时涵盖了近年来主推栽种的辽粳294、盐丰47品种。其中6个增城丝苗米品种为增城市农业科学研究所的科研样品,其余的增城丝苗米由增城市质监局及增城市农业科学研究所协助采集并作现场鉴定,马坝油粘米品种由韶关市曲江区质监局及曲江区农业技术推广中心协助采集并作现场鉴定,盘锦大米委托辽宁省盘锦市质检所采集和鉴定,样品采集时均有农艺专家现场鉴定以保证品种的纯正性和纯度。每个品种采集200g,于-20℃密封保存备用(见表1)。

表1 样品采集资料
2.1.2 主要试剂
Taq酶和100bp DNA ladder Maker,均购自宝生物工程(大连)有限公司;ISSR引物由宝生物工程(大连)有限公司合成;十六烷基三甲基溴化胺(CTAB)和β-巯基乙醇由Amersco分装;琼脂糖购自深圳新拓扑生物科技有限公司。
2.2 方法
2.2.1 DNA 提取
采用改良CTAB法,具体方法为:称取0.5g大米样品于2mL离心管中,加入1mL 65℃预热的2%CTAB提取液(临用前加入2μLβ-疏基乙醇),65℃水浴2h,加等体积氯仿与异戊醇(24∶1)混合液抽提,冰盒中振荡10min,12000rpm离心10min,取出上清,再用等体积氯仿∶异戊醇(24∶1)抽提一次,上清加入2/3体积的异丙醇,于-20℃沉淀DNA 2h,6000rpm离心6min收集沉淀,加入500μL 70%乙醇和100%乙醇洗涤,干燥后,用30μL TE缓冲液(含RNase 10μg/mL)溶解。用紫外分光光度检测DNA样品的浓度和纯度,稀释至50ng/μL作为PCR反应的模板。
2.2.2 ISSR-PCR及琼脂糖凝胶电泳
2.2.2.1 ISSR - PCR
ISSR -PCR 体系为:ddH2O,12.2μL;10 ×buffer,2μL;Mg2+(25mM),1.5μL;ISSR 引物(10μM),0.5μL;dNTP(2.5mM each),1.6μL;Taq 酶(5U/μL,0.2μL;模板(50ng/μL),2μL。
ISSR-PCR反应程序为:94℃ 5min,94℃ 40s,53℃ 35s,72℃ 1min10s,72℃ 5min,循环数为 40。
2.2.2.2 琼脂糖凝胶电泳
ISSR-PCR产物使用2%的琼脂糖凝胶电泳检测,以100-2000bp DNA ladder为 Marker,100V 恒压下电泳90min,凝胶成像系统照像。
2.2.2.3 ISSR - PCR
从增城丝苗米、马坝油粘米、盘锦大米中各选取一个样品提取DNA并以此为模板,对文献中获取的80个ISSR引物(见表2)进行筛选,得出34个引物的扩增效果较好。并用这34个引物对增城丝苗米7个品种、马坝油粘米3个品种、盘锦大米5个品种提取的DNA进行扩增,每一个引物做两次重复实验,最后从34个引物的扩增结果中选出条带清晰,带型稳定的23个引物的扩增结果进行统计,条带统计时只统计带型清晰且在两次重复实验中均出现的条带。

表2 ISSR分析引物序列
2.2.3 数据分析
将琼脂糖凝胶电泳图中的不同样品在相对于marker的不同位置上扩增的产生的条带进行统计,有条带出现的记为1,条带不出现的见为0。用Excel 2003软件计算3种地理标志大米的多态位点数目、多态位点百分率和表型 Shannon指数。表型Shannon指数的计算公式为 H=-∑πi×lnπi,其中πi为条带出现的比率。
对3种地理标志产品进行聚类分析,分析时使用Phyltools软件(Version 1.32)对 ISSR数据构建bootstrap 10000次重新抽样的数据矩阵,并计算品种间的Matching距离,最后用Phylip软件(Version 3.6.1)构建 UPGMA 一致树。
3 结果与分析
使用23个ISSR引物总共扩增出216个条带,多态性条带为103个,单态性条带为113个,多态位点数百分率为47.7%,其中增城丝苗米多态位点百分率为15.7%,马坝油粘米多态位点百分率为10.2%,盘锦大米多态位点百分率为9.7%(见图1)。表型Shannon指数所揭示的总遗传多样性为0.268,其中增城丝苗米为0.086,马坝油粘米为0.065,盘锦大米为0.054。增城丝苗米、马坝油粘米、盘锦大米的多态位点百分率和表型Shannon指数都较低,表明3种地理标志大米产品品种内差异不大。增城丝苗米、马坝油粘米、盘锦大米的多态位点百分率和表型Shannon指数均远远低于总体多态位点百分率和表型Shannon指数,表明遗传差异主要体现在3种地理标志大米产品品种之间。

图1 引物823的ISSR扩增结果
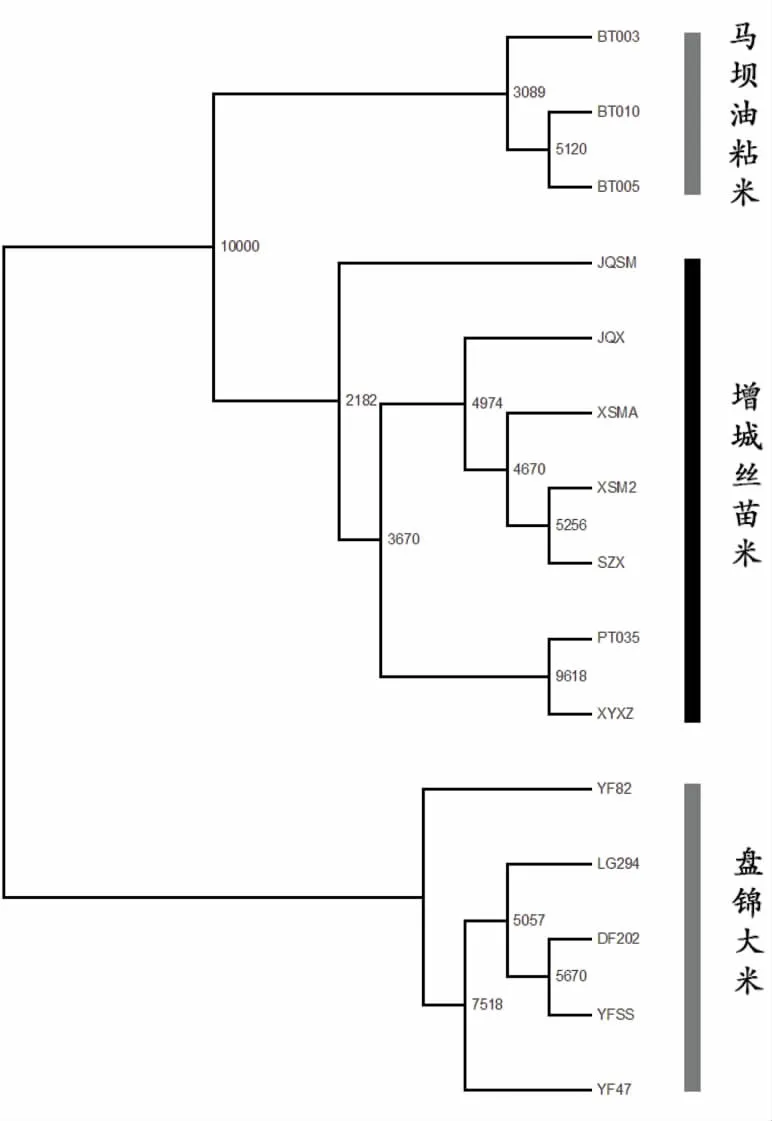
图2 基于Matching遗传距离构建的UPGMA树
UPGMA聚类结果显著支持盘锦大米自成一支,而增城丝苗米与马坝油粘米构成另一分支,支持率高达100%(见图2),并且体现出盘锦大米粳米与另两种籼米在DNA水平上的显著性差异。增城丝苗米与马坝油粘米品种在聚类图上区分为二个独立分支,表现出两种籼米仍存在一定程度的分化。
在3种地理标志大米扩增出的216个条带中,期望寻找到每种地理标志大米的特异性条带,以便可以设计成特异性引物用于地理标志产品的检测。从结果看,216个条带中存在着23个可区分盘锦大米与另两种籼米的特异性条带,其中9个条带在两种籼米10个品种所共有但在盘锦大米中无扩增,有14个条带在盘锦大米5个品种所共有但在另两种籼米中无扩增。此外,还有16个特异性条带为个别品种所特有或为几个品种所特有的条带,但是没发现增城丝苗米7个品种共有但在另两种地理标志大米中无扩增的条带,或是马坝油粘米3个品种所共有但在另两种地理标志大米中无扩增的条带(图1)。
4 讨论
采集了3种受地理标志保护的大米产品的稻谷品种,包括7个增城丝苗米品种、3个马坝油粘米品种和5个盘锦大米品种,使用ISSR标记法分析了大米的遗传分化,由遗传多样性结果及聚类分析结果看,3种大米品种间差异较大,体现出一定程度的分化。
聚类分析结果表明每一种大米内的遗传变异程度远远低于3种大米间的遗传变异,这与样品采集的情况相符。以增城丝苗米为例,其由增城本地野生稻品种经过不断的矮化改良培育而来,不同的品种之间有着或多或少的亲缘关系。香丝苗2号、九七香、穗增香和香丝苗A四个品种亲缘关系相近。香丝苗2号是使用增城高杆香占为母本,增城矮丝苗为父本培育而成。随后,使用香丝苗2号、巴太早香、巴斯马蒂为混合父本,青珍8号为母本,于1997年育成九七香;使用九七香、香丝苗2号为混合父本,增珍占3号或农夫占为母本,于2001年育成穗珍香;使用九七香为父本,增珍占和农夫占为混合母本,于2005年育成香丝苗A。因而这四种增城丝苗米品种在选育谱系中有明显的关联性,亲缘关系相近。以马坝油粘米为例,新银粘是由马坝银粘培育而来的抗病性更强的品种,因而新银粘与马坝银粘的亲缘关系也相近。
3种大米的品种选育多是在本地原有品种的基础上进行培育,如增城丝苗米是由增城本地野生稻品种培育而来,马坝油粘米采用的是在传统马坝油粘米基础上培育而成。据考证,增城丝苗米、马坝油粘米和盘锦大米的最早栽种时期170年、400多年和100多年的栽种历史[7-9],但考虑到本地种野生稻的长期进化,增城丝苗米、马坝油粘米和盘锦大米仍可能由于长期的地理隔离和进化而体现出DNA水平上的差异,这与本实验3种地理标志产品间明显的遗传变异以及在UPGMA聚类分析上3种地理标志产品各成一支的结果相一致。
然而,地理标志大米在其品种的选育及栽种过程中,也会引进外来品种进行栽种或与本地品种杂交育种,以求获得更高的产量、抗性和品质。以增城丝苗米为例,象芽香占是由台山引进的新品种,由于其外观较好、品质较好、稳产,因而在增城市晚稻栽种中广泛使用。另一种增城丝苗米品种九七丝苗为2007年育成,培育过程中引进了外地的占米作为亲本培育。这样情况的存在会对本实验的结果产生影响,从UPGMA树图(图2)上看出,九七丝苗与增城丝苗米的另6个品种的亲缘关系最远,而象芽香占与增城丝苗米的另4个品种有着明显的差异,在聚类图成各成一支。考虑到这种情况,人为地去除九七丝苗与象芽香占的ISSR数据并重新进行UPGMA聚类分析,结果表明增城丝苗米与马坝油粘米形成各自分支的支持率高达90%以上。这种结果暗示着企业在经常性地引进外来品种进行栽种或与本地品种杂交育种时,会影响到本地品种的纯种性能,是否会对本地稻谷品种品质特性的保持产生不利的影响目前还不清楚。
本实验使用23个ISSR引物共扩增出216个条带,依据216个条带可完全区分实验所分析的15个品种。参考 Ken'ichi Ohtsubo 和 Sumiko Nakamura[10]和Kazunori Shinmura等[11]的方法,其均为在构建分子标记谱图的基础上筛选出可以用于鉴定不同大米品种的特异性DNA片段,再在此基础上设计成特异性引物组合。因而,对ISSR引物所扩增出的216个条带进行统计学分析,希望从中寻找3种地理标志产品各自的特异性条带。结果发现,盘锦大米与另两种籼米存在这样的特异性条带,有9个条带为两种籼米共10个品种所共有但在盘锦大米中无扩增,有14个条带为盘锦大米5个品种所共有但在另两种籼米中无扩增。这23个特异性条带应为籼稻与粳稻分化的特异性DNA片段。但无法找到单独一个条带可以完全区分两种籼米即增城丝苗米与马坝油粘米,特异性条带表现为增城丝苗米个别品种特异或是马坝油粘米的个别品种特异,这样的特异性条带有16个,但是通过16个特异性条带协同分析可以完全区分10个籼稻品种。
此次研究将ISSR分子标记技术应用到三种大米品种鉴定中,研究结果找到了盘锦大米区别于另两种籼米的特异性条带,并且找到了16个特异条带可区别增城丝苗米与马坝油粘米,但是用于区分增城丝苗米与马坝油粘米的单独特异条带的查找却还有待深入研究。
[1] Zietkiew icz E,Ra falsk iA ,Labuda D.Genome fingerprinting by simple sequence repeat(SSR)anchored polymerase chain reaction amplification[J].Genomics,1994,20(2):176 - 183.
[2] 朴红梅,李万良,穆楠等.ISSR标记的研究与应用[J].吉林农业科学,2007,32(5):28-30.
[3] 朱岩芳,祝水金,李永平等.ISSR分子标记技术在植物种质资源研究中的应用[J].种子,2010,29(2):55-59.
[4] 欧立军,许栋,廖亚西.水稻籼粳亚种ISSR遗传差异分析[J].怀化学院学报,2009,28(11):22 -25.
[5] 范付华,施文娟,阮仁超,等.23个水稻品种(系)的ISSR指纹图谱及其亲缘关系[J].贵州农业科学,2010,38(9):5 -8.
[6] Su Y J,Wang T,Yang WD,et al.DNA extraction and RAPD analysis of Podocarpus[J].Acta Scientiarum Naturalium Universitis Sunyatseni1998,37:13-18.
[7] 宋东海.利用野生稻培育丝苗米品种的研究[M].广州:广东科技出版社,2007.
[8] 商春艳,王宇.盘锦市优质水稻生产形势与分析[J].北方水稻,2007,3:38-40.
[9] Ohtsubo K,Nakamura S.Cultivar Identification of Rice(Oryza sativa L.)by Polymerase Chain Reaction Method and Its Application to Processed Rice Products[J].Journal of Agricultural and Food Chemistry,2007,55:1501-1509.
[10] Shinmura K,Kanagawa H,Mikami T,et al.Development of multiplex PCR primer sets for the identification of rice varieties[J].Breeding Research,2005,7:87-94.

