易错PCR技术改造扩展青霉脂肪酶*
施碧红,李强,陈明,吴伟斌,施巧琴,吴松刚
1(福建师范大学教育部工业微生物工程研究中心,福建福州,350108)2(福建师范大学生命科学学院,福建福州,350108)
微生物脂肪酶是工业用脂肪酶的一个重要来源。工业催化上要求脂肪酶具有高的活性、稳定性以及对底物的特异性。扩展青霉(Penicillium expansum)FS1884所产碱性脂肪酶是国产商品化的碱性脂肪酶制剂,已被用于洗涤、面包加工、皮革脱脂、鱼类脱脂等领域。但实际应用中仍需进一步提高该脂肪酶的热稳定性和酶活力。扩展青霉FS1884是经过20多代物理化学诱变后的高产突变株,对各种物理化学诱变剂已产生一定的饱和效应,用传统的诱变技术进一步改善其所产脂肪酶的性质或提高酶活已出现困难。且传统的诱变育种方法具有筛选工作量大、突变体遗传背景不清楚等缺点。易错PCR(error-prone PCR)技术直接对目标蛋白质的编码序列进行随机突变,进而定向筛选理想性状的蛋白质,为改良目标蛋白及解析蛋白质的结构与功能关系提供快捷途径。大肠杆菌中,高丝氨酸-琥珀酰转移酶(MetA)是甲硫氨酸合成途径中第1个关键酶,该酶使其无法适应高温的生长环境,Mordukhova[1]通过易错 PCR 技术,筛选到了1株MetA的突变株,其中有5个氨基酸发生了突变:S61T,E213V,I229T,N267D和 N271K,使其能在更高的温度下(44℃)生长。Niu[2]等通过定向进化(易错PCR和DNA Shuffling结合)的研究方法,建立Rhizopus arrhizus脂肪酶随机突变文库,筛选到1株最适作用温度提高10℃的突变株,且突变株在50℃下的半衰期比野生型脂肪酶提高了12倍。Kim[3]通过易错PCR和重叠延伸PCR结合的方法,筛选到了1株突变体,在50℃和55℃放置2h后残余活力分别为30%和10%,而野生型在此条件下均失去活性,突变体的耐热性大大提高。
本研究利用易错PCR技术对扩展青霉脂肪酶基因进行随机突变,建立随机突变文库,通过高通量筛选,期望获得脂肪酶耐热性和酶活力都有所提高的突变株。
1 材料和方法
1.1 材料
1.1.1 菌株和质粒
Pichia pastorisGS115、pPIC3.5K,由福建师范大学林琳教授提供,E.coliJM109由本实验室保存。pPIC3.5k-PEL为本实验室构建并保存[4],PEL为Penicillium expansumlipase的缩写(下同)。
1.1.2 培养基
LB、YPD、MD、BMGY、BMMY、MM 均根据 invitrogen公司Multi-Copy Pichia Expression Kit说明配制,OMM平板为MM平板加上2.5%橄榄油乳化液,橄榄油检验板为含有2.5%橄榄油乳化液的琼脂板。
1.1.3 引物
引物P1,P2用于从pPIC3.5K-PEL质粒上扩增脂肪酶(PEL)基因,其设计原理见参考文献[4]:
P3:5'GACTGGTTCCAATTGACAAGC 3'(5’AOX引物)
P4:5'GGCAAATGGCATTCTGACATCCT 3'(3’AOX引物)
1.2 方法
1.2.1 脂肪酶随机突变库的建立
以含扩展青霉脂肪酶基因的pPIC3.5k-PEL质粒DNA为摸板,P1,P2为引物进行易错 PCR。100μL PCR 体系如下:10 ×PCR Buffer 10 μL,dATP(100 mmol/L)0.2μL,dGTP(100 mmol/L)0.2μL,dCTP(100 mmol/L)1μL,dTTP(100 mmol/L)1μL,MgCl2(25 mmol/L)20 μL ,MnCl2(10 mmol/L)1 μL ,P1Primer(10 μmol/L)1 μL,P2Primer(10μmol/L)1μL,大量抽提 pPIC3.5k-PEL 质粒 DNA 1μL ,TaqDNA Polymerase 2 U,再加入灭菌超纯水到100 μL。扩增程序为:94℃ 2 min;30×(94℃ 1 min,55℃ 1 min,72℃ 1 min);72℃ 7 min。
PCR扩增的突变产物用BamHⅠ和EcoRⅠ双酶切,与经相同双酶切的pPIC3.5k连接,构建表达载体pPIC3.5k-PEL-Mut,,转化大肠杆菌 JM109,构建突变体库。
1.2.2 突变文库的筛选
大量抽提突变体库质粒,经SalⅠ线性化后电转毕赤酵母GS115,涂布于MD平板上,用无菌牙签挑选转化子到含有1%橄榄油乳化液的OMM平板上进行初筛。OMM平板含有0.5%的甲醇,可诱导pPIC3.5K上的AOX启动子,启动脂肪酶的表达,水解橄榄油乳化液后形成水解圈。以含有野生型脂肪酶基因的毕赤酵母GS115(PEL-GS)为对照进行初筛,选择在OMM平板上产生比野生型脂肪酶透明圈大的菌落,进行复筛。
将初筛获得的各菌株分别接种1 mL BMMY液体培养基进行摇管发酵,30℃振荡培养72 h,培养液经过离心后,平板检测酶的活性,耐热性,耐酸性等,筛选优良突变株,摇管复筛中以PEL-GS115为对照。
将摇管复筛获得的优良突变菌株在含甲醇的BMMY培养基中进行摇瓶发酵,诱导毕赤酵母表达脂肪酶,测定脂肪酶活性和耐热性以及最适作用温度等。
1.2.3 表达产物鉴定
平板透明圈法检测脂肪酶活性,在pH9.4的缓冲液中加入2%琼脂粉,并加入2.5%的橄榄油乳化液,制成检验板,用打孔器打孔,每孔加入发酵液10 μL,37℃放置24 h,观察产生的水解圈的大小。
1.2.4 脂肪酶活性的测定
以橄榄油为底物,采用NaOH滴定法[5]测定酶活力。
1.2.5 突变脂肪酶最适作用温度
以橄榄油为底物,在 30、35、40、45、50℃下,采用NaOH滴定法分别测定PEL-GS(野生重组酶)和突变体脂肪酶(ep3-GS)的酶活,以相对酶活确定突变酶的最适作用温度,与对照相比,观察酶活性的提高程度。
1.2.6 突变脂肪酶热稳定性
分别将野生重组酶PEL-GS和突变体脂肪酶在30,35,40,45,50℃ 温育 30 min 后,迅速置冰浴 30 min,在pH9.4,35℃测定酶的残余活力。以未经温度处理的酶液的酶活力为100%,换算不同温度处理后的残余酶活百分比,制作对温度的变化曲线,确定酶的热稳定性。
1.2.7 突变酶的适宜反应pH值
在不同pH的橄榄油平板上,采用透明圈法分别测定突变脂肪酶与野生脂肪酶的酶活力。其中pH 6.0、pH 7.0、pH 8.0橄榄油乳化平板由 K2HPO4-KH2PO4缓冲体系配制,pH 9.4及 pH 10.0橄榄油平板由甘氨酸-NaOH缓冲体系配制。分别以突变酶及野生酶的最大脂肪酶活力为100%,换算相对酶活力,以相对酶活对反应pH值作图,确定突变脂肪酶及野生重组酶的适宜pH值。
1.2.8 正向突变株碱性脂肪酶基因的序列测定、结构预测和突变位点分析
提取高酶活的毕赤酵母突变菌株的基因组DNA[6-7],PCR 扩增及酶切验证突变脂肪酶基因 cDNA,由invitrogen公司进行脂肪酶基因的序列测定。在SWISSMODEL数据库输入突变蛋白的氨基酸序列,预测蛋白的三级结构,分析位点突变效应。
2 结果与分析
2.1 突变脂肪酶基因的获得及突变文库的构建
使用P1、P2为引物在优化后的易错PCR条件下扩增扩展青霉脂肪酶cDNA片段,易错PCR产物如图1,条带大小为900bp左右,符合预期大小。经纯化并双酶切的易错PCR产物,与pPIC3.5k载体连接,转化大肠杆菌JM109,得到了约104个转化子的突变文库。
2.2 突变文库的筛选
将文库的转化子经 LB液体培养,抽提质粒DNA,转化毕赤酵母GS115,涂布在MD平板,经过初筛,筛选了大约4 000株转化了含有目的突变基因的毕赤酵母(图2),其中选择了约300株进行小量的发酵实验,经过72 h的诱导,脂肪酶在毕赤酵母中得到成功表达。分别取10 μL发酵上清液在3种不同条件的检验板进行酶活测定筛选,筛选1株优良突变株ep3。该突变株在橄榄油平板上透明圈达到2.2 cm。
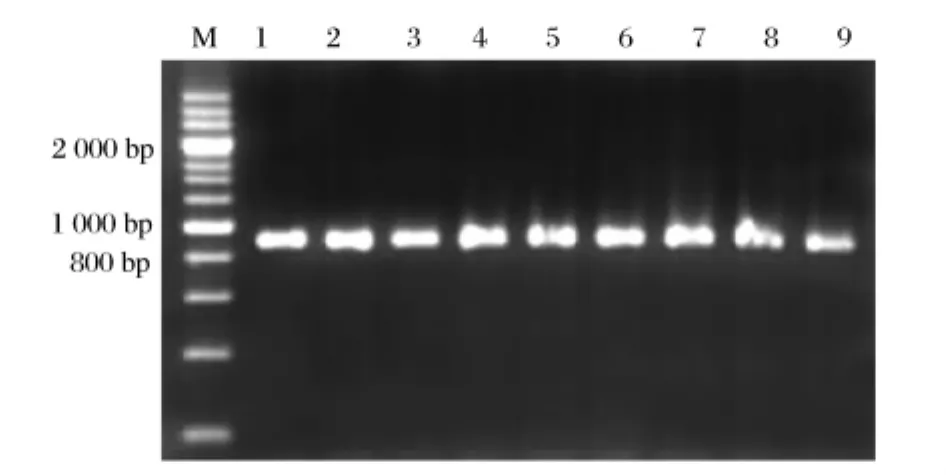
图1 易错PCR产物效果图

图2 突变株在含橄榄油平板上的筛选
2.3 突变脂肪酶的性质
对突变株ep3产生的脂肪酶ep3-GS和野生型重组酶的表达产物(PEL-GS)进行不同温度下酶活测定,发现突变酶的最适作用温度与野生重组酶一致,均为35℃ (图3),35℃时突变酶ep3-GS的活力为3 440 U/mg,比该温度下野生型重组脂肪酶PEL-GS的酶活提高了17%;突变酶及野生重组酶的表达量分别为0.7、0.6 mg/mL。温度对突变脂肪酶的影响显著,50℃时突变酶的活性仅为最适反应温度(35℃)下酶活的30%。
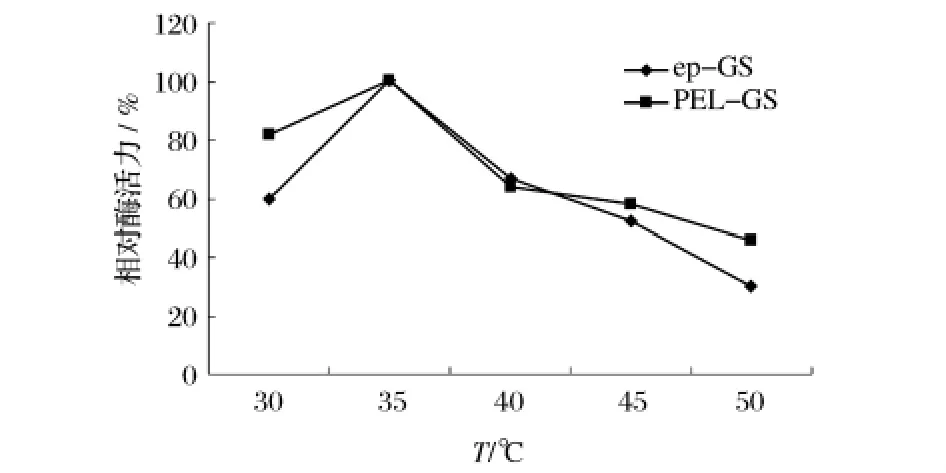
图3 ep3-GS与PEL-GS的最适作用温度
将野生型PEL-GS与突变体脂肪酶ep3-GS分别在不同温度放置30 min,测其残余酶活,结果显示,突变体脂肪酶的热稳定性相比于野生型的脂肪酶有了一定的改善,突变体脂肪酶45℃放置0.5h残余34%的活力,40℃放置0.5 h残余52%的活力,均高于野生型酶的耐热性(图4)。
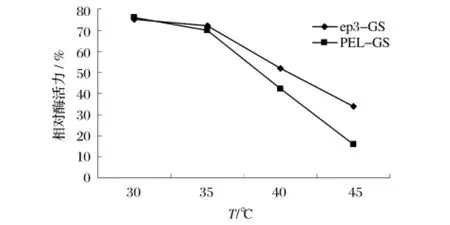
图4 突变体脂肪酶与野生型脂肪酶的耐热性
对突变脂肪酶ep3-GS和野生型重组酶PEL-GS在不同pH值的橄榄油平板上进行酶活测定,发现突变酶的最适pH值没有发生明显变化,与野生重组酶一致,为pH9.4(图5)。

图5 突变体脂肪酶与野生型脂肪酶的最适pH
2.4 突变脂肪酶基因的结构预测与突变位点的检测
利用SWISSMODEL REPOSITORY对突变脂肪酶进行结构预测,发现突变脂肪酶ep3-GS的三级结构与野生型脂肪酶PEL-GS无明显差异。测序结果显示,脂肪酶基因的424位碱基发生突变,即A424G。突变脂肪酶一级结构发生一个氨基酸的变化,142位上的氨基酸由碱性氨基酸赖氨酸突变成酸性氨基酸谷氨酸(LYS142GLU)。
3 讨论
本研究利用易错PCR的随机突变能力,简单快速地在扩展青霉碱性脂肪酶基因上制造核苷酸变化,通过调节易错PCR体系中Mn2+的浓度,调控碱基突变率,使氨基酸突变率稳定在每个基因1~2个的范围内[8],获得了脂肪酶突变基因群,构建扩展青霉碱性脂肪酶基因突变文库。同时利用脂肪酶对橄榄油的水解能力,建立了简单、快捷、经济的高通量平板筛选方法。在含橄榄油底物的各种平板上,直接观察菌落周围或酶液周围的水解圈大小,直观、高效地挑选不同条件下水解能力强的突变株。多个突变体筛选同时进行,提高了筛选效率。
扩展青霉碱性脂肪酶属于α/β型水解酶,含285个氨基酸,其中前27个氨基酸为信号肽和前肽,其余258个氨基酸组成成熟肽[9]。扩展青霉脂肪酶的活性中心是由高度保守的Ser-Asp-His组成的催化三联体,包埋在脂肪酶空间结构中央的β-折叠的环中,形成疏水环境。活性中心外有一段α螺旋形成的盖子,在非激活状态下掩盖催化中心,起保护作用。脂肪酶被激活时,这个盖子就打开,暴露活性中心[9-11]。测序结果显示,142位的赖氨酸突变为谷氨酸(Lys142Glu),由碱性氨基酸变为酸性氨基酸,在空间结构上,该氨基酸位于脂肪酶结构的第4个α螺旋上,该α螺旋紧靠第2个β折叠,142位的赖氨酸突变为酸性的谷氨酸,谷氨酸可与空间位置上靠近的碱性氨基酸形成盐桥,如与第2个β折叠上63位的赖氨酸形成盐桥,盐桥的形成可能对α螺旋的构象翻转起到支持作用,因而促进脂肪酶水解能力提高。
[1] Mordukhova E A,Lee H S,Pan J G.Improved thermostability and acetic acid tolerance ofEscherichia colivia directed evolution of homoserine o-succinyl transferase[J].Appl Environ Microbiol,2008,74(24):7 660 -7 668.
[2] Niu W N,Li Z P,Zhang D W,et al.Improved thermostability and the optimum temperature ofRhizopus arrhizuslipase by directed evolution[J].Journal of Molecular Catalysis B:Enzymatic,2006,43(1 -4):33 -39.
[3] Kim J H,Choi G S,Kim S B,et al.Enhanced thermostability and tolerance of high substrate concentration of an esterase by directed evolution[J].Journal of Molecular Catalysis B:Enzymatic,2004,27(4-6):169-175.
[4] 李强,张建军,蔡容华,等.扩展青霉FS1884脂肪酶基因的定点诱变及其活性分析[J].福建师范大学学报:自然科学版,2009,25(4):105 -108.
[5] 施巧琴.碱性脂肪酶的研究:Ⅰ菌株的分离和筛选[J].微生物学通报,1981(3):108-110.
[6] 高炳淼,长孙东亭,朱晓鹏,等.毕赤酵母重组子PCR模板制备方法的比较[J].中国海洋药物杂志,2009,28(1):7-11.
[7] 剧海,梁东春,郭刚,等.用于PCR实验的毕赤酵母基因组DNA制备方法的比较[J].天津医药,2003,31(5):270-272.
[8] Moore J C,Jin H M,Kuchner O,et a1.Strategies for the in vitro evolution of protein function:enzyme evolution by random recombination of improved sequences[J].Journal of Molecular Biology,1997,272(3):336 -347.
[9] 林琳,谢必峰,杨冠珍,等.扩展青霉PF898碱性脂肪酶cDNA的克隆及序列分析[J].中国生物化学与分子生物学报,2002,18(1):32 -37.
[10] Jaeger K E,Dijkstra B W,Reetz M T.Bacterial biocatalysts:molecular biology,three-dimensional structures,and biotechnological applications of lipases[J].Annu Rev Microbiol,1999,53:315 -351.
[11] Aloulou A,Rodriguez J A,Fernandez S,et al.Exploring the specific features of interfacial enzymology based on lipase studies[J].Biochimica et Biophysica Acta,2006,1 761(9):995-1013.

