稳定表达Cas9蛋白的C2C12细胞株构建







摘要:【目的】优化慢病毒包装体系并建立能稳定表达Cas9蛋白的小鼠成肌细胞系(C2C12),为基因编辑技术研究提供细胞模型和理论参考。【方法】以C2C12细胞为研究对象,设对照组和试验组,调整三质粒(pmd2.g、pspax2和Cas9)用量配比以优化慢病毒包装体系。利用优化后的慢病毒包装体系将表达Cas9蛋白的质粒(lenti-Cas9-puro和lenti-Cas9-Blast)包装成慢病毒感染C2C12细胞。采用药物筛选富集和单克隆分选获得稳定表达Cas9蛋白的C2C12单克隆细胞株。利用基因组鉴定、免疫荧光染色、sgRNA检测等试验验证细胞株构建情况。【结果】慢病毒包装体系优化后,试验组质粒转染效率达90%以上,极显著高于对照组(80%)(Plt;0.01);试验组慢病毒滴度提高至1.8×107 TU/mL,极显著高于对照组(6.2×106 TU/mL)(Plt;0.001)。PCR结果显示,C2C12-Cas9稳转细胞株成功扩增出Cas9、puro和BSD序列,外源Cas9序列成功整合到C2C12细胞基因组中,而野生型的C2C12细胞不存在Cas9序列。免疫荧光染色结果表明,C2C12-Cas9稳转细胞株能表达Cas9蛋白。T7E1酶切和Sanger测序结果表明,整合的Cas9蛋白具有切割活性。【结论】通过调整三质粒用量配比成功优化慢病毒包装体系,提高质粒转染效率和慢病毒滴度。成功构建能稳定表达Cas9蛋白的C2C12细胞株,建立了构建稳转细胞株的相对成熟体系。
关键词:CRISPR/Cas9;C2C12细胞株;慢病毒包装体系;基因编辑
中图分类号:S865.13文献标志码:A文章编号:2095-1191(2024)10-3169-10
Constructing a C2C12 cell line that stably expresses Cas9 protein
LIU Wei-wei1,2,WANG Wei2,YANG Xiao-gan1*,TANG Zhong-lin1,2*
(1College of Animal Science and Technology,Guangxi University,Nanning,Guangxi 530004,China;2Agricultural Genomics Institute at Shenzhen,Chinese Academy of Agricultural Sciences,Shenzhen,Guangdong 518000,China)
Abstract:【Objective】To optimize the lentiviral packaging system and establish a mouse myoblast cell line(C2C12)that stably expressed Cas9 protein,providing a cellular model and theoretical reference for gene editing technology re-search.【Method】Using C2C12 cells as the research subject,control and experimental groups were set up.The dosage ra-tio of three plasmids was adjusted to optimize the lentiviral packaging system,and the plasmid expressing Cas9(lenti-Cas9-puro and lenti-Cas9-Blast)was packaged into lentivirus using the optimized system and infected C2C12 cells.C2C12 monoclonal cell lines with stable Cas9 expression were obtained by drug screening enrichment and monoclonal sorting.Genomic identification,cell immunofluorescence and sgRNA detection were conducted to verify the construction of the cell line.【Result】After optimization of the lentiviral packaging system,the plasmid transfection efficiency of the experimental group reached over 90%,which was extremely significantly higher than the control group(80%)(Plt;0.01);the lentiviral titer of the experimental group increased to 1.8×107 TU/mL,which was extremely significantly higher than the control group(6.2×106 TU/mL)(Plt;0.001).PCR results showed that the C2C12-Cas9 stable cell line suc-cessfully amplified Cas9,puro,and BSD sequences,indicating that the exogenous Cas9 sequence was successfully inte-grated into the C2C12 cell genome,while wild-type C2C12 cells did not contain the Cas9 sequence.Immunofluorescence staining results indicated that the C2C12-Cas9 stable cell line could express Cas9 protein.T7E1 enzyme digestion and Sanger sequencing results indicated that the integrated Cas9 protein had cutting activity.【Conclusion】By adjusting the ra-tiosof the three plasmids,the lentiviral packaging system is successfully optimized,improving plasmid transfection effi-ciency and lentiviral titer.C2C12 cell line that stably expresses Cas9 protein is successfully constructed,establishing a relatively mature system for constructing stable cell lines.
Key words:CRISPR/Cas9;C2C12 cell line;lentiviral packaging system;gene editing
Foundation items:National Key Scientific Research Program of China(2023YFF1001100);National Natural Science Foundation of China(U23A20229);Project of Bama for Talents in Science and Technology(20220027)
0引言
【研究意义】CRISPR/Cas9是生物学和医学领域重要的基因编辑技术(Lander,2016),利用CRISPR/Cas9技术能有目的地改变生物基因组,可用来培育动植物新品种并缩短育种年限(陈璟州等,2022),也为人类疾病治疗提供了新思路(郝静然和王少峡,2021;马孟丹等,2021;史梦然等,2021)。慢病毒载体具有高效的感染能力、广泛的宿主适应性和基因长期稳定表达特性(房恩岳等,2023),是CRISPR/Cas9系统的理想传递工具。优化慢病毒包装体系可提高基因编辑的准确性,减少非特异性整合和基因毒性。构建稳定表达Cas9蛋白的C2C12细胞株,能为骨骼肌发育和基因编辑相关研究提供稳定的细胞模型及技术参考。【前人研究进展】慢病毒载体是以人类免疫缺陷I型病毒(HIV-1)为基础开发的基因治疗载体,与常见的逆转录病毒载体不同,慢病毒载体能感染分裂细胞和非分裂细胞,还能将外源基因有效整合到宿主染色体上,进而实现基因的稳定表达(Tandon et al.,2018;Moreira et al.,2021)。研究表明,慢病毒能有效感染神经元细胞、肝细胞、心肌细胞、肿瘤细胞、内皮细胞和干细胞等多种类型细胞,具有广阔的应用前景(Kiem et al.,2014;Fan et al.,2019;Lamsfus-Calle et al.,2020)。基因编辑技术在动物育种和疾病防治中的应用前景广阔(Kalds et al.,2019)。利用CRISPR/Cas9技术敲除myostatin基因,能提高羊、牛和猪中等家畜的肌肉质量(Han et al.,2014;Lv et al.,2016;Su et al.,2018;Gim et al.,2022);利用CRISPR/Cas9技术敲除CD163后,猪对高致病性猪繁殖与呼吸综合征病毒(PRRSV)表现出完全抗性(Whitworth et al.,2014;Yang et al.,2018);利用慢病毒介导的CRISPR/Cas9基因编辑技术,能成功构建靶向梅花鹿TGF-β1基因敲除载体pBOBI-gRNA2,证明TGF-β1基因缺失能抑制鹿茸软骨细胞的体外增殖(刘明晓等,2019);通过基因编辑敲除ANPEP的猪能抵抗猪传染性胃肠炎病毒(TGEV)感染(Whitworth et al.,2019;Stoian et al.,2020);通过慢病毒感染DF-1细胞筛选稳转Cas9蛋白的阳性细胞,能成功构建具有切割活性的稳定表达Cas9蛋白的DF-1细胞系,有效提高鸡DF-1细胞转染效率(李晓娇等,2023)。【本研究切入点】慢病毒作为基因编辑载体系统展现了巨大潜力,相比于转座子系统和腺病毒系统,慢病毒具有更高的感染效率和稳定性,但也存在包装滴度不高和细胞系构建方法不稳定等问题,但鲜见优化慢病毒包装体系并构建稳定表达Cas9蛋白的小鼠成肌细胞系(C2C12)的研究报道。【拟解决的关键问题】选用C2C12细胞,通过优化慢病毒包装体系,提高慢病毒感染效率,将外源基因敲入C2C12细胞基因组中,构建能稳定表达Cas9蛋白的C2C12细胞株,为基因编辑技术研究提供细胞模型和理论参考。
1材料与方法
1.1试验材料
人胚胎肾细胞系(Human embryonic kidneys 293T,HEK293T)和C2C12细胞购自中国科学院细胞库。慢病毒质粒lenti-Cas9-puro、lenti-Cas9-Blast和辅助质粒pmd2.g、pspax2购自云舟生物科技(广州)股份有限公司;DMED基础培养基、胎牛血清FBS和胰蛋白酶购自美国Gibco公司;细胞转染试剂Jetprime购自法国Polyplus公司;慢病毒助染试剂polybrene购自美国Sigma公司;基因组DNA提取试剂盒购自天根生化科技(北京)有限公司;Cas9蛋白抗体购自美国Cell Signaling Technology公司;T7E1核酸酶购自纽英伦生物技术(北京)有限公司;抗生素Blasticidin S(BSD)和Puromycin(puro)购自美国ThermoFisher Scientific公司。主要仪器设备:PCR仪购自北京东胜创新生物科技有限公司;琼脂糖凝胶电泳仪购自北京六一生物科技有限公司;化学发光成像仪购自广州博鹭腾生物科技有限公司;荧光显微镜购自日本Olympus公司;流式细胞仪和超速离心机购自美国Beckman Coulter公司;克隆环Corning PYREX 6×8mm Cloning Cylinders购自康宁(上海)管理有限公司;超速离心管购自美国Beck-man公司。
1.2引物序列及质粒图谱
细胞株验证所用引物序列信息见表1,慢病毒包装所用辅助质粒和目的质粒图谱见图1。引物委托生工生物工程(上海)股份有限公司合成,质粒图谱信息来源于Addgene网站(https://www.addgene.org/)。
1.3试验方法
1.3.1慢病毒包装体系优化慢病毒包装体系优化步骤:(1)复苏代数靠前且生长状态良好的HEK293T细胞,细胞汇合度达90%左右时进行质粒转染(即慢病毒包装),转染前弃培养基,缓慢加入含2%FBS的无抗生素新鲜培养基。(2)基于预试验慢病毒包装三质粒用量配比摸索及参照Sano等(2019)和Simpson等(2020)的研究结果,将试验组和对照组慢病毒包装三质粒的用量比例分别设为1∶2∶3和1∶3∶4。其中,试验组6孔细胞培养板中各孔转染体系如下:向200μL Jetprime Buffer中按pmd2.g:pspax2∶Cas9=1∶2∶3(pmd2.g质粒0.7μg,pspax2质粒1.4μg,Cas9质粒2.1μg)的比例依次加入对应的去内毒素质粒,轻轻吹打混匀,加入10μL Jetprime regent,再次吹打混匀,室温避光孵育10 min,接种至6孔细胞培养板中,轻轻摇匀放回培养箱。该体系包装的慢病毒上清液可直接感染细胞。试验组100 mm细胞培养皿中转染体系:向500μL Jetprime Buffer中按pmd2.g∶pspax2∶Cas9质粒=1∶2∶3(pmd2.g质粒4.0μg,pspax2质粒8.0μg,Cas9质粒12.0μg)的比例依次加入对应的去内毒素质粒,轻轻吹打混匀,加入40μL Jetprime regent,再次吹打混匀,室温避光孵育10min,接种至100 mm细胞培养皿中,轻轻摇匀放回培养箱。该体系包装的慢病毒经过浓缩后可感染细胞。对照组转染体系:向200μL Jetprime Buffer中按pmd2.g∶pspax2∶Cas9=1∶3∶4(pmd2.g质粒2.5μg,pspax2质粒7.5μg,Cas9质粒10.0μg)的比例依次加入对应的去内毒素质粒,轻轻吹打混匀,加入10μL Jetprime regent,进行慢病毒包装,其余条件不变。(3)慢病毒包装6 h后,6孔细胞培养板各孔和100 mm细胞培养皿分别补加1.0和5.0 mL含1%双抗和2%FBS的培养基,第一次换液24h后观察细胞状态,6孔细胞培养板各孔和100 mm细胞培养皿分别补加1.5和10.0 mL含1%青链霉素和2%FBS的培养基,混匀后放回培养箱。病毒包装后60h时观察细胞状态,显微镜下采集图像。(4)收集上清液(即病毒液),封口膜密封,4℃下3000 r/min离心10 min,0.45μm滤器过滤。6孔细胞培养板中包装的慢病毒于-80℃冰箱保存备用,100 mm细胞培养皿中包装的慢病毒在4℃下30000r/min超高速离心3h,沉淀重悬后于-80℃冰箱保存备用。
1.3.2质粒转染效率及慢病毒感染效率检测慢病毒质粒lenti-Cas9-puro和lenti-Cas9-Blast不携带荧光标签,以表达绿色荧光蛋白(GFP)的慢病毒sgRNA质粒lenti-sgRNA-GFP为参照,分析试验组和对照组慢病毒包装体系的优劣。质粒转染效率检测:质粒转染24h后收集细胞,通过流式细胞仪统计转染效率。慢病毒感染效率检测:将包装好的慢病毒浓缩液感染C2C12细胞,3 d后收集细胞,通过流式细胞仪测定慢病毒的感染效率并计算慢病毒滴度,计算公式如下:
滴度(TU/mL)=细胞数×荧光百分比/病毒体积
式中,病毒体积以mL为单位。
1.3.3 Cas9慢病毒感染C2C12细胞慢病毒浓缩液滴度高,且本研究选用的是易感的C2C12细胞,因此,后续试验仅使用慢病毒上清液进行细胞株构建。C2C12细胞汇合度达30%~50%时进行感染,6孔细胞培养板每孔加入1.5 mL慢病毒上清液、1.5 mL 2%无青链霉素培养基和10.0μg/mL助染试剂poly-brene,充分混匀,每间隔24h更换1次含1%青链霉素和2%FBS的培养基,培养3~5 d。
1.3.4药物筛选及单克隆分选最适药筛浓度摸索:将C2C12细胞接种至6孔细胞培养板中,待细胞汇合度达70%~80%时,弃培养基,PBS洗涤一次,加入含不同浓度梯度puro(0、0.5、1.0、2.0、3.0和4.0μg/mL)和BSD(0、4.0、8.0、12.0、16.0和20.0μg/mL)的10%FBS培养基;培养7 d左右,当出现某个浓度的细胞全部死亡,且小一个浓度的细胞大多数存活时,选择该浓度为最佳致死浓度,用于药筛试验。最终获得puro和BSD的浓度分别为3.0和16.0μg/mL。阳性细胞的药物筛选:药物筛选持续4~7 d仍存活的细胞即为稳定表达Cas9的多克隆C2C12细胞。单克隆分选:采用有限稀释法稀释药物筛选后存活的阳性细胞,通过细胞计数在100mm细胞培养皿中接种50个细胞,待单个细胞生长成细胞团后,利用克隆环和胰蛋白酶消化单克隆细胞团并进行分选扩繁,最终获得稳定表达Cas9的单克隆C2C12细胞,命名为C2C12-Cas9。
1.3.5 C2C12-Cas9稳转细胞株验证(1)提取C2C12-Cas9细胞基因组DNA,使用Cas9、puro和BSD引物进行PCR扩增,产物用2%琼脂糖凝胶电泳检测。(2)免疫荧光染色检测Cas9蛋白是否在C2C12-Cas9细胞内表达。(3)将已验证具有sgRNA活性的sgRNA质粒转染C2C12-Cas9细胞,培养3 d后提取细胞基因组DNA,使用Cas9-KO-test引物进行PCR扩增,产物经Sanger测序和T7E1核酸内切酶检测sgRNA靶标位置是否出现编辑。
1.3.6细胞株构建流程图将表达Cas9的质粒和pmd2.g、pspax2转染HEK293T细胞,培养60 h后收集慢病毒上清液并感染C2C12细胞。药物筛选阳性细胞后,经单克隆分选和扩繁后利用免疫荧光染色、Sanger测序、PCR扩增和T7E1酶切试验获得稳定表达Cas9蛋白的C2C12细胞株(图2)。
2结果与分析
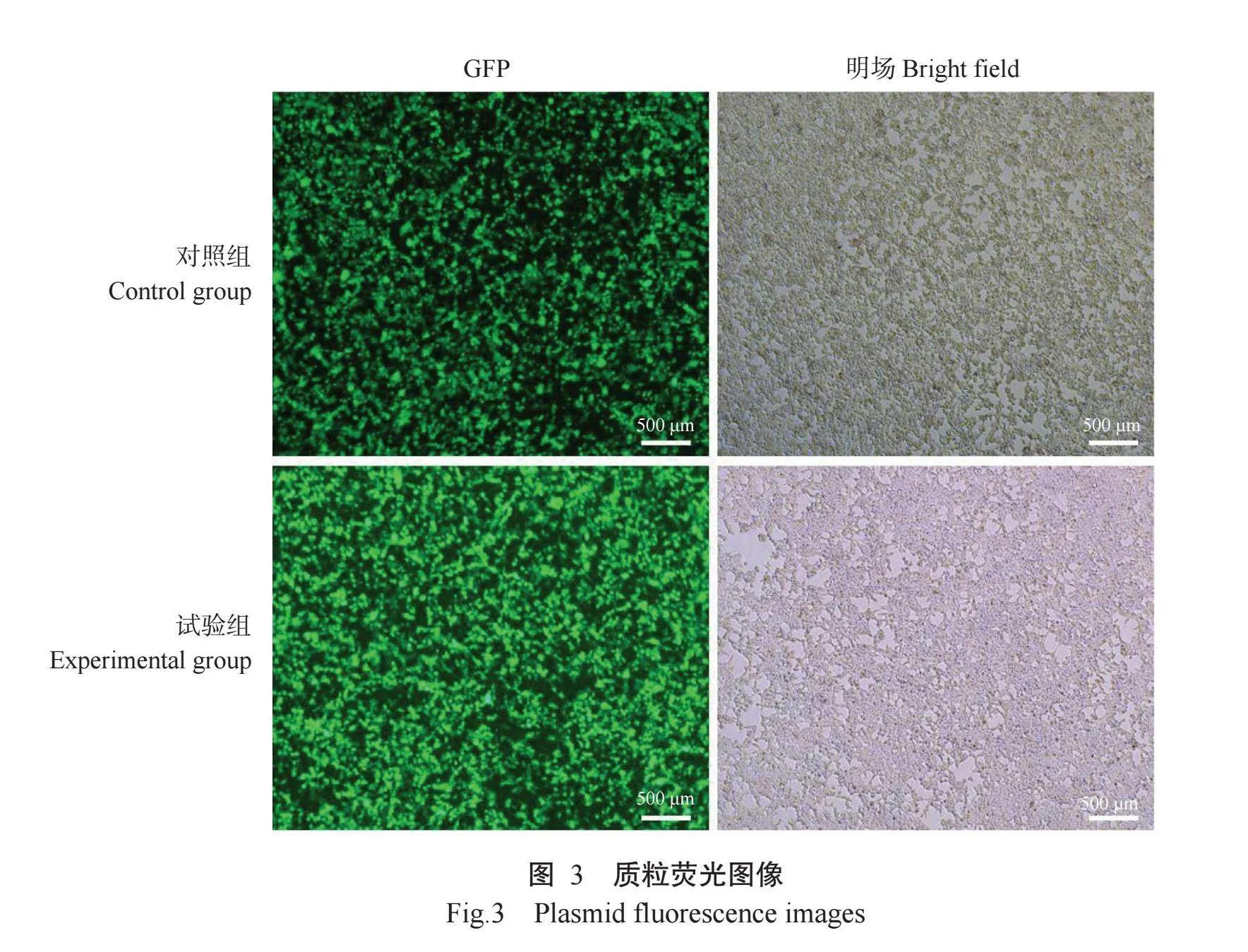
2.1质粒转染效率及慢病毒感染效率检测结果
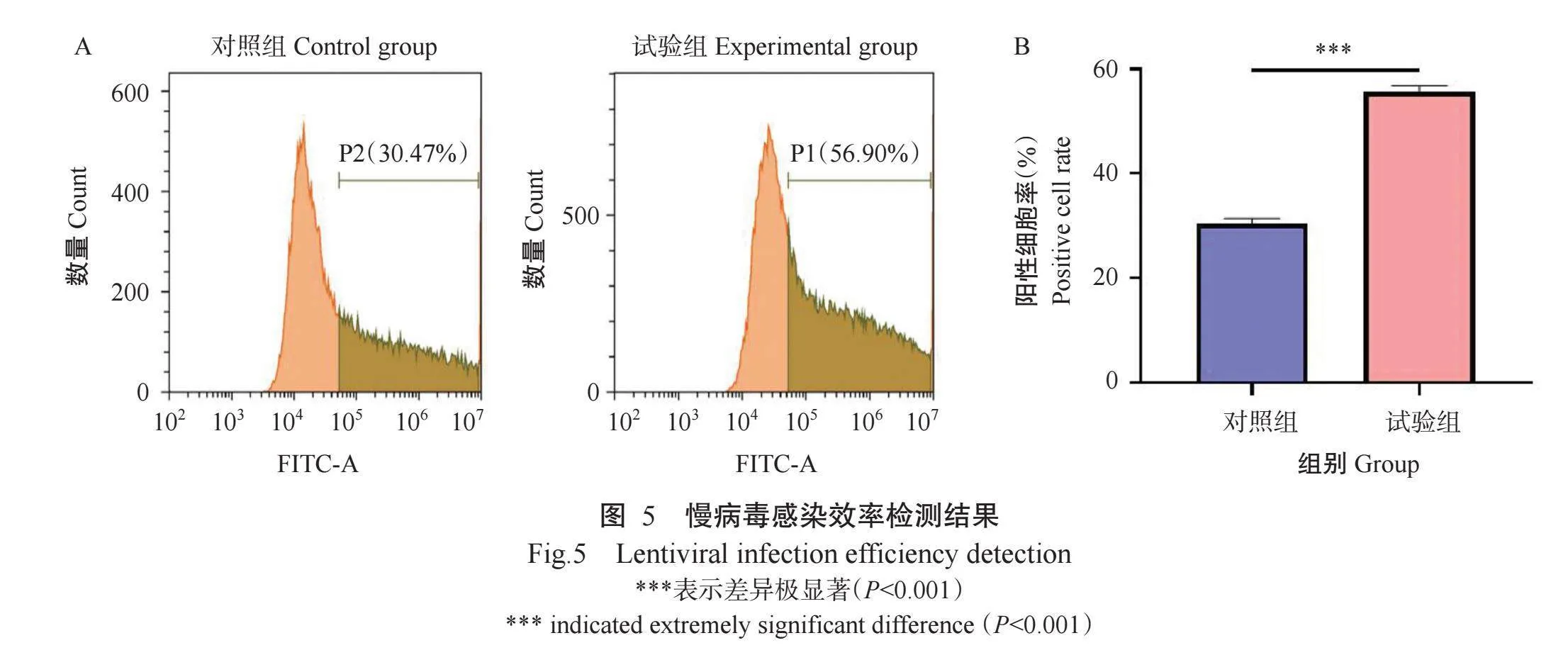
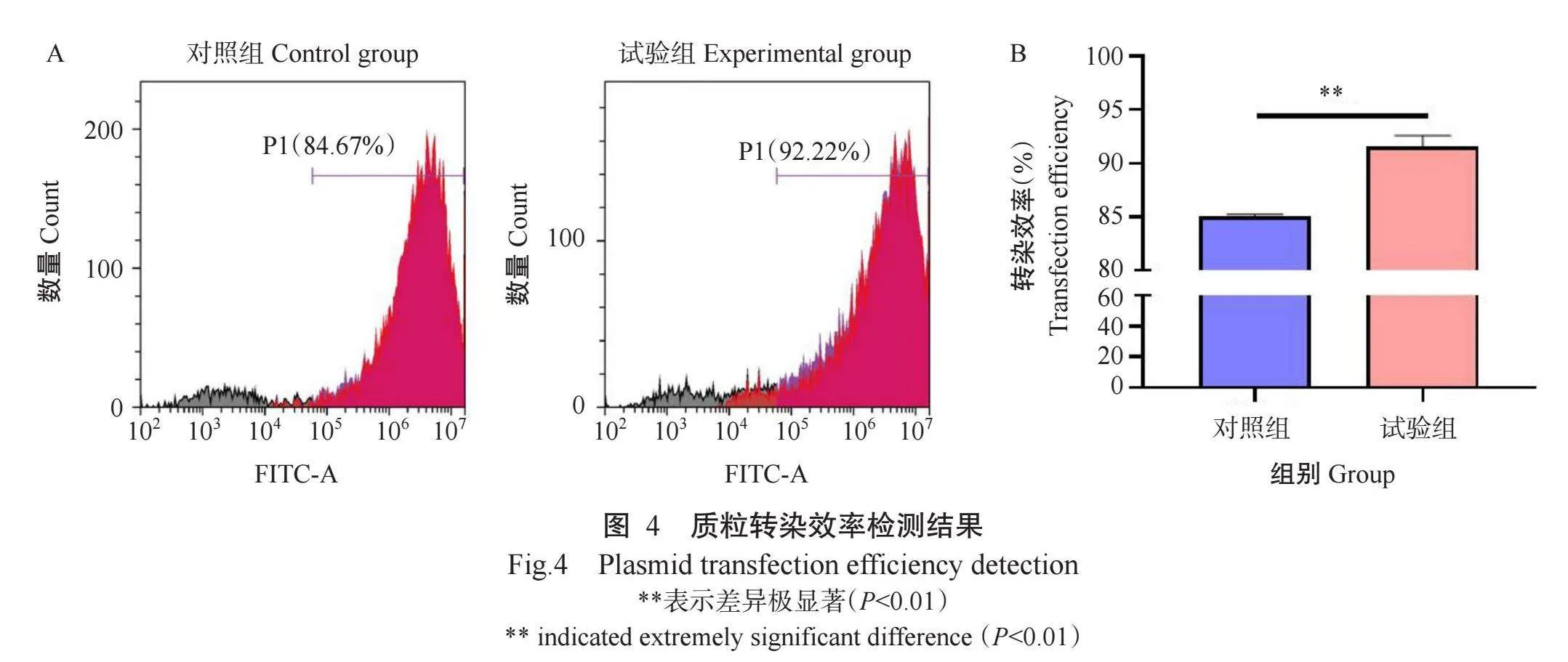
质粒转染效率检测结果如图3和图4所示,试验组转染效率达90%以上,极显著高于对照组(85%左右)(rlt;0.01)。慢病毒感染效率检测结果如图5所示,试验组阳性细胞率极显著高于对照组(rlt;0.001),试验组和对照组慢病毒滴度分别为1.8×107和6.2×106 TU/mL。
2.2 C2C12-Cas9稳转细胞株验证结果
结果如图6所示,C2C12-Cas9稳转细胞株成功扩增出Cas9、puro和BSD序列,而野生型的C2C12不存在Cas9序列。表明成功将Cas9序列整合至C2C12细胞基因组中。
2.3 C2C12-Cas9稳转细胞株免疫荧光检测结果
C2C12-Cas9稳转细胞株免疫荧光检测结果如图7所示,胞质中Cas9呈绿色荧光,细胞核呈蓝色荧光,且绿色和蓝色一一对应。表明C2C12-Cas9稳转细胞株能稳定表达Cas9蛋白,成功分选出单克隆C2C12-Cas9稳转细胞株。
2.4 Cas9编辑活性验证
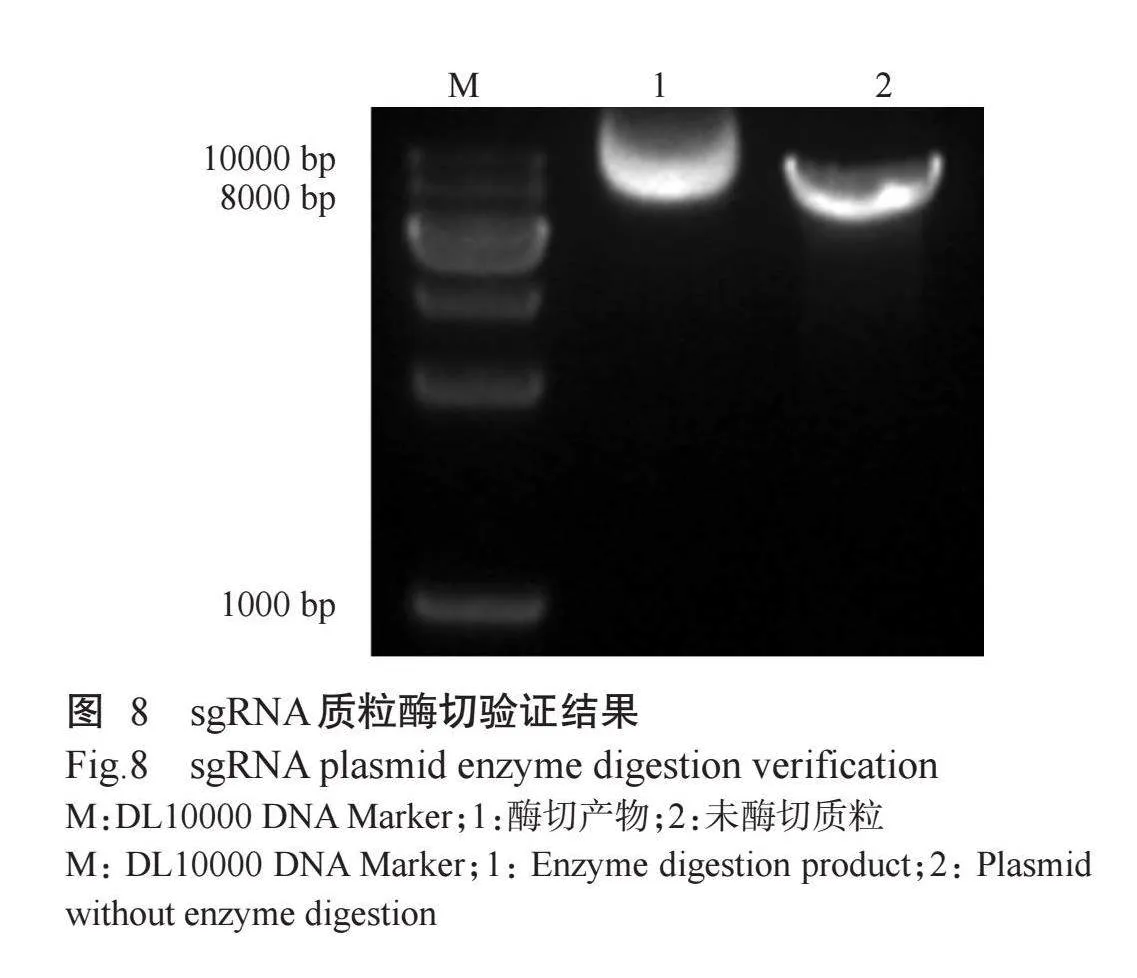
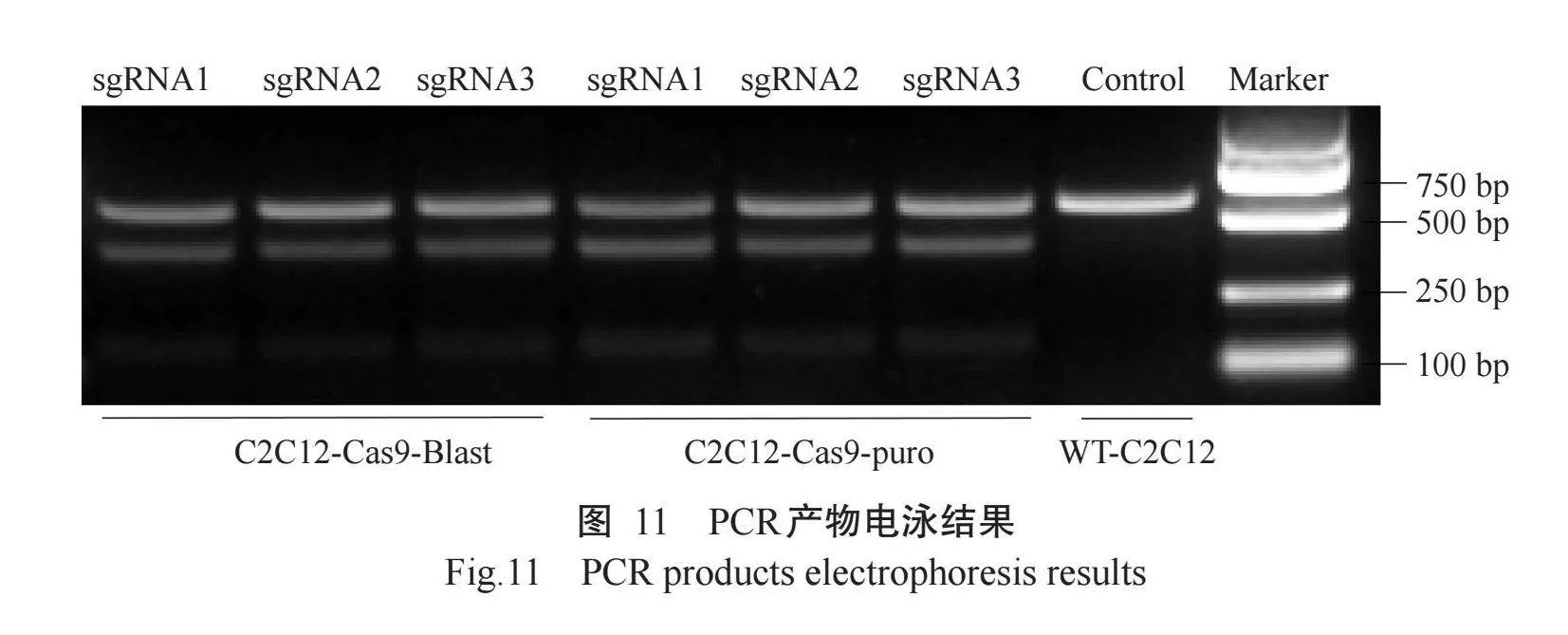
酶切后的sgRNA表达载体经琼脂糖凝胶电泳,确认质粒已被线性化(图8)。随后进行sgRNA引物和酶切载体的酶连反应,Sanger测序结果表明,3条sgRNA序列均已成功构建至sgRNA表达载体上(图9)。将构建好的sgRNA表达载体转染C2C12-Cas9稳转细胞株,提取细胞基因组并进行PCR扩增,扩增产物经测序验证基因编辑位点出现杂峰,表明该位置发生了基因编辑导致的细胞基因型改变(图10)。同时,扩增产物经T7E1酶切后,经琼脂糖凝胶电泳检测,发现部分PCR产物被切割成了2个较短的条带,表明该位置发生了编辑(图11)。以上结果表明,Cas9蛋白发挥了切割作用,利用优化后的慢病毒包装体系成功构建了具有切割活性的C2C12-Cas9稳转细胞株。
3讨论
基因编辑技术能缩短生物育种的年限,同时对疾病的研究具有指导意义(房元杰等,2021;邹菊红等,2021;庄重等,2022)。CRISPR/Cas9技术具有设计简便、易操作、效率高等优势,已成为应用最广泛的基因编辑技术(Knott and Doudna,2018;Anza-lone et al.,2020;Przybyla and Gilbert,2022)。CRISPR/Cas9技术应可用于功能缺失(Loss-of-Function)(如CRISPR KO,CRISPRi)(Gilbert et al.,2014;Zheng et al.,2018)和功能获得(Gain-of-Function)(如CRISPRa)的筛选研究(Chavez et al.,2015)。C2C12细胞是畜禽骨骼肌发育研究中使用最多的细胞系,在该细胞系上进行基因编辑对于了解骨骼肌发育的调控具有重要意义。
与通过转座子系统构建稳转细胞株(Di Matteo et al.,2012;Fonseca et al.,2020)相比,慢病毒包装体系具有许多优势。在外源基因整合到宿主细胞基因组时,转座子系统中的转座酶会造成宿主DNA断裂,给基因组的稳定性带来一定风险(Sandoval-Villegas et al.,2021),而慢病毒系统在整合外源基因时不会出现该现象;转座子系统的成功与否很大程度上取决于系统内2个表达质粒的转染效率,对于原代细胞等难转染的细胞而言,转座子系统的整合效率会大大降低(Holstein et al.,2018;Bishop et al.,2020),而慢病毒包装体系的滴度是可以提升的,使慢病毒几乎可以感染所有细胞(Lundstrom,2018);此外,转座子系统构建的细胞系,外源基因的表达丰度不高,而慢病毒包装体系的整合效率较高,可实现宿主细胞内的外源基因过表达(林馨倩等,2024);CRISPR敲入系统成本较高、方案繁琐、周期长、需设计sgRNA和提供DNA donor等,同时还依赖于Cas9的编辑效率及sgRNA的活性,适用于对外源基因有定点整合需求的细胞系构建(卢媛媛和安艳茹,2023;孙佳鑫,2023),与慢病毒包装体系相比,其适用范围较小。基于慢病毒包装体系的优越性,本研究优化了慢病毒包装体系,结果表明,调整pmd2.g∶pspax2∶Cas9配比为1∶2∶3后,慢病毒质粒的转染效率和滴度明显升高。
慢病毒构建稳转细胞株体系也存在不足,如多克隆细胞株随着传代次数的增丢会失Cas9序列(McCarron etal.,2016),这可能是因为慢病毒整合的细胞相对于野生型细胞在生长活力上无优势,随着传代次数的增加,阳性细胞数目逐渐减少,最终导致Cas9序列丢失;此外,慢病毒细胞株构建过程中还可能产生细胞毒性和免疫反应等问题(Böker et al.,2018;Chavez et al.,2023;Wilk et al.,2024)。本研究通过优化慢病毒包装体系,提高慢病毒滴度,成功构建了能稳定表达Cas9蛋白的C2C12细胞株,为开展外源基因整合至宿主细胞基因组的研究提供了细胞模型。
4结论
通过调整三质粒用量配比成功优化慢病毒包装体系,提高质粒转染效率和慢病毒滴度。成功构建能稳定表达Cas9蛋白的C2C12细胞株,建立了构建稳转细胞株的相对成熟体系。
参考文献(References):
陈璟州,丁一格,宋梓虢,孙珂欣,王小龙.2022.单碱基编辑技术及其在动物育种中的应用研究进展[J].中国畜牧杂志,58(6):53-59.[Chen J Z,Ding Y G,Song Z G,Sun K X,Wang X L.2022.Advances in base editing and research progress in its application in animal breeding[J].Chinese Journal of Animal Science,58(6):53-59.]doi:10.19556/j.0258-7033.20210517-09.
房元杰,张晓爱,魏文康,刘春朋.2021.CRISPR-Cas9技术原理及其在猪的应用研究新进展[J].现代畜牧兽医,(11):92-96.[Fang Y J,Zhang X A,Wei W K,Liu C P.2021.
Principle of CRISPR-Cas9 technology and the new pro-gressofits application in pigs[J].Modern Journal of Ani-mal Husbandry and Veterinary Medicine,(11):92-96.]
房恩岳,张丽,马雪征,胡孔新.2023.慢病毒载体系统及其安全性研究进展[J].病毒学报,39(4):1181-1192.[Fang E Y,Zhang L,Ma X Z,Hu K X.2023.Research progress on lentiviral vector systems and their safety issue[J].Chinese Journal of Virology,39(4):1181-1192.]doi:10.13242/j.cnki.bingduxuebao.004349.
郝静然,王少峡.2021.CRISPR-Cas9技术在疾病治疗中的应用研究进展[J].牡丹江医学院学报,42(6):130-132.[Hao J R,Wang S X.2021.Research progress of CRISPR/Cas9 technology in the treatment of diseases[J].Journal of Mu-danjiang Medical University,42(6):130-132.]doi:10.13799/j.cnki.mdjyxyxb.2021.06.033.
林馨倩,郭紫晶,张瑞,向晗一,林含睿,黃雄挺,陈劲松,张志东,李彦敏.2024.猪源G3BP1基因的稳定敲低PK-15细胞系构建及生物信息学分析[J].微生物学通报,51(3):970-984.[Lin X Q,Guo Z J,Zhang R,Xiang H Y,Lin H R,Huang X T,Chen J S,Zhang Z D,Lin Y M.2024.Con-struction and bioinformatics analysis of a PK-15 cell line with stable knockdown of porcine G3BP1 gene[J].Micro-biology China,51(3):970-984.]doi:10.13344/j.micro-biol.china.230655.
李晓娇,朱新宇,邹娴,严霞,何燕华,罗成龙.2023.慢病毒介导稳定表达Cas9蛋白的鸡成纤维细胞系构建及其活性验证[J].广东农业科学,50(2):125-135.[Li X J,Zhu X Y,Zou X,Yan X,He Y H,Luo C L.2023.Construction of DF-1 cell line of stably expressing Cas9 protein mediated by lentivirus and verification of its activity[J].Guangdong Agricultural Sciences,50(2):125-135.]doi:10.16768/j.issn.1004-874X.2023.02.014.
刘明晓,韩香玉,刘洪运,陈丹阳,李月,胡薇.2019.慢病毒介导的CRISPR/Cas9敲除TGF-β1基因对鹿茸软骨细胞增殖的影响[J].东北林业大学学报,47(1):96-100.[Liu M X,Han X Y,Liu H Y,Chen D Y,Li Y,Hu W.2019.Effects of lentivirus-mediated CRISPR/Cas9 knock-out TGF-β1 gene on proliferation of antler cartilage cells[J].Journal of Northeast Forestry University,47(1):96-100.]doi:10.13759/j.cnki.dlxb.2019.01.019.
卢媛媛,安艳茹.2023.基于CRISPR/Cas9系统构建TIA1基因突变的人诱导多能干细胞系[J].中国细胞生物学学报,45(9):1351-1361.[Lu Y Y,An Y R.2023.Genera-tion of human induced pluripotent stem cell line carrying TlA1 gene mutation by CRISPR/Cas9 system[J].ChineseJournal of Cell Biology,45(9):1351-1361.]doi:10.11844/cjcb.2023.09.0007.
马孟丹,杨育宾,陈延萍,吴果,刘宇辰,黄卫人.2021.CRISPR/Cas9技术及其在肿瘤研究与治疗中的应用[J].生命科学,33(11):1370-1381.[Ma M D,Yang Y B,Cheng Y P,Wu G,Liu Y C,Hang W R.2021.CRISPR/Cas9 and its application in cancer research and therapy[J].Chinese Bulletin of Life Sciences,33(11):1370-1381.]doi:10.13376/j.cbls/2021153.
史梦然,沈宗毅,张楠,王璐瑶,喻长远,杨昭.2021.CRISPR/Cas9系统在疾病研究和治疗中的应用[J].生物工程学报,37(4):1205-1228.[Shi M R,Shen Z Y,Zhang N,Wang L Y,Yu C Y,Yang Z.2021.CRISPR/Cas9 techno-logy in disease research and therapy:A review[J].Chinese Journal of Biotechnology,37(4):1205-1228.]doi:10.13345/j.cjb.200401.
孙佳鑫.2023.基于CRISPR/Cas9系统的外源基因在鸡体细胞中定点敲入的研究[D].呼和浩特:内蒙古大学.[Sun J X.2023.Site-specific knock-in of foreign genesin chicken somatic cellsbased on CRISPR/Cas9 system[D].Hohhot:Inner Mongolia University.]doi:10.27224/d.cnki.gnmdu.2023.001356.
庄重,赵龙,白皓,毕瑜林,黄应权,陈国宏,常国斌.2022.CRISPR/Cas9技术在家禽育种方面的应用[J].中国农业科技导报,24(1):14-23.[Zhuang Z,Zhao L,Bai H,Bi Y L,Huang Y Q,Chen G H,Chang G B.2022.Development of CRlSPR/Cas9 and its application in poultry[J].Journal of Agricultural Science and Technology,24(1):14-23.]doi:10.13304/j.nykjdb.2021.0188.
邹菊红,邹剑伟,申玉建,冯雪萍,连子童,徐建建,宋颖,胡艳,张瑜,黄艳娜,蒋钦杨.2021.基因编辑技术在家畜育种中的研究进展[J].中国畜牧杂志,57(11):45-50.[Zou J H,Zou J W,Shen Y J,Feng X P,Lian Z T,Xu J J,Song Y,Hu Y,Zhang Y,Huang Y N,Jiang Q Y.2021.Research progress of gene editing technology in livestock breeding[J].Chinese Journal of Animal Science,57(11):45-50.]doi:10.19556/j.0258-7033.20201027-04.
Anzalone A V,Koblan L W,Liu D R.2020.Genome editing with CRISPR-Cas nucleases,base editors,transposases and prime editors[J].Nature Biotechnology,38(7):824-844.doi:10.1038/s41587-020-0561-9.
Böker K O,Lemus-Diaz N,Rinaldi Ferreira R,Schiller L,Sch-neider S,Gruber J.2018.The impact of the cd9 tetraspanin on lentivirus infectivity and exosome secretion[J].Molecu-lar Therapy:The Journal of the American Society of Gene Therapy,26(2):634-647.doi:10.1016/j.ymthe.2017.11.008.
Bishop D C,Caproni L,Gowrishankar K,Legiewicz M,Kar-bowniczek K,Tite J,Gottlieb D J,Micklethwaite K P.2020.CAR T cell generation by piggybac transposition from linear doggybone DNA vectors requires transposon DNA-flanking regions[J].Molecular Therapy-Methodsamp;Clinical Development,17:359-368.doi:10.1016/j.omtm.2019.12.020.
Chavez A,Scheiman J,Vora S,Pruitt B W,Tuttle M,Iyer E P R,Lin S L,Kiani S,Guzman C D,Wiegand D J,Ter-
Ovanesyan D,Braff J L,Davidsohn N,Housden B E,Perri-mon N,Weiss R,Aach J,Collins J J,Church G M.2015.Highly efficient Cas9-mediated transcriptional program-ming[J].Nature Methods,12(4):326-328.doi:10.1038/nmeth.3312.
Chavez M,Rane D A,Chen X Y,Qi L S.2023.Stable expres-sion of large transgenes via the knock-in of an integrase-deficient lentivirus[J].Nature Biomedical Engineering,7(5):661-671.doi:10.1038/s41551-023-01037-x.
Di Matteo M,Mátrai J,Belay E,Firdissa T,Vandendriessche T,Chuah M K.2012.PiggyBac toolbox[J].Methods in Mo-lecular Biology,859:241-254.doi:10.1007/978-1-61779-603-6_14.
Fan W H,Shao M Y,Zhang J W,Jin G S,Xu F J,Liu F S.2019.A hybrid nanovector of suicide gene engineered len-tivirus coated with bioreduciblepolyaminoglycosides for enhancing therapeutic efficacy against glioma[J].Advan-ced Functional Materials,29(11):1807104.doi:10.1002/adfm.201807104.
Fonseca J P,Bonny A R,Town J,El-Samad H.2020.Assembly of genetic circuits with the mammalian toolKit[J].Bio-Protocol Journal,10(5):e3547.doi:10.21769/BioProtoc.3547.
Gilbert L A,Horlbeck M A,Adamson B,Villalta J E,Chen Y W,Whitehead E H,Guimaraes C,Panning B,Ploegh H L,Bassik M C,Qi L S,Kampmann M,Weissman J S.2014.Genome-scale CRISPR-mediated control of gene repres-sion and activation[J].Cell,159(3):647-661.doi:10.1016/j.cell.2014.09.029.
Gim G M,Kwon D H,Eom K H,Moon J,Park J H,Lee W W,Jung D J,Kim D H,Yi J K,Ha J J,Lim K Y,Kim J S,Jang G.2022.Production of MSTN-mutated cattle without exogenous gene integration using CRISPR-Cas9[J].Bio-technology Journal,17(7):e2100198.doi:10.1002/biot.202100198.
Han H B,MaY H,Wang T,Lian L,Tian X Z,Hu R,Deng S L,Li K P,Wang F,Li N,Liu G S,Zhao Y F,Lian Z X,2014.One-step generation of myostatin gene knockout sheep via the CRISPR/Cas9 system[J].Frontiers of Agricultural Science and Engineering,1(1):2-5.doi:10.15302/J-Fase-2014007.
Holstein M,Mesa-Nuñez C,Miskey C,Almarza E,Poletti V,Schmeer M,Grueso E,Ordóñez Flores J C,Kobelt D,Wal-ther W,Aneja M K,Geiger J,Bonig H B,Izsvák Z,SchleefM,Rudolph C,Mavilio F,Bueren J A,Guenechea G,Ivics Z.2018.Efficient non-viral gene delivery into human hematopoietic stem cells by minicircle sleeping beauty transposon vectors[J].Molecular Therapy,26:1137-1153.doi:10.1016/j.ymthe.2018.01.012.
Kalds P,Zhou S W,Cai B,Liu J,Wang Y,Petersen B,Son-stegard T,Wang X L,Chen Y L.2019.Sheep and goat genome engineering:From random transgenesis to the CRISPR era[J].Frontiers in Genetics,10:750.doi:10.3389/fgene.2019.00750.
Kiem H P,Arumugam P I,Burtner C R,Fox C F,Beard B C,Dexheimer P,Adair J E,Malik P.2014.Pigtailed macaquesas a model to study long-term safety of lentivirus vector-mediated gene therapy for hemoglobinopathies[J].Mole-cularTherapy-Methodsamp;Clinical Development,1:14055.doi:10.1038/mtm.2014.55.
Knott G J,Doudna J A.2018.CRISPR-Cas guides the future of genetic engineering[J].Science,361(6405):866-869.doi:10.1126/science.aat5011.
Lamsfus-Calle A,Daniel-Moreno A,Ureña-Bailén G,Raju J,Antony J S,Handgretinger R,Mezger M.2020.Hemato-poietic stem cell gene therapy:The optimal use of lentivi-rus and gene editing approaches[J].Blood Reviews,40:100641.doi:10.1016/j.blre.2019.100641.
Lander E S.2016.The heroes of CRISPER[J].Cell,164(1-2):18-28.doi:10.1016/j.cell.2015.12.041.
Lundstrom K.2018.Viral vectors in gene therapy[J].Diseases,6(2):42.doi:10.3390/diseases6020042.
Lv Q Y,Yuan L,Deng J C,Chen M,Wang Y,Zeng J,Li Z J,Lai L X.2016.Efficient generation of myostatin gene mutated rabbit by CRISPR/Cas9[J].Scientific Reports,6:25029.doi:10.1038/srep25029.
McCarron A,Donnelley M,McIntyre C,Parsons D.2016.Chal-lenges of up-scaling lentivirus production and processing[J].Journal of Biotechnol,240:23-30.doi:10.1016/j.jbio-tec.2016.10.016.
Moreira A S,Cavaco D G,Faria T Q,Alves P M,Carrondo M J T,Peixoto C.2021.Advances in Lentivirus Purification[J].Biotechnology Journal,16(1):e2000019.doi:10.1002/biot.202000019.
Przybyla L,Gilbert LA.2022.A new era in functional genom-ics screens[J].Nature Reviews.Genetics,23(2):89-103.doi:10.1038/s41576-021-00409-w.
Sano S,Wang Y,Evans MA,Yura Y,Sano M,Ogawa H,Hori-tani K,Doviak H,Walsh K.2019.Lentiviral CRISPR/Cas9-mediated genome editing for the study of hemato-poietic cells in disease models[J].Journal of Visualized Experiments,(152):10.3791/59977.doi:10.3791/59977.
Sandoval-Villegas N,Nurieva W,Amberger M,Ivics Z.2021.Contemporary transposon tools:A review and guide through mechanisms and applications of Sleeping Beauty,piggyBac and Tol2 for genome engineering[J].Interna-tional Journal of Molecular Sciences,22(10):5084.doi:10.3390/ijms22105084.
Simpson M S,Lencer W I,Luong P.2020.Aquantitative single-cell flow cytometry assay for retrograde membranetraffick-ing using engineered cholera toxin[J].Bio-protocol,10:e3707.doi:10.21769/BioProtoc.3707.
Stoian A,Rowland R RR,Petrovan V,Sheahan M,Samuel MS,Whitworth K M,Wells K D,Zhang J Q,Beaton B,Cigan M,Prather R S.2020.The use of cells from ANPEP knockout pigs to evaluate the role of aminopeptidase N(APN)as a receptor for porcine deltacoronavirus(PDCoV)[J].Virology,541:136-140.doi:10.1016/j.virol.2019.12.007.
Su X P,Cui K Q,Du S S,Li H L,Lu F H,Shi D S,Liu Q Y.2018.Efficient genome editing in cultured cells and embryos of Debao pig and swamp buffalo using the CRISPR/Cas9 system[J].In Vitro Cellularamp;Developmen-tal Biology-Animal,54(5):375-383.doi:10.1007/s 11626-018-0236-8.
Tandon N,Thakkar K N.LaGory E L,Liu Y,Giaccia A J.2018.Generation of stable expression mammalian cell lines using lentivirus[J].Bio-protocol,8(21):e3073.doi:10.21769/BioProtoc.3073.
Whitworth K M,Lee K,Benne J A,Beaton B P,Spate L D,Murphy S L,Samuel M S,Mao J,O'Gorman C,Walters E M,Murphy C N,Driver J,Mileham A,McLaren D,Wells K D,Prather R S.2014.Use of the CRISPR/Cas9 system to produce genetically engineered pigs from in vitro-derived oocytes and embryos[J].Biology of Reproduction,91(3):78.doi:10.1095/biolreprod.114.121723.
Whitworth K M,Rowland R RR,Petrovan V,Sheahan M,Cino-Ozuna A G,Fang Y,Hesse R,Mileham A,Samuel M S,Wells K D,Prather R S.2019.Resistance to coronavirus infection in amino peptidase N-deficient pigs[J].Trans-genic Research,28(1):21-32.doi:10.1007/s 11248-018-0100-3.
Wilk A J,Marceau J O,Kazer S W,Fleming I,Miao V N,Galvez-Reyes J,Kimata J T,Shalek A K,Holmes S,Over-baugh J,Blish C A.2024.Pro-inflammatory feedback loops define immune responses to pathogenic Lentivirus infection[J].Genome Medicine,16(1):24.doi:10.1186/s13073-024-01290-y.
Yang H Q,Zhang J,Zhang X W,Shi J S,Pan Y W,Zhou R,Li G L,Li Z C,Cai G Y,Wu Z F.2018.CD163 knockout pigs are fully resistant to highly pathogenic porcine repro-ductive and respiratory syndrome virus[J].Antiviral Re-search,151:63-70.doi:10.1016/j.antiviral.2018.01.004.
Zheng Y,Shen W,Zhang J,Yang B,Liu Y N,Qi H H,Yu X,Lu S Y,Chen Y,Xu Y Z,Li Y,Gage F H,Mi S L,Yao J.2018.CRISPR interference-based specific and efficient gene inactivation in the brain[J].Nature Neuroscience,21(3):447-454.doi:10.1038/s41593-018-0077-5.
(责任编辑兰宗宝)

