青花菜线粒体基因组组装与序列特征分析
唐征 昂海燕 裴徐梨 荆赞革
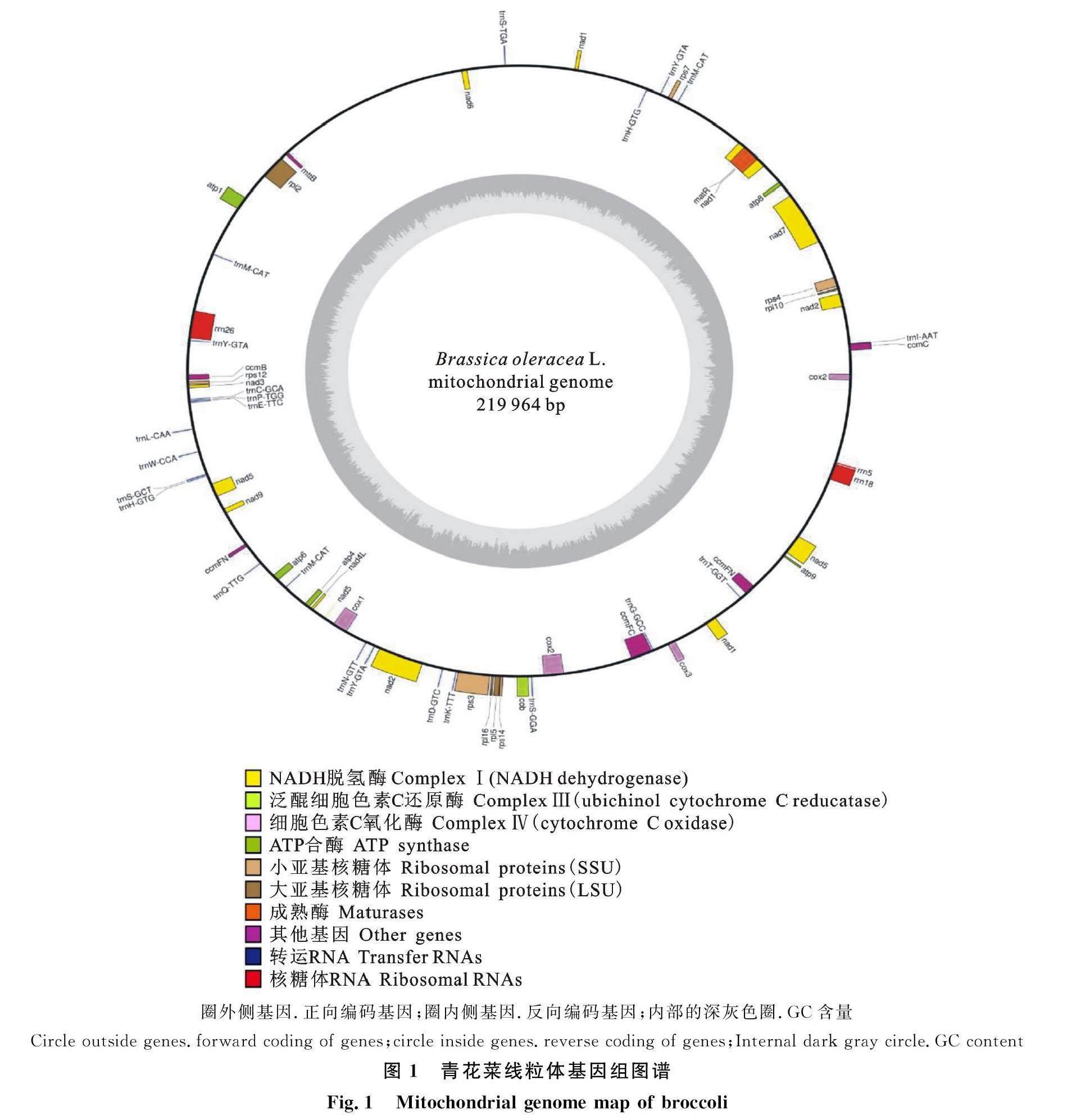

摘 要 为更好地从分子水平了解青花菜,对青花菜进行线粒体基因组组装,并对其序列特征进行分析。青花菜线粒体基因组大小为219 964 bp,GC含量为45.25%。线粒体基因组注释到61个基因,包括33个编码蛋白基因,23个tRNA基因,3个rRNA基因和2个假基因。其中8个基因的核苷酸多样性较高;24个基因无核酸多样性。变异数量最多的基因是 rrn26、 rrn18和 atp1,其变异数量分别为326、277和136。密码子偏好性分析结果显示RSCU值>1的密码子有29个,大多以A/U碱基结尾,表明其密码子更偏向于A/U结尾。基因组序列中共检测到345个重复序列,包括170个正向重复序列和175个回文重复序列。青花菜与花椰菜的线粒体基因组具有极好的共线性,且基因的位置基本一致。青花菜线粒体和叶绿体基因组中共检测到22条同源片段。同源片段总长度为13 355 bp,平均607 bp。
关键词 青花菜;线粒体基因组;组装;序列分析
线粒体是真核生物不可或缺的细胞器,是呼吸作用和能量转换的重要场所。植物线粒体基因组进化慢,保守性高,是对物种进行进化分析的理想工具,可提供系统发育关系、分子进化及物种诊断等信息。自1997年拟南芥线粒体基因组公布以后,研究人员陆续完成了很多重要物种的线粒体基因组测序工作,截至2023年1月,己经有超过3 000多个植物线粒体基因组序列在NCBI中收录。
目前,植物线粒体基因组的研究主要集中在基因组大小、基因注释、密码子偏好性、散在重复序列、核酸多态性、线粒体结构比较[1-3]、叶绿体和线粒体同源序列比较[4]、细胞质雄性不育[5]、遗传多样性[6]以及系统发育分析[7-8]等方面。陈宝玉[9]利用二代测序技术对‘红颊草莓线粒体基因组进行测序和拼接,其大小为109 851 bp,GC含量41.95%,注释到25个基因。栽培莴苣和野生莴苣线粒体的比较分析发现其编码基因富含 A/T碱基,密码子偏好性较弱[10]。辣椒细胞质雄性不育系种质HNUCA00HG的线粒体基因组大小为564 124 bp,注释出77个已知功能基因。同另外两份不育系的线粒体基因组比较发现3个不育系的线粒体可能来自同一个祖先,但明显差异[5]。基于甜瓜线粒体基因组开发SSR标记,对甜瓜种质资源进行遗传多样性分析,结果发现粗网纹厚皮甜瓜类型形成一个独立的分支,其他分支与品种来源相关[11]。
青花菜(Brassica oleracea L. var. italica)是十字花科芸薹属甘蓝种的一个变种,为一二年生植物。目前,关于青花菜线粒体基因组的相关研究还很少。本研究通过组装青花菜线粒体基因组,获得其完整序列,能够更好地从分子水平探究青花菜的遗传和进化等问题,对研究其系统发育关系具有重大意义。
1 材料与方法
1.1 供试材料、DNA提取及测序
以青花菜高代自交系‘KU20-7为试验材料。提取幼嫩叶片的DNA,于南京集思慧远生物科技有限公司进行高通量测序。
1.2 青花菜线粒体基因组序列的组装与注释
使用CANU软件拼接3代数据得到contig序列,利用blast将conting比对到公布植物的线粒体基因组,比对上的conting为种子序列,使用原始数据对序列进行延伸和环化。使用NextPolish对结果进行校正,最后手动校正获得最终的组装结果。
使用blast对青花菜线粒体基因进行比对注释;使用tRNAscanSE(http://lowelab.ucsc.edu/tRNAscan-SE/)对tRNA进行注释;使用OGDRAW(https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)进行青花菜线粒体基因组图谱的绘制。利用自行编写的Perl脚本筛选青花菜线粒体基因的 CDS区域,并计算密码子偏好性。
1.3 青花菜线粒体基因组核酸多样性Pi分析
使用MAFFT软件对不同物种的同源基因序列进行全局比对,采用dnasp5计算每个基因的Pi值,绘制直观图。
1.4 青花菜线粒体基因组散在重复序列分析
将vmatch v2.3.0软件与Perl脚本相结合,以长度≥30 bp,海明距离=3为标准,来鉴定青花菜线粒体基因组的散在重复序列。
1.5 青花菜线粒体序列同源及共线性分析
使用Mauve(http://darlinglab.org/mauve)软件默认参数,对青花菜、花椰菜( KJ820683.1)、甘蓝( KU831325.1)、欧洲油菜( AP018473.1)、埃塞俄比亚芥( JF920287.1)、拟南芥( LUHQ01000021.1)、黑芥( NC029182.1)和芜青( NC-049892.1)等近源物种进行基因组比对,并对其共线性进行分析。
1.6 青花菜线粒体和叶绿体同源序列比较
使用blast软件,设置相似度为70%,E-value为1×10-5查找青花菜叶绿体与线粒体之间的同源序列,利用circos将其可视化。
2 结果与分析
2.1 青花菜线粒体基因组组装与注释
青花菜线粒体基因组大小为219 964 bp,A含量为27.49%,T含量为27.26%,C含量为 22.98%,G含量为22.27%,GC含量为45.25%(图1)。线粒体基因组共注释到61个基因:33个编码蛋白基因,23个tRNA基因,3个rRNA基因,和2个假基因。青花菜线粒体基因组中33个蛋白编码基因总长 29 055 bp,其中 rrn26最长为3 148 bp, atp9和 Rp110最短,为225 bp。
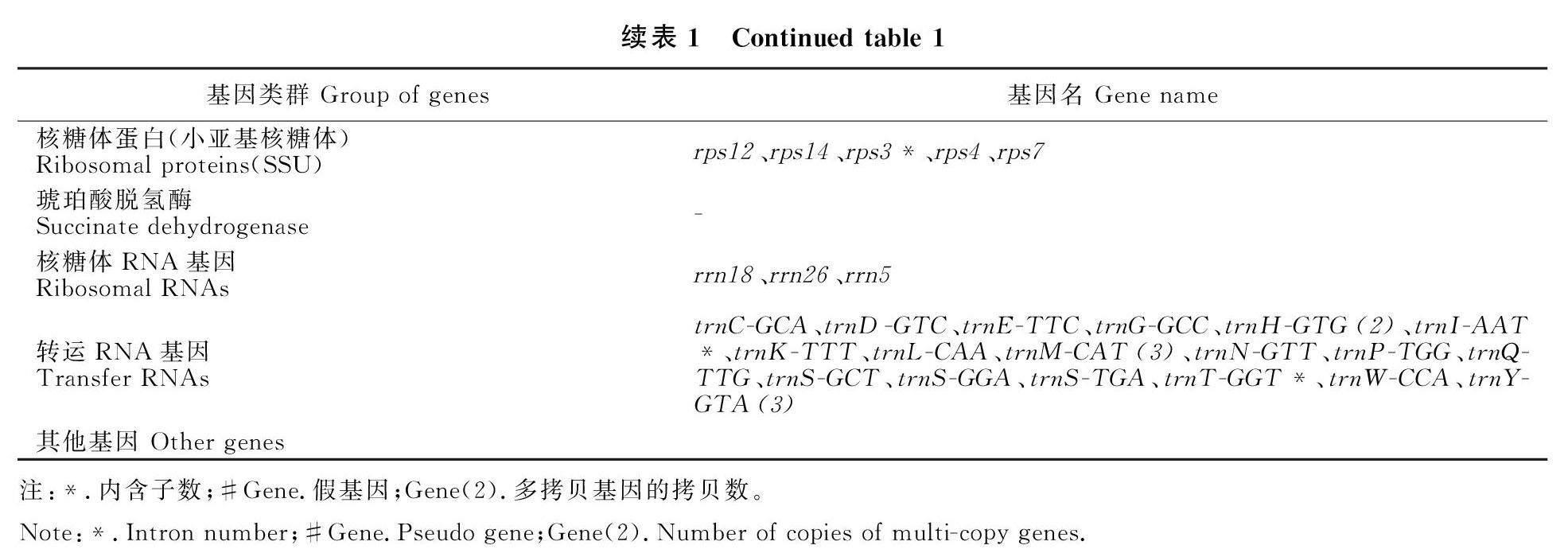
在注释到的61个基因中,复合系统Ⅰ(NADH dehydrogenase)的基因有9个;复合系统Ⅲ的基因为cob;复合系统Ⅳ的基因有 cox1、 cox2和 cox3;ATP合酶基因有 atp1、 atp4、 atp6、 atp8和 atp9;核糖体RNA的基因有 rrn5、 rrn18和 rrn26;转运RNA的基因有trnC-GCA、trnD-GTC、trnE-TTC、trnG-GCC、trnH-GTG、trnI-AAT、trnK-TTT、trnL-CAA、trnM-CAT、trnN-GTT、trnP-TGG、trnQ-TTG、trnS-GCT、trnS-GGA、trnS-TGA、trnT-GGT、trnW-CCA和trnY-GTA;ccmB、ccmC、ccmFc和ccmFn 4个基因参与调控细胞色素c的生物合成;另外, rpl2、 rpl5、 rpl10、 rpl16、 rps12、 rps14、 rps3、 rps4和 rps7基因用于编码核糖体蛋白,余下的基因如matR基因可编码成熟酶,mttB基因可编码转运子。青花菜线粒体基因组中共有11个基因含有内含子(表1)。
2.2 青花菜线粒体基因组密码子偏好性分析
密码子偏好性分析结果表明: RSCU值>1的密码子有29个,其中以A/U碱基结尾的有27个,在青花菜线粒体基因组中的密码子更偏向于A/U碱基结尾。61个基因共有9 209个密码子(除终止密码子外),精氨酸(Arg)、亮氨酸(Leu)和丝氨酸(Ser)的密码子使用量最相近,其中亮氨酸(Leu)的密码子是使用最多的,共995个,约占总密码子数的10.80%;半胱氨酸(Cys)的密码子是使用最少的,共127个,约占总密码子数的 1.38%。丙氨酸的密码子GCU和组氨酸的密码子CAU在青花菜线粒体基因组的使用量最高,偏好性明显;甲硫氨酸的密码子AUG和色氨酸的密码子UGG在青花菜线粒体基因组中无偏好性(表2)。
2.3 青花菜线粒体基因组核酸多样性分析
cox2、 rrn18、 rrn26、 nad2、 atp1、 cox3、trnL-CAA和ccmFC基因的核苷酸多样性较高; trnH-GTG、trnS-TGA、 nad6、trnM-CAT、ccmB、trnC-GCA、trnP-TGG、trnE-TTC、trnW-CCA、trnS-GCT、trnH-GTG、 rpl10、trnQ-TTG、trnM-CAT、 nad4L、trnY-GTA、trnK-TTT、 rpl16、trnS-GGA、trnG-GCC、 atp9、 rrn5、 atp8和trnM-CAT基因无核酸多样性。变异数量较多基因为 rrn26、 rrn18和 atp1,变异数量分别为326、277和136。7个基因的变异数量大于10小于100,21个基因的变异数量小于10,24个基因不存在变异(表3)。
2.4 青花菜线粒体基因组散在重复序列分析
青花菜线粒体基因组共鉴定到345个重复序列,包括170个正向重复和175个回文重复。其中30~39 bp范围内的分布数量最多;40~49 bp范围内的分布数次之。长度为70~79 bp、80~89 bp、90~99 bp和>200 bp范围内的重复序列中的散在重复序列分布数量较少(表4)。
2.5 青花菜线粒体序列同源及共线性分析
线粒体基因组序列同源比对及共线性分析表明:青花菜与花椰菜的线粒体基因组相似度最高,与甘蓝较近,而与其他几个物种相似度相对较远。青花菜与花椰菜的线粒体基因组具有极好的共线性,且基因的排列顺序基本一致。同甘蓝线粒体基因组相比较,可观察到大片段的倒位(图2)。
2.6 青花菜叶绿体和线粒体同源序列分析
青花菜线粒体和叶绿体基因组中共检测到22条同源片段(图3)。同源片段总长度为13 355 bp,平均607 bp。序列长度最长为2 186 bp,最短的仅为42 bp。从长度分布上看,>1 000 bp的有6条,介于100~1 000 bp的有7条,小于100 bp的有9条。7条同源片段E值最小,为零;2条同源片段E值最大,为3.24×10-12。E值为0的同源片段其Bit-score值1 074~3 827,均大于 1 000;E值较大且长度较短的同源片段其Bit-score值较小,介于73.1~163。
3 讨 论
采用形态学方法进行分类和系统发育研究中,由于受到主观性和环境因素的影响,容易得到不一致的结论。自2008年以来,第三代测序技术应运而生,其长测序读长,有效解决了第二代测序读长短和系统偏向性的问题[12]。本研究采用第二代+三代测序技术对青花菜进行线粒体基因组测序,构建了青花菜线粒体基因组图谱,注释了61个基因,明确了青花菜线粒体基因组结构与特征,为之后深入研究青花菜的系统进化提供理论基础。
本研究组装的青花菜线粒体基因组大小为219 964 bp,参考已公开的花椰菜(219 962 bp)和甘蓝(219 975 bp)线粒体基因组的大小,笔者认为此次组装的青花菜线粒体基因组完整度高,且组装质量高。同其近源物种相比较,甘蓝类作物线粒体基因组大小基本相同,而亲缘关系稍远的欧洲油菜(227 181 bp)、埃塞俄比亚芥(232 241 bp)、拟南芥(213 235 bp)和黑芥(232 407 bp)均比青花菜略大一些。参照前人研究结果,不同种类植物的线粒体基因组大小差异较大。本研究中选用的几个物种的大小总体差异不大,可能是由于其为近源物种,线粒体的性质相近造成的。青花菜线粒体基因组的GC含量为45.25%,也符合被子植物GC 含量的一般规律。于前人研究结果相符,植物线粒体基因组的GC含量随着进化而趋于稳定[13]。
有研究表明同源重组是mtDNA快速进化的原因,无论是再塑造还是维持基因组方面[14]。本研究对青花菜线粒体基因多样性进行了研究,结果表明: cox2、 rrn18、 rrn26、 nad2、 atp1、 cox3、trnL-CAA和ccmFC基因的核酸变异度较高,其中 rrn26、 rrn18和 atp1中核酸变异度基因数最多。这为进一步研究同源重组对青花菜mtDNA快速进化提供了一些新的见解。
参考文献 Reference:
[1] 叶丽云,孟国良,何小芳,等.灵芝属不同种线粒体基因组比较分析[J].基因组学与应用生物学,2022,41(8):1723-1731.
YE L Y,MENG G L,HE X F,et al. Comparative analysis of mitochondrial genomes among Ganoderma species[J].Genomics and Applied Biology,2022,41(8):1723-1731.
[2]李玉秋,赵洪锟,谭 化,等.八种植物线粒体基因组结构特征分析与比较[J].生物技术通报,2011(10):156-162.
LI Y Q,ZHAO H K,TAN H,et al.Analysis and comparison on characteristic of mitochondrial genome of eight plant[J].Biotechnology Bulletin,2011(10):156-162.
[3]桂松涛,吴智华,张洪源,等.构建莲线粒体基因组图谱揭示其进化特征[C].2016年全国植物生物学大会摘要集,2016:104.
GUI S T,WU ZH H,ZHANG H Y,et al. Construction of lotus mitochondrial genome map to reveal its evolutionary characteristics[C].2016 National Plant Biology Conference abstract set,2016:104.
[4]王 遂.白桦全基因组测序及分析[D].哈尔滨:东北林业大学,2019.
WANG S.Whole-genome sequencing and analysis of betula platyphylla[D]. Harbin:Northeast Forestry University,2019.
[5]李彩朝,Gagoshidze Zakaria,金威恒,等.辣椒新细胞质雄性不育线粒体基因组的测序及分析[J].分子植物育种,2022,20(16):5250-5257.
LI C CH,ZAKARIA G,JIN W H,et al.Sequencing and analysis of a new cytoplasmic male sterility mitochondrial genome in pepper[J]. Molecular Plant Breeding,2022, 20(16):5250-5257.
[6]ZHAN A,BAILEY S A,HEATH D D,et al.Performance comparison of genetic markers for high-throughput sequencing-based biodiversity assessment in complex communities[J].Molecular Ecology Resources,2014,14:1049-1059.
[7]刘 磊.基于不同基因组序列的十字花科分子系统发育研究[D]. 武汉:武汉大学,2012.
LIU L.Molecular phylogetic relationships of Brassicaceae species inferred from various genomic sequences[D].Wuhan:Wuhan University,2012.
[8]方津津,图尔荪阿依·排祖拉,古丽米热·艾尼瓦尔,等.拉普兰松萝(Usnea lapponica)线粒体基因组特征及其系统进化研究[J].基因组学与应用生物学,2022,42(1):73-83.
FANG J J,PAYZULLA T,ANWAR G,et al. Mitochondrial genome characteristics and phylogeny of Usnea lapponica[J].Genomics and Applied Biology,2022,42(1):73-83.
[9]陈宝玉.‘红颊草莓线粒体基因组测序及序列分析[D]. 南京:南京农业大学,2018.
CHEN B Y. Mitochondrial genome sequencing and sequence analysis of fragaria x ananassa ‘Benihoppe9[D].Nanjing:Nanjing Agricultural University,2018.
[10] 张 扬,孙曙光,李 晴,等.莴苣线粒体基因组密码子使用偏好性分析[J].河南农业科学,2022,51(10):114-124.
ZHANG Y,SUN SH G,LI Q,et al.Codon usage bias analysis of cultivated and wild lettuce mitochondrial genomes [J].Henan Agricultural Science,2022,51(10):114-124.
[11]张跃建,寿伟松,沈 佳.甜瓜(Cucumis melo L.)线粒体基因组标记的开发及应用[J].分子植物育种,2020,18(12):3980-3994.
ZHANG Y J,SHOU W S, SHEN J.Development and application of mitochondrial SSR markers in Cucumis melo L[J]. Molecular Plant Breeding, 2020,18(12):3980-3994.
[12]王久利,郑 旭,邓因子.暴马丁香的叶绿体基因组特征分析[J].阜阳师范大学学报(自然科学版),2022,39(1):55-64.
WANG J L,ZHENG X,DENG Y Z.Characterization of chloroplast genome of Syringa reticulata subsp.amurensis[J].Journal of Fuyang Normal University(Natural Science Edition),2022,39(1):55-64.
[13]叶 楠.银杏线粒体基因组研究[D]. 南京:南京林业大学,2018.
YE N.Study on mitochondrial genome of Ginkgo biloba[D].Nanjing:Nanjing Forestry University,2018.
[14]JOS M,GUALBERTO,DARIA M,et al.The plant mitochondrial genome:dynamics and maintenance[J].Biochimie,2014,100:107-120.
Mitochondrial Genome Assembly and Sequence Characterization in Broccoli
TANG Zheng 1,ANG Haiyan 2,PEI Xuli 2 and JING Zange 2
(1.Wenzhou Vocational College of Science and Technology,Southern Zhejiang Key Laboratory of Crop Breeding,Wenzhou
Zhejiang 325006,China; 2.School of Agriculture and Life Science,Kunming University,Kunming 650214,China)
Abstract In order to better understand it at the molecular level,the mitochondrial genome of broccoli was assembled,and its sequence characteristics were also analyzed in this study. The mitochondrial genome was 219 964 bp,with a GC content of 45.25% in broccoli. 61 genes were annotated,including 33 protein-coding genes,23 tRNA genes,3 rRNA genes,and 2 pseudo genes. Among these genes,eight genes had high nucleotide diversity,while 24 genes had no nucleic acid diversity. The genes rrn26, rrn18 and atp1 had the most variants,with the number of variants being 326,277 and 136,respectively. The codon preference analysis showed that there were 29 codons with RSCU>1,most of them ending with A/U base,indicating that the codon usage tended towards more frequently ending in A/U base. A total of 345 repeats were detected in mitochondrial genome,including 170 forward repeats and 175 palindromic repeats. The mitochondrial genomes of broccoli and cauliflower exhibited excellent collinearity,and the sequence of genes was basically the same.The gene location was also consistent. A total of 22 homologous fragments were detected in the mitochondrial and chloroplast genome in broccoli. The total length of these homologous fragments was 13 355 bp,with an average of 607 bp.
Key words Broccoli;Mitochondrial genome; Assemble; Sequential analysis
Received 2023-02-14 Returned 2023-04-14
Foundation item Joint Basic Research Project for Universities in Yunnan Province(Part)(No.2018FH001-044); Youth Project of Basic Research Project of Yunnan Department of Science and Technology(No.2019-1-C-25318000002236); Natural Science Foundation of Zhejiang Province(No.LY18C150006); Subject of Major Science and Technology Special Project in Zhejiang Province Agriculture(Vegetable New Variety Breeding)(No.2021C02065-4-2);Wenzhou Science and Technology Project(No.2019ZX007-2).
First author TANG Zheng,male,master,associate professor.Research area:molecular biology in vegetables. E-mail:wztangzheng@aliyun.com
Corresponding author JING Zange,male,Ph.D,associate research fellow.Research area:molecular biology in vegetables.E-mail:jingzange@aliyun.com
(责任编辑:潘学燕 Responsible editor:PAN Xueyan)

