艾丁湖可培养好氧嗜盐细菌多样性及抗辐射特征初探
张怡洋 刘阳 阮迪 张璐 刘光琇 陈拓 张威 薛林贵 章高森

摘要:为了解新疆艾丁湖地区中可培养好氧嗜盐细菌的多样性及抗辐射相关特征,以去除盐组分的R2A为基础培养基,在NaCl浓度为50~300 g/L范围内对艾丁湖地区湖边滩涂沉积物和湖周盐碱地中可培养好氧嗜盐细菌进行分离。结果表明,经16S rRNA序列测定和系统进化分析,共得到80株好氧嗜盐细菌。湖周盐碱地的可培养好氧嗜盐细菌数量显著高于湖边滩涂沉积物,物种丰富度也显著高于湖边滩涂沉积物;其中,中度嗜盐菌不仅数量多且多样性丰富。分离所得的好氧嗜盐细菌隶属于厚壁菌门、放线菌门和变形菌门的22个属;其中,厚壁菌门(70.00%)是优势菌门,Bacillus(45.00%)是优势菌属。此外,还分离得到19株高度嗜盐的潜在新种。分离所得的80株可培养好氧嗜盐细菌中有34株在100 J/m2的UVC辐照后存活率(SR)大于10%,属于Bacillus的嗜盐-抗辐射细菌占25.00%;分离自100 g/L NaCl的Streptomyces sp. ALP-8和Streptomyces sp. ASS-29的抗辐射能力最强,SR分别为67.23%和65.73%;极端嗜盐细菌Staphylococcus sp. ASS-5(分离自300 g/L NaCl)、Sediminibacillus sp. ALP-17(分离自250 g/L NaCl)和Kocuria sp. ASS-27(分离自250 g/L NaCl)的SR也在50%以上。
关键词:可培养细菌;嗜盐细菌;多样性;抗辐射;艾丁湖
中图分类号:Q939.9 文献标识码:A
文章编号:0439-8114(2024)06-0044-09
DOI:10.14088/j.cnki.issn0439-8114.2024.06.007 开放科学(资源服务)标识码(OSID):
The preliminary study of the diversity and radiation-resistant characteristics of culturable aerobic halophilic bacteria in the Aiding Lake
ZHANG Yi-yang1,2,3, LIU Yang2,4,5, RUAN Di2,6, ZHANG Lu2,3,5, LIU Guang-xiu2,3,
CHEN Tuo2,4,5, ZHANG Wei2,3, XUE Lin-gui1,2, ZHANG Gao-sen 2,3
(1.School of Biological and Pharmaceutical Engineering, Lanzhou Jiaotong University, Lanzhou 730070, China; 2.Gansu Key Laboratory of Extreme Environmental Microbial Resources and Engineering, Lanzhou 730000, China; 3. Northwest Institute of Eco-Environment and Resources/Key Laboratory of Desert and Desertification,Chinese Academy of Sciences, Lanzhou 730000, China; 4.Northwest Institute of Eco-Environment and Resources/State Key Laboratory of Cryospheric Science,Chinese Academy of Sciences, Lanzhou 730000, China; 5.University of Chinese Academy of Sciences, Beijing 100049, China; 6.College of Geography and Environmental Science, Northwest Normal University, Lanzhou 730070, China)
Abstract:In order to reveal the diversity and radiation-resistant related characteristics of the culturable aerobic halophilic bacteria in Aiding Lake of Xinjiang, R2A with the salt fraction removed was used as the basal medium to isolate aerobic halophilic bacteria in the mudflat sediment and saline land around the Aiding Lake area in the range of 50~300 g/L NaCl concentration. The results showed that 80 strains of aerobic halophilic bacteria were obtained by 16S rRNA sequence and phylogenetic analysis. The number of culturable aerobic halophilic bacteria and the species richness in the saline land around the lake were significantly higher than those in the mudflat sediment. Among them, moderate halophilic bacteria were not only more abundant but also rich in diversity. The isolated aerobic halophilic bacteria belonged to 22 genera of the phyla Firmicutes, Actinobacteria and Proteobacteria. Among them, the Firmicutes (70.00%) was the dominant phylum and the Bacillus (45.00%) was the dominant genus. In addition, 19 potential new species with high salinity were isolated. 34 strains from the isolated 80 aerobic halophilic bacteria had a survival rate (SR) of more than 10% after UVC irradiation at 100 J/m2, and 25.00% of the halophilic radiation-resistant bacteria were Bacillus. Streptomyces sp. ALP-8 and Streptomyces sp. ASS-29 isolated from 100 g/L salt concentration were the most radiation-resistant, and SR was 67.23% and 65.73% respectively. The SR of extreme halophilic bacteria Staphylococcus sp. ASS-5(isolated from 300 g/L NaCl), Sediminibacillus sp. ALP-17 (isolated from 250 g/L NaCl) and Kocuria sp. ASS-27 (isolated from 250 g/L NaCl) was all above 50%.
Key words: culturable bacteria; halophilic bacteria; diversity; radiation-resistant; Aiding Lake
嗜盐菌是一类在高盐环境中生长繁殖的极端微生物,拥有独特的生存机制和多样的代谢产物[1]。根据其对盐的需求和生长模式分为中度嗜盐菌和极端嗜盐菌,中度嗜盐菌能够在5%~20%的NaCl下生长,极端嗜盐菌能够在20%~30%的NaCl下生长。许多嗜盐菌可以在很宽的盐浓度范围内生长,取决于环境和营养因素的影响[2,3]。嗜盐菌能够耐受高盐浓度的主要原因是其拥有特殊的适应性机制,可以在高盐环境中生存和繁殖,这些生存机制使其在制药、药物输送、农业、盐废水处理、可生物降解塑料生产、生物燃料生产等领域都具有一定的应用价值[4-7]。
嗜盐菌广泛存在于细菌和古菌中,已报道的嗜盐菌主要分布于各种高盐环境(海水、盐湖、盐沼、盐田、盐碱地等)[8]。极端嗜盐菌大多是古菌,Tindall等[9]于1984年从东非索达湖中分离出了第一株嗜盐古菌,Zheng等[10]从西藏的盐碱地中分离出6株极端嗜盐古菌。中度嗜盐菌中细菌占比较大,如Sediminibacillus dalangtanensis分离自盐湖沉积物[11],Gracilibacillus salitolerans分离自盐碱地[12],Microbulbifer salipaludis分离自盐沼[13]。中度嗜盐菌可用于降解工业残留污染物或有毒化学品[14]。极端嗜盐菌具有产生水解酶如淀粉酶和脂肪酶的能力,在皮革工业和食品保鲜等领域具有潜在的应用价值[15]。
艾丁湖位于新疆吐鲁番盆地的最低点,海拔低至-153.4 m[16],属于典型的大陆性暖温带干旱沙漠气候,降水稀少,蒸发强烈[17],是中国西北地区著名的高盐湖泊,其湖水含盐量高达300~350 g/L,矿化度最高可达20%,是嗜盐微生物生长的理想环境[18]。由于艾丁湖地处高原干旱区,因此太阳辐射非常强烈,分布其中的嗜盐菌除了应对高盐度挑战外,还存在高度暴露于太阳紫外线辐射的挑战,艾丁湖及其周边地区太阳年辐射量达5 940~6 120 MJ/m2[19]。研究者们对艾丁湖微生物的研究主要集中在嗜盐细菌多样性、抗菌活性[20]、苯酚降解特性[21]、纤维素酶活性[22,23]等方面,尚未见关于艾丁湖地区嗜盐-抗辐射细菌的报道。本研究在对新疆艾丁湖的湖边滩涂沉积物和湖周盐碱地可培养嗜盐细菌分离及多样性研究的基础上,对分离所得的嗜盐细菌进行抗辐射能力筛选,并研究其抗辐射特征和耐盐能力之间的联系。对艾丁湖嗜盐细菌多样性和抗辐射特性的研究不仅有助于深入了解高盐、强辐射环境下微生物的适应机制,还可以为相关抗逆领域的应用开发和抗逆耦联机制的研究提供重要的科学依据和资源。
1 材料与方法
1.1 样品采集
样品于2022年6月采集自新疆艾丁湖(89°26′E,42°67′N)的湖边滩涂沉积物(ASS)和湖周盐碱地(ALP)2个样点,分别取表层5~10 cm的湖边滩涂沉积物和湖周盐碱土样品,每个样点均采集3份平行样品,将采集的样品装于无菌离心管中,于4 ℃低温保存,运至实验室后保存于-20 ℃环境下以供后续菌株分离。
1.2 好氧嗜盐细菌分离培养
培养基选择:以去除盐组分的R2A琼脂培养基(R2A,DSMZ No. 830)为基础培养基,配制成NaCl浓度为50、100、150、200、250、300 g/L的盐基培养基,以此培养基作为筛选好氧嗜盐细菌的功能培养基。菌株培养分离的主要步骤如下:称取5 g沉积物或盐碱土样品于含有10~20颗无菌小玻璃珠的100 mL锥形瓶中;加入45 mL 9 g/L的无菌生理盐水;30 ℃、200 r/min振荡培养30 min后,取上清梯度稀释至10-2、10-3、10-4后吸取100 μL菌悬液均匀涂布于不同盐度的R2A培养基;30 ℃恒温培养2周左右,记录菌落生长情况,根据菌落的形态特征进行纯化,将纯化的菌株用20%的甘油(V/V)保存在-80 ℃冰箱中,以供后续试验使用。以上所有培养试验均设置3个平行。
1.3 基于16S rRNA基因的系统发育分析
分离得到的好氧嗜盐细菌DNA使用DNA提取试剂盒(Omega,美国)提取;使用16S rRNA基因序列通用引物(Forward 27F:5′-AGAGTTTGATCCTGGCTCAG-3′;Reverse 1492R:5′-GGTTACCTTGTT? ACGACITT-3′)对每株可培养细菌进行PCR扩增。PCR反应体系(25 μL):1.0 μL通用引物(0.5 μL 27F + 0.5 μL 1492R),12.5 μL Taq DNA聚合酶,10.5 μL去离子水,1.0 μL单菌落DNA。PCR扩增条件:94 ℃预变性5 min;94 ℃变性1 min,55 ℃退火45 s,72 ℃延伸90 s,循环30次;72 ℃延伸10 min。扩增得到的PCR产物由北京擎科生物科技股份有限公司西安分公司完成16S rRNA基因序列的测定,获得的16S rRNA基因序列在NCBI GenBank保藏获得可培养好氧嗜盐细菌的16S rRNA基因序列保藏编号。利用EZBioCloud在线服务器(http://www.ezbiocloud.net)[24],选取与分离菌株具有较高相似性的16S rRNA作为参比菌株,利用MEGA11.0[25]中的邻近法[26]构建系统发育树。
1.4 好氧嗜盐细菌的抗UVC能力评估
以OD600 nm=1的菌悬液作为试验样品,将细菌悬液逐级稀释至浓度为10-3、10-4、10-5,各取100 μL均匀涂布于R2A琼脂平板上,然后放入紫外灯箱进行UVC辐照处理,剂量为100 J/m2。以大肠杆菌(Escherichia coli)为阴性对照,耐辐射奇异球菌(Deinococcus radiodurans)为阳性对照,设置3组平行试验。将平板放置于30 ℃恒温培养箱培养7~14 d后,计算其存活率(Survival rates, SR),计算式如下。
SR = [NrN0] (1)
式中,Nr为辐照后菌落个数;N0为辐照前菌落个数[27]。
1.5 数据分析
用CLUSTAL X软件进行16S rRNA基因序列比对[28],系统发育树由MEGA11.0软件绘制;其余统计分析与图的绘制由SPSS 24.0和Origin 2023b version软件完成。
2 结果与分析
2.1 可培养好氧嗜盐细菌的分离与类群分析
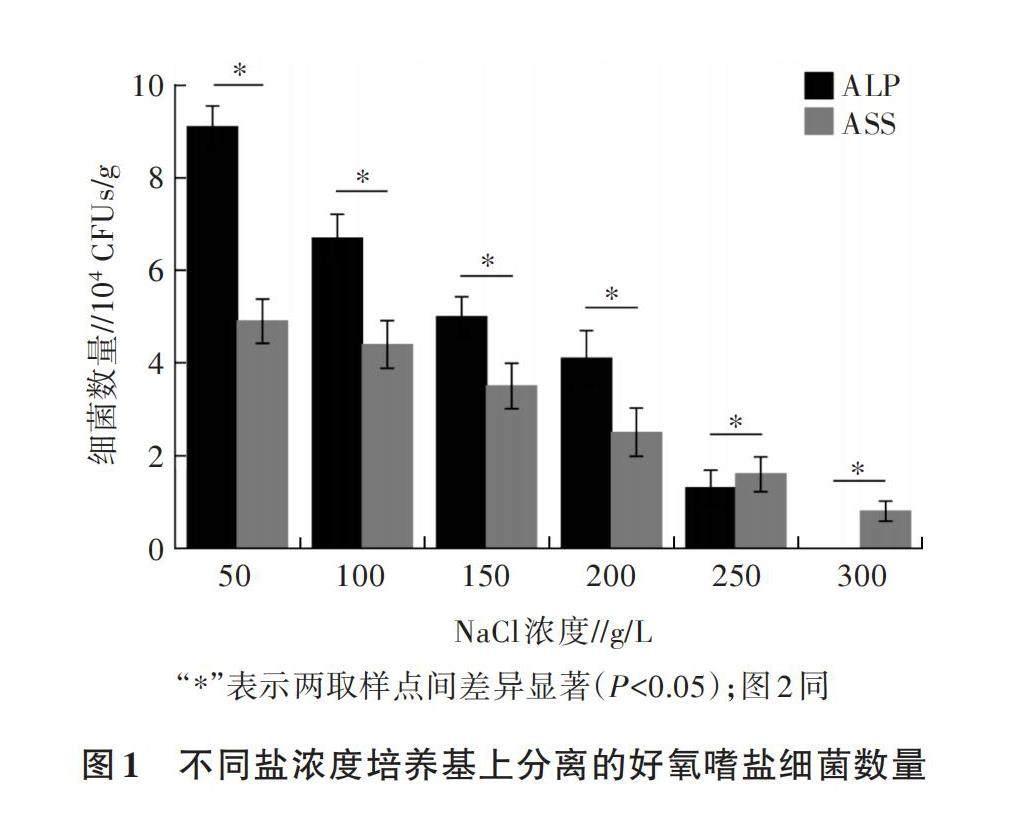
使用添加50、100、150、200、250、300 g/L NaCl的R2A盐基培养基进行好氧嗜盐细菌的分离筛选。对不同浓度NaCl培养基上可培养好氧嗜盐细菌的数量进行统计发现,ASS和ALP两个样地培养的好氧嗜盐细菌数量随着培养基中NaCl浓度的增加逐渐减少,具体表现为50 g/L>100 g/L>150 g/L>200 g/L>250 g/L>300 g/L(图1)。当NaCl浓度为50~200 g/L时(图1),ALP的可培养细菌数量均显著大于ASS(P<0.05),但随着NaCl浓度的升高,ASS可培养好氧嗜盐细菌的数量显著高于ALP(P<0.05);NaCl浓度为250 g/L和300 g/L时,ASS的可培养嗜盐细菌数量分别为1.63×104 CFUs/g和0.82×104 CFUs/g,ALP在 NaCl浓度为300 g/L的基础培养基上未分离得到菌株。综上可知,艾丁湖湖边滩涂沉积物和湖周盐碱地都是嗜盐细菌的良好生态栖息地;相较于湖周盐碱地,湖边滩涂沉积物中分离得到的嗜盐细菌对盐浓度具有更强的耐受能力。
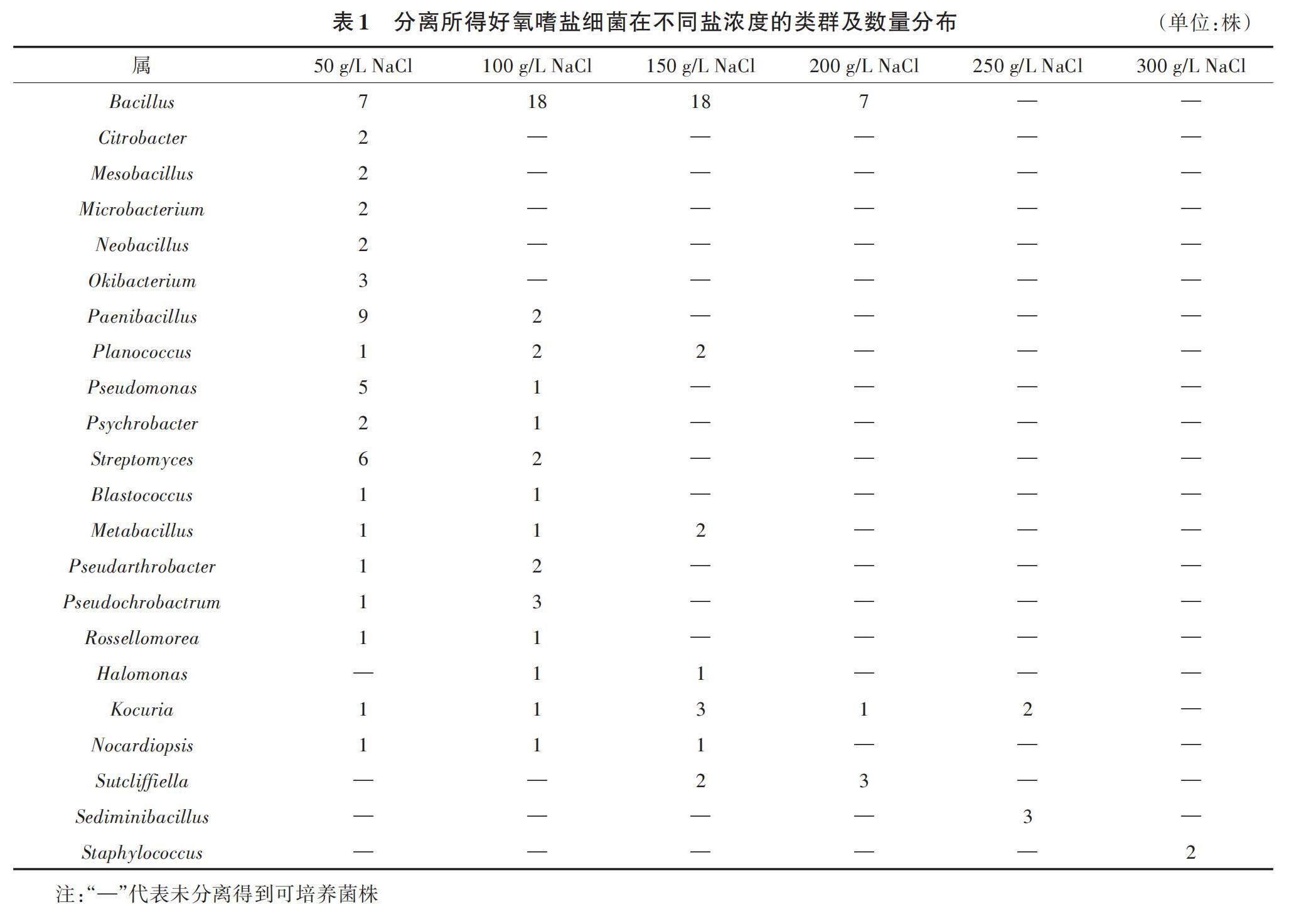
基于ASS和ALP分离得到的所有嗜盐细菌菌株形态、颜色等特征的不同,用平板划线法对不同的菌落进行纯化,共获得132株好氧嗜盐细菌(表1)。50~300 g/L NaCl分别分离得到48株(36.36%)、37株(28.03%)、29株(21.97%)、11株(8.33%)、5株(3.79%)、2株(1.52%)菌株,根据16S rRNA基因序列采用EZBioCloud(http://www.ezbiocloud.net)对132株菌株进行物种鉴定。类群分析表明,在属水平上,50 g/L NaCl分离得到的类群最多,共有18个属,分别为Bacillus(芽孢杆菌属)、Citrobacter(柠檬酸杆菌属)、Mesobacillus、Microbacterium(微杆菌属)、Neobacillus、Okibacterium、Paenibacillus(类芽孢杆菌属)、Planococcus(游动球菌属)、Pseudomonas(假单胞菌属)、Psychrobacter(嗜冷杆菌属)、Streptomyces(链霉菌属)、Blastococcus、Metabacillus(副芽孢杆菌属)、Pseudarthrobacter(类节杆菌属)、Pseudochrobactrum(假苍白杆菌属)、Rossellomorea、Kocuria(考克氏菌属)、Nocardiopsis(拟诺卡氏菌属),其中Paenibacillus的菌株数量最多。100 g/L NaCl分离得到的类群有14个属,分别为Bacillus、Paenibacillus、Planococcus、Pseudomonas、Psychrobacter、Streptomyces、Blastococcus、Metabacillus、Pseudarthrobacter、Pseudochrobactrum、Rossellomorea、Halomonas、Kocuria、Nocardiopsis。150 g/L NaCl分离得到的类群有7个属,分别为Bacillus、Planococcus、Metabacillus、Halomonas、Kocuria、Nocardiopsis、Sutcliffiella。200 g/L NaCl分离得到的类群有3个属,分别为Bacillus、Kocuria、Sutcliffiella。100、150、200 g/L NaCl分离得到的嗜盐细菌中,均为Bacillus的细菌数量最多。250 g/L NaCl分离得到的类群有2个属,分别为Kocuria、Sediminibacillus(沉积物芽孢杆菌属)。300 g/L NaCl分离得到的类群只有1个属,为Staphylococcus(葡萄球菌属)。
通过比较分析发现,Kocuria的菌株能够生存的盐浓度范围较广,能够耐受50~250 g/L的NaCl;其次是Bacillus的菌株,能够耐受50~200 g/L的NaCl;Planococcus和Metabacillus均可在50~150 g/L的NaCl下生长。Halomonas在较低盐浓度下未分离得到,却可以耐受100~150 g/L NaCl;同样,Sutcliffiella菌株在50~100 g/L NaCl下未分离得到,却能够在150~200 g/L NaCl下生长。Sediminibacillus和Staphylococcus分别仅在250 g/L和300 g/L NaCl的条件下分离得到(表1)。以上结果表明,不同盐浓度所分离菌株的数量和类群均有不同,能耐高盐浓度的极端嗜盐细菌在数量和类群上相对较少,而50、100、150 g/L NaCl分离得到的中度嗜盐细菌在属水平上的物种丰富度较高。此外,高盐环境中不同系统发育水平的可培养细菌对不同盐浓度的耐受能力不同,生长适应性也不同。
2.2 可培养好氧嗜盐细菌的多样性和系统发育分析
对ASS和ALP的可培养细菌的菌落数量进行统计发现,ALP的可培养数量(2.62×105 CFUs/g)显著高于ASS(1.81×105 CFUs/g)(P<0.05,图2)。基于16S rRNA基因测序结果,以16S rRNA基因序列相似度100%为相同菌株的标准,去除132株嗜盐细菌中的重复菌株后,最终得到80个系统发育型(Genotype),16S rRNA基因序列结果保藏到NCBI GenBank。采用EZBioCloud进行序列比对后进行系统发育分析发现,80个系统发育型分属于厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)和变形菌门(Proteobacteria)。其中,厚壁菌门是优势菌门,占总分离菌株的70.00%,放线菌门次之,占总分离菌株的16.25%,变形菌门仅占总分离菌株的13.75%(图2)。
图3为用邻近法构建的系统发育树,分析表明,厚壁菌门分离到的56株好氧嗜盐细菌分属于Bacillus、Paenibacillus、Planococcus、Metabacillus、Sutcliffiella、Mesobacillus、Neobacillus、Rossellomorea、Sediminibacillus和Staphylococcus共10个属,Bacillus是其中的优势菌属。变形菌门的13个系统发育型分属于Kocuria、Streptomyces、Blastococcus、Microbacterium、Nocardiopsis、Okibacterium和Pseudarthrobacter共7个属,Kocuria和Streptomyces是其中的优势菌属。变形菌门包括11株嗜盐细菌,分属于Pseudomonas、Pseudochrobactrum、Psychrobacter、Citrobacter和Halomonas共5个属,Pseudomonas是其中的优势菌属。艾丁湖中可培养好氧嗜盐细菌的多样性丰富,是蕴藏嗜盐细菌的天然优质菌种资源库,Bacillus的嗜盐细菌最多,占分离菌株的45.00%。
2.3 潜在新型好氧嗜盐细菌分析
通过16S rRNA基因序列比对和系统发育分析,发现有19株可培养好氧嗜盐细菌为潜在新种,与其最相似模式菌株的16S rRNA基因序列相似度低于原核生物的物种划分阈值(98.65%)[29](图3)。所有潜在新种在ASS中的数量明显高于ALP;ASS发现潜在新种13株,ALP仅有6株(图4)。其中,ASS发现的潜在新种分属于Bacillus、Halomonas、Nocardiopsis、Paenibacillus、Pseudochrobactrum、Rossellomorea、Staphylococcus和Streptomyces共8个属;ALP发现的潜在新种分属于4个属,分别为Bacillus、Neobacillus、Planococcus、Sutcliffiella。潜在新种的系统发育分析表明,Bacillus和Paenibacillus的潜在新种较多;Sutcliffiella和Bacillus的潜在新种对200 g/L的极端高盐浓度具有良好的耐受性。综上,艾丁湖湖边滩涂和湖周盐碱地均发现大量对高盐浓度有抗性的潜在新种,是发掘新型嗜盐细菌资源的有利环境。
2.4 好氧嗜盐细菌的抗辐射与耐盐能力相关性分析
将分离得到的80株好氧嗜盐细菌进行辐射强度为100 J/m2的UVC辐照并以其存活率评估菌株的抗辐射能力。结果表明,阴性对照大肠杆菌(Escherichia coli)在辐射强度为100 J/m2 UVC辐照下的SR为0,阳性对照耐辐射奇异球菌(Deinococcus radiodurans)的SR为67.10%,UVC辐照后SR在10%以上的菌株有34株(图5)。SR在10%以上的菌株与其分离盐度的分析结果表明(图5),抗辐射能力最强的2株菌分离自含100 g/L NaCl的培养基,分别为Streptomyces sp. ASS-29和Streptomyces sp. ALP-8,在辐射强度为100 J/m2的UVC辐照下SR分别为65.73%和67.23%;其次是菌株Staphylococcus sp. ASS-5、Sediminibacillus sp. ALP-17和Kocuria sp. ASS-27,SR均高于50%,其中Staphylococcus sp. ASS-5分离自300 g/L NaCl,Sediminibacillus sp. ALP-17和Kocuria sp. ASS-27分离自250 g/L NaCl。SR在40%~50%的菌株均分离自200 g/L NaCl。SR在30%~40%的菌株均分离自100~150 g/L的NaCl。SR在20%~30%的菌株,除了Streptomyces sp. ASS-3分离自50 g/L NaCl,SR为22.36%,其余均分离自100 g/L NaCl。SR在10%~20%的菌株均分离自50 g/L NaCl的R2A盐基培养基上。以上结果表明,常年受到强太阳辐照的盐湖艾丁湖分离得到的嗜盐细菌均具有较强的抗辐射能力,强抗辐射能力的菌株大多分离自高盐浓度的培养基,并且嗜盐细菌的抗辐射能力与盐耐受浓度有关,与其系统发育地位也有紧密的联系。
3 讨论
本研究对新疆艾丁湖的湖边滩涂沉积物和湖周盐碱地的好氧嗜盐细菌进行分离筛选,使用含有50、100、150、200、250、300 g/L NaCl的R2A盐基培养基对其好氧嗜盐细菌的多样性进行分析;此外,对分离出的所有可培养好氧嗜盐细菌进行抗辐射能力筛选,并研究其抗辐射和耐盐能力之间的相关性。研究发现,湖周盐碱地的可培养好氧嗜盐细菌数量显著高于湖边滩涂沉积物。不同盐浓度所分离菌株的数量和系统发育型均有不同。随着培养盐浓度的增加,好氧嗜盐细菌多样性和数量显著降低,极端嗜盐细菌的数量和系统发育型相对较少,中度嗜盐细菌不仅数量多且多样性丰富。在高盐浓度下(250、300 g/L NaCl),湖周盐碱地可培养好氧嗜盐细菌数量显著低于湖边滩涂沉积物,湖周盐碱地在NaCl浓度为300 g/L的培养基上未分离得到菌株,以上结果表明湖边滩涂沉积物中的好氧嗜盐细菌具有更强的盐耐受能力。这可能是因为极端嗜盐细菌的生长不仅需要相对较高的Na+,而且其中大多数细菌的生长需要Mg2+等其他盐基阳离子的参与,而轻度嗜盐细菌的生长通常不依赖于Na+以外的其他盐基阳离子[30,31]。此外,湖边滩涂和湖周盐碱地环境的异质性也是影响嗜盐细菌生存的关键因素[32,33]。
根据16S rRNA基因测序比对结果,分离得到的好氧嗜盐细菌隶属于厚壁菌门、放线菌门和变形菌门,其中厚壁菌门(70.00%)是优势菌门,这与其他干旱高盐环境中的研究结果相似[34,35]。这可能是因为厚壁菌门大多为革兰氏阳性菌,且在干旱的沙漠环境中也存在大量的厚壁菌门的细菌,其厚的细胞壁能够耐受极强的膜渗透压变化,高盐浓度胁迫下能够进行良好的渗透来保护膜内免受渗透胁迫的影响,保证细胞的正常生命活性[27,36]。此外,厚壁菌门嗜盐细菌分泌的独特碱性蛋白、促进植物生长的次级代谢产物和独特的耐盐胁迫基因参与的代谢通路也能够保护其免受高盐浓度胁迫[37-39]。其中,Bacillus的嗜盐细菌最多,占全部分离菌株的45.00%,表明高盐碱环境是Bacillus spp.能够良好适应并生存的生态位[40];例如,Bagheri等[41]从伊朗高盐湖的盐泥中分离得到中度嗜盐菌Bacillus iranensis,Chen等[42]从海胆中分离得到中度嗜盐菌Bacillus hemicentroti,Menes等[43]从马加迪湖的沉积物中分离得到嗜盐碱菌Bacillus natronophilus,Lei等[44]从新疆沙车县的盐碱土中分离得到中度嗜盐菌Bacillus shacheensis等。
研究表明,沙漠中强抗辐射能力的菌株以厚壁菌门中的Bacillus为主[45,46]。刘阳等[47]发现沙漠中抗辐射能力较强的菌株均来自放线菌门。本研究发现,经100 J/m2的UVC辐照后存活率在10%以上的菌株有34株(占比为42.50%),Bacillus的细菌占比最高,达25.00%,抗辐射能力最强的2株菌分离自含100 g/L NaCl的培养基,分别为Streptomyces sp. ALP-8和Streptomyces sp. ASS-29,均为放线菌门,这与之前的研究相似[27]。分离自300 g/L NaCl的菌株Staphylococcus sp. ASS-5 UVC辐照后存活率为55.61%;分离自250 g/L NaCl的菌株Sediminibacillus sp. ALP-17和Kocuria sp. ASS-27在100 J/m2的UVC辐照后的存活率均在50%以上,分别为54.64%和52.18%。综上,分离自高盐浓度的细菌均具有强的抗辐射能力,说明好氧嗜盐细菌的抗辐射能力与其耐盐能力有一定相关性。这可能与极端环境的生境特点有关,生存在极端环境的微生物一般都能够进化出多元的抗逆特性,例如Georgiou等[48]认为沙漠中大多数抗辐射细菌的辐射耐受性与其抗干旱和抗氧化能力相关;此外也有研究表明干旱、低碳且高盐的环境中嗜盐厚壁菌门的细菌表现出较丰富的多样性,干旱和高盐环境共同提供了嗜盐细菌的优势生态位[49]。极端环境的异质性或许是驱动抗逆细菌进化出独特多元抗逆通路的关键因素[48]。另外,厚壁菌门和放线菌门的嗜盐-抗辐射细菌可以形成孢子进入保护性休眠,保护细胞免受高盐和辐照胁迫[49,50];由于细胞壁的屏蔽作用,革兰氏阳性菌中UV诱导的DNA损伤程度低于革兰氏阴性菌,厚细胞壁的存在同样也可以保护细胞免受高盐胁迫[51]。不仅如此,嗜盐杆菌产生的类胡罗卜素不仅对紫外线具有良好的吸收作用,也能够在光催化下保护细胞,抵抗盐胁迫[52];中度和极端嗜盐杆菌和球菌中多胺类物质(如putrescine、permidine和cadaverine)可以在光效应下提高细胞摄入的高浓度盐调节通道的效率,从而尽可能减少高盐胁迫对细胞内部的损伤[53];嗜盐菌对紫外暴露和高盐导致的渗透胁迫损伤后强大的修复系统也是嗜盐-抗辐射细菌抗逆能力的体现[54]。
4 小结
新疆艾丁湖地区湖周盐碱地的可培养好氧嗜盐细菌数量显著高于湖边滩涂沉积物,分别为2.62×105 CFUs/g和1.81×105 CFUs/g。基于16S rRNA基因序列比对和系统发育分析得到的80株可培养好氧嗜盐细菌隶属于厚壁菌门、放线菌门和变形菌门,其中厚壁菌门(相对占比70.00%)是优势菌门;属水平上,分离得到22个属,其中Bacillus(相对占比45.00%)是优势菌属。在高盐浓度的培养基中分离得到19株潜在新种,其中Bacillus分离得到的潜在新种最多。此外,分离得到的80株可培养好氧嗜盐细菌有34株细菌的D10(存活率大于10%的辐照剂量)大于100 J/m2 UVC 辐照。研究发现,分离自高盐浓度培养基的嗜盐细菌具有强抗辐射能力,分离自300 g/L NaCl隶属厚壁菌门的Staphylococcus sp. ASS-5(存活率为 55.61%)以及分离自250 g/L NaCl隶属厚壁菌门的Sediminibacillus sp. ALP-17(存活率为54.64%)和放线菌门的Kocuria sp. ASS-27(存活率为52.18%)在辐射强度为100 J/m2 UVC辐照下的存活率均在50%以上。
综上所述,艾丁湖地区的可培养好氧嗜盐细菌多样性高,并且均具有强的抗辐射能力,说明该研究区域蕴藏着大量嗜盐-抗辐射细菌极端微生物资源,为盐耐受-抗辐射相关的耦联抗逆机制研究提供了菌株资源,同时也对深入研究细菌在高盐、强辐射环境中的适应机制奠定了一定的理论基础。
致谢:感谢云南大学吴明辉博士和青海大学李娟博士对此次艾丁湖科考采样的帮助与支持。
参考文献:
[1] EDBEIB M F, WAHAB R A, HUYOP F, et al. Halophiles: Biology, adaptation, and their role in decontamination of hypersaline environments[J]. World journal of microbiology & biotechnology, 2016, 32(8): 135.
[2] DAS S, LYLA P S, KHAN S A. Marine microbial diversity and ecology: Importance and future perspectives[J]. Current science association, 2006, 90(10): 1325-1335.
[3] ATLAS R, BYNUM W, COOPER D, et al. Encyclopaedia of life sciences[M]. London:Nature publishing group, 2001.
[4] DUTTA B, BANDOPADHYAY R. Biotechnological potentials of halophilic microorganisms and their impact on mankind[J]. Beni Suef Univ J Basic Appl Sci, 2022, 11(1): 75.
[5] BRUIJN F D. Use of repetitive (repetitive extragenic palindromic and enterobacterial repetitive intergeneric consensus) sequences and the polymerase chain reaction to fingerprint the genomes of Rhizobium meliloti isolates and other soil bacteria[J]. Applenvironmicrobiol, 1992, 58(7): 2180-2187.
[6] GHASEMI Y, RASOUL-AMINI S, KAZEMI A, et al. Isolation and characterization of some moderately halophilic bacteria with lipase activity[J]. Mikrobiologiia, 2010, 80(4): 477-481.
[7] GHASEMI Y, RSOUL-AMINI S, EBRAHIMINEZHAD A, et al. Screening and isolation of extracellular protease producing bacteria from the Maharloo salt lake[J]. Iranian journal of pharmaceutical sciences, 2011, 7(3): 175-180.
[8] VENTOSA A, MáRQUEZ M C, GARABITO M J, et al. Moderately halophilic gram-positive bacterial diversity in hypersaline environments[J]. Extremophiles, 1998, 2(3): 297-304.
[9] TINDALL B J, ROSS H N M, GRANT W D. Natronobacterium gen. nov. and Natronococcus gen. nov., two new genera of haloalkaliphilic archaebacteria [J]. Systematic and applied microbiology, 1984, 5(1): 41-57.
[10] ZHENG X W, WU Z P, SUN Y P, et al. Halorussus vallis sp. nov., Halorussus aquaticus sp. nov., Halorussus gelatinilyticus sp. nov., Halorussus limi sp. nov., Halorussus salilacus sp. nov., Halorussus salinisoli sp. nov.: Six extremely halophilic archaea isolated from solar saltern, salt lake and saline soil[J]. Extremophiles, 2022, 26(3): 32.
[11] HUANG T, LAI Q, XIAO L, et al. Sediminibacillus dalangtanensis sp. nov., a moderate halophile isolated from hypersaline sediments of the Qaidam Basin in Northwest China[J]. Int J Syst Evol Microbiol, 2022, 71(8): 1137-1140.
[12] GAN L, LI X, CHEN J, et al. Gracilibacillus salitolerans sp. nov., a moderate halophile isolated from saline soil in Northwest China[J]. Int J Syst Evol Microbiol, 2020, 70(6): 3701-3710.
[13] YOON J H,KIM I G,SHIN D Y,et al. Microbulbifer salipaludis sp. nov., a moderate halophile isolated from a Korean salt marsh[J]. Int J Syst Evol Microbiol, 2003, 53(Pt1): 53-57.
[14] DAOUD L,ALI M B. Halophilic microorganisms: Interesting group of extremophiles with important applications in biotechnology and environment[A]. SALWAN R,SHARMA V. Physiological and biotechnological aspects of extremophiles[M]. Pittsburgh:Academic press, 2020.51-64.
[15] VENTOSA A. Unusual micro-organisms from unusual habitats: Hypersaline environments[J]. Society for general microbiology symposia, 2006, 66: 223-254..
[16] 王亚俊, 吴素芬.新疆吐鲁番盆地艾丁湖的环境变化[J].冰川冻土, 2003, 25(2): 229-231.
[17] 刘 亮,任世昌, 薛 挺, 等.基于Shannon-Wiener指数的艾丁湖流域植被分布及物种多样性研究[J].地下水,2020,42(4):120-123.
[18] 郑喜玉, 张明刚, 徐 昶, 等.中国盐湖志[M].北京:科学出版社, 2002.
[19] 胡小韦.太阳辐射气象站实测数据与NASA数据对比分析——以吐鲁番地区光资源为例[J].西北水电, 2016(3): 75-78.
[20] 夏占峰, 关统伟, 阮继生, 等.艾丁湖沉积物放线菌多样性[J].微生物学报, 2011, 51(8): 1023-1031.
[21] 王 芸, 倪 萍, 蒋刚强, 等.新疆艾丁湖中度嗜盐苯酚降解菌多样性研究[J].微生物学杂志, 2018, 38(4): 24-30.
[22] 蔡 曼, 胡松楠, 职晓阳, 等.新疆艾丁湖嗜盐放线菌纤维素酶特性初探[J].云南大学学报(自然科学版), 2007, 29(S1): 420-423.
[23] 欧尔比特, 徐晓晶.艾丁湖极端嗜碱菌碱性纤维素酶的初步研究[J].干旱区研究, 2000,17(1): 27-31 .
[24] YOON S H, HA S M, KWON S, et al. Introducing EzBioCloud: A taxonomically united database of 16S rRNA gene sequences and whole-genome assemblies[J]. Int J Syst Evol Microbiol, 2017, 67(5): 1613-1617.
[25] TAMURA K, STECHER G, KUMAR S. MEGA11: Molecular evolutionary genetics analysis version 11[J]. Mol Biol Evol, 2021,38(7): 3022-3027.
[26] SAITOU N, NEI M. The neighbor-joining method: A new method for reconstructing phylogenetic trees[J]. Mol Biol Evol, 1987, 4(4): 406-425.
[27] LIU Y, CHEN T, LI J, et al. High proportions of radiation-resistant strains in culturable bacteria from the Taklimakan Desert[J]. Biology (Basel), 2022, 11(4): 501.
[28] LARKIN M A, BLACKSHIELDS G, BROWN N P, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948.
[29] KIM M, OH H S, PARK S C, et al. Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J]. Int J Syst Evol Microbiol, 2014, 64(Pt2): 346-351.
[30] VENTOSA A, NIETO J J, OREN A. Biology of moderately halophilic aerobic bacteria[J]. Microbiol Mol Biol Rev, 1998, 62(2):504-544.
[31] GRANT W D, KAMEKURA M, MCGENITY T J, et al. Class III. Halobacteria class nov[J/OL]. [2023-02-05].https://api.semanticscholar.org/CorpusID:87801354.
[32] KRAWCZYK K T,LOCHT C,KOWALEWICZ-KULBAT M. Halophilic archaea halorhabdus rudnickae and natrinema salaciae activate human dendritic cells and orient T helper cell responses[J]. Frontiers in immunology, 2022, 13: 833635.
[33] ZHAO D, ZHANG S, KUMAR S, et al. Comparative genomic insights into the evolution of Halobacteria-associated “Candidatus Nanohaloarchaeota”[J]. mSystems, 2022, 7(6): e0066922.
[34] BARICZ A, CRISTEA A, MUNTEAN V, et al. Culturable diversity of aerobic halophilic archaea (Fam. Halobacteriaceae) from hypersaline, meromictic Transylvanian lakes[J]. Extremophiles, 2015, 19(2): 525-537.
[35] YADAV A N, SACHAN S G, VERMA P, et al. Culturable diversity and functional annotation of psychrotrophic bacteria from cold desert of Leh Ladakh (India)[J]. World J Microbiol Biotechnol, 2015, 31(1): 95-108.
[36] MOLINA-MENOR E, GIMENO-VALERO H, PASCUAL J, et al. High culturable bacterial diversity from a European desert: The Tabernas Desert[J]. Front Microbiol, 2020, 11: 583120.
[37] HUANG R, WU F, ZHOU Q, et al. Lactobacillus and intestinal diseases: Mechanisms of action and clinical applications[J]. Microbiol Res, 2022, 260: 127019.
[38] RAWAL C M,RAVAL V H,BHIMANI H D,et al. Whole-genome shotgun sequencing of the extremophile Alkalibacillus haloalkaliphilus C-5, of Indian origin[J]. J Bacteriol,2012,194(17):4775.
[39] HYUN D W,WHON T W,KIM J Y,et al. Genomic analysis of the moderately Haloalkaliphilic bacterium Oceanobacillus kimchii Strain X50T with improved high-quality draft genome sequences[J]. J Microbiol Biotechnol, 2015, 25(12): 1971-1976.
[40] QUESADA E, VENTOSA A, RODRIGUEZ-VALERA F, et al. Types and properties of some bacteria isolated from hypersaline soils [J]. Journal of applied bacteriology, 1982, 53(2): 155-161.
[41] BAGHERI M,DIDARI M, AMOOZEGAR M A, et al. Bacillus iranensis sp. nov., a moderate halophile from a hypersaline lake[J]. Int J Syst Evol Microbiol, 2012, 62(Pt4): 811-816.
[42] CHEN Y G, ZHANG Y Q, HE J W, et al. Bacillus hemicentroti sp. nov., a moderate halophile isolated from a sea urchin[J]. Int J Syst Evol Microbiol, 2011, 61(Pt12): 2950-2955.
[43] MENES R J, MACHIN E V, IRIARTE A, et al. Bacillus natronophilus sp. nov., an alkaliphilic bacterium isolated from a soda lake[J]. Int J Syst Evol Microbiol, 2020, 70(1): 562-568.
[44] LEI Z, QIU P, YE R, et al. Bacillus shacheensis sp. nov., a moderately halophilic bacterium isolated from a saline-alkali soil[J]. J Gen Appl Microbiol, 2014, 60(3): 101-105.
[45] PAULINO-LIMA I G, FUJISHIMA K, NAVARRETE J U, et al. Extremely high UV-C radiation resistant microorganisms from desert environments with different manganese concentrations[J]. J Photochem Photobiol B, 2016, 163: 327-336.
[46] PAULINO-LIMA I G, AZUA-BUSTOS A, VICU?A R, et al. Isolation of UVC-tolerant bacteria from the hyperarid Atacama Desert, Chile[J]. Microb Ecol, 2013, 65(2): 325-335.
[47] 刘 阳, 田 茂, 王馨悦, 等.可培养细菌多样性及抗辐射-抗氧化相关性特征——以库姆塔格沙漠东缘为例[J].中国环境科学, 2021, 41(12): 5921-5932.
[48] GEORGIOU C D,SUN H J,MCKAY C P,et al. Evidence for photochemical production of reactive oxygen species in desert soils[J]. Nat Commun, 2015, 6: 7100.
[49] GENDERJAHN S, ALAWI M, MANGELSDORF K, et al. Desiccation- and saline- tolerant bacteria and archaea in Kalahari Pan sediments [J]. Front Microbiol, 2018, 9: 2082.
[50] GAO J, LUO Y, WEI Y, et al. Effect of aridity and dune type on rhizosphere soil bacterial communities of Caragana microphylla in desert regions of northern China [J]. PLoS one, 2019, 14(10): e0224195.
[51] BOUDJELAL M, WANG Z, VOORHEES J J, et al. Ubiquitin/proteasome pathway regulates levels of retinoic acid receptor gamma and retinoid X receptor alpha in human keratinocytes [J]. Cancer Res, 2000, 60(8): 2247-2252.
[52] LAKE J A,CLARK M W,HENDERSON E,et al. Eubacteria, halobacteria, and the origin of photosynthesis:The photocytes[J]. Proc Natl Acad Sci U S A, 1985, 82(11): 3716-3720.
[53] HAMANA K,KAMEKURA M,ONISHI H,et al. Polyamines in photosynthetic eubacteria and extreme-halophilic archaebacteria[J]. J Biochem, 1985, 97(6): 1653-1658.
[54] JONES D L,BAXTER B K. DNA Repair and photoprotection: Mechanisms of overcoming environmental ultraviolet radiation exposure in halophilic archaea[J]. Front Microbiol, 2017, 8: 1882.

