藜麦CqGAI 基因密码子偏好性与进化分析
丰扬 郭凤根 王仕玉 刘正杰 龙雯虹

摘要:为了阐明藜麦CqGAI 基因密码子使用特性,克隆获得藜麦CqGAI 基因序列,运用CodonW、SPSS软件及EMBOSS在线程序分析藜麦CqGAI 基因密码子使用偏好性,并与25种植物的GAI 基因进行中性绘图、ENC分析和奇偶偏好偏差性分析。结果表明,藜麦CqGAI 基因编码序列(coding sequence,CDS)全长1 782 bp,编码593个氨基酸,包含DELLA基因家族特有结构域DELLA、TVHYNP、NLS、VHIID、LHR、RVER;CqGAI基因能够迅速响应赤霉素(gibberellins,GA),在GA信号通路中起关键作用;密码子偏好性分析显示,CqGAI 基因的有效密码子数(effective number of codon,ENC)、密码子适应指数(codon adaptation index, CAI)及GC含量分别为54.14、0.21和46.18%,密码子偏好性较弱,偏好以A/T结尾,有27个高频密码子。聚类分析表明,藜麦CqGAI 基因与石竹目植物的亲缘关系较近。碱基组成与相关性分析发现,CqGAI 基因密码子偏好性受碱基突变和选择效应影响。密码子使用频率表明,大肠杆菌与酵母菌均适用于藜麦CqGAI 基因异源表达,拟南芥、烟草、甜菜均可作为藜麦CqGAI 基因功能分析的遗传转化受体。以上研究结果为进一步研究藜麦CqGAI 基因功能和异源表达提供了重要参考。
关键词:藜麦;CqGAI 基因;密码子偏好性;进化分析
doi:10.13304/j.nykjdb.2023.0125
中图分类号:S516 文献标志码:A 文章编号:10080864(2024)04002710
藜麦(Chenopodium quinoa Willd.)原产于南美洲安第斯山脉,其果实营养丰富,蛋白质含量高,各种氨基酸比例均衡,富含膳食纤维、矿质元素及维生素等,适合各类人群食用,因此被称为“全营养作物”[12],但藜麦在生产上也遇到穗发芽等诸多阻碍。穗发芽是指作物在收获前遇到连绵阴雨或在潮湿环境下种子在穗上发芽的现象[3],在水稻、小麦、藜麦等作物中均有发生,严重影响作物的产量和品质[4-6]。种子从休眠到萌发阶段,其生理变化由多种植物内源激素共同调控[7]。DELLA蛋白是多种植物内源激素信号转导的作用因子,而DELLA蛋白的作用通过赤霉素(gibberellins,GA)信号调控[89]。
GAI 属于DELLA 蛋白家族,其N 端具有DELLA和TVHYNP 2个GA3 信号感知结构域,中部含有核定位信号(nuclear localization signal,NLS),C端的VHIID、SH2、SAW是阻遏结构域[10]。DELLA 蛋白缺少DNA 结合结构域,因此DELLA蛋白常与其他调控蛋白互作,进而调控植物的生理过程[11]。不同物种中DELLA 蛋白的数量及作用方式不同,同一物种中DELLA蛋白家族成员间功能既有冗余,又有互补[1213]。在种子萌发与休眠中,DELLA蛋白通过介导激素和光信号来调控种子的萌发,DELLA蛋白可直接与ABI3和ABI5互作,使下游SOM 基因表达上调,进而调控种子萌发[14]。光敏色素接收到光信号后,PIL5蛋白被降解,GAI和RGA的转录受到抑制,内源GA3含量升高,促进种子萌发[15]。目前,关于DELLA蛋白家族在植物内源激素、逆境响应等方面已有一些研究,但DELLA 蛋白的调控机理复杂,不同植物DELLA蛋白的数量与功能不同,还需进一步研究。
密码子是遗传信息从DNA到蛋白质的传递中枢,在生物进化过程中由于环境等因素形成密码子偏好。密码子偏好性是由于生物同义密码子使用不均衡造成的,其存在物种及基因差异[1617];导致基因在碱基组成和表达水平存在差异,因此密码子偏好性分析对基因的进化、功能预测、蛋白质表达等具有重要参考价值[1819]。很多物种的遗传转化体系不成熟,需要借助异源表达分析基因的功能。在异源表达过程中,由于物种间存在密码子使用偏好性,容易造成甲基化、表达水平低等,因此需要根据目标基因密码子使用特性及受体密码子使用特性选择合适的受体系统或对目标基因密码子进行优化或改造[20]。近年关于密码子的使用偏好性在多个植物中已有报道,但未见藜麦GAI 基因密码子偏好性的相关研究。因此,本研究克隆藜麦GAI 基因,对其进行生物信息学分析,初探其在GA3处理下种子萌发中的功能,并对其密码子偏好性进行分析,旨在为深入研究藜麦GAI 基因在种子萌发中的作用机制奠定基础。
1 材料与方法
1.1 试验材料
供试藜麦为本课题组前期经穗发芽抗性评价获得的不同穗发芽抗性的藜麦种质资源SBQ1、WQ8、BBQ3,抗性表现为SBQ1>WQ8>BBQ3。以其萌发的种子及幼苗作为试验材料,干种子置于4 ℃保存[21]。
1.2 藜麦CqGAI 基因cDNA 的全长克隆
取3 叶期SBQ1、WQ8、BBQ3 藜麦的幼嫩叶片,液氮研磨,使用TransZol(北京全式金生物技术股份有限公司)提取总RNA。按照TransScript?One-Step gDNA Removal and cDNA Synthesis SuperMix逆转录试剂盒(北京全式金生物技术股份有限公司)合成cDNA,产物于?20 ℃冰箱保存。根据GeneBank公布的藜麦CqGAI-like(LOC110694429)全长序列设计特异引物CqGAI-F 和CqGAI-R(表1)。以幼苗叶片cDNA 为模板,用Phanta Flash MasterMix聚合酶(南京诺唯赞生物科技有限公司)扩增CqGAI 基因全长。PCR体系为: Phanta Flash MasterMix聚合酶25 μL、模板1 μL、上下游引物各2 μL,加ddH2O至总体积50 μL。PCR程序:98 ℃ 30 s;98 ℃ 10 s,58 ℃ 5 s,72 ℃ 10 s,35 个循环;72 ℃1 min。1.0% 琼脂糖凝胶电泳切胶回收连接pEASY克隆载体(北京全式金生物技术股份有限公司),转化DH5ɑ感受态,挑取阳性单菌落扩大培养后送至生工生物工程(上海)股份有限公司测序。
1.3 藜麦CqGAI 基因的生物信息学分析
利用ExPASy ProtParam分析CqGAI 基因编码蛋白的理化性质;使用TMHMM 和Protscale 预测蛋白质跨膜结构域和亲疏水性;通过PSORT 和SignalP-5.0进行蛋白亚细胞定位及信号肽分析;使用SOPMA与SWISS-MODEL预测蛋白的二级结构和三维结构;通过NCBI-Conserved Domains 预测蛋白的保守结构域[22]。
1.4 藜麦CqGAI 基因在种子萌发中的表达分析
分别将藜麦种子置于含蒸馏水(对照)和10、25、50 mg·L-1赤霉素(GA3)处理的滤纸上萌发6、12、24、36 h,分别提取总RNA并反转录。根据克隆获得的藜麦CqGAI 基因序列设计qRT-PCR引物CqGAI-qF和CqGAI-qR(表1),以Actin 基因作为内参基因,使用qRT-PCR技术分析不同处理的藜麦种子萌发24 h 时CqGAI 基因的相对表达量和25 mg·L-1 GA3处理下不同藜麦种子萌发过程中的CqGAI 基因的相对表达量。
1.5 GAI 基因的密码子偏好性分析
甜菜(Beta vulgaris)、菠菜(Spinacia oleracea)、苋菜(Amaranthus tricolor) 、番茄(Solanumlycopersicum)等24种植物的GAI 基因序列来源于GenBank(http://www.ncbi.nlm.nih.gov/ genebank/)。拟南芥(Arabidopsis thaliana)、烟草(Nicotianatabacum)、甜菜、水稻(Oryza sativa)、酵母菌(Saccharomyces cerevisiae)和大肠杆菌(Escherichiacoli)等模式生物的基因组密码子偏好性数据来源于密码子使用数据库Codon Usage Database (http://www.kazusa.or.jp/codon/)。使用Codon W 软件和CHIPS 网站(https://www.bioinformatics.nl/embossexplorer/)分析25种植物GAI 基因的有效密码子数(effective number of codon,ENC)、密码子适应指数(codon adaptation index, CAI)、最优密码子使用频率(frequency of optiomal codons, FOC)、同义密码子相对使用度(relative synonymous codon usage,RSCU)、密码子第1、2和3位上的G/C含量(GC1、GC2和GC3)和密码子第3位上各碱基含量(A3s、T3s、G3s 和C3s)等密码子偏好性参数;采用EMBOSS分析密码子使用频率。
1.6 数据分析
采用SPSS 27.0 和Excel 2019 对数据进行统计分析,并对密码子参数相关性、ENC绘图和奇偶偏性分析(parity rule 2,PR2)进行运算分析。同时,基于不同物种中GAI 基因的同义密码子相对使用度(RSCU)进行密码子使用偏性聚类分析,用欧氏平方距离表示基因间的进化距离,采用邻近法利用MEGA Ⅺ构建25个物种GAI 基因的系统进化树。
2 结果与分析
2.1 藜麦CqGAI 基因cDNA 全长克隆
在预测序列的基础上设计引物对目的基因进行扩增,获得约1 782 bp大小的PCR产物(图1)。连接pEASY克隆载体,转化DH5ɑ,阳性单菌落经M13通用引物测序表明,与GeneBank中预测的藜麦GAI 基因CDS区域相似度达99%,但在C 端缺失75 bp。在SBQ1、WQ8 和BBQ3 中扩增获得的序列均与GAI 基因一致,因此命名为CqGAI,登录号OQ067480。
2.2 藜麦CqGAI 基因生物信息学分析
CqGAI 基因编码593 个氨基酸,蛋白分子量64.65 kD,分子式C2 828H4 438N776O905S27,等电点4.93;含有49个碱性氨基酸(Arg+Lys),72个酸性氨基酸(Asp+Glu),平均疏水系数?0.272,脂溶指数79.12,不稳定指数48.95,属于酸性亲水的不稳定蛋白。对CqGAI蛋白二级结构和三级结构进行分析,其二级结构由43.68% 的α-螺旋、9.78% 的延伸链、3.71%的β-转角、42.83%的无规则卷曲组成;其三级结构与DELLA亚基因家族中的GAI蛋白相似度达67.33%,且含有GA3的识别位点。信号肽预测显示,CqGAI蛋白无信号肽,不具有跨膜区,亚细胞定位主要存在于细胞核。对CqGAI蛋白进行保守结构域及与近缘种蛋白序列进行比对分析,结果(图2)发现,CqGAI蛋白含有DELLA亚基因家族特有的结构域,其中N 端有DELLA、TVHYNP酸性结构域;中部有核定位信号;C端有VHIID、LHR、RVER等保守结构域。
2.3 藜麦CqGAI 基因对GA3的表达响应
利用qRT-PCR分析CqGAI 基因在GA3处理下不同穗发芽抗性藜麦种子萌发过程中的相对表达量,结果(图3)表明,在10、25、50 mg·L-1 GA3处理下种子发芽24 h时,CqGAI 基因在SBQ1、BBQ3中的表达量与对照均无显著差异,而WQ8在10和25 mg·L-1 GA3处理下CqGAI 基因的表达量较对照分别显著上调和下调。在25 mg·L-1 GA3处理下,随着种子的萌发,CqGAI 基因表达量在SBQ1、WQ8和BBQ3中均下调,说明CqGAI 基因能够响应GA3。在种子萌发24 h时,CqGAI 基因表达量在SBQ1、WQ8和BBQ3中均趋于平稳,说明在GA3处理下,CqGAI 基因能够迅速响应GA3,进而行使其生物学功能,促进种子萌发。
2.4 藜麦CqGAI 基因密码子偏好性分析
2.4.1 藜麦CqGAI 基因密码子相关分析
有效密码子数(ENC)是指基因的密码子使用频率与同义密码子平均使用频率偏差的量化值,其数值一般在20~61之间,越接近61表示同义密码子偏倚越小,基因表达量越低。高表达基因密码子偏好程度越大,密码子适应指数(CAI)值越大,ENC值越小;低表达基因包含的稀有密码子类型较多,因此偏好度低,CAI 值较小,ENC 值较高。运用CodonW 软件和EMBOSS 在线网站获得了藜麦CqGAI 基因密码子使用偏性相关参数,如表2 所示。CqGAI 基因的ENC为54.14,CAI为0.21,表明藜麦CqGAI 基因的密码子偏好性较弱,属于低表达基因,其密码子中GC、GC1、GC2、GC3含量分别为46.18%、52.02%、42.76%、43.77%,说明CqGAI基因密码子偏好以A/T结尾。
相对同义密码子使用度(RSCU)能够体现某一密码子偏性的强弱,RSCU=1表示密码子不存在偏好;RSCU>1表示该密码子较其他同义密码子出现的频率更高;RSCU<1表明该基因使用此密码子的频率较低。藜麦CqGAI 基因的RSCU值如图4所示,高频密码子有27个,其中以A/T结尾的有18个,以G/C结尾的有9个,说明藜麦CqGAI基因密码子偏好以A/T结尾,与GC含量分析结果一致。密码子AGG、GUU、GCU 的RSCU>2,具有较强的使用偏好性;其余31个密码子的RSCU<1,说明其在使用时被选择的频率较低。
2.4.2 不同物种GAI 基因的聚类分析
通过邻近法对25种植物的GAI基因的CDS序列构建系统进化树(图5A),将单子叶植物和双子叶植物分为2大类。在双子叶植物中,同为石竹目的藜麦、菠菜、苋菜、甜菜聚为一小类,这4种植物GAI 基因的GC含量和ENC值较相近,其中藜麦与菠菜的GC含量和ENC值差异更小,因此菠菜与藜麦亲缘关系最近;在蔷薇科植物中,沙梨、苹果、李、杂交玫瑰聚为一小类,而樱桃、水蜜桃、梅花聚为另一小类,这7种植物中沙梨、苹果、李、杂交玫瑰GAI 基因的GC含量和ENC值差异较小,樱桃、水蜜桃、梅花间差异较小。由此表明,密码子使用偏好性越近,其物种在进化上的亲缘关系越近。
基于GAI 基因的RSCU值进行聚类分析,结果(图5B)表明,将25种植物也分为2大类,但与基于CDS序列的进化树存在差异。石竹目植物与橡胶树、马铃薯聚为一小类;4个单子叶植物依然聚为一小类;7个蔷薇科植物间的距离较近。因此,在基于RSCU值的聚类分析中,密码子使用频率相近的物种亲缘关系可能较近,也可能较远。对比2种聚类结果,基于CDS序列的聚类分析更接近传统的物种进化关系;基于RSCU值的聚类结果仅能体现密码子使用偏好的丰富性,而密码子使用偏好性与物种亲缘关系并没有直接的相关性。
2.4.3 不同物种GAI 基因中性分析
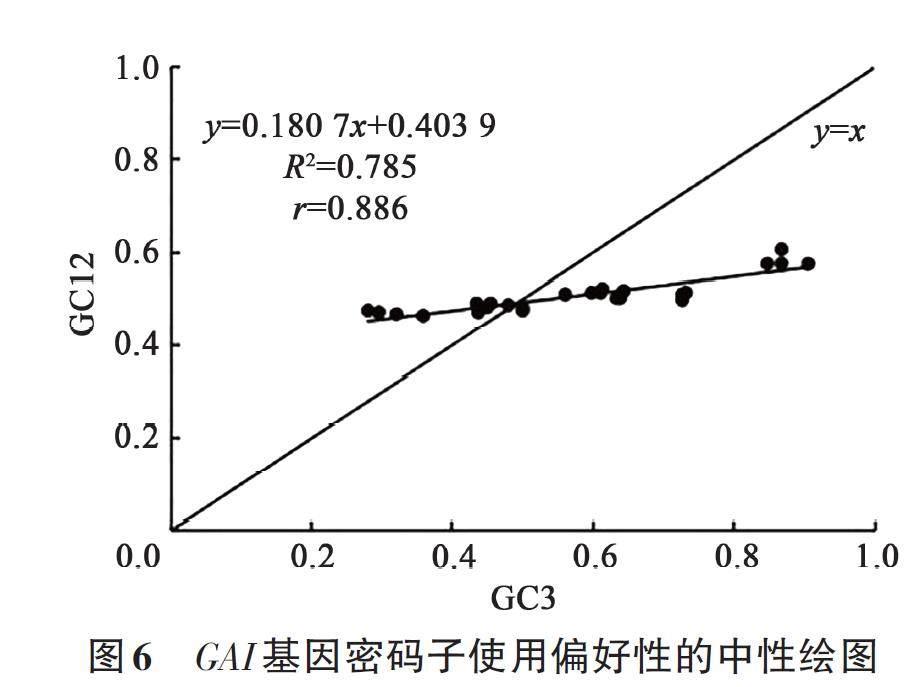
对25个物种GAI 基因的GC含量和ENC相关性进行分析,结果(表3)表明,GC、GC1、GC2、GC3间均呈极显著正相关;而GC含量和ENC呈极显著负相关。对不同物种GAI 基因进行中性分析,结果(图6)表明,GC3为0.281 8~0.905 4,GC12为0.466 5~0.606 3,GC12的变幅较小,大部分GAI 基因分布于回归线的两侧,线性回归系数为0.180 7;GC3与GC12呈极显著正相关,相关系数(r)为0.886,与相关性分析结果一致。由此表明,GAI 基因的碱基组成差异较小,主要受突变压力影响。
2.4.4 不同物种GAI 基因的ENC 和PR2 分析
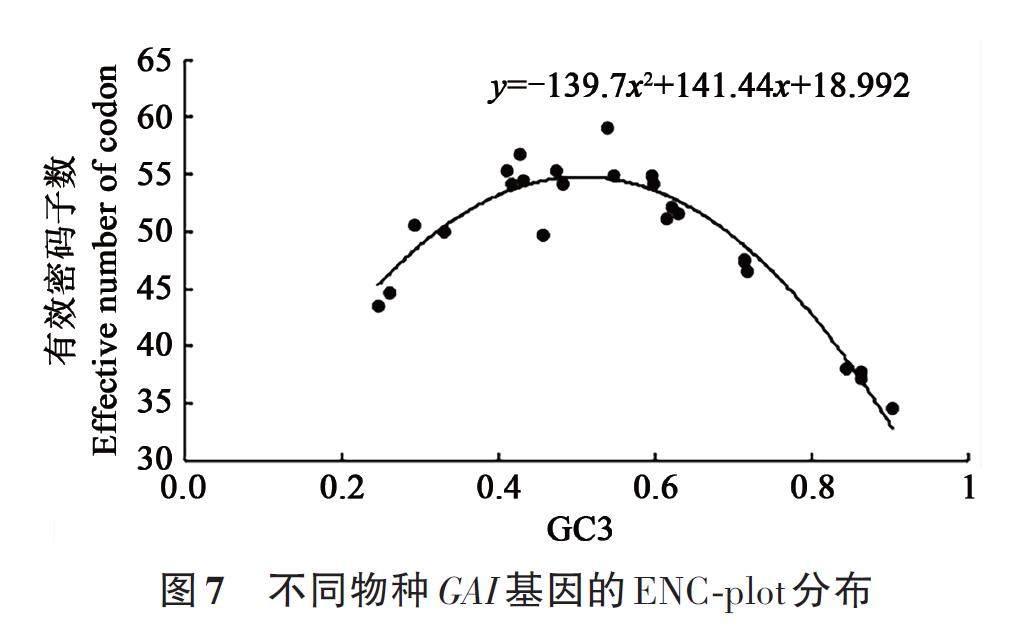
ENC 分析可检测碱基组成对密码子偏好性的影响,若基因沿标准曲线分布或在标准曲线附近,表示该基因密码子偏好性受突变压力的影响;若基因在标准曲线下方较远的位置则表示该基因密码子偏好性受选择压力的影响。由图7可知,25种植物GAI 基因的ENC分布趋势与标准曲线一致,其中苋菜、牡丹、橡胶树、毛白杨分布于标准曲线上方;拟南芥、番茄、水稻分布于标准曲线下方;其余物种分布于标准曲线附近。由此说明,突变是影响GAI 基因偏好性的主要因素,但选择压力也产生了一定的影响。
奇偶偏性分析(PR2)中若基因均匀地分布在坐标轴中心,说明4种碱基的使用频率一致,其密码子偏好性主要受碱基突变影响;若偏离中心位置,说明受其他因素影响。由图8可知,GAI 基因的G3/(G3+C3)和A3/(A3+T3)值未在0.5 左右。其中,大多数GAI 基因的G3/(G3+C3)值小于0.5,表明GAI基因密码子选用G与C时,偏向于碱基C结尾;大多数GAI 基因的A3/(A3+T3)值均匀分布于0.5区域的上、下侧,表明GAI 基因密码子选用A与T时没有明显的偏好性。由此推断,突变不是影响GAI 基因密码子使用偏好性的唯一原因,选择压力等其他因素也在其进化过程中发挥了一定作用。
2.4.5 藜麦CqGAI 基因受体系统的选择
藜麦遗传转化体系尚未完善,其基因的功能分析需要借助具有完善遗传转化体系的物种进行异源表达。通常认为,物种间密码子使用频率在0.5~2.0 之间,说明种间在使用该密码子时偏好性较小;使用频率≤0.5或≥2.0,说明种间在使用该密码子时偏好性较大。将藜麦CqGAI 基因密码子使用频率与拟南芥、烟草、甜菜、水稻、酵母菌和大肠杆菌的基因组密码子使用频率进行比较,结果(表4)表明,藜麦CqGAI 基因与拟南芥、烟草、甜菜、水稻在使用频率上差异较大的密码子个数分别为9、9、10、19,说明拟南芥、烟草和甜菜可作为藜麦CqGAI 基因遗传转化的受体;藜麦CqGAI 基因与酵母菌、大肠杆菌在使用频率上差异较大的密码子个数均为13,说明酵母菌、大肠杆菌都可用于藜麦CqGAI 基因的异源表达。
3 讨论
DELLA蛋白参与多种植物激素信号转导,对植物的生长发育至关重要,如种子萌发、茎伸长、开花时间等[2324]。它作为GA信号的抑制因子,经泛素化降解后激活GA 信号通路[2526]。藜麦穗发芽现象严重影响其品质与产量,GA3作为种子萌发与休眠的主要植物激素,其生物活性与DELLA蛋白有直接关系[3,25]。GA3是受体GID1与DELLA相互作用的关键,GA-GID1-DELLA复合物形成后,SCF E3 泛素连接酶SLEEPY1(SLY1)特异性识别DELLA蛋白,并通过26S蛋白酶体途径泛素化降解DELLA,进而发挥其生物活性[2728]。藜麦种子的休眠是初级休眠和生理休眠的组合,或没有初级休眠[29]。因此,对DELLA家族基因的研究有利于深入了解藜麦种子的休眠与萌发机制。
本研究克隆了藜麦DELLA家族基因CqGAI,编码区全长1 782 bp,编码593个氨基酸。生物信息学分析表明,其含有DELLA基因家族特有结构域DELLA、TVHYNP、NLS、VHIID、LHR、RVER等,但C端无阻遏基序SAW。水稻SLR1的SAW基序突变导致GID1与DELLA蛋白的结合能力降低[30],对于CqGAI 基因SAW基序缺失是否会影响藜麦中GID1-DELLA蛋白相互作用还有待进一步研究。为探究CqGAI 基因在种子萌发中的作用,分析了CqGAI 基因在GA3处理下不同穗发芽抗性藜麦种子萌发过程中的相对表达量,表明CqGAI基因能够响应GA3,表达量迅速降低,GA3促进种子萌发;但在不同水平GA3处理下,CqGAI 基因在不同穗发芽抗性藜麦中并没有表现出显著的表达差异,推测藜麦种子的穗发芽抗性可能与种子自身内源激素含量、种皮厚度等因素有关。
密码子使用偏好性是生物进化过程中适应性选择的结果,广泛存在于自然界中。分析CqGAI基因密码子偏好性,探索其在进化过程中的规律及合适的异源转化受体,可为深入研究藜麦CqGAI 基因的功能奠定基础。根据密码子的简并性,可将特定密码子进行同义突变,以提高目的基因的异源表达效率[31]。石竹目植物GAI 基因的ENC值均大于50,说明石竹目植物的密码子使用偏好性较弱,在密码子的选择上具有较高的随机性。对藜麦CqGAI 基因密码子进行分析发现,其密码子偏好使用A/T并使用A/T结尾,这符合双子叶植物的密码子使用特性[32]。在聚类分析中,亲缘关系较近的物种其密码子使用偏好性较一致,但二者之间并不存在相关性;基于CDS序列与基于RSCU 的聚类分析结果存在一定差异,基于CDS序列的聚类结果更接近系统分类中的物种亲缘关系,而基于RSCU的聚类结果仅反映出GAI 基因的进化规律,将2种聚类结果相结合可在一定程度上反映出物种的进化规律[3334]。
密码子使用偏好性主要受突变压力和自然选择的影响,对GAI 基因进行中性绘图、ENC分析和PR2分析发现,GAI 基因在密码子偏好性选择上,除受碱基突变影响外,选择效应等其他因素也有一定影响。在异源表达中,物种间密码子使用频率的比值差异越小越有利于目的基因的表达[35]。本研究结果表明,大肠杆菌原核表达系统与酵母真核表达系统均适用于藜麦CqGAI 基因的异源表达,同时拟南芥、烟草、甜菜也可作为藜麦CqGAI基因功能分析的异源受体,但试验中仍需对部分密码子进行优化,同时也要考虑转化效率、转录后调控及mRNA 结构等诸多因素,对于藜麦CqGAI基因的异源高效转化表达仍需深入研究。
参考文献
[1] TANG Y, LI X H, CHEN P X, et al .. Characterisation of fattyacid, carotenoid, tocopherol/tocotrienol compositions andantioxidant activities in seeds of three Chenopodium quinoaWilld. genotypes [J]. Food Chem., 2015, 174(5):502-508.
[2] TANG Y, LI X H, ZHANG B, et al .. Characterisation ofphenolics, betanins and antioxidant activities in seeds of threeChenopodium quinoa Willd. genotypes [J]. Food Chem., 2015,166(1):380-388.
[3] MCGINTY E M, MURPHY K M, HAUVERMALE A L. Seeddormancy and preharvest sprouting in quinoa [J/OL]. Plants,2021, 10(3):458 [2023-01-10]. https://doi.org/10.3390/plants10030458.
[4] CECCATO D V, BERTERO H D, BATLLA D. Environmentalcontrol of dormancy in quinoa (Chenopodium quinoa) seeds:two potential genetic resources for pre-harvest sproutingtolerance [J]. Seed Sci. Res., 2011, 21(2):133-141.
[5] VETCH J M, STOUGAARD R N, MARTIN J M, et al ..Revealing the genetic mechanisms of pre-harvest sprouting inhexaploid wheat (Triticum aestivum L.) [J]. Plant Sci., 2019,281:180-185.
[6] NONOGAKI H, BARRERO J M, LI C D. Seed dormancy,germination, and pre-harvest sprouting [J/OL]. Front Plant Sci.,2018, 9:1783 [2023-01-10]. https://doi.org/10.3389/978-2-88945-762-5.
[7] 徐恒恒,黎妮,刘树君,等.种子萌发及其调控的研究进展[J].作物学报,2014,40(7):1141-1156.
XU H H, LI N, LIU S J, et al .. Research progress in seedgermination and its control [J]. Acta Agron. Sin., 2014, 40(7):1141-1156.
[8] PHOKAS A, COATES J C. Evolution of DELLA function andsignaling in land plants [J]. Evol. Dev., 2021, 23(3):137-154.
[9] VELDE K V, RUELENS P, GEUTEN K, et al .. ExploitingDELLA signaling in cereals [J]. Trends Plant Sci., 2017,22(10):880-893.
[10] SERRANO M A, BENCIVENGA S, BUSH M, et al .. DELLAgenes restrict inflorescence meristem function independentlyof plant height [J]. Nat. Plants, 2017, 3(9):749-754.
[11] DAVIERE J, ACHARD P. A pivotal role of DELLAs inregulating multiple hormone signals [J]. Mol. Plant, 2016, 9(1):10-20.
[12] KELLER J, DELCROS P, LIBOUREL C, et al .. DELLA familyduplication events lead to different selective constraints inangiosperms [J]. Genetica, 2020, 148(5-6):243-251.
[13] BLANCO T N, SERRANO M A, ALABADI D. Regulation ofDELLA proteins by post-translational modifications [J]. PlantCell Physiol., 2020, 61(11):1891-1901.
[14] PAN J J, HU Y R, WANG H P, et al .. Molecular mechanismunderlying the synergetic effect of jasmonate on abscisic acidsignaling during seed germination in Arabidopsis [J]. PlantCell, 2020, 32(12):3846-3865.
[15] LI K L, YU R B, FAN L M, et al .. DELLA-mediated PIFdegradation contributes to coordination of light and gibberellinsignalling in Arabidopsis [J/OL]. Nat. Commun., 2016, 7:11868[2023-01-10]. https://doi.org/10.1038/ncomms11868.
[16] 张以忠,曾文艺,邓琳琼,等.甘蓝S-位点基因SRK、SLG 和SP11/SCR 密码子偏好性分析[J].作物学报,2022,48(5):1152-1168.
ZHANG Y Z, ZENG W Y, DENG L Q, et al .. Codon usage biasanalysis of S-locus genes SRK, SLG, and SP11/SCR in Brassicaoleracea [J]. Acta Agron. Sin., 2022, 48(5):1152-1168.
[17] 任桂萍,董璎莹,党云琨.密码子中的密码:密码子偏好性与基因表达的精细调控[J].中国科学:生命科学,2019,49(7):839-847.
REN G P, DONG Y Y, DANG Y K. Codon in codon:preference and fine regulation of gene expression [J]. Sci. Sin.Vitae, 2019, 49(7):839-847.
[18] WEI L, HE J, JIA X, et al .. Analysis of codon usage bias ofmitochondrial genome in Bombyx mori and its relation toevolution [J]. BMC Evol. Biol., 2014, 14(1):1-12.
[19] PEK H B, KLEMENT M, ANG K S, et al .. Exploring codoncontext bias for synthetic gene design of a thermostableinvertase in Escherichia coli [J]. Enzyme Microb. Technol.,2015, 75-76:57-63.
[20] GUN L, YUMIAO R, HAIXIAN P, et al .. Comprehensiveanalysis and comparison on the codon usage pattern of wholemycobacterium tuberculosis coding genome from different area[J/OL]. Biomed. Res. Int., 2018, 2018:3574976 [2023-01-10].https://doi.org/10.1155/2018/3574976.
[21] 毛琪,晏兴珠,王仕玉,等.30 份藜麦资源的穗发芽抗性评价[J].种子,2021,40(10):62-66.
MAO Q, YAN X Z, WANG S Y, et al .. Evaluation of paniclegermination resistance of 30 quinoa resources [J]. Seed, 2021,40(10): 62-66.
[22] 丰扬,郭凤根,王仕玉,等.藜麦Cq6GT 基因的克隆与表达分析[J].植物生理学报,2022,58(10):2017-2024.
FENG Y, GUO F G, WANG S Y, et al .. Cloning and expressionanalysis of Cq6GT gene from Chenopodium quinoa [J]. PlantPhysiol. J., 2022, (58)10:2017-2024.
[23] XU H, LANTZOUNI O, BRUGGINK T, et al .. A molecularsignal integration network underpinning Arabidopsis seedgermination [J]. Curr. Biol., 2020, 30(19):3703-3712.
[24] CHAHTANE H, NOGUEIRA F T, ALLARD P M, et al .. Theplant pathogen pseudomonas aeruginosa triggers a DELLAdependentseed germination arrest in Arabidopsis [J/OL]. Elife,2018, 7:e37082 [2023-01-10]. https://doi.org/10.7554/eLife.37082.
[25] WANG Y J, DENG D X. Molecular basis and evolutionarypattern of GA-GID1-DELLA regulatory module [J]. Mol. Genet.Genomics, 2014, 289(1):1-9.
[26] PONNU J. Repressing a repressor: E3 ligase COP1/SPApromotes seed germination by targeting the DELLA proteinRGL2 [J]. Plant Physiol., 2022, 189(3):1192-1193.
[27] SUN T P. The molecular mechanism and evolution of the GAGID1-DELLA signaling module in plants [J]. Curr. Biol., 2011,21(9):338-345.
[28] 宋松泉,刘军,黄荟,等.赤霉素代谢与信号转导及其调控种子萌发与休眠的分子机制[J]. 中国科学:生命科学,2020,50(6):599-615.
SONG S Q, LIU J, HUANG H, et al .. Gibberellin metabolismand signaling and its molecular mechanism in regulating seedgermination and dormancy [J]. Sci. Sin. Vitae, 2020, 50(6):599-615.
[29] BASKIN J M, BASKIN C C. A classification system for seeddormancy [J]. Seed Sci. Res., 2007, 14(1):1-16.
[30] HIRANO K, ASANO K, TSUJI H, et al .. Characterization of themolecular mechanism underlying gibberellin perception complexformation in rice [J]. Plant Cell, 2010, 22(8):2680-2696.
[31] WANG L Y, XING H X, YUAN Y C, et al .. Genome-wideanalysis of codon usage bias in four sequenced cotton species[J/OL]. PLoS One, 2018, 13(3):e0194372 [2023-01-10]. https://doi.org/10.1371/journal.pone.0194372.
[32] MURRAY E E, LOTZER J, EBERLE M. Codon usage in plantgenes [J]. Nucl. Acids Res., 1989, 17(2):477-498.
[33] 宋芸,贾孟君,陈亮,等.忍冬ICE1 基因密码子偏好性分析及受体系统选择[J].植物生理学报,2020,56(11):2459-2468.
SONG Y, JIA M J, CHEN L, et al .. Codon bias analysis andreceptor system selection of ICE1 gene in Lonicera japonicaThunb [J]. Plant Physiol. J., 2020, 56(11):2459-2468.
[34] 赵春丽,彭丽云,王晓,等.苋菜AtGAI 基因密码子偏好性与进化分析[J].中国农业大学学报,2019,24(12):10-22.
ZHAO C L, PENG L Y, WANG X, et al .. Codon bias andevolution analysis of AtGAI in Amaranthus tricolor [J]. J. Chin.Agric. Univ., 2019, 24(12):10-22.
[35] 李翔,范作义,王井源,等.红松查尔酮合成酶基因CHS 密码子偏好性分析[J].植物研究,2020,40(3):447-457.
LI X, FAN Z Y, WANG J Y, et al .. Codon usage bias ofchalcone synthase gene CHS in Pinus koraiensis [J]. Bull. Bot.Res., 2020, 40(3):447-457.
(责任编辑:张冬玲)
基金项目:国家自然科学基金项目(31960417);云南省教育厅科学研究基金项目(2023Y1019)。

