茶树叶片黄化变异相关的CAB基因家族鉴定及关键基因挖掘
钟思彤 张亚真 游小妹 陈志辉 孔祥瑞 林郑和 伍慧妮 金珊 陈常颂
摘要:捕光叶绿素a/b结合蛋白(Light-harvesting chlorophyll a/b binding protein,CAB)基因家族成员在植物叶片黄化变异中具有重要作用。本研究利用铁观音(Camellia sinensis Tieguanyin)基因组数据对CAB基因家族进行了筛选,并进行了生物信息学和表达模式分析;进一步以不同黄化、绿叶茶树品种(系)为材料,通过基因克隆和qRT-PCR,分析CABs基因的表达特性,结合叶色参数和叶绿素SPAD值的相关性分析筛选出与茶树黄化变异相关的关键CAB基因。结果表明,共鉴定到25个CAB基因家族成员,其氨基酸長度为167~337个,蛋白分子质量为18.5~37.1 kDa,大部分CAB成员属于稳定性蛋白和疏水性蛋白,并且亚细胞定位预测在叶绿体上;根据进化关系25个CAB家族成员分为13个亚家族,Lhcb1亚族的成员数量最多;启动子分析显示,CAB家族成员启动子中包含大量的光响应元件,还有其他与植物生长发育、激素和逆境胁迫响应相关的元件。从茶树中克隆Lhcb1亚家族成员,通过序列比对筛选出CAB1、CAB6和CAB7基因;表达分析显示,CAB1、CAB6和CAB7基因都具有组织表达特异性,在芽、叶和果实中表达量较高,并能响应多种逆境胁迫。qRT-PCR分析发现,CAB1、CAB6和CAB7基因在黄、绿茶树的叶片中具有一致的表达特性:与正常绿叶相比,黄化叶片中的CABs基因表达均显著下调;通过与叶色参数和叶绿素SPAD值的相关性分析,发现CAB1、CAB6和CAB7表达量与叶色参数a、b、L值以及叶绿素SPAD值均呈极显著相关(P<0.01),其中CAB1的基因表达量与叶色相关参数和叶绿素SPAD值的相关性最显著;烟草亚细胞定位结果显示,CAB1在细胞核、细胞膜和叶绿体上均有分布。以上研究初步解析了茶树CAB家族成员的基本特征,挖掘出与茶树叶色变异紧密相关CAB基因,后续可作为候选关键基因进行深入研究,以期为茶树叶色变异的分子调控机制的研究提供理论基础。
关键词:茶树;捕光叶绿素a/b结合蛋白;叶色黄化变异;基因克隆;表达分析
中图分类号:S571.1;S326 文献标识码:A 文章编号:1000-369X(2024)02-175-18
Identification of CAB Gene Family and Excavation of
Key Genes Related to Leaf Yellowing Variationin
Tea Plants (Camellia sinensis)
ZHONG Sitong1,2, ZHANG Yazhen2, YOU Xiaomei2, CHEN Zhihui2, KONG Xiangrui2,
LIN Zhenghe2, WU Huini1,2, JIN Shan1*, CHEN Changsong1,2*
1. College of Horticulture, Fujian Agriculture and Forestry University, Fuzhou 350002, China; 2. Tea Research Institute, Fujian Academy of Agricultural Sciences/Fujian Branch, National Center for Tea Improvement, Fuzhou 350012, China
Abstract: Members of the light-harvesting chlorophyll a/b binding protein (CAB) gene family play an important role in plant leaf yellowing variation. In this study, the CAB family members were identified from tea plant ‘Tieguanyin genomic data. The bioinformatics and expression patterns were analyzed. Furthermore, the expression patterns of the CABs gene were analyzed by gene cloning and qRT-PCR in tea cultivars with different leaf colors. The key CAB genes related to tea yellowing were screened by correlation analysis of leaf color parameters and chlorophyll SPAD values. The results show that 25 members of the CAB gene family were identified, their amino acid length ranged from 167-337 and the protein molecular weight ranged from 18.5-37.1 kDa. Most CAB members were stable and hydrophobic proteins, and distributed in chloroplast by the subcellular localization prediction. According to the evolutionary relationship, CAB family members are divided into 13 subfamilies, and the Lhcb1 subfamily has the most members. Cis-acting element analysis of promoter shows that CAB family members have a lot of light-responsive elements, as well as other elements related to growth and development, hormone response, and adversity stress. The members of Lhcb1 subfamily were cloned from tea plants, CAB1, CAB6, and CAB7 genes were screened by sequence alignment. The expression analysis shows that CAB1, CAB6, and CAB7 genes had tissue expression characteristics with higher expression levels in buds, leaves and fruits, and could respond to various stresses. Finally, the qRT-PCR indicates that the expressions of CAB1, CAB6, and CAB7 genes were consistent in the yellow and green leaves. Compared with green leaves, the expression of CAB genes in yellow leaves were significantly down-regulated. The correlation analysis of gene expressions and related leaf color parameters shows that the gene expressions of CAB1, CAB6, and CAB7 were significantly correlated with leaf color parameters a, b, L, and chlorophyll SPAD values (P<0.01). Among them, the expression of CAB1 shows the highest correlation coefficient. The subcellular localization analysis shows that CAB1 was distributed in the nucleus, cytoplasm, and chloroplasts. The studies analyzed the basic characteristics of CAB family members in tea plants and the key genes related to tea color variation were identified, which provided a theoretical basis for the molecular regulation mechanism of tea color variation.
Keywords: Camellia sinensis, CAB, yellowing variation, gene cloning, expression analysis
光合作用是植物生命活动之一,为植物的生长发育提供所需的能量和物质基础。叶绿体作为植物进行光合作用的主要场所,依赖类囊体膜上的光系统I(Photosystem I,PSI)、光系统Ⅱ(Photosystem Ⅱ,PSⅡ)以及细胞色素b6f等多个蛋白复合物协同作用,进行光能捕获、吸收和转换等过程[1]。捕光叶绿素a/b结合蛋白(Light-harvesting chlorophyll a/b binding protein,CAB)是分布在PSI和PSⅡ上的类囊体膜蛋白,通过与叶绿素、类胡萝卜素和叶黄素等色素分子结合,发挥光能吸收、能量分布,以及维持类囊体结构等作用[2]。根据结构和进化关系,高等植物的CAB蛋白家族被分成Lhca(Light-harvesting chlorophyll a/b binding proteins of photosystem I)和Lhcb(Light-harvesting chlorophyll a/b binding proteins of photosystem Ⅱ)两个蛋白亚族,分别由Lhca和Lhcb两个多基因家族编码。其中,Lhca又被细分为6个基因亚族Lhca1-6,是编码PSI中LHCI(Light harvesting complex I)的主要成员;Lhcb分为8个基因亚族Lhcb1-8,参与编码PSII中的LHCII(Light harvesting complex Ⅱ)[3]。
以往研究表明,CAB基因在植物叶绿体中存在特异表达,并具有叶片优先表达模式,对调控叶片的生长发育十分关键[4]。在拟南芥中,敲除或下调Lhcb基因,都会影响植株光合速率和叶绿素含量,从而出现浅绿色或泛白的叶色表型以及生长延迟的现象[5-7]。沉默陆地棉GhLhcb2.3和猕猴桃Lhcb3.1/3.2同样发现转基因植株叶绿素含量降低,尤其是影响叶绿素a的合成[8-9]。反之,过表达SaLhcb2的转基因烟草则能表现出更好的植株生长势、更高的生物量和叶绿素含量[10]。番茄LeLhcb2和枇杷EjLhc4.1/5/6的转基因植株也有类似地表现,并且在低温胁迫下也能维持优于野生型植株的生长状态[11-12]。此外,当植物面对不同的逆境胁迫时,CABs基因的表达水平也会通过上调或下调变化作出应答,在毛竹、东南景天和水稻等植物中已相继证明了CAB在植物抗逆方面起着至关重要的作用[13-15]。
黄化茶树是一类珍稀的茶树叶色突变体,更是制茶品质优异的宝贵种质资源,通过开展黄化茶树的分子机制和代谢机理相关研究,对改善黄化茶树品质和提高其生产利用具有重要意义。Ma等[16]对英红九号的黄化突变体的黄叶和绿叶进行蛋白组学分析,发现CAB蛋白、细胞色素b6f复合物和F型ATP酶等与光合作用相关的蛋白差异最显著。Liu等[17]对勐海黄叶的黄化叶、绿叶和返绿叶研究同样发现,与光合作用相关的基因和与叶绿素合成相关的基因存在显著差异表达,其中,编码CAB蛋白的多个基因在黄化叶中显著下调。中黄1号、白叶1号和福黄1号等其他黄化茶树中也有类似发现,即CAB基因家族成员在黄化茶树中的表达量会显著低于绿叶茶树[18-20]。上述研究表明,CAB基因表达量的变化与茶树叶色变异紧密相关。然而,目前少有研究针对茶树完整的CAB基因家族成员进行深入分析,并且在以往针对茶树CAB基因家族成员的克隆研究中,试验材料通常采用单一绿叶或黄化茶树,需进一步扩大样本量,在更多的材料中进行验证。与茶树黄化密切相关的CAB基因家族成员还有待挖掘,关于茶树CAB基因的作用机制也有待研究。因此,本研究首先用铁观音茶树基因组筛选出CAB基因家族成员,通过生物信息学方法对其基因结构、启动子和系统发育等方面进行分析。其次,基于前人研究结果,重点研究与叶色调控密切相关的Lhcb1亚家族成员,通过克隆Lhcb1亚家族的CABs基因,研究其在不同黄、绿茶树品种间的表达模式,并评估CABs基因表达量与叶色参数和叶绿素含量的相关性,以期挖掘出与茶树叶色变异紧密相关的关键CAB基因,为深入研究茶树CAB基因的作用机制提供试验基础。
1 材料与方法
1.1 试验材料
以种植于福建省农业科学院茶叶研究所的茶树种质资源为试验材料,于2023年4月(15~27 ℃)选取绿叶茶树种质福鼎大白茶、黄棪、福云6号和白鸡冠自然杂交后代0306A、0317D、0309E;黄、白化茶树种质白叶1號、白鸡冠及其自然杂交后代0309C、0317L、0317F、0317H、0306E、0306C、0306D的一芽一叶新梢,液氮速冻,置于﹣80 ℃冰箱保存备用。
试验材料白叶1号采样期间属于返绿阶段。经多年田间性状调查0306A、0317D、0309E 3个材料属于表型稳定的绿叶茶树,而白鸡冠及其自然杂交后代的7个黄化株系材料在自然生长状态下,一芽一叶新梢也具有稳定的黄化表型,无返绿现象。
本氏烟草(Nicotiana benthamiana)种植:播种烟草种子若干于土壤中,在25 ℃、12 h光照/12 h黑暗条件下培养1个月后用于试验。
1.2 试验方法
1.2.1 茶树CAB基因家族成员鉴定
从铁观音茶树数据库中获取全基因组蛋白序列文件和基因注释文件,以拟南芥CAB家族成员的蛋白序列为索引,在铁观音基因组中比对出CAB基因家族的候选成员。结合CAB家族的隐马可夫模型(http://pfam.sanger. ac.uk/search)PF00504,以及NCBI中的BLAST结果共同筛选出CAB家族成员。
1.2.2 茶树CABs生物信息学分析
利用ExPASy网站(https://web.expasy.org/
protparam)对CAB基因家族成员的理化性质进行分析,并利用WoLF PSORT在线网站(https://wolfpsort.hgc.jp)预测成员的亚细胞定位;使用MEME软件(https://meme-suite.org/meme)在线预测CAB家族成员的保守基序,保守基序数量设置为10;通过NCBI-CDD数据库(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)分析CAB蛋白的结构域信息;从基因组中获取CAB家族成员转录起始位点上游2 000 bp序列,使用PlantCARE网站预测其启动子顺式作用元件。然后,采用TBtools软件对候选CABs的系统进化树、保守基序、启动子顺式作用元件和基因结构进行可视化;根据基因组注释文件,用TBtools获取CAB基因家族成员的染色体位置信息并绘制基因染色体定位图。
从Phyutozome v13数据库(https:// phyto-zome-next.jgi.doe.gov)中下载蓖麻、番木瓜、水稻和拟南芥的全基因组数据,参照文献[21-23]筛选出CAB基因家族成员。利用MEGA 7.0软件中的Muscle完成茶树CAB基因家族和不同物种CAB基因家族的氨基酸序列比对,并通过邻接算法(自检举1 000次)构建系统进化树。
1.2.3 茶树CABs基因克隆及序列分析
采用RNA提取试剂盒(FastPure Universal Plant Total RNA Isolation Kit,南京Vazyme公司)提取茶树叶片的总RNA,使用BioSpec-nano(岛津,日本)紫外分光光度计检测RNA 浓度,利用1.2%凝胶电泳检测RNA质量。按照反转录试剂盒(FastKing gDNA Dispelling RT SuperMix,北京Tiangen公司)进行逆转录,将获得的cDNA置于﹣20 ℃冰箱保存。
根据筛选出的候选CABs基因的开放阅读框(ORF)设计上、下游引物进行扩增(表1)。PCR扩增体系为50 μL,包括2×Phanta Max Master Mix酶25 μL,ddH2O 19 μL,模板cDNA 2 μL,上、下游引物各2 μL,反应程序为95 ℃预变性3 min;95 ℃ 15 s,48 ℃ 15 s,72 ℃ 1 min,共进行40个循环;72 ℃延伸5 min。PCR产物利用DNA纯化回收试剂盒(TIANGEN,北京)回收目的片段,根据5 min TA/Blunt-Zero Cloning Kit(Vazyme,南京)说明书将目的片段与载体连接后,热激转化大肠杆菌DH5α,挑取阳性单克隆送生工生物工程(上海)股份有限公司测序。
采用DNAMAN 6.0软件对CABs测序结果进行多序列比对,并用MOTIFSCAN软件对CABs蛋白的功能结构域进行分析。
1.2.4 茶树CABs基因表达分析
使用NCBI在线软件设计茶树CAB1、CAB6和CAB7的qRT-PCR引物(表1),以GAPDH为内参基因,参照Taq Pro Universal SYBR qPCR Master Mix(Vazyme,南京)试剂盒说明配制反应体系。利用CFX Connect荧光定量PCR仪(Bio-Rad,美国)进行qRT-PCR反应,使用 方法分析3次生物重复的数据。
通过TPIA(http://tpdb.shengxin.ren)数据库获取茶树CAB1、CAB6和CAB7基因的转录组数据,以TPM(Transcripts per million)值评估CAB1、CAB6和CAB7基因在茶树根、茎、叶、花和果实等组织以及在非生物胁迫下的表达量,利用Tbtools软件绘制热图。
1.2.5 与叶色的相关性分析
叶片色泽测定:使用色差计(ltraScanVIS1195 HunterLab colorimeter)测定茶树叶片的L、a和b值。L值表示明亮度,数值越大越鲜亮;a值表示红绿色彩程度,正值偏红,负值偏绿;b值表示黄蓝色彩程度,正值偏黄,负值偏蓝。使用色差计前用白色标准板校正,并以白色标准板为背景,随机选取供试茶树种质的10个芽下第二叶进行3次重复测定,记录和计算叶片的L、a和b数值。
叶绿素SPAD值测定:使用叶绿素计SPAD-502Plus测定茶树资源的叶绿素含量,选取茶树芽下的第三叶进行测定,避开叶脉,10次重复,取平均值。
采用SPSS软件的Pearson评估CABs基因的表达量与茶树叶片色泽和叶绿素含量的相关性。
1.2.6 亚细胞定位
根据CAB1的基因全长序列设计带有酶切位点的引物,具体序列见表1。参照BioRun Seamless Cloning Kit(#RDA01)试剂盒(武汉伯远公司)说明书将CAB1酶切后的DNA片段连接到pBWA(V)BS-ccdb-linker-gfp载体上,进行酶切鉴定和测序验证。将构建好35S::CAB1:GFP重组质粒转化到农杆菌GV3101中,30 ℃扩大培养2 d,将重悬菌液注射至生长状况良好的烟草叶片下表皮,弱光培养2 d。取農杆菌注射的烟草叶片,制作成玻片,激光共聚焦显微镜下观察并拍照记录。
2 结果与分析
2.1 茶树CAB家族基因鉴定及理化性质分析
通过BLATSP和HMMER共同检索,在茶树中筛选出25个CAB基因家族成员(表2)。CAB家族编码167~337个氨基酸,多数家族成员编码250~270个氨基酸;CAB家族蛋白相对分子质量范围为18.5~37.1 kDa;等电点为5~8.54,等电点大于7的成员只有TGY022607.t1、TGY067398.t1、TGY031151.t1、TGY043134.t1和TGY031987.t1,说明CAB家族成员大多为酸性蛋白;平均亲水性为0.03208,除TGY091686.t1、TGY001087.t1、TGY050556.t1、TGY043134.t1、TGY014634.t1和TGY125121.t1这6个CAB成员是亲水性蛋白外,其余成员都是疏水性蛋白;蛋白不稳定系数在16.35~49.16,大部分CAB成员都属于稳定性蛋白,只有TGY067398.t1、TGY031151.t1、TGY001087.t1、TGY125121.t1和TGY020806.t1不稳定系数大于40,为不稳定性蛋白;亚细胞定位预测结果显示,只有TGY067398.t1和TGY031151.t1分布在细胞膜上,其余CAB家族成员都分布在叶绿体上。
2.2 茶树CAB基因家族生物信息学分析
对25个茶树CAB基因家族成员进行生物信息学分析(图1),蛋白结构域结果显示(图1B),CAB家族成员都具有捕光叶绿素a/b结合蛋白功能域,且蛋白保守结构域包含的氨基酸序列长度占总长度的80%以上;基因结构分析发现(图1C),茶树CAB基因家族成员的外显子有1~2个,内含子有1~6个;通过保守基序预测发现(图1D),CAB家族成员的基序分布整体偏向于N端,其中基序motif2和motif8的保守性校强,在25个CAB成员中均有出现,其次是motif3、motif7和motif10,而motif4、motif6和motif9只分布在部分CAB成员。结合系统发育分析(图1A),发现聚为一类群的CAB成员都具有相似的基因结构和保守基序,说明不同CAB亚家族之间可能因为存在基因结构和基序种类的差异而具有不同的生物学功能。进一步通过染色体定位分析发现,茶树CAB家族成员在染色体上分布不均匀。如图2所示,25个CAB基因分布在12条染色体上,其中,有7个成员定位在第8号染色体,4个成员定位在第7号染色体,第2号、4号、5号和10号染色体上均有2个CAB基因分布,其余染色体上只有1个CAB基因。
利用PlantCARE对茶树25个CAB基因上游2 000 bp序列的启动子区进行顺式作用元件分析,除去一般性转录调控元件和功能未知元件外,获得与植物生长发育、激素及逆境胁迫相关的顺式作用元件(图3)。与植物生长发育相关的元件有6种,包括大量的光响应元件(Box 4、G-Box和TCT-motif等)、昼夜节律调控元件(circadian)、分生组织表达(CAT-box)、参与胚乳表达元件(GCN4-motif)、参与叶肉细胞分化元件(HD-Zip 1)和细胞周期调节元件(MSA-like);激素响应元件有5种,包括赤霉素(TATC-box、P-box和gibberellin-responsive element,GARE-motif)、生长素(TGA-element和auxin responsiveness,AuxRR-core)、水杨酸(TCA-element)、脱落酸(Abscisic acid responsive element,ABRE)和茉莉酸甲酯(CGTCA-motif);逆境胁迫响应元件有4种,包括低温(Low temperature responsiveness,LTR)、厌氧诱导(Antioxident response element,ARE)、干旱(MYB binding site involved in drought inducibility,MBS)、防御和胁迫响应(TC-rich repeats)。其中,光响应元件(Box 4、G-Box和TCT-motif等)数量最多,普遍存在于每一个CAB基因的启动子区,表明茶树CAB基因主要受光信号调控,且光响应元件可能在CABs基因调控茶树叶色方面发挥着重要作用。脱落酸响应元件、茉莉酸甲酯响应元件和厌氧诱导响应元件的数量也较多,表明茶树CAB基因也会受脱落酸和茉莉酸信号调控。此外,赤霉素响应元件、生长素响应元件、低温响应元件和干旱响应元件也在大部分CAB基因启动子中被鉴定到,说明这些元件在茶树应对逆境胁迫方面可能具有重要作用。
为了解茶树CAB基因家族的分类和进化关系,利用MEGA 7.0软件对茶树、水稻、拟南芥、蓖麻、番木瓜中的CAB家族进行系统发育分析。如图4所示,CAB基因家族聚为14个分枝,分别对应Lhcb的8个亚家族和Lhca的6个亚家族。茶树CAB家族的25个成员分布在13个亚家族中,只有Lhcb8亚家族未聚类到茶树CAB成员。其中,Lhcb1亚家族聚类到的CAB成员数目最多,茶树有7个,拟南芥有5个,水稻、蓖麻和番木瓜均有3个。其次是Lhcb2亚家族,包含3个茶树CAB成员。Lhcb4、Lhcb6、Lhcb7、Lhca2和Lhca4亚家族均包含2个茶树CAB成员,其余亚家族只含1个CAB成员。此外,茶树CAB基因家族与蓖麻和番木瓜的CAB基因家族亲缘关系较近,暗示茶树的CABs可能具有与蓖麻、番木瓜相似的功能。在过往研究中,植物Lhcb1是CAB家族中研究最广泛和最深入的亚家族,并且已有研究表明Lhcb1与植物叶色调控密切相关,因此,本研究选取Lhcb1亚族的7个CAB基因进行后续试验,并依据基因ID号分别命名为CAB1、CAB2、CAB3、CAB4、CAB5、CAB6、CAB7。
2.3 茶树CABs基因克隆及序列分析
以铁观音基因组中获得的CAB基因序列为参考,福鼎大白茶新梢叶片的cDNA为模板,设计全长引物,进行CABs基因克隆。测序结果如图5所示,5个CAB基因(CAB1、CAB2、CAB3、CAB4和CAB5)的开放阅读框序列及其编码的氨基酸序列高度一致,为同一基因。
进一步对CAB1、CAB6和CAB7的氨基酸序列进行序列相似度及保守结构域分析(图6),结果表明,3个基因编码的氨基酸序列相似度为97.36%;MOTIFSCAN分析结果显示,在第64~232位包含一个捕光叶绿素a/b结合蛋白功能域(Chlorophyll a/b binding domain,CB);第23~26位有酪氨酸蛋白激酶Ⅱ磷酸化位点(Casein kinase Ⅱ phosphorylation site,CK2);第108~113、122~127、150~155、169~174和178~183位为N-肉豆蔻酰位点(N-myristoylation site);第34~36和234~236位为蛋白激酶C磷酸化位点(Protein kinase C phosphorylation site,Pc)。CAB1、CAB6和CAB7的多个结构域高度保守,说明三者行使的功能存在相似性。
2.4 茶樹CABs表达模式分析
从TPIA数据库中下载CAB1、CAB6和CAB7茶树不同组织以及不同逆境胁迫下的表达量数据,对其表达模式进行分析(图7)。结果显示,CABs在茶树芽、叶和果实中的表达量较高,茎、根和花中的表达量较低。在叶片不同的生长发育阶段,CAB1和CAB6在嫩叶中的表达量最高,CAB7则在老叶中表达量最高,表明CABs可能在茶树叶片生长发育过程中发挥不同的作用。在4种非生物胁迫处理下CABs的表达水平呈现了相似的变化趋势,在冷处理下,CABs表达水平均受抑制,在恢复常温后表达上调;在茉莉酸甲酯(MeJA)胁迫中,处理12 h时CABs表达显著上调,而后随着胁迫时间增加表达水平逐渐下降;在盐处理下,CABs均下调表达,其中CAB6在处理48 h时表达水平呈先上升后下降趋势,CAB7则在处理72 h时表达量上升;在干旱处理下,CAB1和CAB7的表达水平均随处理时间增加而下降,而CAB6的表达水平在处理48 h时,有明显的上调。
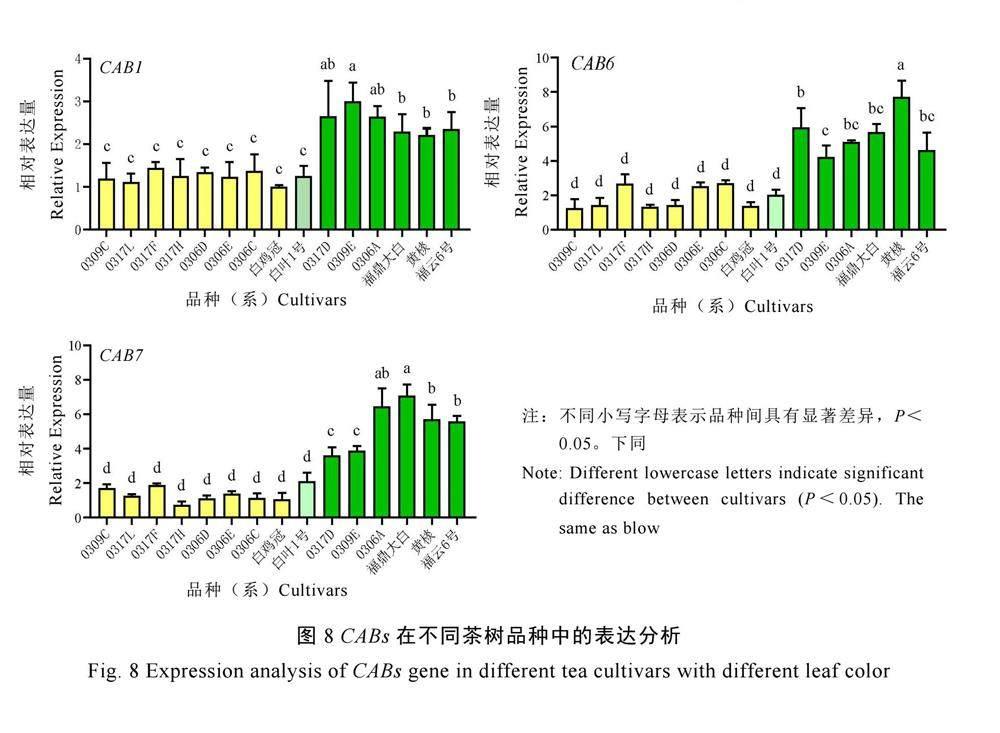
为验证茶树CABs基因在不同黄、绿茶树品种(系)之间的表达差异,本研究使用qRT-PCR技术,测定了不同叶色茶树叶片中的CABs基因表达量。如图8所示,CAB1在绿叶茶树0309E中的表达量最高,在黄化茶树白鸡冠中的表达量最低,差异达3倍;而CAB6在绿叶茶树黄棪的表达量最高,是白鸡冠表达量的5.6倍;CAB7则在福鼎大白茶中的表达量
最高,在黄化茶树0317最低。整体来看,CAB1、CAB6和CAB7在黄、绿叶中的表达水平都具有显著性差异,与绿色叶片相比,3个基因都在黄化叶片中显著低表达。
2.5 与叶色的相关性分析
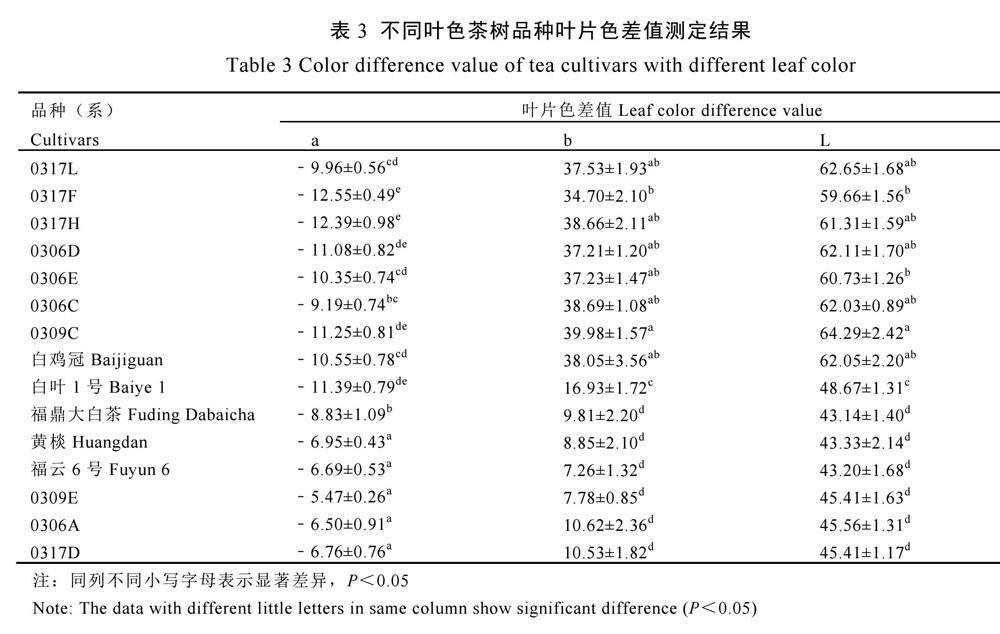
利用叶片色差计和叶绿素仪对15个黄、绿叶茶树品种(系)的叶片色泽性状进行分析。叶片色差值结果如表3所示,与叶色表型一致,黄叶的明亮度L值均高于绿叶的明亮度,其中,以黄化茶树0309C的明亮度最高;a值反映叶片绿色程度,黄叶的a值普遍低于绿叶a值;b值反映叶片黄色程度,黄叶b值普遍高于绿叶b值,其中0309C的b值最高。茶树叶片的色泽與叶片的叶绿素含量是密切相关的,叶绿素SPAD值结果显示(图9),福6号的SPAD值最高,其次是0309E和0306A,黄化茶树0306C、0309C和0317L的SPAD值最低。整体来看,绿叶的SPAD值均比黄叶的SPAD值高,与叶片色差参数反映结果一致。
进一步利用Pearson评估茶树CABs基因的表达量与茶树叶色的相关性。结果如表4所示,CAB1、CAB6和CAB7基因表达量与茶树叶片叶绿素含量(SPAD值)均呈极显著正相关,CAB1基因的相关性系数最高(0.913**),其次是CAB7,CAB6的相关性最低。此外,CAB1、CAB6和CAB7表达量与叶片色差值的a、b和L值也都呈极显著相关,其中与a值呈正相关,相关性表现为CAB1> CAB6>CAB7;与b值呈负相关,相关性表现为CAB1>CAB7>CAB6;与L值呈负相关,相关性表现为CAB7> CAB1>CAB6。综合以上结果,发现CAB1基因与茶树叶色性状的相关性最为显著,后续可作为关键基因进行深入探究。
2.6 茶树CAB1克隆及亚细胞定位
为进一步探究CAB1基因在不同叶色茶树品种中是否存在差异,本研究分别以黄棪、白叶1号、白鸡冠及其后代黄叶的0306E和绿叶的0306A茶树cDNA为模板,通过PCR扩增,再次克隆了CAB1基因。经测序比对可知(图10),CAB1在不同黄、绿茶树品种中的氨基酸序列一致性较高,序列相似度达到99.69%,说明CAB1在不同茶树品种中高度保守。
为明确CAB1的亚细胞定位模式,构建了带有35 S启动子及GFP标签的过表达载体35S::CAB1:GFP,通过质粒电转化法转入农杆菌GV3101中,再分别转化至烟草叶片进行瞬时表达,并使用激光共聚焦显微镜观察表达情况。结果如图11所示,空载体35S:GFP(对照)的绿色荧光信号分布在烟草的细胞质、细胞核和质膜中,叶绿体则呈自发红色荧光,绿色荧光与红色荧光无法重叠;而在35S::CAB1:GFP的绿色荧光能与叶绿体自发荧光信号重叠,呈现出黄色荧光(图中白色箭头标注),结果与WoLF PSORT的预测一致,CAB1定位于叶绿体,且在细胞核和细胞膜上也均有表达。
3 讨论
目前,已经从水稻[21]、蓖麻[22]、拟南芥[24]、木薯[25]和苹果[26]等植物中分别鉴定出21、17、20、16和27个CAB基因家族成员。本研究通过对铁观音的全基因组进行分析,从中筛选出了25个茶树CAB基因家族成员,其中Lhcb亚家族成员有17个,Lhca亚家族成员有8个,除了Lhcb8亚家族未聚类到茶树CAB成员外,其他成员在Lhcb1~Lhcb7和Lhca1~Lhcb6亚族中均有分布,这可能是因为Lhcb8是由Lhcb4进化而来,因此Lhcb8亚族与Lhcb4亚族属于同源,需要进行蛋白质水平鉴别才能区分其是否存在[24,27]。与拟南芥、番木瓜、蓖麻和水稻的CAB家族成员相比,茶树的Lhcb1和Lhcb3亚家族的成员数目最多,这种不同物种的CAB家族成员数量差异,与CAB基因进化过程中不同亚基因组内所发生基因复制事件有关,从而导致同源基因对出现拷贝数的扩增[28]。Ye等[29]利用舒茶早和龙井43茶树数据库筛选出了20个Lhcb亚家族成员,比本研究多2个CAB家族成员,数量差异主要出现在Lhcb1和Lhcb3亚族中,该研究鉴定到Lhcb1亚族有4个CAB成员,Lhcb3有7个CAB成员。Luo等[9]对两个猕猴桃品种(Actinidia chinensis和A. eriantha)的CAB基因家族进行研究,同样发现两个品种的CAB家族成员数存在差异。CAB基因在铁观音茶树基因组上分布较为广泛,遍布12条染色体,且在第7、8号染色体上分布数目较多,这与CAB家族在舒茶早和龙井43基因组染色体上分布情况一致,表明第7、8号染色体上存在CAB基因簇且这些基因可能为连锁遗传。
CAB蛋白是植物叶绿体内含量最为丰富的一类膜蛋白,约占总叶绿体膜蛋白的1/3,其结合了类囊体膜上50%的色素[30]。因此,CAB基因属于与叶绿体发育密切相关的功能基因,在植物光合作用中发挥着重要作用。本研究对茶树CAB基因家族进行了亚细胞定位预测,结果显示大部分CAB家族成员都定位于叶绿体上,且茶树CAB基因家族成员的启动子区域都包含有大量与光响应相关的作用元件,如Box II、I-box、Box 4、GT1-box和G-box等,说明CAB基因在茶树光合作用中的重要性以及在调控叶色方面具有重要作用。在众多光响应元件中,Box 4和G-box元件的数量较多。研究表明,G-box元件的核心序列ACGT在CAB基因中高度保守,能与HY5、GBF2和PIF3等转录因子特异结合,调控CAB基因的表达[31]。水稻的mEmBP-1a转录因子也能直接结合到G-box结构域上促进CAB基因的表达,进而提高水稻光合作用和产量[32]。Liu等[33]研究发现,茶树CsHY5能直接与CsLhcb的G-box(位于ATG上游﹣750 bp)结合,在绿叶中促进Lhcb表达,在黄叶中则抑制Lhcb表达,从而影响黄魁的季节性返绿。还有MYB家族的CsRVE1转录因子能结合到CsLhcb的G-box区域,在黄魁返绿过程中,CsRVE1通过过表达能激活CsLhcb,促进叶绿素的积累[34]。可见,G-box是CAB基因发挥作用的重要元件,本研究筛选的CABs基因也可能通过G-box元件受转录因子调控,参与调控茶树叶色变异。此外,茶树CAB基因启动子区还包含响应植物激素、干旱和冷害等非生物胁迫的顺式作用元件,说明CAB基因也可能在茶树抗逆过程中发挥重要作用。Li等[35]对茶树Lhcb2和Lhcb5亚家族基因进行克隆,发现各CAB成员在脱落酸、茉莉酸甲酯、机械损伤和冷胁迫等处理中具有不同的表达调控模式,Lhcb5亚族成员普遍受抑制。还有研究同样通过克隆茶树CAB基因,发现其参与茶树低温和干旱胁迫过程[36-37]。
Lhcb1蛋白是目前结构和功能研究最為深入的色素结合蛋白,其成员数最多,含量最为丰富,可以结合12个叶绿素(包含7个叶绿素a,5个叶绿素b)和2个叶黄素分子[30]。已有大量研究表明,Lhcb1亚家族成员的表达水平能直接反映叶绿素的含量及叶绿体的功能状态,如橡胶树HbCAB1在衰老期叶片中的表达量显著降低,与叶片叶绿素含量的减少呈正相关[38];紫薇无叶绿素显型突变体叶片的黄化也与CAB1基因表达量降低有关[39];敲除拟南芥编码Lhcb1蛋白的AtLhcb1.1~1.5 5个基因,其他Lhcb成员虽然会积累,但不能完全取代Lhcb1的作用以及挽救叶绿素的损失,最终导致植株叶片黄化和生长延迟[40]。根据前人研究结果,推测茶树Lhcb1亚族基因也在调控叶色方面具有重要作用,因此本研究选取Lhcb1亚家族的7个CABs基因进一步深入研究。首先,从福鼎大白茶中克隆了Lhcb1亚家族的7个CAB基因,通过核酸序列比对发现有5个CAB基因的核酸序列完全一致,其他亚族成员是否存在同样情况有待验证。然后,从中筛选出CAB1、CAB6和CAB7基因进行氨基酸序列比对,发现这3个基因氨基酸序列一致性较高。最后,通过茶树不同组织部位的基因表达特性分析发现,这3个基因在茶树芽和叶中均大量表达,在茎、根和花中的表达量则较低,与马尾松[41]和苎麻[42]的CAB基因表达模式相似。此外,CAB1、CAB6和CAB7基因可能在抵御逆境胁迫过程中起重要作用,3个基因能对激素、干旱、低温和盐胁迫有不同程度的应答,并在同一胁迫下三者的表达水平呈现出相似的变化趋势,推测CAB1、CAB6和CAB7基因可能具有相似的功能。
为明确CAB1、CAB6和CAB7基因在不同叶色茶树中的表达差异,分析了3个基因在9个黄化茶树材料和5个绿叶茶树材料中的表达水平,发现3个基因在黄叶中的表达量均显著低于在绿叶中的表达量,表达量差异达到3~9.6倍。随后测定了黄、绿茶树品种(系)的叶色相关参数和叶绿素SPAD值,结果与叶色表型一致,黄化茶树的叶色参数a、b、L值和叶绿素SPAD值都与绿叶茶树有显著性差异。通过相关性分析,发现CAB1、CAB6和CAB7基因表达量与叶色参数L、a、b和叶绿素SPAD值都呈极显著相关,其中以CAB1的相关性最为显著,说明CAB1基因的表达与茶树叶色性状密切相关。
为了探究CAB1基因编码区序列在黄化叶与绿色叶片中是否存在差异,进一步以2种绿叶茶树和3种黄化茶树为材料,再次克隆了CAB1基因,发现CAB1基因在黄、绿茶树品种中只存在个别单核苷酸位点差异,翻译后的氨基酸序列呈现高度一致。这与Ye等[29]以2个绿叶茶树和5个温度敏感型白化茶树为材料克隆的Lhcb1亚族基因的结果相同,说明Lhcb1亚族的CAB基因成员在不同叶色茶树中的氨基酸序列高度保守。进一步通过烟草亚细胞定位分析,证实了CAB1在叶绿体中的分布。茶树叶色的变异往往涉及叶绿体形态结构的变化,特别是体现在叶绿体的超微结构上。前人通过对茶树黄化叶片叶绿体超微结构的观察发现,与绿色正常叶色相比,黄化叶片中叶绿体结构发育异常,主要表现为基粒片层堆叠减少,类囊体结构被破坏,甚至消失[43-45]。结合本研究的试验数据及CAB1的亚细胞定位结果,推测可能是由于黄化叶片中CAB1表达受抑制,使CAB1蛋白无法与叶绿素结合,难以形成稳定的类囊体膜结构,从而导致叶色黄化变异。
本研究首次对铁观音基因组中的CAB基因家族进行鉴定和分析,丰富了茶树CAB家族基因库资源。在25个CAB家族成员的启动子区发现了大量的光响应元件以及激素响应、逆境胁迫相关的作用元件,暗示CAB基因家族在茶树的光合作用和抗逆方面具有重要作用。本研究发现CAB1、CAB6和CAB7表达量与叶色参数a、b、L值以及叶绿素SPAD值均呈极显著相关(P<0.01),其中以CAB1相关性最为显著。烟草亚细胞定位结果显示,CAB1在细胞膜、细胞核和叶绿体上均有分布。以上结果表明,CAB1与茶树叶色黄化变异紧密相关,后续可作为关键基因进行深入探究。
参考文献
[1] 苏小东, 李梅. 高等植物光系统复合物结构生物学研究进展[J]. 自然杂志, 2021, 43(3): 165-175.
Su X D, Li M. Advances in structural biology of photosystem complexes in higher plants [J]. Chinese Journal of Nature, 2021, 43(3): 165-175.
[2] 石兰馨, 张晓平, 梁厚果. 捕光叶绿素a/b结合蛋白和cab基因[J]. 植物生理学通讯, 1995, 31(6): 470-476.
Shi L X, Zhang X P, Liang H G. Light harvesting chlorophyll a/b binding protein and cab gene [J]. Plant Physiology Communications, 1995, 31(6): 470-476.
[3] 羅玲. 捕光色素蛋白复合物的研究进展[J]. 现代农业科技, 2008, 1(22): 270-273.
Luo L. Research progress of light-harvesting pigment protein complexes [J]. Modern Agricultural Science and Technology, 2008, 1(22): 270-273.
[4] Sheen J Y, Bogorad L. Differential expression of six light-harvesting chlorophyll a/b binding protein genes in maize leaf cell types [J]. Proceedings of the National Academy of Sciences, 1986, 83(20): 7811-7815.
[5] Andersson J, Wentworth M, Walters R G, et al. Absence of the lhcb1 and lhcb2 proteins of the light-harvesting complex of photosystem Ⅱ: effects on photosynthesis, grana stacking and tness [J]. The Plant Journal, 2003, 35(3): 350-361.
[6] Kim E H, Li X P, Razeghifard R, et al. The multiple roles of light-harvesting chlorophyll a/b-protein complexes define structure and optimize function of Arabidopsis chloroplasts: A study using two chlorophyll b-less mutants [J]. Biochimica et Biophysica Acta (BBA)-Bioenergetics, 2009, 1787(8): 973-984.
[7] Ilíková I, Ilík P, Opatíková M, et al. Towards spruce-type photosystem II: consequences of the loss of light-harvesting proteins LHCB3 and LHCB6in Arabidopsis [J]. Plant Physiology, 2021, 187(4): 2691-2715.
[8] Zhang Q, Ma C, Wang X, et al. Genome-wide identification of the light-harvesting chlorophyll a/b binding (Lhc) family in Gossypium hirsutum reveals the influence of GhLhcb2.3 on chlorophyll a synthesis [J]. Plant Biology, 2021, 23(5): 831-842.
[9] Luo J, Abid M, Tu J, et al. Genome-wide identification of the LHC gene family in kiwifruit and regulatory role of AcLhcb3.1/3.2 for chlorophyll a content [J]. International Journal of Molecular Sciences, 2022, 23(12): 6528. doi: 10.3390/ijms23126528.
[10] Zhang M, Senoura T, Yang X, et al. Lhcb2 gene expression analysis in two ecotypes of Sedumalfredii subjected to Zn/Cd treatments with functional analysis of SaLhcb2 isolated from a Zn/Cd hyperaccumulator [J]. Biotechnology Letters, 2011, 33(9): 1865-1871.
[11] Deng Y S, Kong F Y, Zhou B, et al. Heterology expression of the tomato LeLhcb2 gene confers elevated tolerance to chilling stress in transgenic tobacco [J]. Plant Physiology and Biochemistry, 2014, 80: 318-327.
[12] 李韵佳. 低温胁迫下三倍体枇杷微量捕光天线蛋白基因EjLhcb4.1/5/6的功能分析[D]. 重庆: 西南大学, 2020.
Li Y J. Functional characterization of light harvesting chlorophyll a/b binding antenna proteins EjLhcb4.1/5/6 genes in triploid loquat (Eriobotrya japonica) under cold stress [D]. Chongqing: Southwest University, 2021.
[13] 唐文莉. 毛竹Lhca基因的克隆和光照在转录水平对其表达的调控[D]. 北京: 中国林业科学研究院, 2008.
Tang W L. Cloning Lhca genes and at the transcriptional level light intensity regulating their expression of Phyllostachys edulis [D]. Beijing: Chinese Academy of Forestry, 2008.
[14] 李真, 刘明英, 韩小娇, 等. 东南景天捕光叶绿素a/b结合蛋白基因SaLhcb2的分离及功能[J]. 浙江农林大学学报, 2014, 31(6): 838-846.
Li Z, Liu M Y, Han X J, et al. Characterization of a light-harvesting chlorophyll a/b binding protein (LHCB) geneSaLhcb2 in Sedum alfredii [J]. Journal of Zhejiang A & F University, 2014, 31(6): 838-846.
[15] Liu M, Zhang S, Hu J, et al. Phosphorylation-guarded light-harvesting complex II contributes to broad-spectrum blast resistance in rice [J]. Proceedings of the National Academy of Sciences, 2019, 116(35): 17572-17577.
[16] Ma C, Cao J, Li J, et al. Phenotypic, histological and proteomic analyses reveal multiple differences associated with chloroplast development in yellow and variegated variants from Camellia sinensis [J]. Scientific Reports, 2016, 6(1): 1-15.
[17] Liu Y, Pang D, Jiang H, et al. Identifying key genes involved in yellow leaf variation in ‘MenghaiHuangye based on biochemical and transcriptomic analysis [J]. Functional & Integrative Genomics, 2022, 22(2): 251-260.
[18] Wang L, Cao H, Chen C, et al. Complementary transcriptomic and proteomic analyses of a chlorophyll-deficient tea plant cultivar reveal multiple metabolic pathway changes [J]. Journal of Proteomics, 2016, 130: 160-169.
[19] Ma C L, Chen L, Wang X C, et al. Differential expression analysis of different albescent stages of ‘AnjiBaicha (Camellia sinensis (L.) O. Kuntze) using cDNA microarray [J]. Scientia Horticulturae, 2012, 148: 246-254.
[20] 林馨穎, 王鹏杰, 杨如兴, 等. 高茶氨酸茶树新品系‘福黄1号黄化变异机理[J]. 中国农业科学, 2022, 55(9): 1831-1852.
Lin X Y, Wang P J, Yang R X, et al. The albino mechanism of a new high theanine tea cultivar Fuhuang 1 [J]. Scientia Agricultura Sinica, 2022, 55(9): 1831-1852.
[21] Umate P. Genome-wide analysis of the family of light-harvesting chlorophyll a/b-binding proteins in Arabidopsis and rice [J]. Plant Signaling & Behavior, 2010, 5(12): 1537-1542.
[22] Zou Z, Huang Q, An F. Genome-wide identification, classification and expression analysis of Lhc supergene family in castor bean (Ricinus communis L.) [J]. Agricultural Biotechnology, 2013, 2(6): 44. https://www.
proquest.com/scholarly-journals/genome-wide-identification-classification/docview/1476279495/se-2.
[23] Zou Z, Li M, Jia R, et al. Genes encoding light-harvesting chlorophyll a/b-binding proteins in papaya (Carica papaya L.) and insight into lineage-specific evolution in Brassicaceae [J]. Gene, 2020, 748: 144685. doi: 10.1016/j.gene.2020.144685.
[24] Klimmek F, Sjo?din A, Noutsos C, et al. Abundantly and rarely expressed Lhc protein genes exhibit distinct regulation patterns in plants [J]. Plant Physiology, 2006, 140(3): 793-804.
[25] Zou Z, Yang J. Genomics analysis of the light-harvesting chlorophyll a/b-binding (Lhc) superfamily in cassava (Manihot esculenta Crantz) [J]. Gene, 2019, 702: 171-181.
[26] Zhao S, Gao H, Luo J, et al. Genome-wide analysis of the light-harvesting chlorophyll a/b-binding gene family in apple (Malus domestica) and functional characterization of MdLhcb4.3, which confers tolerance to drought and osmotic stress [J]. Plant Physiology and Biochemistry, 2020, 154: 517-529. doi: 10.1016/j.plaphy.2020.06.022.
[27] Wang Y, Tan X, PatersonA. Different patterns of gene structure divergence following gene duplication in Arabidopsis [J]. BMC Genomics, 2013, 14(1): 652. doi: 10.1186/1471-2164-14-652.
[28] Cannon S B, Mitra A, Baumgarten A, et al. The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana [J]. BMC Plant Biology, 2004, 4(10): 1-21.
[29] Ye J, Lin X, Yang Z, et al. The light-harvesting chlorophyll a/b-binding proteins of photosystem Ⅱ family members are responsible for temperature sensitivity and leaf color phenotype in albino tea plant [J]. Journal of Advanced Research, 2023. doi: 10.1016/j.jare.2023.12.017.
[30] 孫钦秒, 冷静, 李良璧, 等. 高等植物光系统Ⅱ捕光色素蛋白复合体结构与功能研究的新进展[J]. 植物学通报, 2000, 17(4): 289-301.
Sun Q M, Leng J, Li L B, et al. Recent advances of studies on the structure and function of the light-harvesting chlorophyll a/b protein complex [J]. Chinese Bulletin of Botany, 2000, 17(4): 289-301.
[31] 张毅, 尹辉, 李丹, 等. 植物环境响应启动子的诱导元件及转录因子[J]. 中国生物工程杂志, 2007(7): 122-128.
Zhang Y, Yin H, Li D, et al. The cis-elements and transcription factors of plant environmental response promoters [J]. China Biotechnology, 2007(7): 122-128.
[32] Perveen S, Qu M, Chen F, et al. Overexpression of maize transcription factor mEmBP-1 increases photosynthesis, biomass, and yield in rice [J]. Journal of Experimental Botany, 2020, 71(16): 4944-4957.
[33] Liu X, Cheng X, Cao J, et al. UV-B regulates seasonal greening of albino leaves by modulating CsHY5-inhibiting chlorophyll biosynthesis in Camellia sinensis cv. Huangkui [J]. Plant Science, 2023, 328: 111569. doi: 10.1016/j.plantsci.
2022.111569.
[34] Liu X, Cao J, Cheng X, et al. CsRVE1 promotes seasonal greening of albino Camellia sinensis cv. Huangkui by activating chlorophyll biosynthesis [J]. Tree Physiol. 2023, 43(8):1432-1443.
[35] Li X W, Zhu Y L, Chen C Y, et al. Cloning and characterization of two chlorophyll A/B binding protein genes and analysis of their gene family in Camellia sinensis [J]. Scientific Reports, 2020, 10(1): 4602-4633.
[36] 胡志航, 秦志远, 李静文, 等. 茶树捕光色素蛋白复合体基因CsLhcb2的鉴定及低温响应分析[J]. 茶叶科学, 2023, 43(2): 183-193.
Hu Z H, Qin Z Y, Li J W, et al. Identification of the light-harvesting chlorophyll-protein complex gene CsLhcb2 and its response to low temperature in tea plants [J]. Journal of Tea Science, 2023, 43(2): 183-193.
[37] 秦志远, 胡志航, 杨妮, 等. 茶树捕光色素蛋白复合体CsLhcb1基因的鉴定及干旱胁迫响应分析[J]. 茶叶学报, 2023, 64(5): 10-18.
Qin Z Y, Hu Z H, Yang N, et al. Identification and drought stress response of CsLhcb1in tea plants [J]. Acta Tea Sinica, 2023, 64(5): 10-18.
[38] 鄒智, 杨礼富, 安峰, 等. 橡胶树AtCAB1同源基因的克隆及其在稳定与衰老期叶片中的差异分析[J]. 热带农业科学, 2013, 33(4): 30-35.
Zou Z, Yang L F, An F, et al. Molecular cloning of a cDNA homologue to Arabidopsis CAB1 from Hevea brasiliensis and its different expression in mature and senescent rubber tree leaves [J]. Chinese Journal of Tropical Agriculture, 2013, 33(4): 30-35.
[39] Wang S, Wang P, Gao L, et al. Characterization and complementation of a chlorophyll-less dominant mutant GL1 in Lagerstroemia indica [J]. DNA and Cell Biology, 2017, 36(5): 354-366.
[40] Sattari Vayghan H, Nawrocki W J, Schiphorst C, et al. Photosynthetic light harvesting and thylakoid organization in a CRISPR/Cas9 Arabidopsis thaliana LHCB1 knockout mutant [J]. Frontiers in Plant Science, 2022, 13: 833032. doi: 10.3389/fpls.2022.833032.
[41] 赵渊祥, 王好运, 梁大曲, 等. 马尾松捕光复合体Ⅱ(Lhcb)基因家族鉴定及表达分析[J]. 植物生理学报, 2023, 59(8): 1583-1595.
Zhao Y X, Wang H Y, Liang D Q, et al. Identification and expression analysis of light-harvesting complex Ⅱ (Lhcb) gene family of Pinus massoniana [J]. Plant Physiology Journal, 2023, 59(8): 1583-1595.
[42] 王云鹤. 苎麻Lhc基因的鉴定和表达模式研究[D]. 武汉: 华中农业大学, 2020.
Wang Y H. Identification and expression profiling of Lhc genes in ramie [D]. Wuhan: Huazhong Agricultural University, 2020.
[43] 成浩, 陈明, 虞富莲, 等. 茶叶片阶段性返白过程中色素蛋白复合体的变化[J]. 植物生理学通讯, 2000(4): 300-304.
Chen H, Chen M, Yu F L, et al. The variation of pigment-protein complexes in the albescent stage of tea [J]. Plant Physiology Journal, 2000(4): 300-304.
[44] Du Y, Chen H, Zhong W, et al. Effect of temperature on accumulation of chlorophylls and leaf ultrastructure of low temperature induced albino tea plant [J]. African Journal of Biotechnology, 2008, 7(12): 1881-1885.
[45] Wu Q, Chen Z, Sun W, et al. De novo sequencing of the leaf transcriptome reveals complex light-responsive regulatory networks in Camellia sinensis cv. Baijiguan [J]. Frontiers in plant science, 2016, 7: 332. doi: 10.3389/fpls.2016.00332.

