1株引起食物中毒事件的肠炎沙门菌定量蛋白质组学分析
徐本锦 侯竹如 刘玲 严荣荣 张金晶 杜淼 宣焱 李卓禧 范蕾

摘要:目的 基于蛋白質组学,深入揭示1株引起食物中毒事件的肠炎沙门菌21A的分子特征,从而更好地防控食源性疾病的发生。方法 利用定量蛋白质组学技术对肠炎沙门菌株21A进行分析,使用TimsTOF Pro仪器在数据非依赖采集模式下采集质谱数据,利用MSstats软件完成肽段与蛋白的定量以及差异蛋白统计,并对差异表达蛋白的生物学功能进行GO、KOG功能富集、KEGG通路富集分析与CAZY注释、互作分析。结果 菌株21A中共鉴定出3183种蛋白质,其中差异蛋白300种。GO分析表明,差异蛋白主要与催化、结合、细胞内过程和代谢有关;KOG和KEGG分析显示,差异蛋白主要富集在6种代谢通路中;CAZY分析发现,37.96%的蛋白质为糖苷水解酶。结论 本研究揭示了肠炎沙门菌株21A的蛋白质组学特征,该菌株在入侵和感染过程中采用多种生存策略,包括增强毒力因子表达,增加脂质降解和诱导铁获取等。对代谢相关蛋白质的深入分析,有助于深入了解该菌的传播与感染机制,更好地防控食源性疾病的发生。
关键词:肠炎沙门菌;蛋白质组学;食物中毒;感染;代谢
中图分类号:R378 文献标志码:A
Quantitative proteomics analysis of a Salmonella enteritidis strain
causing food poisoning incident
Abstract Objective Based on proteomics, the study was used to reveal the molecular genetic characteristics of a Salmonella enteritidis 21A that caused a food poisoning event, so as to better prevent and control the occurrence of foodborne diseases and ensure human life. Methods The quantitative proteomics technology was used to analyze Salmonella enteritidis 21A, and the TimsTOF Pro instrument was used to collect mass spectrometry data in the data independent collection mode. The MSstats software was used to complete the quantitative analysis of peptides and proteins as well as the statistics of differential proteins, and the biological functions of the differentially expressed proteins were examined using the GO, KOG function enrichment, KEGG pathway enrichment analysis, CAZY annotation, and interaction analysis. Results The quantitative study of 21A revealed 3183 proteins, including 300 differential proteins. The GO database functional annotation indicated that differential proteins were primarily associated with catalytic processes, binding, intracellular processes, and metabolism, whereas the KOG and KEGG databases functional annotation demonstrated that difference proteins were mainly associated with six metabolic pathways. The CAZY database analyzed the carbohydrate-active enzymes in 21A, 37.96% of which were glycoside hydrolases. Conclusion This work characterized the proteins of S. enteritidis 21A, which used a number of survival tactics during the invasion and infection processes, including raising the production of virulence factors, promoting lipid breakdown, and driving iron acquisition. This in-depth examination of metabolism-related proteins may aid in the understanding of S. enteritidis invasion and infection mechanisms and better prevent and control the occurrence of foodborne illness.
Key words Salmonella enteritidis; Proteomics; Food poisoning; Infection; Metabolism
近年来,随着物流行业的快速发展、食品生产工艺的更新换代以及人们饮食习惯的改变,食源性疾病的发生呈上升趋势,食源性病原微生物已成为影响食品安全的主要因素[1]。沙门菌作为全球最主要的食源性病原菌之一,严重危害人类生命健康和食品安全[2]。
肠炎沙门菌是一种宿主广泛的人畜共患病病原体,在人类、动物、植物,甚至环境中都可以定植和传播[3]。肠炎沙门菌的感染始于肠腔,主要依赖于III型分泌系统(type Ⅲ secretion system, TTSS)实施对肠上皮细胞的侵袭。TTSS主要由沙门菌致病性岛1(SPI-1)和致病性岛2(SPI-2)编码,SPI-1参与沙门菌入侵,进入肠上皮细胞后利用SPI-2编码的毒力因子在沙门菌液泡(Salmonella containing vacuole,SCV)中存活[4]。此外,细菌分泌的效应蛋白可以拦截和修饰肠上皮细胞,从而逃避宿主先天免疫受体的监测并建立起一个适宜生存的细胞内生态位[5]。沙门菌的入侵和感染是一个复杂的过程,宿主细胞为沙门菌提供营养,沙门菌消耗某些代谢物来维持自身生存,同时释放废物,产生脂多糖等刺激成分,并将许多毒力因子直接分泌到宿主细胞,从而扰乱宿主代谢网络[6]。
蛋白质是生命活动的功能执行者,蛋白质组成、结构、表达水平以及翻译后修饰等信息不能简单地从基因组或转录本中读取。因此,蛋白质组学是对基因组学和转录组学的有力补充,对于了解生命活动规律意义重大。基于液相色谱-串联质谱(liquid chromatography-tandem mass spectrometry,LC-MS/MS)的蛋白质组学技术为定量复杂蛋白质混合物提供了一种高通量方法,其定量蛋白质的能力比传统的基于免疫亲和力的定量方法高出几个数量级[7]。这些技术已被广泛用于绘制细菌蛋白质组和蛋白质翻译后修饰,有助于揭示细菌-宿主相互作用的分子机制,从而发现新的生物标志物[8]。同时,蛋白质组学方法为检测细菌细胞对抗生素损伤等攻击性刺激的分子反应的快速变化提供了机会,捕捉引起抗生素耐药性发展的代谢途径的变化。这些方法将提供更多关于细菌耐药性机制和菌株在食品生产中的生命周期的信息[9]。蛋白组学在阐明细菌生活方式、开发食品安全生物标志物和创新食品保护策略方面具有重要相关性。
本研究在蛋白质组学水平揭示了临床食物中毒患者来源的肠炎沙门菌的分子特征,分析沙门菌在宿主体内存活和适应的分子机制,研究结果為进一步阐明肠炎沙门菌的传播途径、感染和毒力机制奠定基础,有助于更好地防控食源性和院内感染性疾病的发生,助力我国医药卫生和食品安全建设。
1 材料与方法
1.1 样本选择
从临床食物中毒患者的粪便中分离到肠炎沙门菌株,编号21A,下文简称21A,以沙门菌ATCC14028为对照菌株。将菌株21A以2%的比例接种至10 mL LB液体培养基,37 ℃、220 r/min活化培养12 h;再取2 mL活化的菌液转接至100 mL LB液体培养基,37 ℃、220 r/min继续培养12 h;6500 r/min离心5 min收集菌体后用灭菌去离子水清洗1次;加入5倍体积的甲醇,4 ℃静置1 h灭活菌体;离心收集菌体,液氮速冻30 min后置-80 ℃保存,每个样品平行收集3份。
1.2 蛋白样品的提取和质控
取适量样品到1.5 mL离心管中;加入一颗5 mm磁珠和适量Lysis Buffer 3(8 mol/L Urea, 4% CHAPS, 50 mmol/L DTT, 0.5% pharmalyte),分别添加终浓度为1 mmol/L的PMSF,2 mmol/L的EDTA,涡旋振荡后静置5 min,然后添加终浓度为10 mmol/L的DTT溶液;用组织研磨仪震荡2 min(50 Hz,120 s);4 ℃、25,000 g离心20 min,取上清;加入终浓度为10 mmol/L的DTT,56 ℃水浴1 h;恢复至室温后加入终浓度为55 mmol/L的IAM(碘代乙酰胺)暗室静置45 min;加入4倍体积冷丙酮,-20 ℃静置2 h;重复上一步2~3次,直至上清无色;4 ℃、25,000 g离心
20 min,弃上清液;往沉淀中加入适量Lysis Buffer 3,然后超声处理使沉淀溶解;4 ℃、25,000 g离心
20 min,取上清,做蛋白浓度定量。使用Bradford蛋白质定量试剂盒,将具有不同浓度的BSA标准蛋白质溶液和具有不同稀释比例的测试样品溶液添加到96孔板中。每个梯度重复3次。各孔加入180 μLG250染色溶液,在595 nm处测量吸光度。根据标准曲线和标准蛋白溶液的吸光度计算21A样品的蛋白质浓度。
每个样取10 μg蛋白溶液加入适量loading buffer,混匀后95 ℃加热5 min,25,000 g离心5 min,取上清点入12% SDS聚丙烯酰胺凝胶的点样孔中,80 V恒压电泳30 min后再120 V恒压电泳120 min;电泳结束后,将胶放入快速染脱仪器中10 min后,取出胶图扫描。
1.3 蛋白酶解及High pH RP(reversed-phase)分离
每个样品取100 μg蛋白溶液;按蛋白:酶=40:1的比例加入Trypsin酶2.5 μg,37 ℃酶解4 h;酶解的肽段利用Strata X柱进行除盐,真空抽干。将所有样本各取等量肽段进行混合后,用流动相A(5% ACN,pH9.8)稀释并进样,采用岛津LC-20AD液相系统,分离柱为Gemini C18柱(4.6 mm×250 mm, 5 μm)对样品进行液相分离。以1 mL/min的流速梯度洗脱:5%流动相B(95% ACN,pH9.8)10 min,5%至35%流动相B 40 min,35%至95%流动相B 1 min,流动相B持续
3 min,5%流动相B平衡10 min。在214 nm波长下监测洗脱峰并每分钟收集1个组分,结合色谱洗脱峰图合并样品得到10个组分,然后冷冻抽干。
1.4 DDA建库和DIA定量检测(Nano-LC-MS/MS)
将抽干的肽段样品用流动相A(100% H2O,0.1% FA)復溶,20,000 g离心10 min,然后取上清进样。通过Bruker公司的nanoElute进行分离。样品首先进入trap柱富集并除盐,随后与自装C18柱(75 μm内径,1.8 μm柱料粒径,约25 cm柱长)串联,以300 nL/min流速通过如下有效梯度进行分离:0 min,2%流动相B(100% ACN,0.1% FA);0~45 min,流动相B从2%线性升至22%;45~50 min,流动相B从25%升至35%;50~55 min,流动相B从35%升至80%;55~60 min,80%流动相B。纳升液相分离末端直接连接质谱仪并进行DDA(Data Dependent Acquisition)建库检测和DIA(Data Independent Acquisition)质谱检测。
1.5 蛋白质组学生物信息分析
DDA数据使用MaxQuant整合的Andromeda引擎完成鉴定,利用该结果建立谱图库。通过对DIA数据去卷积,结合DDA谱图库,得到肽段和蛋白的定性定量信息。使用MSstats包[10]对数据进行显著性差异的统计学评估,以Fold change>2和Pvalue<0.05两个条件作为显著性差异蛋白的筛选标准,并对差异蛋白的生物学功能进行分析。使用GO[11]、KOG和KEGG[12]数据库完成功能注释。根据对差异蛋白的功能注释,筛选与代谢有关的差异蛋白,使用STRING软件用于预测潜在的蛋白质-蛋白质相互作用[13],使用cytoscape软件绘制网络互作图。使用chiplot网站(https://www.chiplot.online/)分析、绘制差异表达蛋白热图。最后,基于以上的蛋白质组学分析结果,构建了一个肠炎沙门菌的入侵和感染模型。
2 结果与分析
2.1 样品质量控制与技术路线
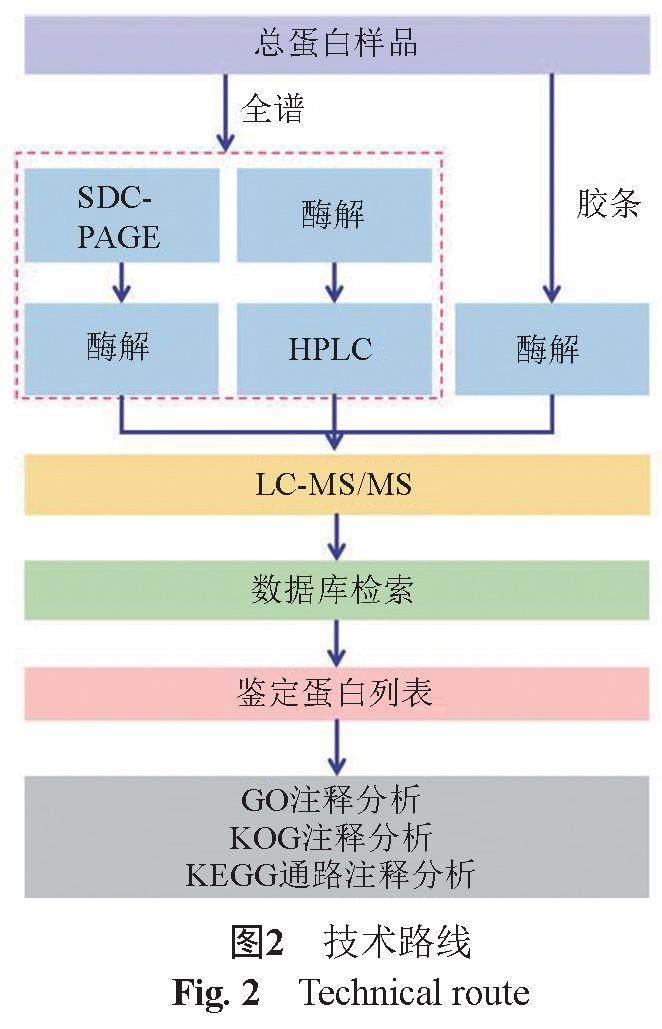
收集菌体21A并提取总蛋白,SDS-PAGE检测结果显示,胶图完整,蛋白质条带丰富,样品重复性好(图1)。质检合格的蛋白质样品经酶解得到肽段,并进行质谱检测和建库检测。基于高分辨率质谱仪产生样本数据,建立谱图库,使用UniProt数据库鉴定蛋白,得到蛋白列表。最后,对这些鉴定到的蛋白进行GO、COG和KEGG数据库的功能注释,并基于定量结果,完成不同比较组间差异蛋白的查找,进行差异富集蛋白的功能分析和差异蛋白的相互作用分析(图2)。
2.2 蛋白质鉴定及功能注释
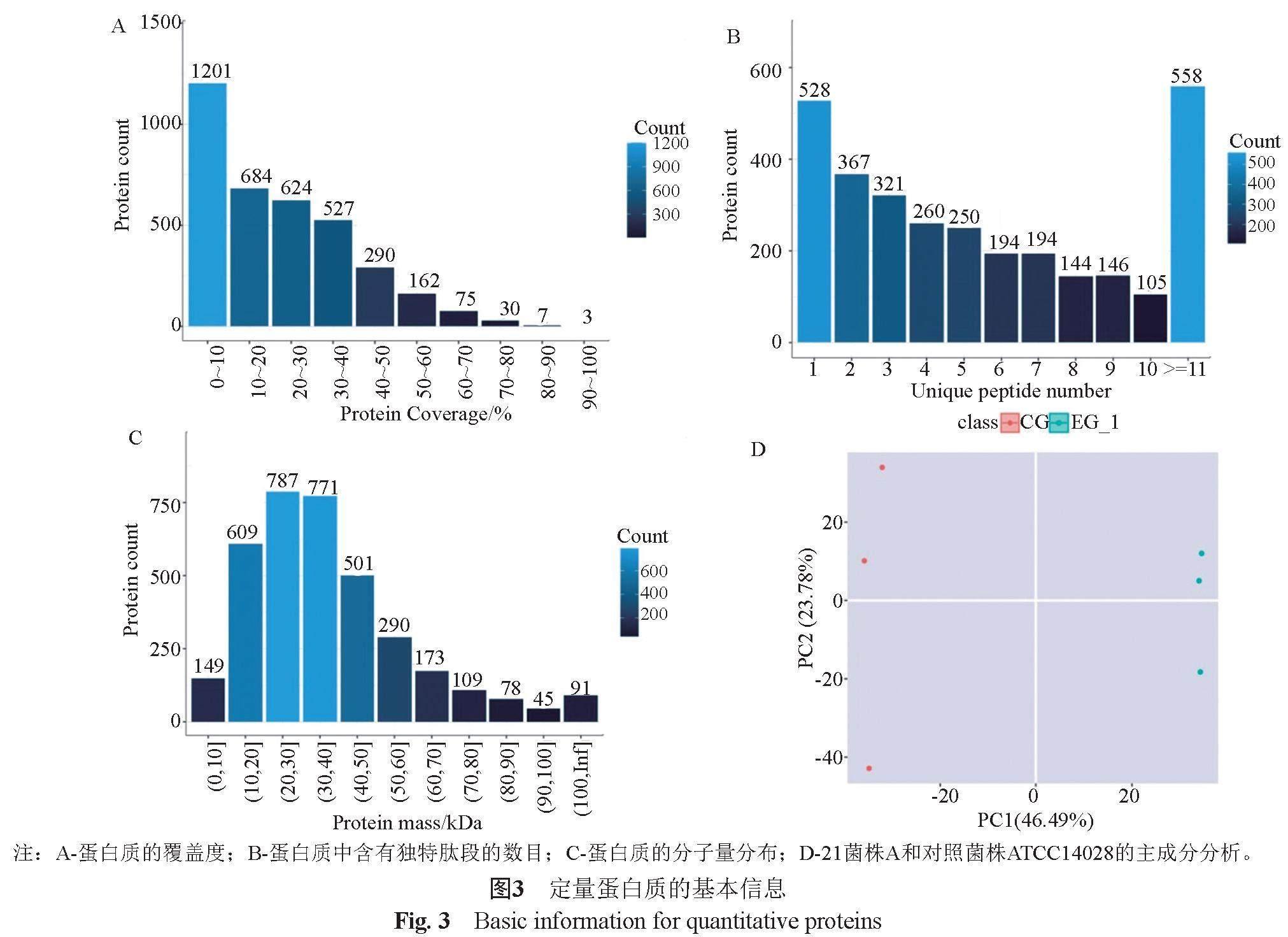
在该实验中共定量到37221个肽段和3604个蛋白,参考菌株平均鉴定出3214种蛋白质,菌株21A平均鉴定出3183种蛋白质。69.6%的蛋白质覆盖率不到30%,表明蛋白质鉴定的可信度仍有待提高(图3A)。在所鉴定到的蛋白质中,含有独特肽段的数目大多为1个(17.22%)或是大于11个(18.19%)(图3B)。所鉴定蛋白质的分子量大多在10~50 kDa的范围内(图3C)。主成分分析表明,菌株21A和参考菌株的表达水平存在显著差异(图3D)。
将鉴定到的所有蛋白质进行GO、KOG和KEGG数据库功能注释,共注释到2879种蛋白质(图4A)。KOG注释表明,蛋白质参与运输代谢、翻译、核糖体结构和生物发生、能量生产和转换、翻译后修饰、蛋白质转换、信号转导、RNA加工和修饰等,11.90%的蛋白质参与氨基酸运输与代谢,8.64%参与翻译、核糖体结构和生物发生,8.58%参与能源生产和转换(图4B)。基于GO功能注释,生物过程分析表明,蛋白质主要参与细胞内过程(11.80%)和代谢过程(11.32%);分子功能分析表明,蛋白质主要参与催化(13.67%)和结合(10.81%);细胞成分分析表明,这些蛋白主要定位于细胞内(10.35%)和细胞膜(10.24%)上(图4C)。KEGG通路注释表明,蛋白质共参与到细胞过程、环境信息处理、遗传信息处理、人类疾病、新陈代谢,以及生物体系统等6大类通路,其中代谢占据71.9%,主要的代谢途径是碳水化合物代谢、氨基酸代谢、辅因子与维生素的代谢和能量代谢(图4D)。
将鉴定到的所有蛋白质进行碳水化合物活性酶(carbohydrate-active enzymes database,CAZY)数据库功能注释,共注释到137个蛋白质。这些蛋白质共分为6个大类,即糖苷水解酶(glycoside hydrolases, GHs)、糖基转移酶(glycosyl transferases, GTs)、多糖裂解酶(polysaccharide lyases, PLs)、糖类酯解酶(carbohydrate esterases, CEs)、辅助功能(auxiliary activities, AAs)以及与碳水化合物相关的modules(carbohydrate-binding modules, CBMs)(图4E)。其中,37.96%的蛋白质注释为糖苷水解酶,是碳水化合物酶中最多的酶类;GTs次之,占33.58%。此外,共有5个蛋白质有两种功能,蛋白A4R40_06970、CysK、LFZ55_19225、MltD、PluTT01m_09850,既注释到CBMs,也注释到GHs。蛋白A4R40_06970、CysK、LFZ55_19225、PluTT01m_09850均被注释为几丁质酶和溶菌酶,A4R40_06970和CysK属于纤维素结合结构域家族V(cellulose-binding domain family V, CBD V),PluTT01m_09850属于CBDⅡ。蛋白MltD,也称为LysM结构域,注释为G型溶菌酶、肽聚糖裂解酶和几丁质酶。
2.3 差异蛋白的定量和功能注释
以鼠伤寒沙门菌ATCC14028为对照,以Fold change>2和P<0.05两个条件作为显著性差异蛋白的筛选标准。在菌株21A的所有蛋白中鉴定出300个差异蛋白,其中119个显著上调,181个显著下调(图5A)。将差异蛋白进行GO数据库功能注释,共富集到3大类34小类,在结合(11.53%)、催化过程(13.32%)、细胞内过程(13.52%)和代谢(14.51%)中富集的差异蛋白数量最多(图5B)。KEGG富集显示,菌株21A主要改变代谢活动来参与入侵和感染机体,参与代谢活动的共鉴定出44种蛋白质,其中6种蛋白质显著上调,包括硫代酯酶YigI、亚硝酸盐还原酶NirD、四亚硫酸还原酶TtrB、半胱氨酸脱硫酶SufS、胞苷酸激酶Cmk、脂多糖核心生物合成蛋白RfaZ;38种蛋白质显著下调,主要参与氨基酸代谢、能量代谢、碳水化合物代谢、脂类代谢和核苷酸代谢(图5C)。此外,还注释到27种蛋白质参与细胞过程、环境信息处理、遗传信息处理、人类疾病和生物体系统(表1)。基于KOG数据库的功能注释,每种蛋白质参与不同的生物过程,差异蛋白主要参与氨基酸的代谢(图5D)。从所有蛋白质中筛选出90种差异蛋白,以构建蛋白质-蛋白质相互作用网络(图5E),表明不同通路的协调调控参与了肠炎沙门菌对机体的入侵和感染。这些蛋白质主要聚类为代谢过程和氨基酸代谢、有机物质代谢、硫代谢、乙醇胺代谢,其中氨基酸代谢和乙醇胺代谢的相关蛋白质均为显著下调蛋白。
2.4 代谢相关的差异表达蛋白
相当数量的蛋白质与代谢过程相关,包括碳水化合物代谢、氨基酸代谢、辅因子和维生素代谢、能量代谢、脂类代谢和核苷酸代谢(图6)。多个蛋白质可以参与不同的代谢过程,其中有一个蛋白质A0A0D6H408参加4项代谢过程,碳水化合物代谢、氨基酸代谢、能量代谢和脂质代谢;有4个蛋白参加3项代谢过程,蛋白A0A718PRP1(碳水化合物代谢、氨基酸代谢、脂质代谢),A0A0D6HNW0(氨基酸代谢、核苷酸代谢、能量代谢),A0A0F6B8V6(碳水化合物代谢、氨基酸代谢、辅助因子和维生素代谢),A0A4U8K550(氨基酸代谢、核苷酸代谢、辅助因子和维生素代谢)。这表明细菌代谢过程中不同蛋白质之间相互协调,调节细菌的入侵和感染过程。并且在130个蛋白质中,有85.38%的蛋白的log2Foldchange的值位于-2~2之间,表明在沙门菌感染人体的过程中,与代谢有关的蛋白表达差异较小。
2.5 差异蛋白的时间序列分析
根据蛋白的表达量信息,鉴定到的3604个蛋白可以聚类成与时间相关联的蛋白簇,表达模式一致的蛋白会被聚到同一个簇,这些蛋白共分为9个聚类,随着时间的变化,蛋白的表达量发生显著变化(图7)。在聚类1、5中,随着时间的推移,蛋白表达量逐渐增加;聚类3、4、8中,蛋白表达量呈下降趋势。
2.6 基于蛋白质组学结果的感染模型
为了更好地理解肠炎沙门菌在宿主机体内的存活机制,本课题进一步建立了基于蛋白质组学结果的生存策略(图8)。沙门菌入侵机体后,通过SPI-1编码的TTSS-1和侵袭因子Rck、PagN进入宿主细胞。沙门菌还含有多种毒力因子,如SPI-2,鞭毛蛋白等,并且SPI-1的蛋白显著上调伴随着SPI-2的蛋白显著下调。当沙门菌进入宿主细胞后,代谢过程的调节发生显著变化,如氨基酸代谢,辅助因子和维生素的代谢,脂质代谢等。此外,细胞内沙门菌的其他蛋白质组学特征包括Fe-S簇蛋白显著上调,DNA复制和修复蛋白显著下调。
3 讨论
本研究前期从山西某三甲医院的食物中毒患者中分离到1株肠炎沙门菌21A,为了解肠炎沙门菌如何入侵并感染机体,对其进行定量蛋白质组学分析。
人类感染肠炎沙门菌主要是通过粪口传播,当摄入被该菌污染的食物后,沙门菌通过人体消化系统发生了一系列复杂的耐酸反应,在胃酸的强酸环境下定植下来[14]。进入机体的沙门菌具有侵袭性,通过侵袭因子TTSS-1、Rck和PagN入侵宿主细胞[15],
这3种侵袭机制在21A菌株中均存在。SPI-1编码TTSS-1,其效应子主要包括SptP、SipABCD、SopABDD2EE2和AvrA。21A注释到了除AvrA以外的其他效应子,这些效应子通过参与宿主细胞骨架的重排、免疫细胞募集和宿主炎症反应的调节来改变宿主细胞的环境,从而使沙门菌在宿主细胞内存活[16]。其中,Sops蛋白有助于沙门菌入侵并导致炎症和腹泻[17],这与先前21A患者的临床表现一致,分离出21A菌株的患者因感染性腹泻入院,并且经检查,C-反应蛋白增加。在本研究中,与TTSS-1相关的蛋白无显著变化,而Rck和PagN均显著上调。不同于TTSS-1的入侵机制,Rck和PagN是两种外膜蛋白,通过拉链机制诱导细菌入侵,即Rck与宿主细胞膜上的表皮生长因子受体相互作用,PagN与硫酸肝素蛋白聚糖相互作用,激活磷脂酰肌醇3-激酶和磷酸化酪氨酸蛋白,导致肌动蛋白聚合和胞膜重排,从而导致细菌内化[18]。此外,Rck还可干扰主要的细胞周期调节器,增加细胞周期S期的时间,从而阻碍宿主细胞的周期进展,创造一个适宜的生态位,促进细菌的入侵[19]。21A菌株中,显著上调的Rck、PagN蛋白和TTSS-1效应子相互作用,一起促进沙门菌对人体宿主细胞的入侵。
除TTSS-1、Rck和PagN以外,沙门菌还含有多种毒力因子,参与沙门菌的传播和感染。在本研究中,肠炎沙门菌的毒力蛋白发生显著变化,与SPI有关的蛋白SpvABCR和SsaQ、菌毛蛋白FlgE、与脂多糖有关的蛋白MsbB和RfaZ显著上调,菌毛蛋白FliZ、FliA、FliC、FlgN显著下调。Spv是位于质粒上的一段高度保守的序列,SpvB可干擾巨噬细胞和上皮细胞中的自噬和铁稳态,也可以破坏肠道上皮细胞的完整性,增加肠道的通透性,实现沙门菌的易位[20-21]。SpvC是磷酸苏氨酸裂解酶,可以通过β消除使双磷酸化的丝裂原活化蛋白激酶失活而抑制肠道炎症,也可以抑制NLRP3和NLRC4而抑制宿主细胞的焦亡,促进机体内的细菌传播。SPI-2是沙门菌在上皮细胞和巨噬细胞内感染和复制的重要蛋白,21A中注释到其6个效应蛋白SifA,SopD2,PipB2,SteA,SseJ和SseF,它们操纵宿主细胞内的运输并建立细菌的细胞内复制生态位[22]。效应蛋白SopD2,SteA,SseJ和SseF无明显变化,SifA和PipB2显著下调,这些蛋白质的表达水平决定了沙门菌感染的后果,可能在致病的组织特异性方面至关重要。菌毛蛋白除FlgE以外均显著下调,这与之前的研究菌毛过表达会减弱沙门菌的发病机制相一致[23]。
在人体的肠道内,分布着多种营养物质,如氨基酸、脂类、碳水化合物等,入侵的沙门菌在机体内增殖需利用这些营养素以获取能量和合成新的生物成分,沙门菌与宿主细胞相互作用,从而形成了复杂的代谢网络[24]。21A中差异激活的途径主要包括氨基酸代谢、碳水化合物代谢、辅因子和维生素代谢和脂类代谢等。本研究与色氨酸(TrpC)、精氨酸(ArcA、ArcB、ArcC)、鸟氨酸(ArgI)、天冬酰胺(AsnA)、谷氨酰胺(GlmS)、脯氨酸(PutA)和天冬氨酸(PyrB、PyrI)等氨基酸合成与代谢有关的蛋白均显著下调。据之前的研究报道,对腹泻感染患者的肠道微生物进行代谢组分析,L-组氨酸生物合成和降解途径以及L-鸟氨酸生物合成途径的水平降低,从而调节细菌的致病性,但是具体的机制仍待进一步研究[25]。经碳水化合物酶分析,21A中主要含有糖苷水解酶,其中幾丁质酶占据重要作用,它们属于GH18和GH19,促进沙门菌的体内入侵、存活和发病[26]。这些蛋白在差异分析中无显著变化。本研究中与脂质β氧化途径有关的蛋白FadAB、YdiR显著上调。β氧化途径是脂质的主要降解途径,在葡萄糖受限和氨基酸丰富的情况下,脂类代谢是沙门菌在促炎巨噬细胞中的重要代谢途径,并且脂质降解基因是沙门菌定植组织所必须的[27]。为更全面地探究细菌脂膜的适应机制,需联合运用蛋白质组学、代谢组学以及脂质组学,以鉴定、表征和量化脂质和蛋白,从而阐明特定应激与单个脂质和特定脂代谢酶改变之间的关系[28]。
沙门菌中有一种蛋白质细胞器为细菌微腔(bacterial microcompartments,MCPs),它由包裹在选择性渗透蛋白质外壳上的代谢酶组成。MCP代谢与肠道系统疾病的发病机制有关,沙门菌中主要有2种分解代谢的MCPs,即Pdu和Eut MCP[29]。在本研究中,发现了Pdu和Eut MCP,它们均显著下调,功能注释显示分别参与1,2-丙二醇和乙醇胺的代谢。据之前的报道,肠炎沙门菌中一般同时存在Pdu和Eut MCP,在本研究中也证明了一点。这两种MCP具有高度相似的外壳,Pdu MCP通过控制1,2-丙二醇的代谢,增加沙门菌在哺乳动物胃肠道的定植,这是沙门菌感染发病机制的关键阶段[30]。Eut MCP蛋白联合作用分解代谢乙醇胺,在有氧条件下,乙醇胺是提供沙门菌生存所需碳、能量和氮的唯一来源[31]。根据对肠道内的沙门菌的研究,1,2-丙二醇可以诱导pdu操纵子,阻遏eut操纵子,从而防止了两种系统的有害混合[32]。MCPs对特定碳底物的代谢使致病菌在宿主环境中具有选择优势,并以影响人类健康的方式影响人体肠道生态。此外,由于肠炎沙门菌感染引起的炎症,肠道中发生一系列化学反应支持乙醇胺和丙二醇的厌氧呼吸,从而为沙门菌的生长提供了便利,促进沙门菌的传播[29]。
此外,21A中SUF蛋白SufABCDES的表达全部显著上调,SUF蛋白是Fe-S簇组装系统的一种。沙门菌侵染宿主细胞后,细胞铁流出增加,细胞质中不稳定铁和巨噬细胞内铁蛋白的储存大大减少,造成铁限制的宿主环境,SUF蛋白可以帮助沙门菌来获取铁去维持沙门菌的生产[33]。过表达的SUF蛋白帮助沙门菌应对氧化应激,并帮助沙门菌在巨噬细胞中存活[34]。
4 结论
本研究从食物中毒患者的粪便中筛选分离出肠炎沙门菌,对其中的21A菌株进行蛋白质组学分析,了解其蛋白质功能特征。经GO、KOG、KEGG数据库对蛋白质组进行功能注释,显示与代谢有关的蛋白发生显著变化。肠炎沙门菌通过TTSS-1、Rck和PagN等3种入侵机制侵袭宿主细胞,在感染机体过程中采用了多种生存策略,包括增强毒力因子表达,增加脂质降解,诱导铁获取等。新陈代谢对于病原微生物的毒力至关重要,本研究在蛋白质组学水平上为肠炎沙门菌的入侵和感染机制提供了新的见解,有助于快速检测试剂、疫苗和新型抗感染药物的研发。
参 考 文 献
张彪. 沙门氏菌感染肠上皮细胞外泌体宿主蛋白质组学分析[J]. 食品科学, 2022, 43(6): 132-140.
Soubeiga A P, Kpoda D S, Compaore M K A, et al. Molecular characterization and the antimicrobial resistance profile of Salmonella spp. isolated from ready-to-eat foods in Ouagadougou, Burkina Faso[J]. Int J Microbiol, 2022, 2022: 9640828.
Knodler L A, Elfenbein J R. Salmonella enterica[J]. Trends Microbiol, 2019, 27(11): 964-965.
Li Q. Mechanisms for the invasion and dissemination of Salmonella[J]. Can J Infect Dis Med Microbiol, 2022, 2022: 2655801.
Walch P, Selkrig J, Knodler L A, et al. Global mapping of Salmonella enterica-host protein-protein interactions during infection[J]. Cell Host Microbe, 2021, 29(8):1316-1332.e12.
Bumann D, Schothorst J. Intracellular Salmonella metabolism[J]. Cell Microbiol, 2017, 19(10): e12766.
Li J, Smith L S, Zhu H J. Data-independent acquisition (DIA): An emerging proteomics technology for analysis of drug-metabolizing enzymes and transporters[J]. Drug Discov Today Technol, 2021, 39: 49-56.
Saleh S, Staes A, Deborggraeve S, et al. Targeted proteomics for studying pathogenic bacteria[J]. Proteomics, 2019, 19(16): e1800435.
Tarbeeva S, Kozlova A, Sarygina E, et al. Food for thought: Proteomics for meat safety[J]. Life (Basel), 2023, 13(2): 255.
Choi M, Chang C Y, Clough T, et al. MSstats: An R package for statistical analysis of quantitative mass spectrometry-based proteomic experiments[J]. Bioinformatics, 2014, 30(17): 2524-2526.
Ashburner M, Ball C A, Blake J A, et al. Gene ontology: Tool for the unification of biology[J]. Nature Genetics, 2000, 25(1): 25.
Jones P, Binns D, Chang H Y, et al. InterProScan 5: Genome-scale protein function classification[J]. Bioinformatics, 2014, 30(9): 1236-1240.
Franceschini A, Szklarczyk D, Frankild S, et al. STRING v9.1: Protein-protein interaction networks, with increased coverage and integration[J]. Nucleic Acids Res, 2013, 41(database issue): D808-815.
Diard M, Hardt W D. Basic processes in Salmonella-host interactions: Within-host evolution and the transmission of the virulent genotype[J]. Microbiol Spectr, 2017, 5(5): 1-11
Holbert S, Barilleau E, Roche S M, et al. Murine AML12 hepatocytes allow Salmonella typhimurium T3SS1-independent invasion and intracellular fate[J]. Sci Rep, 2021, 11(1): 22803.
Fattinger S A, Sellin M E, Hardt W D. Salmonella effector driven invasion of the gut epithelium: breaking in and setting the house on fire[J]. Curr Opin Microbiol, 2021, 64: 9-18.
Lou L, Zhang P, Piao R, et al. Salmonella pathogenicity island 1 (SPI-1) and its complex regulatory network[J]. Front Cell Infect Microbiol, 2019, 9: 270.
Barilleau E, Vedrine M, Koczerka M, et al. Investigation of the invasion mechanism mediated by the outer membrane protein PagN of Salmonella typhimurium[J]. BMC Microbiol, 2021, 21(1): 153.
Mambu J, Barilleau E, Fragnet-Trapp L, et al. Rck of Salmonella typhimurium delays the host cell cycle to facilitate bacterial invasion[J]. Front Cell Infect Microbiol, 2020, 10: 586934.
Sun L, Yang S, Deng Q, et al. Salmonella effector SpvB disrupts intestinal epithelial barrier integrity for bacterial translocation[J]. Front Cell Infecti Microbi, 2020, 10: 606541.
Wang L, Li Y, Liu Y, et al. Salmonella spv locus affects type I interferon response and the chemotaxis of neutrophils via suppressing autophagy[J]. Fish Shellfish Immunol, 2019, 87: 721-729.
Knuff-Janzen K, Serapio-Palacios A, McCoy J, et al. Quantitative proteomic screen identifies annexin A2 as a host target for Salmonella pathogenicity island-2 effectors SopD2 and PipB2[J]. Sci Rep, 2021, 11(1): 23630.
Yang X, Thornburg T, Suo Z, et al. Flagella overexpression attenuates Salmonella pathogenesis[J]. PLoS One, 2012, 7(10): e46828.
ibinelli-Sousa S, de Araujo-Silva AL, Hespanhol JT, et al. Revisiting the steps of Salmonella gut infection with a focus on antagonistic interbacterial interactions[J]. FEBS J, 2022, 289(14): 4192-4211.
Gallardo P, Izquierdo M, Vidal RM, et al. Gut microbiota-metabolome changes in children with diarrhea by diarrheagenic E. coli[J]. Front Cell Infect Microbiol, 2020, 10: 485.
Chandra K, Roy Chowdhury A, Chatterjee R, et al. GH18 family glycoside hydrolase Chitinase A of Salmonella enhances virulence by facilitating invasion and modulating host immune responses[J]. PLoS Pathog, 2022, 18(4): e1010407.
Reens A L, Nagy T A, Detweiler C S. Salmonella enterica requires lipid metabolism genes to replicate in proinflammatory macrophages and mice[J]. Infect Immun, 2019, 88(1): e00776-00719.
Kralj T, Nuske M, Hofferek V, et al. Multi-omic analysis to characterize metabolic adaptation of the E. coli lipidome in response to environmental stress[J]. Metabolites, 2022, 12(2): 171.
Stewart K L, Stewart A M, Bobik T A. Prokaryotic organelles: Bacterial microcompartments in E. coli and Salmonella[J]. EcoSal Plus, 2020, 9(1): ESP-0025-2019.
Yang M, Wenner N, Dykes G F, et al. Biogenesis of a bacterial metabolosome for propanediol utilization[J]. Nat Commun, 2022, 13(1): 2920.
Kaval K G, Garsin D A. Ethanolamine utilization in bacteria[J]. mBio, 2018, 9(1): e00066-00018.
Sturms R, Streauslin N A, Cheng S, et al. In Salmonella enterica, ethanolamine utilization is repressed by 1,2-propanediol to prevent detrimental mixing of components of two different bacterial microcompartments[J]. J Bacteriol, 2015, 197(14): 2412-2421.
Karash S, Jiang T, Kwon Y M. Genome-wide characterization of Salmonella typhimurium genes required for the fitness under iron restriction[J]. BMC Genom Data, 2022, 23(1): 55.
Wang M, Qi L, Xiao Y, et al. SufC may promote the survival of Salmonella enterica serovar Typhi in macrophages[J]. Microb Pathog, 2015, 85: 40-43.

