应用噬菌体展示随机12肽库筛选诺如病毒抗原模拟表位
周飞园 王璐△ 梁芷妍 林璧慧 李佳恒 王宇 井多娜 张绪富 戴迎春
(1.南方医科大学公共卫生学院流行病学系,广州 510515;2.南方医科大学中医药学院,广州 510515)
诺如病毒(Norovirus,NoV)是导致非细菌性胃肠炎散发及暴发的主要病原体,约占全球急性胃肠炎发病率的1/5[1]。NoV属于杯状病毒科,是单股正链小RNA病毒,全长7.5~7.7 kb,由3个开放阅读框(open reading frames,ORFs)组成,分别为ORF1、ORF2和ORF3,其中ORF2编码主要衣壳蛋白(major capsid protein,VP1)。VP1包括衣壳区(shell domain,S)和突出区(protruding domain,P)[2]。其中P区可进一步分为相对保守的P1区和高度变异的P2区,前者能够增强病毒颗粒稳定性,后者位于VP1最外层,具有高度变异性,含有抗原决定簇和潜在的中和抗原表位,存在与人类组织血型抗原(histoblooding group antigens,HBGAs)的结合位点,是构成免疫原性的关键区域[3-5]。抗原表位决定了病毒的抗原特异性,能够刺激人体产生抗体或淋巴细胞,对新型表位疫苗设计具有重要意义[6-7]。目前由于NoV的基因多样性及高度变异性,且缺乏成熟的体外培养体系,严重阻碍了NoV疫苗发展[8]。因此,了解NoV的抗原表位及免疫原性对NoV疫苗发展有重要意义。
本研究利用课题组前期制备的全人源抗NoV中和性单链可变片段抗体(single-chain variable fragment antibody,scFv)[9],应用噬菌体12肽库技术淘选获得NoV抗原模拟表位,并对抗原模拟表位进行初步鉴定,为后续NoV治疗性单链抗体药物和广谱保护性疫苗研制奠定基础。
1 材料与方法
1.1 材料 Ph.D.-12噬菌体展示肽库试剂盒购自美国NEB公司;M13噬菌体单链基因组DNA快速提取试剂盒购自北京庄盟国际生物基因科技有限公司;HRP-羊抗鼠IgG购自美国Abcam公司;TMB辣根过氧化物酶底物显色试剂盒购自碧云天生物技术有限公司;源于GⅡ.6 NoV及GⅡ.17暴发感染者恢复期PBMC制备的NoV人源中和性scFv、GⅡ.4/GⅡ.6/GⅡ.17 NoV P蛋白、抗GⅡ.4/GⅡ.6/GⅡ.17小鼠血清及B型唾液由南方医科大学流行病学实验室保存。
1.2 方法
1.2.1 噬菌体展示随机十二肽库淘选 选取课题组前期制备的源于GⅡ.6及GⅡ.17 NoV患者恢复期外周血的NoV人源中和性scFv[9]各6株,抗体来源见表1。噬菌体展示文库筛选参照试剂盒说明书(E8110S)操作,以scFv与噬菌体展示文库分别作用,进行三轮“吸附-洗涤-洗脱”生物淘选即可获得与ScFv特异性结合的噬菌体。

表1 用于生物淘选NoV抗原模拟表位的scFv来源Tab.1 Source of scFv antibodies for biopanning of NoV antigen mimic epitopes
1.2.2 ELISA初步鉴定单克隆噬菌体 随机挑取第3轮洗脱后的多个单克隆噬菌体进行扩增纯化,鉴定噬菌体与scFv亲和活性以及与NoV P蛋白的竞争作用。
1.2.2.1 Phage-ELISA鉴定噬菌体亲和性 将scFv(2 μg/ml)包被于酶标板,4 ℃过夜后用5%脱脂奶粉封闭,以纯化的噬菌体为一抗,阴性对照为淘选过程中洗涤下来未与scFv结合的噬菌体。加入anti-M13-HRP抗体(1∶3 000)作为二抗,TMB避光显色,2 mol/L磷酸终止反应,读取各孔OD450。若实验孔OD450>阴性孔OD4502倍,则判定为阳性噬菌体。实验孔均设两孔平行重复。
1.2.2.2 阳性噬菌体与P蛋白竞争作用鉴定 将scFv(2 μg/ml)包被于酶标板,4 ℃过夜后用5%脱脂奶粉封闭,加入阳性噬菌体50 μl和NoV P蛋白50 μl(GⅡ.6 P蛋白4 μg/ml,GⅡ.17 P蛋白0.4 μg/ml)混合液作为一抗,以anti-M13-HRP抗体(1∶3 500)为二抗,TMB避光显色,2 mol/L磷酸终止反应,读取各孔OD450。抑制率(%)=(阳性对照OD450-实验组OD450)/阳性对照OD450×100%。实验孔均设置两孔重复。
1.2.3 阳性噬菌体DNA提取分析 参照M13噬菌体单链基因组DNA快速提取试剂盒说明书,提取阳性噬菌体DNA,送华大基因公司测序,以Ph.D.-12噬菌体展示肽库试剂盒的-96gⅢ为测序引物。获得的12肽序列用DNASTAR MegAlign程序与NoV VP1的氨基酸序同源性对比分析,选取出现次数较多且同源性高的多肽序列进行后续分析。
1.2.4 生物信息学分析 利用DNAstar软件protean程序分析多肽抗原模拟表位在VP1中所在区域的β转角、无规卷曲等二级结构情况,并分析其抗原性指数、亲水性指数及表面可及性指数。使用蛋白结构预测SwissModell server(https://swissmodel.expasy.org/)在线软件,对GⅡ.4/GⅡ.6/GⅡ.17 NoV VP1进行同源建模获得其三维结构,PyMol软件分析抗原模拟表位在VP1中的定位。
1.2.5 ELISA检测多肽与GⅡ.6 NoV P蛋白竞争抑制 多肽MGLDLWENFQVL(纯度98%)由广州睿博生物技术有限公司合成,分别包被与GⅡ.6 NoV P蛋白结合最优的唾液类型(B型;1∶1 000),4 ℃过夜后用5%脱脂奶粉封闭,加入梯度稀释的多肽和GⅡ.6 NoV P蛋白混合液37 ℃孵育1 h。以鼠抗GⅡ.6血清(1∶3 500)为一抗,HRP-羊抗鼠IgG(1∶5 000)为二抗,TMB避光显色后测定OD450。实验孔均设置复孔,以无关多肽为阴性对照,计算多肽抑制率,公式同1.2.2.2。
2 结果
2.1 噬菌体随机12肽库淘选 经过3轮“吸附-洗涤-洗脱”生物淘选,产出率逐轮升高,说明有效富集了可与scFv特异性结合的噬菌体,淘选结果见表2。
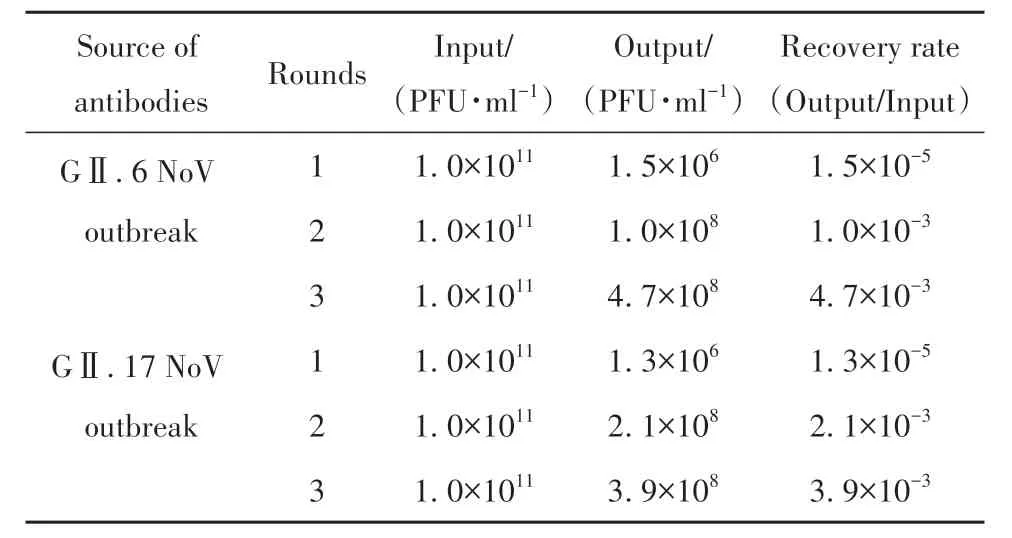
表2 噬菌体随机12肽库对源于G Ⅱ.6 NoV暴发和GⅡ.17 NoV暴发的scFv淘选结果Tab.2 Panning results of phage display peptide library against scFv derived from a GⅡ.6 NoV outbreak and a G Ⅱ.17 NoV outbreak
2.2 单克隆噬菌体初步鉴定
2.2.1 Phage-ELISA鉴定噬菌体亲和活性 包被与生物淘选过程中相应的scFv,以未与抗体结合的噬菌体为阴性对照,进行Phage-ELISA鉴定,阳性噬菌体与抗体的亲和活性结果见图1。

图1 阳性噬菌体克隆与源于G Ⅱ.6 NoV、GⅡ.17 NoV暴发的scFv的结合活性分析Fig.1 Affinity analysis of positive phage clones with scFv derived from GⅡ.6 NoV and GⅡ.17 NoV outbreak
2.2.2 竞争抑制ELISA筛选NoV抗原模拟表位对阳性噬菌体进行竞争抑制ELISA试验,发现GⅡ.6 NoV P蛋白能不同程度地抑制阳性噬菌体与scFv结合,抑制率为19.12%~89.61%(图2A)。GⅡ.17 NoV P蛋白也能抑制阳性噬菌体与scFv结合,抑制率为10.70%~83.76%(图2B)。

图2 G Ⅱ.6 NoV、GⅡ.17 NoV P蛋白对阳性噬菌体克隆与scFv抗体结合的竞争抑制作用Fig.2 Competitive inhibition of GⅡ.6 NoV and GⅡ.17 NoV P proteins on binding of positive phage clones and scFv
2.3 阳性噬菌体克隆的DNA序列分析 将对NoV P蛋白有抑制作用的阳性噬菌体进行DNA提取并送测序分析,最终得到27条不同碱基序列并将其翻译为氨基酸序列(表3、表4)。其中MGLDLWENFQVL和FHWPSYYLTPWV出现频次较多,分别为47次和3次,提示其展示了scFv与NoV结合的高亲和力高肽段。同源性对比这两条肽段与NoV VP1蛋白有3段有高同源性的氨基酸序列(图3、图4):MGXDXWWXXPXY和YXXXXXXXLXPXXWV(其中X代表任一氨基酸)。

图3 G Ⅱ.6 NoV VP1区域与多肽MGLDLWENFQVL的高同源性片段Fig.3 High homology fragment between G Ⅱ.6 NoV VP1 region and MGLDLWENFQVL

图4 GⅡ.4/GⅡ.6/G Ⅱ.17 NoV VP1区域与多肽FHWPSYYLTPWV的高同源性片段Fig.4 High homology fragment between GⅡ.4/GⅡ.6/G Ⅱ.17 NoV VP1 region and FHWPSYYLTPWV

图5 GⅡ.4/GⅡ.6/G Ⅱ.17 NoV VP1中部分区域二级结构分析Fig.5 Secondary structure analysis of some regions in GⅡ.4/GⅡ.6/G Ⅱ.17 NoV VP1

图6 “MG-D-W”“W-P-Y”“Y-L-P-WV”在G Ⅱ.6 NoV VP1三维结构的位置Fig.6 Location of "MG-D-W""W-P-Y""Y-L-P-WV" in 3D structure of G Ⅱ.6 VP1

表3 源于G Ⅱ.6 NoV暴发的scFv的阳性噬菌体克隆氨基酸序列Tab.3 Amino acid sequences of positive phage clones for scFv derived from G Ⅱ.6 NoV outbreak

表4 源于GⅡ.17 NoV暴发的scFv的阳性噬菌体克隆氨基酸序列Tab.4 Amino acid sequences of positive phage clones for scFv derived from GⅡ.17 NoV outbreak
2.4 生物学信息分析
2.4.1 多肽抗原模拟表位与VP1二级结构的关系 蛋白潜在的抗原表位具有亲水性、柔韧性、抗原指数和表面可及性较高的特征且常位于转角或无规则卷曲区域,其中抗原指数、亲水性>0及表面可及性>1形成表位的可能性较大[10]。Protean分析发现,序列“MG-D-W”位于GⅡ.6 VP1蛋白402~407氨基酸区域,该区域中具有丰富的无规则卷曲结构且亲水性>0、抗原性>0及表面可及性>1,该序列为GⅡ.6抗原表位的可能性较大。而在GⅡ.4/GⅡ.6/GⅡ.17这三株NoV的VP1蛋白中序列“W-P-Y”和序列“Y-L-P-WV”所在区域分别有抗原性<0、表面可及性<1,抗原性<0等情况(Protean分析结果通过VP1整条序列获得的,图4仅截取部分结果)。
2.4.2 多肽抗原模拟表位与VP1三维结构的关系 “MG-D-W”处于无规则卷曲结构,与Protean分析结果一致,这4个氨基酸间距离较近,并暴露于GⅡ.6 VP1蛋白P2区表面,而P2区是存在NoV与HBGA受体结合位点和主要抗原位点的主要区域[3]。此外,“MG-D-W”高度暴露且具有良好的嵌合结构,易被宿主免疫系统作为产生中和抗体的目标。“W-P-Y”和“Y-L-P-WV”分别位于VP1蛋白P2区域和P1区域,分别有色氨酸、酪氨酸暴露于VP1蛋白表面,其余氨基酸深埋于VP1蛋白,不利于抗体结合(绿色区域为VP1蛋白P1区,灰色为P2区,蓝色为S区,“MG-D-W”“W-P-Y”及“Y-L-P-WV”分别以红色、紫色、橙色显示;“MG-D-W”仅出现于GⅡ.6VP1,而“W-P-Y”“Y-L-P-WV”在GⅡ.4和GⅡ.17 VP1三维结构的位置及二级结构均与GⅡ.6相同,因此仅展示GⅡ.6)。综上,“MG-D-W”为抗原表位的可能性较大,且可能为GⅡ.6的特异性抗原表位。
2.5 多肽与P蛋白的竞争抑制ELISA 在合成多肽MGLDLWENFQVL与GⅡ.6 NoV P蛋白的竞争抑制ELISA实验中,随着多肽浓度逐渐升高,对P蛋白和HBGA的结合抑制作用逐渐增强(图7)。

图7 梯度稀释多肽对GⅡ.6 NoV P蛋白与HBGA受体结合的竞争抑制作用Fig.7 Competitive inhibition of GⅡ.6 NoV P protein binding to HBGA receptors by gradient dilution peptides
3 讨论
抗原表位又称抗原决定簇,对其进行分析鉴定能够为疫苗和抗病毒药物研发提供重要信息[11]。传统的抗原表位分析方法是通过人工合成一系列抗原蛋白肽段,再用ELISA、Western blot等方法鉴定这些肽段与相应抗体的结合能力,以此获得抗原表位氨基酸序列、位置信息等。这种方法工作量大、费用高,且人工合成的蛋白肽段仅能模拟线性表位[12-13]。或用X射线衍射技术进行抗原表位研究,通过晶体学信息准确分析抗原表位的氨基酸构成及表位类型,该方法操作过程复杂,费用高,且仅在已知合适晶体情况下才能应用[14]。噬菌体展示技术是目前抗原表位筛选主流方法之一,是基于外源多肽或蛋白与噬菌体衣壳蛋白融合的一种技术,包被目的抗体后利用噬菌体随机12肽库进行生物淘选,即可获得与目的抗体特异性结合的抗原表位[15-17]。噬菌体展示技术具有高亲和选择、高分辨率等优点,能够识别抗NoV多克隆抗体及scFv中的多个不同抗原表位,有助于了解NoV表位分布[18]。具有中和作用的抗体是筛选和鉴定NoV抗原表位的重要工具,与传统单克隆抗体相比,本研究所用的scFv体积较小,具有较高分辨率,能够发现单克隆抗体未能识别的抗原表位[19]。
本研究利用Ph.D.-12噬菌体展示肽库,以与GⅡ.4/GⅡ.6/GⅡ.17 NoV P蛋白具有高特异性且中和能力较强的scFv作为靶标蛋白进行生物淘选,获得了与scFv特异性结合的噬菌体。以ELISA鉴定噬菌体,并对阳性噬菌体进行测序获得其递呈的十二肽序列,有2条出现频次较多的序列MGLDLWENFQVL和FHWPSYYLTPWV。生物信息学分析结果提示“MG-D-W”为GⅡ.6抗原表位的可能性较大,可能模拟了GⅡ.6 NoV与scFv结合的抗原表位。通过合成多肽进一步验证以排除噬菌体自身的影响,发现随着多肽浓度升高其阻断P蛋白与HBGA受体结合的能力增强。
GⅡ.6 NoV以散发为主,较少引起暴发流行[20]。2016-2018年,我国疫情监测数据显示:GⅡ.6引起的NoV暴发共有16起(3.4%)[21]。同时,GⅡ.6在美国、巴西、阿根廷、日本等国引起了多起NoV胃肠炎暴发[22-25],成为幼儿感染NoV的第二大常见毒株,提示GⅡ.6发病率在全球范围内呈现一定上升趋势。因此,有学者根据河北省正定县50多年的腹泻监测数据分析结果,提出GⅡ.6应纳入NoV疫苗候选毒株[26]。但目前NoV候选疫苗主要集中于GⅠ.1、GⅠ.3、GⅡ.3、GⅡ.4和GⅡ.17 NoVs几种基因型,尚无GⅡ.6疫苗研发,且关于GⅡ.6 NoV抗原表位的研究较少,QIU等[27]首次提出GⅡ.6 NoV与HBGA的结合区域位于286~312氨基酸残基之间。因此本研究成功筛选并鉴定的GⅡ.6 NoV的抗原表位“MG-D-W”,能够为NoV疫苗设计提供重要信息。